真核生物α-甘露糖苷酶生物信息学分析
2018-08-02孙春玉
王 铎,孙春玉,陈 静,王 义
(吉林农业大学生命科学学院,中国吉林长春 130118)
α-甘露糖苷酶(α-mannosidase,α-Man)参与真核生物蛋白质糖基化过程,其对N-聚糖的修饰,在有机体糖蛋白复杂化过程中发挥着至关重要的作用[1]。α-Man分布广泛、种类繁多,在真核生物胞质、内质网、高尔基体、溶酶体中都有发现,不同种类、不同功能的α-Man共同参与N-聚糖的修饰过程[2]。根据甘露糖苷酶的功能特异性、序列同源性和酶机制,α-Man可分为GH47家族和GH38 家族[3]。GH47 家族为 I型 α-甘露糖苷酶(αmannosidase I,Man I),包括内质网I型α-甘露糖苷酶 (endoplasmic reticulum Man I,ERMan I)、高尔基体I型α-甘露糖苷酶(Golgi α-mannosidase I,GM I)两种;GH38家族为Ⅱ型α-甘露糖苷酶(αmannosidaseⅡ,ManⅡ),包括高尔基体Ⅱ型α-甘露糖苷酶、溶酶体Ⅱ型α-甘露糖苷酶、内质网Ⅱ型α-甘露糖苷酶、胞质Ⅱ型α-甘露糖苷酶4种。
α-Man与植物的生长发育有密切关系,不仅影响植物根系的生长,同时影响果实的成熟。在植物细胞内,细胞壁糖蛋白含有大量的N-聚糖结构,α-Man对N-聚糖的修饰直接影响细胞壁的组成,细胞壁结构的改变关系到果实的成熟与软化[4]。在动物细胞内,α-Man功能异常会引起N-聚糖代谢异常,造成蛋白质的空间结构、理化性质改变,同时影响细胞粘附、细胞迁移、细胞生长及细胞分化等生理过程[5]。一旦α-Man功能出现异常,就会造成病毒感染、炎症反应、癌细胞扩散、甘露糖代谢异常等症状[6]。ERMan I是真核生物所共有的酶,属于N-聚糖修饰过程的限速酶,参与了一个重要的蛋白质质量控制途径——内质网相关蛋白降解(ER-associated protein degradation,ERAD),对真核生物蛋白质代谢过程起到至关重要的作用[7]。
目前,生命科学已经步入后基因组时代,通过生物信息学手段挖掘α-Man基因的生物学功能,有助于进一步了解α-Man在有机体生命过程中所发挥的作用,对调控由N-聚糖代谢引起的果实软化,治愈由于α-Man功能异常所造成的疾病有重大意义,同时也可对未来生命科学、医学的发展起推动作用。本文通过生物信息学手段,对α-Man的功能、进化、保守性进行分析,同时对不同物种中ERMan I的代谢途径、蛋白质结构、理化性质等进行分析,以期为后续实验提供研究基础。
1 材料与方法
1.1 材料
通过NCBI网站下载已注释的动物、植物及真菌α-Man的mRNA序列各100条,具体信息如表1所示。
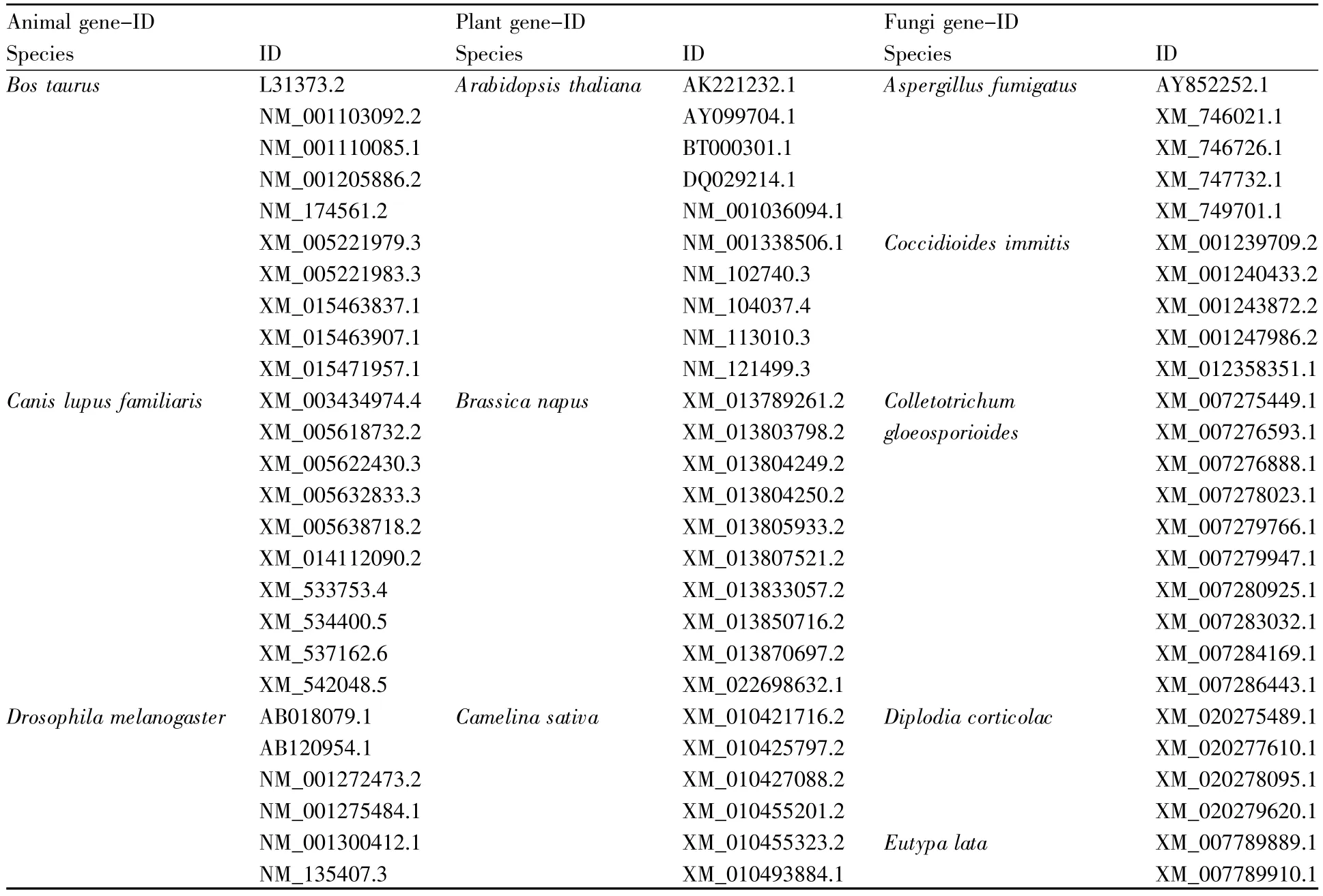
表1 真核生物α-Man基因来源Table 1 The source of α-Man genes in eukaryotes
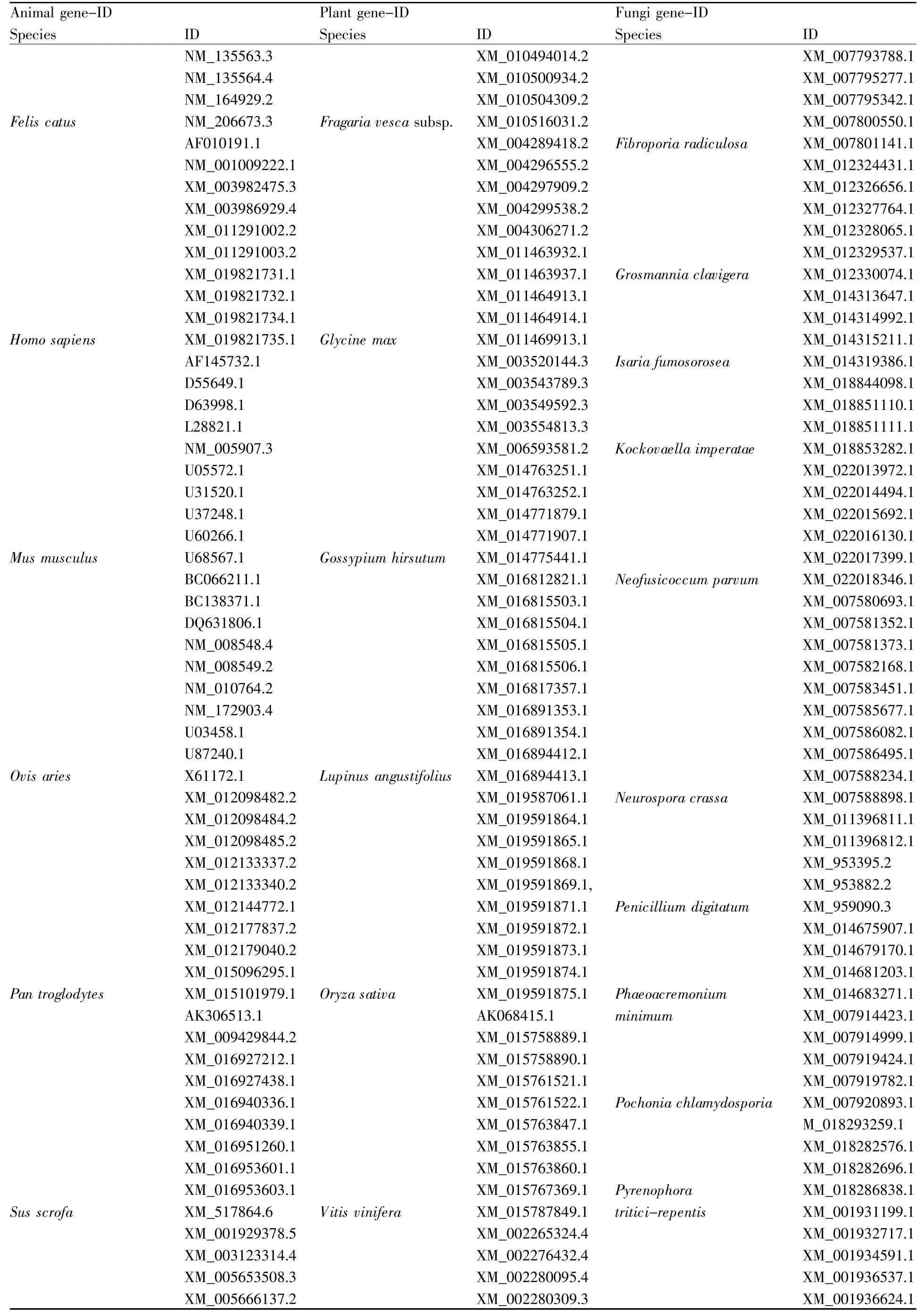
(接上表)
1.2 方法
1.2.1 网络资源
NCBI序列查找:https://www.ncbi.nlm.nih.gov/;
NCBI在线比对:https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi;
结构域分析:http://www.ebi.ac.uk/interpro/search/sequence-search;
Motif分析:http://meme-suite.org/;
蛋白质序列理化性质分析:http://web.expasy org/protparam;.
信号肽分析:http://www.cbs.dtu.dk/services/TMHMM/;
磷酸化位点分析:http://www.cbs.dtu.dk/services/NetPhos;
二级结构分析:https://npsa-prabi.ibcp.fr/cgibin/npsa_automat.pl?page=npsa_sopma.html;
三级结构分析:https://swissmodel.expasy.org/;
酶编号查询网站:http://enzyme.expasy.org/;
KEGG代谢途径查找:http://www.genome.jp/kegg/pathway.html。
1.2.2 分析软件
利用Blast2GO软件进行基因功能预测;使用Vector NTI Adance软件进行序列比对;利用ClustalX、MEGA 6.06软件进行进化树绘制。
2 结果与分析
2.1 α-甘露糖苷酶基因长度分析
Blast2GO软件为常用的生物信息学分析软件,其可通过基因序列查询到该基因的功能、代谢途径、近缘物种等注释信息。通过Blast2GO软件对动物、植物、真菌α-Man基因序列长度进行分析发现,动物α-Man基因序列长度分布在1 300~3 780 bp之间,主要集中分布在2 000~3 500 bp之间,平均长度2 783 bp;植物α-Man基因序列长度分布在200~3 500 bp之间,主要集中分布在1 700~1 800 bp、3 000~3 100 bp 及 3 450~3 500 bp之间,平均长度2 561 bp;真菌α-Man基因序列长度分布在360~3 500 bp之间,主要集中分布在1 190~1 260 bp、1 745~1 825 bp 及 2 870~3 455 bp之间,平均长度2 213 bp。通过基因序列长度分析可知,α-Man基因在不同物种中长度存在明显差异,物种越高等基因平均长度越长。
2.2 α-甘露糖苷酶基因GO功能注释分析
在level-2水平上对动物、植物、真菌α-Man基因进行GO功能注释,分别在生物学过程(biological process,BP)、细胞组分(cellular component,CC)、分子功能(molecular function,MF)3种功能方向得到注释信息。其中,动物α-Man基因在BP中注释到11种功能,CC中注释到4种功能,MF中注释到3种功能;植物α-Man基因在BP中注释到7种功能,CC中注释到7种功能,MF中注释到3种功能;真菌α-Man基因在BP中注释到4种功能,CC中注释到6种功能,MF中注释到3种功能。具体信息如表2所示。
通过Blast2GO软件对α-Man的酶功能进行分析,结果如表3所示。动物、植物及真菌α-Man基因共注释到 EC:3.2.1.24、EC:3.2.1.113、EC:3.2.1.114、EC:3.2.1.130及 EC:3.2.1.101五种酶功能,其中外切酶3种,内切酶2种。由结果可知,真核生物共同注释到GH47家族的Man I和GH38家族的Man II。
2.3 α-甘露糖苷酶进化分析
通过Blast结果可知,动物、植物、真菌都存在GH38、GH47家族基因,选取具有完整结构域的动物、植物及真菌GH38家族氨基酸序列各10条,动物、植物及真菌GH47家族氨基酸序列各10条,共60条α-Man氨基酸序列通过MEGA 6.06软件绘制进化树。结果如图1所示,真菌GH38家族聚在一起与动物GH38家族TtGH38、SsGH38、OaGH38及ClfGH38亲缘关系较近;植物GH38家族聚在一起与动物GH38家族PtGH38、Hs-GH38、DmGH38、MmGH38、FcGH38 及 BtGH38 亲缘关系较近。真菌GH47家族聚在一起,并且相对动物、植物进化较早,动物GH47家族HsGH47、MmGH47及DmGH47与真菌GH47家族亲缘关系较近;植物GH47家族聚在一起与动物GH47家族 BtGH47、TtGH47、PtGH47、ClfGH47、SsGH47、FcGH47及OaGH47亲缘关系较近。由进化树可知,GH47家族在进化上早于GH38家族,而且所有的α-Man氨基酸序列按GH38、GH47家族分开,这表明两种糖基水解酶家族氨基酸序列排布存在较大差异,序列同源性较低。同时发现,真菌GH38、GH47家族在进化关系上早于动物和植物等高等生物。
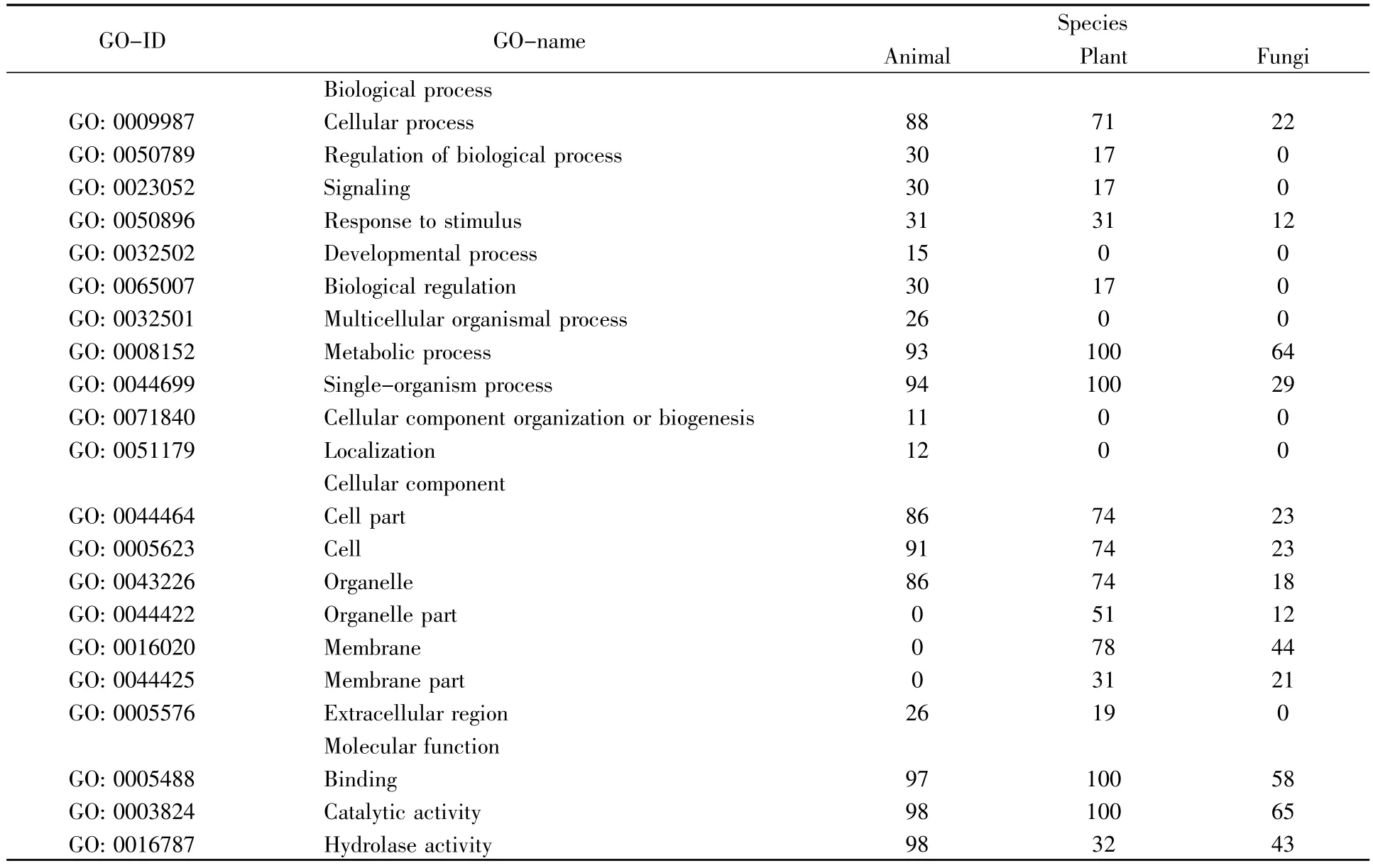
表2 真核生物α-Man基因在level-2水平GO功能注释分布Table 2 The GO function distribution of eukaryotic α-Man genes at level-2

表3 真核生物α-Man基因在level-2水平酶功能注释分布Table 3 The enzyme function distribution of eukaryotic α-Man genes at level-2
2.4 GH38及GH47家族保守性分析
MEME网站常用于氨基酸序列保守性分析,本研究通过MEME网站对动物、植物、真菌的GH38及GH47家族氨基酸序列进行motif查询。结果显示,GH38家族共找到6个motif,如图2所示。其中,所有物种都具有1、5、6三种motif,动物具有6 种 motif,植物具有 1、2、5、6 四种 motif,真菌具有 1、3、4、5、6 五种 motif。GH47 家族也找到 6 个motif,如图3所示,而且所有物种都具有这6种motif。综合保守性分析结果可知,GH47家族比GH38家族保守性更好。
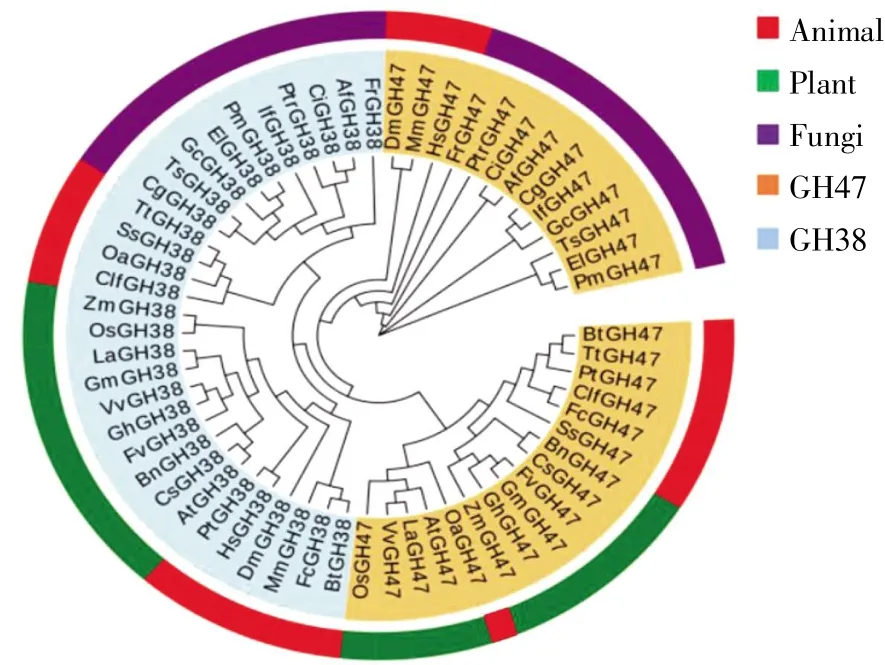
图1 真核生物α-Man系统进化树Fig.1 The phylogenetic tree of eukaryotic α-Man
2.5 GH47家族ERMan I的生物信息学分析
在真核生物N-聚糖修饰过程中存在着一种重要的限速酶——ERMan I,该酶催化水解Man9GlcNAc2生成Man8GlcNAc2,是N-糖基化过程的关键酶。ERMan I属于GH47家族,主要在N-聚糖修饰过程前期发挥作用。由于ERMan I在进化上高度保守,所以该酶在不同真核生物蛋白质N-聚糖修饰过程中具有相同的催化方式[8,9]。为了进一步分析真核生物细胞中的ERMan I,选取拟南芥(Arabidopsis thaliana,At,NM_104037.4)、小鼠(Mus musculus,Mm,NM_008548.4)、酿酒酵母(Saccharomyces cerevisiae,Sc,NM_001181789.3)3种模式生物的ERMan I进行生物信息学分析,分别对其结构域、蛋白质理化性质、磷酸化位点、信号肽、蛋白质二级结构及三级结构进行预测。
2.5.1 ERMan I结构域分析
通过NCBI网站对拟南芥、小鼠、酿酒酵母的ERMan I进行Blast,结果显示 ERMan I为 I型α-甘露糖苷酶(EC 3.2.1.113),属于GH47家族。进一步通过InterProScan程序对拟南芥、小鼠、酿酒酵母ERMan I氨基酸序列进行结构域查找,发现拟南芥ERMan I在氨基酸序列86~536 aa处显示GH47家族结构域,小鼠ERMan I在氨基酸序列189~642 aa处显示GH47家族结构域,酿酒酵母ERMan I在氨基酸序列37~545 aa处显示GH47家族结构域(图4),表明3种物种中的ERMan I都带有GH47家族结构域,属于糖基水解酶47家族。
2.5.2 ERMan I的KEGG代谢途径预测
通过KEGG网站的KEGG PASSWAY Database程序对拟南芥、小鼠、酿酒酵母的ERMan I进行代谢途径预测,结果如图5所示。3种ERMan I都参与N-糖基化过程,其功能都是水解N-聚糖,不同物种中ERMan I发挥作用相同,这一结果说明了ERMan I在功能上高度保守。
2.5.3 ERMan I蛋白的理化性质分析
利用ExPASy网站的ProtParam tool程序对拟南芥、小鼠、酿酒酵母ERMan I的氨基酸序列进行分析。结果显示,3种ERMan I的等电点(isoelectric point,pI)都小于7,这表明ERMan I为酸性蛋白质;3种ERMan I的氨基酸序列亲水指数都为负值,说明氨基酸序列存在亲水区域,进一步结合其脂肪族系数可判定ERMan I为亲水性蛋白质。此外,拟南芥、小鼠、酿酒酵母ERMan I的氨基酸序列不稳定指数都小于40,280 nm时消光系数很大,这表明ERMan I为不稳定蛋白质(表 4)。
2.5.4 ERMan I信号肽预测
信号肽通常情况下是由20~30个疏水性氨基酸残基形成的α-螺旋结构,可引导蛋白质跨膜运输。通过信号肽的预测能够较准确地了解蛋白质的特性。本研究利用TMHMM Server在线预测,对拟南芥、小鼠、酿酒酵母ERMan I氨基酸序列进行信号肽预测,结果如图6所示。3种ERMan I氨基酸序列都具有信号肽,拟南芥ERMan I氨基酸序列第28~47位为跨膜区域,小鼠ERMan I氨基酸序列第37~56位为跨膜区域,酿酒酵母第5~24位为跨膜区域。所有ERMan I氨基酸序列N末端都处于质膜内侧,C末端处于质膜外侧。由此推测真核生物ERMan I蛋白为跨膜蛋白。
2.5.5 ERMan I潜在磷酸化位点预测

图2 GH38家族保守motifFig.2 The distribution of conserved motifs for GH38 family

图3 GH47家族蛋白质保守motifFig.3 The distribution of conserved motifs for GH47 family

图4 拟南芥、小鼠、酿酒酵母ERMan I氨基酸序列GH47家族结构域示意图Fig.4 GH47 family domain diagram of A.thaliana,M.musculus and S.cerevisiae ERMan I amino acid sequences

图5 拟南芥、小鼠、酿酒酵母ERMan I KEGG代谢途径Fig.5 The metabolic pathway of ERMan I proteins from A.thaliana,M.musculus and S.cerevisiae

图6 拟南芥、小鼠、酿酒酵母ERMan I氨基酸序列信号肽预测图Fig.6 Predicition of the gene signal peptide of ERMan I proteins from A.thaliana,M.musculus and S.cerevisiae

表4 拟南芥、小鼠、酿酒酵母ERMan I蛋白的理化性质Table 4 The physico-chemical property of ERMan I proteins from A.thaliana,M.musculus and S.cerevisiae
通过CBS网站的NetPhos 3.1 Server程序对拟南芥、小鼠、酿酒酵母ERMan I的氨基酸序列进行磷酸化位点预测,结果如图7所示。拟南芥ERMan I氨基酸序列中可与ATP磷酸基团结合的氨基酸残基为:Ser 35个、Thr 21个、Tyr 12个。其中,第 114 位 Ser、116 位 Ser、289 位 Thr、357 位Ser、589位Ser的预测分数值接近1,均达到0.95以上。小鼠ERMan I氨基酸序列中可与ATP磷酸基团结合的氨基酸残基为:Ser 14个、Thr 4个、Tyr 8 个。其中,第 33 位 Ser、334 位 Ser、372 位Ser、406 位 Ser、501 位 Thr、707 位 Ser、707 位 Tyr预测分数值接近1,均达到0.95以上。酿酒酵母ERMan I氨基酸序列中可与ATP磷酸基团结合的氨基酸残基为:Ser 24个、Thr 7个、Tyr 9个。其中,第37、40、151、168、222、391、395、419、451、474、492、536、600位Ser预测分数值达到0.95以上,753位Thr、278位Tyr、358位Tyr预测分数值也均达到0.95以上。3种ERMan I都存在多个磷酸化位点,且磷酸化位点多存在于Thr、Tyr、Ser上,这说明不同物种中的ERMan I都可被苏氨酸、酪氨酸、丝氨酸激酶磷酸化。以上结果提示,3种ERMan I蛋白可能通过相应位点的磷酸化来实现其功能的调控。
2.5.6 ERMan I二级结构预测
蛋白质生物学功能与其结构有密切关系,研究蛋白质空间结构对其功能探讨有重要指导作用。利用Prabi网站的SOPMA程序对拟南芥、小鼠、酿酒酵母ERMan I蛋白二级结构进行预测,具体数据见图8。对比拟南芥、小鼠、酿酒酵母ERMan I蛋白二级结构,发现3种ERMan I的二级结构都由α-螺旋(alpha helix)、延伸链(extended strand)、β-转角(beta turn)、无规则卷曲(random coil)4种二级结构原件组成。相比于其他物种,α-螺旋在小鼠ERMan I二级结构中所占比例最多(47.94%),延伸链在拟南芥ERMan I二级结构中所占比例最多(19.29%),β-转角在拟南芥ERMan I二级结构中所占比例最多(10.89%)。
2.5.7 ERMan I三级结构的预测和分析
三级结构是由α-螺旋、β-折叠等二级结构原件进一步折叠行成的一个包裹紧密的立体空间结构,是蛋白质发挥正常生理功能的基础。本研究参考王兆松等[10]对氨基酸序列的预测方法,运用SWISS-MODEL程序对3种真核生物ERMan I蛋白氨基酸序列的三级结构进行同源建模,结果如图9所示。3种ERMan I由α-螺旋、β-折叠等二级结构原件共同折叠形成中空桶形结构,其中空部分存在Ca2+结合部位,拟南芥ERMan I第145位 Trp、217位 Arg,小鼠 ERMan I第 164位 Trp、166位 Trp、167位 Ala、173位 Ile,酵母 ERMan I第 135位 Asp、138位 Ala、191位 Asn、194位 Try,形成ERMan I蛋白的活性部位,它们识别Man9GlcNAc2并与之结合,进行N-糖基化修饰过程。
3 讨论
3.1 GH38、GH47家族生物信息学分析
在基因长度方面,α-Man基因长度存在物种差异,物种越高等基因平均长度越长。基因长度决定了基因功能,基因长度的增加意味着功能的增加[11]。以ERMan I为例,其在动植物等高等真核生物中,不仅特异性水解Man9GlcNAc2中的一个α-1,2糖苷键,同时参与了ERAD降解途径,但是在酵母等低等真核生物中,ERMan I不能参与ERAD过程[12]。这说明随着物种的进化,α-Man基因长度也随之变化,其编码蛋白质的功能也有所改变。

图7 拟南芥、小鼠、酿酒酵母ERMan I氨基酸序列磷酸化位点分析图Fig.7 Analysis of phosphorylation of ERMan I proteins from A.thaliana,M.musculus and S.cerevisiae

图8 ERMan I蛋白二级结构组成图Fig.8 The composition diagram of ERMan I protein secondary structure
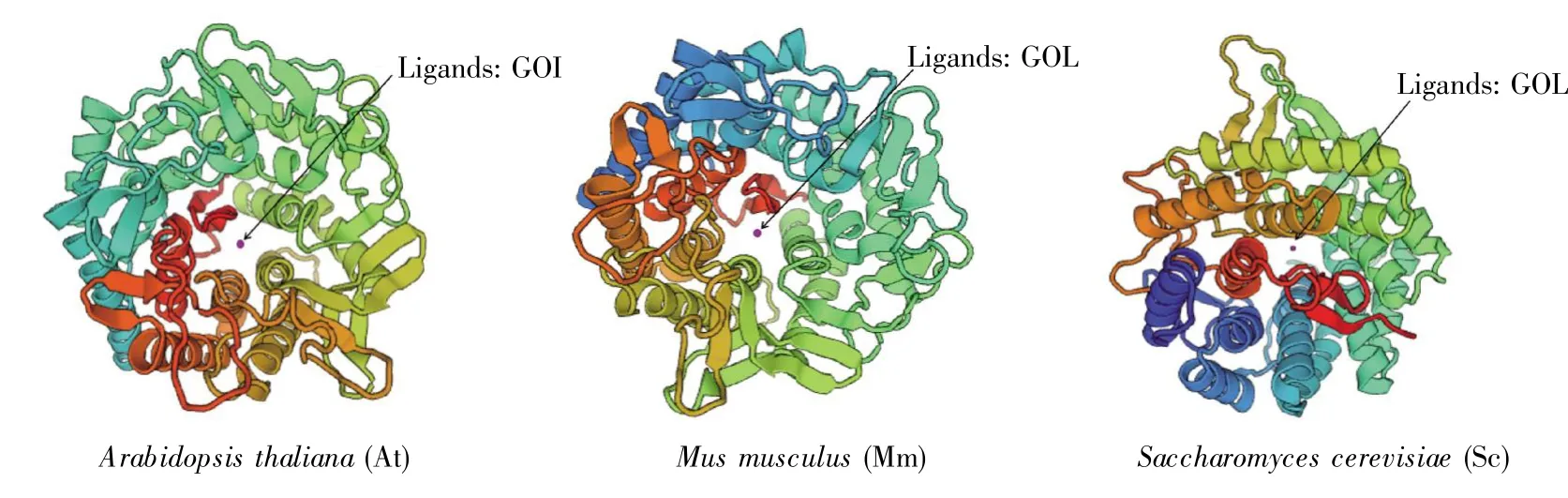
图9 ERMan I蛋白三级结构预测图Fig.9 The tertiary structure prediction of ERMan I proteins
在功能方面,α-Man在生物进程中主要表现为细胞过程、代谢过程;在细胞组分中表现为细胞和细胞部分,在分子功能中表现为结合功能、催化活性及水解酶活性。这说明该酶编码基因定位于胞内,在细胞内部起催化作用,在基础代谢和蛋白质合成中表达量比较多。α-Man属于N-糖基化过程的关键酶,参与基础代谢过程,其定位于内质网、高尔基体等细胞器[13],这些信息都与GO功能注释结果一致。文中所有真核生物α-Man基因都注释到Man I、Man II两种酶功能,即所有真核生物都具有GH47、GH38家族成员。这一结果与GH47、GH38家族所参与的代谢过程有关,真核生物N-聚糖修饰过程分为高甘露糖修饰、复杂甘露糖合成两种,其中GH47、GH38家族共同参与的高甘露糖修饰过程在所有真核生物细胞一致,代谢过程的相同决定了酶种类的相同[6,12]。
在进化方面,GH47家族早于GH38家族,低等动物早于高等动物,同家族基因同源性更高,同家族中相同物种同源性更高,这是符合进化规律的。对比GH38、GH47家族motif查询结果可知,不同物种GH47家族的氨基酸保守基序相似度更高。现有研究有两点证明了这一结论:第一,家族成员种类上的保守与氨基酸结构的保守是对应的,GH47家族只有ERMan I和GM I两种I型α-甘露糖苷酶,并且这两种酶在所有真核生物中都有发现。而GH38家族中的酶种类更多,不同物种中所包含的酶种类不同。例如高尔基体Ⅱx型α-甘露糖苷酶,该酶由Man2a2基因编码,与高尔基体Man II的DNA序列具有高度相似性,但其只存在于动物细胞当中[14];第二,在功能上的保守源自于氨基酸结构的保守,GH47家族功能高度保守,其所有成员都是水解α-1,2-甘露糖苷键,而GH38家族成员可水解α-1,2/1,6两种甘露糖苷键[15]。
3.2 ERMan I生物信息学分析
在功能方面,ERMan I属于GH47家族,于N-聚糖修饰过程初期发挥功能,在不同物种中功能相同,这不仅说明ERMan I在功能上高度保守,也是对GH47家族高度保守的证明。已有研究报道,真核生物ERMan I普遍存在于动物、植物及真菌的内质网中,其具有特异性强、保守性高等特点,所有物种中的ERMan I都是水解Man9GlcNAc2中的 α-1,2-甘露糖苷键,产物为 Man8GlcNAc2[16~18]。
在结构方面,不同物种中ERMan I二级结构组成相同,都包含α-螺旋、延伸链、β-转角、无规则卷曲4种结构,其中α-螺旋为主要的二级结构原件。三级结构预测结果与二级结构预测结果相符,3种ERMan I的三级结构均是由α-螺旋、延伸链、β-转角、无规则卷曲等二级结构原件共同折叠形成的桶状结构,桶状结构中心为Ca2+结合区域,其可与Ca2+结合形成聚糖复合物,这符合ERMan I的结构特征。ERMan I的催化过程对Ca2+有依赖性,研究证明,脱氧野尻霉素可抑制ERMan I的活性,这种抑制是由于脱氧野尻霉素与内质网中的Ca2+不可逆结合,造成内质网腔内Ca2+平衡紊乱,致使ERMan I无法正常与Ca2+结合,造成其水解功能丧失[19]。
在蛋白质理化性质方面,ERMan I为酸性蛋白质,稳定性较差,多肽链表现为亲水性。信号肽预测结果显示,不同物种中ERMan I氨基酸序列都存在跨膜α-螺旋,这与二级结构预测结果一致。研究证明,ERMan I蛋白在细胞质中合成后,经跨膜运输至内质网,催化N-聚糖的修饰过程[20]。磷酸化位点预测结果显示,不同物种中ERMan I都存在磷酸化位点,可通过磷酸化实现功能上的调控,但现有研究尚未发现ERMan I在催化过程中伴随有磷酸化的发生,这一点需继续研究。
综上分析可知,本研究对GH38、GH47家族及ERMan I开展的生物信息学分析所获得的预测结果真实可靠,对于深入研究α-Man对N-聚糖修饰过程的调控作用、通过生物技术手段延长果实储藏时间、治疗由于α-Man功能异常所造成的疾病具有重要的指导意义。