新型基因编辑技术发展及在植物育种中的应用
2018-06-06王艳芳苏婉玉曹绍玉张应华许俊强云南农业大学云南省滇台农业特色产业化工程研究中心昆明650201
王艳芳,苏婉玉,曹绍玉,张 琳,吕 霞,张应华,许俊强(云南农业大学 云南省滇台农业特色产业化工程研究中心,昆明 650201)
目前,以基因编辑(Gene editing)为基础的反向遗传学技术是基因改造与研究的手段之一[1],促进生物学研究的迅猛发展。近年来,以序列特异核酸酶(Sequence-specific nucleases,SSNs)为工具的基因编辑技术已在全世界掀起研究热潮。2012年《Science》将其列为年度十大科学进展之一;以TALEN为代表的SSNs被誉为“基因组巡航导弹”;2013年基因编辑技术的新星CRISPR/Cas9又被《Science》列为年度十大科学进展之一;2014年《Nature Methods》将基因编辑技术评为过去10 a对生物学研究最具影响力的10项研究方法之一[2]。2016年NgAgo基因编辑技术被首次公布,理论上更加先进、高效但技术尚未成熟且不能被多数试验证明[3]。
基因组编辑通过特异性结构识别目标基因,改造基因DNA来改变基因组中的特定基因,从而定点修复致病基因[4]或定点插入一个基因或DNA元件[5-6];同时该技术也可对正常基因进行编辑,实现基因的敲除、敲入、结构变异等修饰,从而研究其在体内的功能。
1 人工核酸内切酶介导的新型基因编辑技术原理
人工核酸内切酶包含两个结构域:DNA结合域和内切酶结构域。第1个结构域负责识别特定的DNA位点;后者负责切割DNA。核酸内切酶在特定位点切割使DNA双链断裂(DSB),诱发细胞产生非同源末端连接(NHEJ)和同源重组(HR)修复机制对受损DNA实现修复,因而在特定位点插入、删除或缺失,产生特定位点的基因突变,从而实现对基因的定点修饰[7-9]。
1.1 ZFN
ZFNS是最先用于基因编辑技术的特异性人工核酸酶。ZFN是人工设计且具有锌指结构的蛋白,由锌指蛋白(Zinc finger protein,ZFP)构成的特异性DNA结合域和特异性核酸内切酶FokⅠ构成[10-11]。ZFN的N末端为锌指蛋白DNA结合域,可识别特定的DNA序列;其C末端为FokⅠ切割结构域。FokⅠ与锌指结构域相连,当FokⅠ形成二聚体时才具备酶切活性[12],ZFP在FokⅠ的指导下识别靶基因位点将DNA双链切开(图1),诱导细胞的修复机制对损伤DNA进行自主修复[13]。
1.2 TALENS
TALEN是由转录激活因子样效应物(Transcription activator-like effector,TALE)替代ZF作为DNA结合域,与FokⅠ切割域组成的基因编辑技术。与ZFN相似,由TALE蛋白DNA结合域和FokⅠ两部分发挥作用,前者负责目标序列的特异性识别,后者进行靶位点的切割[15]。

图1 ZFN类工程核酸酶的作用原理[14]
TALE蛋白是植物病原体黄单胞菌分泌的一类效应蛋白因子,能够特异结合DNA[15-16]。TALE蛋白由3部分组成[17],第1部分来自于天然TALE蛋白,其N端含有Ⅲ型分泌系统所需的分泌和易位信号,即易位结构域;第2部分是DNA结合结构域,是一段重复氨基酸序列,由多个长度不同的TALE单元组成,且每34个氨基酸组成1个TALE单元,其中高度保守的氨基酸有32个,只有第12、13位氨基酸是变化的,能够特异地结合1个碱基序列,因此这两个氨基酸又被称为重复变异双残基(Repeat variant diresidue,RVD)。不同的RVD能够特异性识别1种或多种碱基[17];第3部分为天然TALE蛋白的C端,含有1个核定位信号和转录激活域,可帮助TALE蛋白定位于细胞核,同时发挥转录激活作用[15,18-20]。FokⅠ的切割活性依赖于其切割结构域的二聚化,两个FokⅠ发生二聚化后才会对DNA双链进行切割(图2),从而诱发细胞DNA损伤后的两种修复过程[21]。
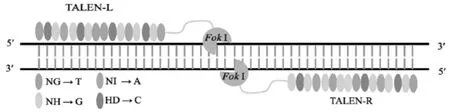
图2 TALEN类工程核酸酶的作用原理[14]
1.3 CRISPR/Cas
CRISPR系统发现于细菌自身免疫系统的过程中,主要存在细菌和古生菌中。CRISPR系统由3部分构成:CRISPR基因座(CRISPR locus)、前导序列(Leader sequence,LS) 以及一类基因家族Cas基因(Casgenes)。CRISPR基因座由一些正向重复序列(Direct repect,DR)和非重复间隔序列(Spacer)两部分间隔排列组成。Cas基因是CRISPR位点附近的一类家族基因,主要承担核酸酶切割作用。1个Cas基因簇往往包含4~10个保守基因[22],它表达出的Cas蛋白在免疫系统过程中发挥重要作用。前导序列由300~500 bp碱基组成,位于CRISPR的5′端,与第1个重复序列直接相连,是相对保守的AT富集区[23]。
CRISPR/Cas系统工作的核心在于人工设计的sgRNA(Single-guide RNA),sgRNA是由crRNA(CRISPR RNA)与反式激活crRNA即tracrRNA(Trans-activating CRISPR RNA)两种非编码RNA通过碱基配对结合而成[24]。其作用原理:核酸内切酶Cas在sgRNA的引导下,在目标基因的特定区域对特定DNA进行定点切割,造成双DNA链断裂,细胞实行自主修复机制,从而实现特定位点的定向编辑[25]。由于Cas基因的不同,CRISPR/Cas系统可分为3种类型:Ⅰ型、Ⅱ型和Ⅲ型。其中Ⅰ型和Ⅲ型因使用较复杂,应用较少,而Ⅱ型最为简单,研究的也较多,也是目前最常用的人工核酸酶。CRISPR/Cas9就是最常用的一种,Cas9酶在sgRNA的引导下定向切割目标位点,产生DSB,从而实现基因的定向编辑(图3)。
1.4 新型基因编辑技术的比较
从ZFN到CRISPR/CAS技术来看,在基因编辑的过程中每种技术都具有自己的特点(表1)。

图3 CRISPR/Cas9类工程核酸酶的作用原理[14]

表1 新型基因编辑技术的差异比较
2 新型基因编辑技术在植物中应用
备受欢迎的新型基因编辑技术已成功在多种植物中得到普遍运用,不仅在模式植物中成熟应用,且随着不断发展、完善,该技术逐步走向成熟。近年来也在非模式植物如小麦、大豆、番茄等中得到广泛应用。
2.1 模式植物
2.1.1 拟南芥 2005年,ZFN初次诱导其染色体基因发生突变,提高后代突变率,且有一半以上突变体可以稳定遗传至下一代[26]。利用雌激素诱导系统对基因进行高效突变,获得丙烯醇抗性突变体 adh1(alcoholdehydrogenase1)和种皮不含花青素突变体 tt4(transparenttesta),其中有69%的 adh1突变及33%的 tt4突变可稳定遗传给后代[27]。Osakabe等[28]对拟南芥的ABSISCICACIDINSENSITIVE4基因进行突变,获得预期表型,如对脱落酸及葡萄糖的不敏感性的abi4(ABSISCIC ACID INSENSITIVE 4)突变体。种种例子可证明,ZFN可提高植物基因打靶效率,在今后研究中有望克服植物基因组靶向修饰的难题[29]。TALEN在拟南芥的原生质体中,首次实现对内源靶基因的敲除[30]。Cermak等[31]利用一套基于Golden gate方法组装TALEN,对拟南芥原生质体中ADH1(alcoholdehydrogenase1 ) 基因进行高效定点突变,得到6个独立的突变体。针对TT4 (transparenttesta4 )基因设计间隔230 bp的两个sg RNAs,实现大片段缺失[32]。Feng等[33]对拟南芥耐油菜素类固醇基因BRIL(BRASSINOSTEROIDINSENSITIVE1) 和赤霉素基因GAI(GIBBERELLICACIDINSENSITIVE) 进行编辑,获得50株T1代转基因植株,大部分转基因植株在早期生长阶段表现出多种生长表型,其中BRIL转基因植株有一半表现出生长阻滞、叶片卷曲,GAI转基因植株中有1/4的植株外形矮小。
2.1.2 烟草 利用ZFN对烟草细胞的单个基因序列进行突变,使其耐除草剂[34]。Wright等[35]将部分序列缺失的Gus:NptⅡ报告基因(β-葡糖酸糖苷酶:新霉素磷酸转移酶)随机整合到烟草基因组的不同位点,并修复该基因使其能够正常表达,不仅获得转基因植物,也证明ZFNs对基因有修复功能。对烟草原生质体乙酰乳酸合成酶基因SuRA(tobaccoacetohydroxyacidsynthase) 和SuRB编辑后,内源基因替换效率可达40%,且对咪唑啉酮和磺酰脲类除草剂具备抗性[36]。利用TALEN技术定向敲除体内eIF4E-6(eu-karyotictranslationinitiationfactors4E)基因,提高其对马铃薯Y病毒(PVY) 的抗性[37]。利用CRISPR/Cas9 技术编辑转基因本氏烟草16C (16C transgenic Nicotiana benthamiana,16C-NB)的GFP(greenfluorescentprotein)基因,用PCR-RE (polymerase chain reaction-restrictionenzyme)的方法检测其突变体,成功建立本氏烟草CRISPR/Cas9基因敲除瞬时表达系统[38]。针对其NbPDS(Nicotianabenthamianaphytoenedesaturase)基因设计间隔50 bp的两个sg RNAs,实现基因大片段的缺失、突变[39]。利用CRISPR/Cas9技术成功的在烟草中敲除eIF4E-6基因,从而提高对PVY病毒的抗性[40]。
2.1.3 水稻 Shan等[41]将两对TALENs转入水稻原生质体及愈伤组织中,实现OsBADH2a、b(Oryzasativabetainealdehydedehydrogenase2a,b)两个基因的同时突变。随后又对水稻不同性状的基因进行突变,包括香味、大穗、半矮化等基因,直接用于水稻育种。利用TALENs使水稻感病基因 Os SWEET14的启动子发生突变,在不影响其生长发育的条件下提高水稻对白叶枯病的抗性[42]。利用CRISPR/Cas系统定点敲除其PDS(phytoenedesaturase)基因,基因突变的效率高于转基因水稻的[43]。定点突变水稻基因组中已知的苯达松(Bentazone)抗性基因CYP81A6(cytochromeP450monooxgenase),突变效率约为2.73%,可为以后苯达松敏感突变体育种提供材料[44]。在水稻品种改良中,利用CRISPR技术分别对水稻OsBADH2(Oryzasativabetainealdehydedehydrogenase2)和LOC_Os10g05490编码区进行编辑,获得水稻香米株系和耐冷性株系[45-46]。
2.1.4 玉米 玉米内源基因IPK1编码其体内植酸生物合成的关键酶,Shukla等[47]利用ZFN实现对该基因的插入失活,使植酸合成途径受阻,从而降低植酸含量。之后又将除草剂抗性基因PAT插入玉米内源基因IPK,获得抗除草剂植株[47]。将天然存在的优良糯玉米性状通过CRISPR技术直接敲入高产杂交品种中,获得优良品种的同时也解决糯玉米产量低的问题[48]。
2.2 在其他植物中应用
利用ZFN成功地对大豆基因组中高度重复序列进行定点敲除[49]。在衣藻中实现用ZFN敲除内源基因[50]。基于Golden Gate方法构建的TALEN技术成功且高效地敲除短柄草中的一系列基因[41]。通过TALEN技术成功敲除大麦中的grf(growthhormonereleasingfactor)基因,不仅效率高且可稳定地遗传给后代[51]。利用TALEN敲除两个脂肪酸去饱和酶基因FAD2-1a(TheFattyAcidDesaturase2-1a)和FAD2-1b(TheFattyAcidDesaturase2-1b),使大豆单饱和脂肪酸含量大大提高,延长保质期[52]。通过TALEN技术定向突变土豆Vinv(VacuolarInvertase)基因,改进土豆的冷藏性及加工品质[53]。在小麦中利用TALEN获得抗白粉病的小麦株系[54]。利用TALEN技术编辑1个马铃薯品系基因,降低天门冬酰胺和单糖的含量的同时也提高其耐储藏性[55]。
作为基因组编辑技术的新方向,CRISPR已被广泛运用。运用CRISPR/Cas9系统可对多倍体小麦基因进行多位点同时突变或单位点定向突变[54]。在甜橙中通过农杆菌介导法转入Cas9以及合成的sgRNA,编辑靶向CsPDS(Citrussinensisphytoenedesaturase) 基因,成功使该基因发生突变[56]。成功地在香蕉体内定点敲除内源基因MaPDS(Musaphytoenedehydrogenase),获得突变体株系,为以后进一步研究奠定基础[57]。对杨树(Populusto-mentosa)的PDS基因进行多靶点修饰,在 T0代转化体获得白化突变体[58-59]。Fan等[58]设计4个不同的gRNA分别编辑毛白杨不同基因组位点的八氢番茄红素脱氢酶基因8PtoPDS(poplarphytoenedesaturasegene)以及相邻的基因,农杆菌介导转化后,转基因杨树表现出明显的白化表型。利用CRISPR 技术删除双孢菇(Agaricusbisporus)中编码导致褐变的多酚氧化酶(polyphenoloxidase,PPO)基因家族,PPO酶的活性降低30%,使双孢菇延缓褐变或抗褐变[60]。CRISPR/Cas9 系统成功地应用于编辑大豆的乙酰乳酸合成基因1ALS1(Acetylatedlacticacidsynthesis)并获得氯磺隆抗性基因[61]。通过CRISPR系统,对调控番茄果实成熟基因RIN(Ripeninginhibitor) 进行编辑,延长保质期[62];2017年,对番茄体内SIACS2(SolanumLycopersicum1-aminocyclopropane-1-carboxylicacidsynthase)进行修饰,SIACS2是番茄系统Ⅱ乙烯合成的限速酶,从而调控系统Ⅱ乙烯的过量表达,可避免番茄过熟[63]。利用CRISPR/Cas9技术对控制番茄叶片形态的 SlAGO7基因进行敲除,将突变植株的叶片与野生型番茄叶片相比,其有些窄小甚至呈针状,由此可证明番茄SlAGO7基因的功能[64]。此外,CRISPR/Cas9系统也被广泛应用于如高粱[65]、马铃薯[66-70]、西瓜[71]、黄瓜[72]等植物中。
3 展 望
近年来,在基因编辑技术不断发展的过程中,尽管每种技术不可避免存在一些有待解决和发展的问题,如ZFN-如何增强锌指蛋白的特异性以及如何精确调控ZFN的表达;TALEN-如何避免重复构建载体和缩短其重复序列避免序列太长而限制应用等;CRISPR/Cas-怎样降低严重的脱靶效应和解决PAM(protospacer adjacent motif)限制打靶位点的问题;但这些人工核酸酶所介导的新型基因编辑技术凭借其自身独特的优势仍取得突飞猛进的发展,且极大地推进科学研究的进展。
新型基因编辑技术存在一系列问题,尤其是各基因编辑技术的脱靶效应,当然人们也在不断寻求解决的方法。例如,可针对不同目标基因设计不同的ZFP蛋白组合、TALE蛋白组合以及sgRNA,增强其识别特异性的同时也可降低脱靶效应;各基因编辑技术在设计编辑靶位点时选择特异性较高的位点,最大程度上降低脱靶效应;在ZFN和TALEN应用中务必保证核酸内切酶FokⅠ为二聚体,这样不仅具有切割作用还可降低脱靶效应;而在CRISPR/Cas应用时,利用Cas9单切口酶和成对的sgRNA以及对sgRNA的正确选择在基因组编辑中可能对脱靶效应有所帮助;另外,针对那些已出现脱靶的位点进行研究,找出其原因和解决措施;当然也可针对切割的目标片段选择合适的修复途径或者根据自身所能切割的片段大小来降低脱靶效应等。更值得一提的是最近《Plant Cell》报道一篇有关解决植物基因组编辑的整体方案。其核心是存在于植物体内的一种多功能基因编辑“工具箱”,该“工具箱”以TALEN和CRISPR/Cas两个系统为依据,不仅可实现同时编辑多个基因和删除大片断基因,还可定点修饰和编辑单子叶和多子叶植物的基因等,且目前这种方案已成功在西红柿、烟草、小麦、大麦以及苜蓿中得到应用[73]。由此可见,通过新型基因编辑技术对植物体内重要的基因进行定点修饰及编辑,如基因敲除、敲入或替换等,可以实现对植物品种和性状的精准改良,培育出适合各种不良环境条件和人们所需求的植物新品种,促进农业生产快速发展。
总而言之,新型基因编辑技术在未来科学发展的道路上会扮演越来越重要的角色,也会成为许多领域研究与应用的重要工具。
参考文献Reference:
[1] VOYTAS D F.Plant genome engineering with sequence-specific nucleases[J].AnnualReviewofPlantBiology,2013,64(1):327-350.
[2] 单齐伟,高彩霞.植物基因组编辑及衍生技术最新研究进展[J].遗传,2015,37(10):953-973.
SHAN Q W,GAO C X.Research progress of genome editing and derivative technologies in plants[J].Hereditas,2015,37(10):953-973.
[3] GAO F,SHEN X Z,JIANG F,etal.DNA-guided genome editing using theNatronobacteriumgregoryiArgonaute[J].NatureBiotechnology,2016,34(7):768-773.
[4] CHAMBERLAIN J K,Schwarze U,Ussell K D,etal.Genetargeting in stem cells from individuals with osteogenesis imperfecta[J].Science,2004,303(5661):1198-1201.
[5] JOHNSON L,MERCER K,JACKS T,etal.Somatic activation of the K-ras oncogene causes early onset lung cancer in mice[J].Nature,2001,410(6832):1111-1116.
[6] BEARD C,HOCHEDLINGER K,JAENISCH R,etal.Efficient method to generate single-copy transgenic mice by site-specific integration inembryonic stem cells[J].Genesis,2006,44(1):23-28.
[7] SYMINGTON L S,GAUTIER J.Double-strand break end resection and repair pathway choice[J].AnnualReviewofGenetics,2011,45(1):247-271.
[8] WOOD A J,LO T W,ZEITLER B,etal.Targeted genome editing across species using ZFNs and TALENs[J].Science,2011,333(6040):307.
[9] LO T W,PICKLE C S,LIN S,etal.Precise and heritable genome editing in evolutionarily diverse nematodes using TALENs and CRISPR/Cas9 to engineer insertions and deletions[J].Genetics,2013,195(2):331-348.
[10] KPM Y G,CHA J,CHANDRASEGARAN S.Hybrid restriction enzymes:zinc finger fusions toFokⅠ cleavage domain[J].ProceedingsoftheNationalAcademyofSciences,1996,93(3):1156-1160.
[11] 王 令,张存芳,张智英.锌指核酸酶在基因组靶向修饰中的应用[J].中国生物化学与分子生物学报,2009,25(7):585-589.
WANG L,ZHANG C F,ZHANG ZH Y.Application of zinc finger nucleases in genome targeting modification[J].ChineseJournalofBiochemistryandMolecularBiology,2009,25(7):585-589.
[12] BITINAITE J,WAH D A,AGGARWAL A K,etal.FokⅠ dimerization is required for DNA cleavage[J].ProceedingsoftheNationalAcademyofSciences,1998,95(18):10570-10575.
[13] KIM S,LEE M J,KIM H,etal.Preassembled zinc-finger arrays for rapid construction of ZFN[J].NatureMethods,2011,8(1):7.
[14] 周想春,邢永忠.基因组编辑技术在植物基因功能鉴定及作物育种中的应用[J].遗 传,2016,38(3):227-242.
ZHOU X CH,XING Y ZH.The application of genome editing in identification of plant gene function and crop breeding[J].Hereditas,2016,38(3):227-242.
[15] MOSCOU M J,BOGDANOVE A J.A simple cipher governs DNA recognition by TAL effectors [J].Science,2009,326(5959):1501.
[16] BOCH J,SCHOLZE H,SCHOMACK S,etal.Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors[J].Science,2009,326(5959):1509-1512.
[17] 杨发誉,葛香连,谷 峰.新型靶向基因组编辑技术研究进展[J].中国生物工程杂志,2014,34(2):98-103.
YANG F Y,GE X L,GU F.Reserach progress of new targeted gene editing technique[J].ChinaBiotechnology,2014,34(2):98-103.
[18] MUSSOLINO C,CATHOMEN T.TALE nucleases:tailored genome engineering made easy[J].CurrentOpinioninBiotechnology,2012,23(5):644-650.
[19] BONAS U,STALL R E,STASKAWICZ B,etal.Genetic and structural characterization of the avirulence gene avr Bs3 fromXanthomonascampestrispv.vesicatoria[J].Molecular&GeneralGenetics,1989,218(1):127-136.
[20] KAY S,HAHN S,MAROIS E,etal.Abacterial effector acts as a plant transcription factor and induces a cell size regulator[J].Science,2007,318(5850):648-651.
[21] URNOV F D,REBAR E J,HOLMES M C,etal.Genome editing with engineered zinc finger nucleases[J].NatureReviewsGenetics,2010,11(19):636-646.
[22] GRISSA I,VERGNAUD G,POURCEL C.The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J].Bioinformatics,2007,8(1):172.
[23] JANSEN R,VANEMBDEN J D,GAASTRA W,etal.Identificationof a novel family of sequence repeats among prokaryotes[J].Omics-aJournalofIntegrativeBiology,2002,6(1):23-33.
[24] JINEK M,CHYLINSKIh K,FONFARA I,etal.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J].Science,2012,337(6096):816-821.
在低压微电网系统中,线路阻抗通常呈现阻性,而微电网孤岛运行时呈低压特性,因此本文系统为阻性,可忽略虚拟电感。本文在微电网的分布式电源变流器中添加虚拟电阻控制回路,使系统呈现阻性,具体如图2所示。图2中,两个简化变换器框图表示两个DG单元带有虚拟电阻并考虑线路电阻的戴维南等效简化模型。
[25] SAPRANAUSKAS R,GASIUNAS G,FREMAUX C,etal.TheStreptococcusthermophilesCRISPR/Cas system provides immunity inEscherichiacoli[J].NucleicAcidsResearch,2011,39(21):9275-9282.
[26] LLOYD A,PLAISIER C L,CARROLL D,etal.Targeted mutagenesis using zinc-finger nucleases inArabidopsis[J].ProceedingsoftheNationalAcademyofSciences,2005,102(6):2232-2237.
[27] ZHANG F,MAEDER M L,UNGERWALLAC E,etal.High frequency targeted mutagenesis inArabidopsisthalianausing zinc-finger nucleases[J].ProceedingsoftheNationalAcademyofSciences,2010,107(26):12028-12033.
[28] OSAKABE K,OSAKABE Y,TOKI S.Site-directed mutagenesis inArabidopsisusing custom-designed zinc finger nucleases[J].ProceedingsoftheNationalAcademyofSciences,2010,107(26):12034-12039.
[29] 李 乐,朱晔荣,王 勇.植物基因打靶研究进展[J].生物学通报,2007,42(12):8-10.
LI L,ZHU Y R,WANG Y.Advances in gene targeting of plants[J].BulletinofBiology,2007,42(12):8-10.
[30] 张金脉,任兆瑞.TALENs:一种新的基因定点修饰技术[J].生命科学,2013,25(1):126-132.
[31] CERMAK T,DOYLE E L,CHRISTIAN M,etal.Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J].NucleicAcidsResearch,2011,39(17):7879.
[32] MAO Y F,ZHANG H,XU N F,etal.Application of the CRISPR-Cas system for efficient genome engineering in plants[J].MolecularPlant,2013,6(6):2008-2011.
[33] FENG Z Y,ZHANG B T,DING W N,etal.Efficient genome editing in plants using a CRISPR/Cas system[J].CellResearch,2013,23(10):1229-1232.
[34] CAI C Q,DOYON Y,AINLEY W M,etal.Targeted transgene integration in plant cells using designed zinc finger nucleases[J].PlantMolecularBiology,2009,69(6):699-709.
[35] WRIGHT D A,THIBODEAU-BEGANNYH S,SANDER J D,etal.Standardized reagents and protocols for engineering zinc finger nucleases by modular assembly[J].NatureProtocols,2006,1(3):1637-1652.
[36] TOWNSEND J A,WRIGHT D A,WINFREY R J,etal.High-frequency modification of plant genes using engineered zinc-finger nucleases[J].Nature,2009,459(7245):442-445.
[37] 宋琳娜,杨鹏九,万秀清,等.应用TALENs技术定向敲除烟草eIF4E-6 基因[J].安徽农业科学,2017,45(4):142-146.
SONG L N,YANG P J,WAN X Q,etal.The directional knockout ofeIF4E-6 gene using TALENs technique[J].JournalofAnhuiAgricultureSciences,2017,45(4):142-146.
[38] 杨学飞,刘亚娟,高 原,等.CRISPR/Cas9系统在本氏烟草基因敲除中的应用[J].分子植物育种,2017,15(1):30-38.
YANG X F,LIU Y J,GAO Y,etal.The application of CRISPR/Cas9 system in gene knock-out ofNicotianabenthamiana[J].MolecularPlantBreeding,2017,15(1):30-38.
[39] BELHAJ K,CHAPARRO-GARCIAH A,KAMOUN S,etal.Plant genome editing made easy:targeted mutagenesis in model and crop plants using the CRISPR/Cas system[J].PlantMethods,2013,9(1):39.
[40] 潘红杏,刘 侠,万秀清,等.利用CRISPR-Cas9基因组编辑技术定向敲除烟草eIF4E-6 基因[J].分子植物育种,2017,15(2):538-544.
PAN H X,LIU X,WAN X Q,etal.Directional knockout ofeIF4E-6 gene using CRISPR-Cas9 genome editing technique[J].MolecularPlantBreeding,2017,15(2):538-544.
[41] SHAN Q W,WANG Y P,CHEN K L,etal.Rapid and efficient gene modification in rice andBrachypodiumusing TALENs[J].MolecularPlant,2013,6(4):1365-1368.
[42] LI T,LIU B,SPALDING M H,etal.High-efficiency TALEN-based gene editing produces disease-resistant rice [J].NatureBiotechnology,2012,30(5):390-392.
[43] SHAN Q,WANG Y,LI J,etal.Targeted genome modifi cation of crop plants using a CRISPR-Cas system [J].NatureBiotechnology,2013,31(8):686-688.
[44] 姜明君,常振仪,卢嘉威,等.应用TALEN技术定点突变水稻苯达松抗性基因CYP81A6[J].农业生物技术学报,2016,24(8):1225-1232.
JIANG M J,CHANG ZH Y,LU J W.TALEN-mediated mutagenesis of bentazone resistance geneCYP81A6 in rice[J].JournalofAgriculturalBiotechnology,2016,24(8):1225-1232.
[45] 邵高能,谢黎虹,焦桂爱,等.利用CRISPR/CAS9技术编辑水稻香味基因Badh2[J].中国水稻科学,2017,31(2):216-222.
SHAO G N,XIE L H,JIAO G A,etal.CRISPR/CAS9-mediated editing of the fragrant geneBadh2 in rice[J].ChineseJournalofRiceScience,2017,31(2):216-222.
[46] 沈春修.水稻LOC_Os10g05490位点冷胁迫条件下表达分析CRISPR/CAS定向编辑[J].浙江农业学报,2017,29(2):177-185.
SHEN CH X.CRISPR/ Cas9 editing and expression analysis of LOC_Os10g05490 in rice under cold stress[J].ActaAgriculturaeZhejiangensis,2017,29(2):177-185.
[47] SHUKLA V K,DOYON Y,MILLER J C,etal.Precise genome modification in the crop speciesZeamaysusing zinc-finger nucleases[J].Nature,2009,459(7245):437-441.
[48] LEDFORD H.Gene-editing surges as us rethinks regulation[J].Nature,2016,532(7598):158-159.
[49] CURTIN S J,ZHANG F,SANDER J D,etal.Targeted mutagenesis of duplicated genes in soybean with zinc-finger nucleases [J].PlantPhysiology,2011,156 (2):466-473.
[50] SIZOVA I,GREINER A,AWASTHI M,etal.Nuclera gene targeting inChlamydomonasusing engineered zinc-finger nucleases[J].PlantJournal,2013,73(5):873-882.
[51] CHEN K,SHAN Q,GAO C.An efficient TALEN mutagenesis system in rice[J].Methonds,2014,69(1):2-8.
[52] HAUN W,COFFMAN A,CLASEN B M,etal.Improved soybean oil quality by targeted mutagenesis of the fatty acid desaturase 2 gene family[J].PlantBiotechnolJournalogy,2014,12(7):934-940.
[53] CLASEN B M,STODDARD T J,LUO S,etal.Improving cold storage and processing traits in potato through targeted gene knockout[J].PlantBiotechnologyJournal,2016,14(1):169-176.
[54] WANG Y P,CHENG X,SHAN Q W,etal.Simultaneous editing of three homoeo-alleles in hexaploid bread wheat confers heritable resistance to powdery mildew[J].NatureBiotechnology,2014,32(9):947-951.
[55] JONES H D.Regulatory uncertainty over genome editing[J].NaturePlants,2015,1(1):14011.
[56] JIA H,WANG N.Targeted genome editing of sweet orange using Cas/sgRNA[J].PLosone,2014,9(4):e93806.
[57] 胡春华,邓贵明,孙晓玄,等.香蕉 CRISPR/Cas9 基因编辑技术体系的建立[J].中国农业科学,2017,50(7):1294-1301.
HU CH H,DENG G M,SUN X X,etal.Establishment of an efficient CRISPR/Cas9-mediated gene editing system in banana[J].ScientiaAgriculturaSinica,2017,50(7):1294-1301.
[58] FAN D,LIU T T,LI C F,etal.Efficient CRISPR/Cas9-mediated targeted mutagenesis in populus in the first generation[J].ScientificReports,2015,5(12217):12217.
[59] 刘婷婷,范 迪,冉玲玉,等.应用CRISPR/Cas9 技术在杨树中高效敲除多个靶基因[J].遗 传,2015,37(10):1044-1052.
LIU T T,FAN D,RAN L Y,etal.Highly efficient CRISPR/Cas9-mediated targeted mutagenesis of multiple genes inPopulus[J].Hereditas,2015,37(10):1044-1052.
[60] WALTZ E.Gene-edited CRISPR mushroom escapes US regulation[J].Nature,2016,5329(7599):293.
[61] LI Z S,LIU Z B,XING A Q,etal.Cas9-Guide RNA directed genome editing in soybean[J].PlantPhysiology,2015,169(2):960-970.
[62] ITO Y,NISHIZAWA-YOKOI A,ENDO M,etal.CRISPR/Cas9-mediated mutagenesis of the RIN locus that regulates tomato fruit ripening[J].BiochemicalandBiophysicalResearchCommunications,2015,467(1):76-82.
[63] 白云凤,张爱萍,闫建俊,等.靶向番茄SLACS2基因CRISPA/CAS sgRNA的设计和分析[J].生物信息学,2017,15(1):7-15.
BAI Y F,ZHANG A P,YAN J J,etal.Design and evaluation for sgRNAs target editingSLACS2 gene in CRISPR/Cas system[J].ChineseJournalofBioinformatics,2017,15(1):7-15.
[64] BROOKS C,NEKRASOV V,LIPPMAN Z B,etal.Efficient gene editing in tomato in the first generation using the clustered regularly interspaced short palindromic repeats/CRISPR associated 9 system[J].PlantPhysiology,2014,166(3):1292-1297.
[65] JIANG W Z,ZHOU H B,BI H H,etal.Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification inArabidopsis,tobacco,Sorghumand rice[J].NucleicAcidsResearch,2013,41(20):e188.
[66] MUTHONI J.Tetrasomic inheritance in cultivated potato and implications in conventional breeding [J].AustralianJournalofCropScience,2015,54(3):185-190.
[67] WANG S,ZHANG S,WANG W,etal.Efficient targeted mutagenesis in potato by the CRISPR/Cas9 system [J].PlantCellReports,2015,34(9):1473-1476.
[68] 李祥栋,石 明,魏心元.禾谷类作物胚乳淀粉合成及Waxy基因研究进展[J].中国农学通报,2015,31(12):181-186.
LI X D,SHI M,WEI X Y.Advances for starch biosynthesis andWaxygene in cereal crop[J].ChineseAgriculturalScienceBulletin,2015,31(12):181-186.
[69] ANDERSSON M,TURESSON H,NICOLIA A,etal.Efficient targeted multiallelic mutagenesis in tetraploid potato(Solanumtuberosum) by ytansient CRISPR-Cas9 expression in protoplasts[J].PlantCellReports,2017,36(1):117-128.
[70] BUTLER N M,BALTES N J,VOYTAS D F,etal.Geminivirus-mediated genome edting in potato(SolanumtuberosumL.) using sequence-specific nucleases[J].FrontiersinPlantScience,2016,7(1045):1-13.
[71] TIAN S,JIANG L,GAO Q,etal.Efficient CRISPR/Cas9-based gene knockout in watermulon[J].PlantCellReport,2016,36(3):399-406.
[72] CHANDRASEKARAN J,BRUMIN M,WOLF D,etal.Development of broad virus resistance in non-transgenic cucumber using CRISPR/Cas9 technology[J].MolecularPlantPathology,2016,17(7):1140-1153.
[73] CERMAK T,CURTIN S J,GIL-HUMANES J,etal.A multi-purpose toolkit to enable advanced genome engineering in plants[J].PlantCell,29(6):1196-1217.
