过表达谷子SiANT1对水稻耐盐性的影响
2018-05-31宁蕾王曙光琚鹏举柏星轩葛林豪齐欣姜奇彦孙现军陈明孙黛珍
宁蕾,王曙光,琚鹏举,柏星轩,葛林豪,齐欣,姜奇彦,孙现军,陈明,孙黛珍
过表达谷子对水稻耐盐性的影响
宁蕾1,王曙光1,琚鹏举1,柏星轩1,葛林豪2,齐欣2,姜奇彦2,孙现军2,陈明2,孙黛珍1
(1山西农业大学农学院,山西太谷 030800;2中国农业科学院作物科学研究所,北京 100081)
【目的】高盐胁迫是影响作物产量的主要因素之一,谷子(L.)具有耐逆性强的特点,从谷子中筛选耐盐基因对于利用基因工程的手段培育耐盐作物新品种具有重要意义。【方法】分析谷子AP2/ERF类转录因子类基因在豫谷一号幼苗期在高盐、低氮、PEG模拟干旱处理条件下的表达模式;进而利用农杆菌转化法将转入模式作物水稻品种Kitaake;分别用0.9% 盐水和自来水浸泡过表达水稻和野生型Kitaake的种子,观察其萌芽期的表型并统计发芽率;用含0.9% NaCl的Hogland营养液室内处理水稻幼苗10 d,65℃烘48 h之后称量干重;同时将其种植在大田,在水稻孕穗前用0.9%盐水灌溉一次,统计成熟后过表达水稻各株系和野生型的单株籽粒重、株高、单株分蘖数,对其耐盐性进行评价;利用定量Real time-PCR方法,验证分析转基因水稻株系中及其可能的下游基因的相对表达情况。【结果】谷子SiANT1蛋白(XP004985124.1,SiANT1)属于AP2亚族,与高粱(XP021318293.1,SbANT1)和玉米(XP008658933.1,ZmAP2)亲缘关系较近;豫谷一号中的表达受高盐胁迫(100 mmol·L-1NaCl)诱导;将和LP047 1118-Bar-ubi-EDLL载体连接,利用农杆菌转化法将转入到水稻基因组中,筛选得阳性T0植株,繁殖两代得T3代种子;自来水浸泡下,过表达水稻种子萌发率和野生型相差不大,萌芽率为90%以上;0.9%盐水浸泡下,过表达水稻种子萌发明显受到抑制,与野生型相比,过表达水稻种子露白推迟,但最终(处理120 h)萌芽率达到80%以上;室内水稻苗期用0.9% NaCl处理后,过表达水稻的单株干重较受体Kitaake增重14.11%—37.42%,在1%水平上呈显著差异;大田水稻成熟后过表达株系单株籽粒重较受体Kitaake增产56.12%—76.58%,在5%水平上呈显著差异,单株分蘖数增多、株高增加,但与野生型无显著差异;半定量RT-PCR分析表明,过表达的3个水稻株系都在RNA水平上表达;定量Real time-PCR结果表明,过表达的3个水稻株系间的相对表达量有所差异,但较受体而言,都极显著增加,并且其内源耐盐相关基因和的相对表达量分别较受体提高1.1—1.7倍和1.6—2.3倍。【结论】和是耐盐相关基因已被证实,具有一定的耐盐性,过表达水稻可能是通过增加下游基因和的表达从而提高耐盐性。
谷子;;AP2/ERF类转录因子;耐盐性;耐盐胁迫相关基因
0 引言
【研究意义】植物在生长发育过程中不可避免的会受到干旱、高温、土壤盐碱化等非生物胁迫因素的影响,其中盐胁迫是影响作物生长发育并最终导致减产的主要因素之一[1]。植物为了减少土壤中高盐离子浓度胁迫对自身造成的伤害,通过感知环境中离子浓度变化,精确调控相关基因表达,产生一系列的应答反应以应对盐胁迫。逆境胁迫诱导的相关基因表达主要通过转录因子进行调控,转录因子抑制或激活逆境胁迫相关基因的表达是植物响应逆境胁迫的重要过程[1]。谷子起源于中国,是耐贫瘠、抗逆性强的单子叶作物,已成为研究单子叶作物抗逆机制的重要材料[2],因此,研究谷子相关抗逆基因的功能对于筛选优良抗逆基因用于作物抗逆遗传改良具有重要意义。【前人研究进展】AP2/ERF转录因子家族的主要特征是蛋白序列中含有一个高度保守的58或59个氨基酸的AP2结构域[3]。Sakuma等[4]根据AP2/ERF结构域将拟南芥基因组中的AP2/ERF类蛋白分为5个亚家族,分别为AP2、RAV、DREB、ERF亚家族及一个特异蛋白AL079349[5]。作为蛋白质,AP2亚家族调节生长发育系统中的糖含量,并且涉及运输、信号传递[6]。拟南芥AP2类蛋白在花发育的ABC模型中起着重要的作用,其中ANT(AINTEGUMENTA)类转录因子,是植物特有的一类DNA结合蛋白,由与乙烯应答元件结合的2个AP2结构域组成[7],在转录水平上调节基因的表达。ANT类基因是器官生长的关键调节剂,参与胚珠发育、花器官生长发育和器官大小的调节,在调节植物生长发育的过程中起着非常重要的作用[7-12]。拟南芥突变体显示多效的作用,包括减少花器官和叶子的大小[7-8,11],过表达导致细胞增殖的持续时间增加,并增大叶、花和长角果中的器官[11]。在拟南芥转录因子RAV1中,N端AP2结构域结合5′-CAACA-3′序列,而C端高度保守的B3结构域结合5′-CACCTG-3′序列[13],参与响应生物胁迫和非生物胁迫[14]。DREB亚家族成员可以识别干旱和冷诱导响应元件(DRE/ CRT,A/GCCGAC),在植物抵抗非生物胁迫过程中起着非常重要的作用[15-16],如水稻显著影响转基因大豆耐盐性[17]。ERF亚家族成员能识别GCC盒(AGCCGCC)[18],在水稻中过表达增加了其对盐胁迫的敏感性[19],水稻ERF转录因子OsERF922负调控稻瘟病菌的耐药性和耐盐性[20]。从谷子中克隆到ABA响应DREB(脱水响应元件结合)蛋白基因,谷子过表达增强了抗旱性,拟南芥组成型表达增强了其种子萌发和幼苗发育期的抗旱性和耐盐性[21]。目前关于谷子耐旱性的报道,还有NAC类基因等,如谷子转录因子SiNAC18通过ABA信号途径正向调控干旱条件下的种子萌发[22]。在功能标记开发方面,谷子受激素诱导表达、参与器官大小控制的基因,从其启动子区开发的SSR标记AP-2,可用于谷子穗重和穗粒重等产量性状相关优异等位变异的鉴定和筛选[23]。【本研究切入点】中国农业科学院作物科学研究所马有志课题组从谷子品种豫谷一号中克隆ANT类转录因子基因(Si034722m.g,XP 004985124.1),位于第9染色体,全长4 196 bp,包含相间分布的6个外显子和5个内含子,cDNA序列长1 863 bp,编码620个氨基酸,蛋白质等电点为6.82,分子量为66.86 kD,该蛋白有2个AP2的DNA结合区,分别在280—346和382—440区域,长度为67个和59个氨基酸,即有一个ANT结构域。目前,关于AP2/ERF转录因子参与植物生长发育和增强抗逆性的报道较多,而ANT类转录因子是否参与植物胁迫响应过程,尚不清楚。【拟解决的关键问题】本研究通过在水稻中过表达,分析过表达水稻与野生型水稻分别在萌芽期、室内苗期、田间成熟期经0.9%NaCl处理后的表型及部分农艺性状的差异,确定过表达提高了水稻的耐盐性,进一步通过分子试验验证其可能的耐盐过程。
1 材料与方法
1.1 谷子SiANT1的分析及进化树构建
使用生物信息学工具、在线网站Phytozome(https:// phytozome.jgi.doe.gov/pz/portal.html)分析在search in搜索框中选中,并且keywords搜索框中输入序列号Si034722m.g,查找到基因组序列、CDS序列、蛋白质序列,2个AP2结构域的位置。使用DNAMAN软件查询该蛋白分子量大并预测等电点。在NCBI(https://www.ncbi.nlm. nih.gov/)中的Protein blast比对该蛋白序列,下载比对结果中同源性较高并含有ERF、AP2、DREB、RAV结构域的蛋白序列,用MEGA6软件打开蛋白序列并使用Neighbor- joining方法构建进化树。
1.2 不同胁迫条件下谷子幼苗中SiANT1的表达分析
将豫谷一号种子(由山西农业大学农学院孙黛珍课题组赠送)25℃清水浸泡4 d后(每天换2次水)移栽至配制的营养土(营养土﹕蛭石=2﹕1)中,置于地下温室(22℃,相对湿度65%、光照周期16 h/8 h)培养2周,然后取生长健康并且大小一致的幼苗分别用100 mmol·L-1NaCl培养液、0.2 μmol·L-1低氮培养液、10% PEG培养液进行胁迫处理,并于处理后的0、1、6、12、24和48 h分别取样,液氮冷冻,-80℃保存备用。
采用RNA提取试剂盒(TIANGEN公司)提取总RNA,并反转录成cDNA。通过NCBI的Primer Blast设计和引物(F:5′-CAAGTCT GGTGGCGTCAACT-3′,R:5′-AAAGGGAGG TGGGAAACGAC-3′;-F:5′-GGCAAACAGGGA GAAGATGA-3′,-R:5′-GAGGTTGTCGGTAAG GTCACG-3′)。反应体系为10 µl 2×Super Mix、0.5 µl 50×reference Dye Ⅱ、正反向引物各0.5 µl、7.5µl ddH2O和1µl cDNA。采用三步法反应程序:95℃ 15 min;95℃ 5 s,60℃ 20 s,72℃ 32 s后收集荧光信号,40个循环。Real-time PCR使用Real Master Mix(SYBR Green)试剂盒,ABI7500进行扩增。采用2-△△Ct法根据各样品在特定荧光阈值下的Ct值计算基因在不同样品中的相对表达量。
1.3 谷子SiANT1的表达载体构建及水稻的遗传转化
使用HⅠ酶切体系将适量LP047 1118-Bar- ubi-EDLL载体质粒30℃恒温酶切5 h以上,以豫谷一号的cDNA为模板,设计引物进行扩增含有HⅠ接头的CDS序列。用In-Fusion连接法将上述2个片段连接(50℃、20 min),转化TOP10大肠杆菌感受态,筛选阳性克隆测序。使用冻融法转化农杆菌,在农杆菌感受态中加入1.5 µl上述质粒,冰浴45 min,液氮中速冻1 min,并在37℃恒温水浴3 min。加800 µl的LB,28℃,200 r/min,培养3 h。涂于含利福平(50 mg·L-1)和卡那霉素(50 mg·L-1)的LB固体培养基上。筛选阳性菌落测序。按照蔗糖﹕siwet=5%﹕0.03%(v/v)配制农杆菌悬浮液。取5 µl的阳性菌液与1.5 mlLB液体培养基(含50 mg·L-1利福平和50 mg·L-1卡那霉素)混合,28℃ 250 r/min培养10—12 h。按1﹕100转移到三角瓶中。培养至OD600=0.6—0.8,6 000 r/min室温离心15 mim。再用悬浮液悬浮菌液调至OD600=0.8,用于水稻的遗传转化。利用农杆菌转化方法[22],将剥去壳的水稻成熟种子消毒、洗净后,利用组织培养的方法,经过诱导培养基-继代培养基-共培养培养基-选择培养基-生根培养基-移植到花盆,得到T0代植株,繁种后收获T1种子,本研究过表达水稻是T3代种子。
1.4 过表达SiANT1水稻的耐盐性鉴定
1.4.1 萌芽期盐处理试验 用浓度为0.9%的NaCl溶液分别浸泡过表达水稻3个株系及野生型Kitaake种子,各50粒。同时设空白对照,自来水浸泡上述种子。期间每天换2次相应溶液,直至0.9%的NaCl溶液处理的水稻开始露白,统计发芽率,每隔12 h统计1次,取5个时间点,并拍照记录相应差异。
1.4.2 培养室内水稻盐处理 将过表达水稻各株系及受体发芽至5 cm左右移至多孔板上,每个株系40株,Hogland营养液培养10 d,之后用含有0.9%NaCl的Hogland营养液培养10 d,将各株系每5株烘干称重、重复3组并记录数据。试验于2016年在中国农业科学院作物科学研究所品种资源楼进行。
1.4.3 田间水稻盐处理 2016年在河北唐海自然盐碱地上进行耐盐性鉴定,土壤盐碱度相对均匀。每个株系20粒种子,旱播,待水稻苗长到30 cm左右开始灌水(60 d),在水稻孕穗前进行一次盐处理,正常水(545 μs·cm-1)﹕海水(59 ms·cm-1)=4﹕1,约为0.9% NaCl浓度,处理后,同一地块盐碱度提高。待水稻成熟后调查水稻籽粒产量,评价转基因水稻的耐盐能力,筛选耐盐株系。每个株系收取5株,统计每株的单株分蘖数、株高、单株籽粒重,筛除各株系极端值,即每个株系统计3株。
1.5 谷子SiANT1及可能的下游基因在转基因水稻中的表达分析
将过表达水稻株系和野生型Kitaake种子在30℃条件下,用清水浸泡3 d后(每天换2次水),移至截去底部的96孔板上,置于22℃、相对湿度65%、光照周期16 h/8 h的温室水培至三叶一心期,取生长健康且大小一致的幼苗,液氮冷冻,-80℃保存备用。过表达水稻和野生型Kitaake种子均来自于中国农业科学院作物科学研究所马有志课题组。
在DNA水平上检测过表达水稻各株系是否属于阳性。设计水稻(F:5′-GAAGATCA CTGCCTTGCTCC-3′,R:5′-CGATAACAGCTCCTC TTGGC-3′[25])和的RT-PCR引物。利用半定量RT-PCR的方法检测过表达水稻的3个株系是否在RNA水平上表达,进一步做Real time-PCR确定3个株系间相对表达量是否有差异。
通过在NCBI(https://www.ncbi.nlm.nih.gov/)查找相关的盐胁迫基因并下载相应CDS序列(salt overly sensitive 1、protein kinase complex、calcium sensing protein、cation transporter、mitogen-activated protein kinases、zinc finger protein等),设计引物进行Real-time PCR分析,明确过表达水稻与受体Kitaake中的上述盐胁迫相关内源基因的相对表达情况。
2 结果
2.1 谷子SiANT1蛋白与AP2/ERF类转录因子各亚族间亲缘关系
谷子全长4 196 bp,CDS长为1 863 bp,编码620个氨基酸,蛋白质等电点为6.82,分子量为66.86 kD,该蛋白有2个AP2的DNA结合区,分别在280—346和382—440区域,长度为67个和59个氨基酸,即有一个ANT结构域。
利用MEGA6软件分析谷子SiANT1蛋白在AP2/ERF类转录因子各亚族间的进化位置结果表明,水稻、小麦、玉米、高粱、谷子的AP2/ERF类转录因子分为DREB、ERF、AP2、RAV等4个亚族(图1),其中DREB亚族和ERF亚族之间亲缘关系较近。谷子SiANT1蛋白(XP004985124.1,SiANT1)属于AP2亚族,与高粱(XP021318293.1,SbANT1)和玉米(XP008658933.1,ZmAP2)亲缘关系较近。
2.2 逆境胁迫下谷子SiANT1的表达模式
通过对100 mmol·L-1NaCl、0.2 μmol·L-1低氮、10% PEG处理的豫谷一号幼苗进行实时荧光定量PCR分析,结果表明,在100 mmol·L-1NaCl处理下(图2-A),与对照相比,谷子在1和6 h时的表达量明显下降,在12 h时的表达量显著上升,在24 h时的表达量达到100倍以上,之后在48 h谷子的表达量明显下降,说明谷子受到高盐胁迫诱导上调表达。在0.2 μmol·L-1低氮(LN)处理下(图2-B),与对照相比,谷子在1 h时表达量有所下降,在6 h时表达量达到1.5倍,之后12和48 h下降明显,并且24 h之后表达量几乎不变。在10%PEG处理下(图2-C),与对照相比,谷子表达量在1 h时明显下降,6 h稍微上升,但仍远低于0 h,12 h之后相对表达量几乎不随处理时间变化,说明在10%PEG6000条件下表达下调。
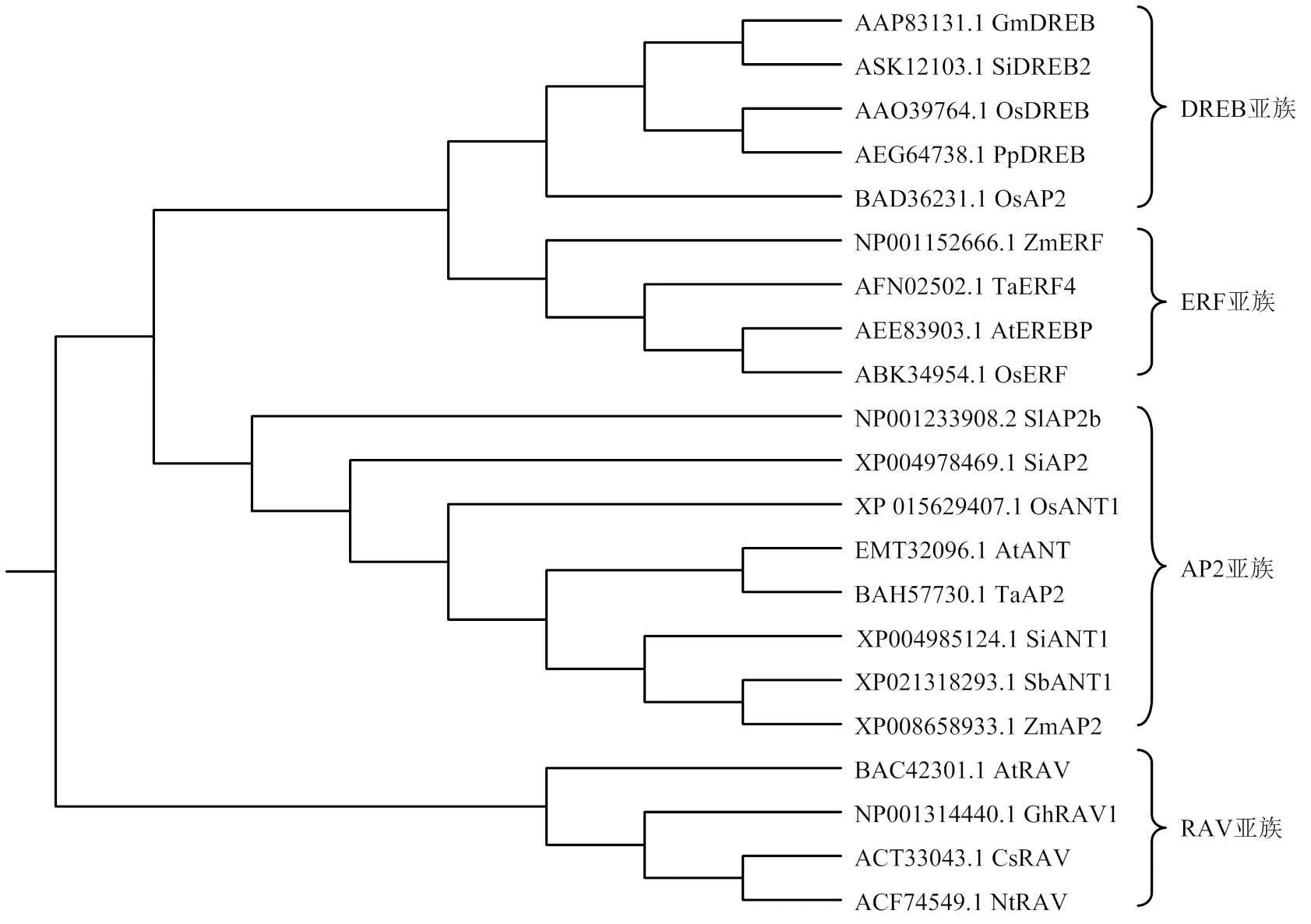
图1 利用部分植物AP2/ERF类转录因子构建的进化树

** 在0.01水平上差异显著(n=3);***在0.001水平上差异显著(n=3)。下同
** indicates significantly different at 0.01 level (n = 3); *** indicates significantly different at 0.001 level (n =3). The same as below
图2 不同胁迫处理三叶一心期豫谷一号后的相对表达量
Fig. 2 The expression profile ofgene in Yugu 1 under various stresses conditions including 100 mmol·L-1NaCl, 0.2 μmol·L-1LN, 10% PEG6000
2.3 谷子SiANT1的表达载体构建与水稻遗传转化
经过HⅠ酶切的基因和LP047 1118-Bar-ubi-EDLL载体连接,利用农杆菌转化法将目的基因片段转移到水稻的基因组中,通过组织培养最终筛选过表达水稻阳性T0植株,进而繁殖两代得到T3代种子。
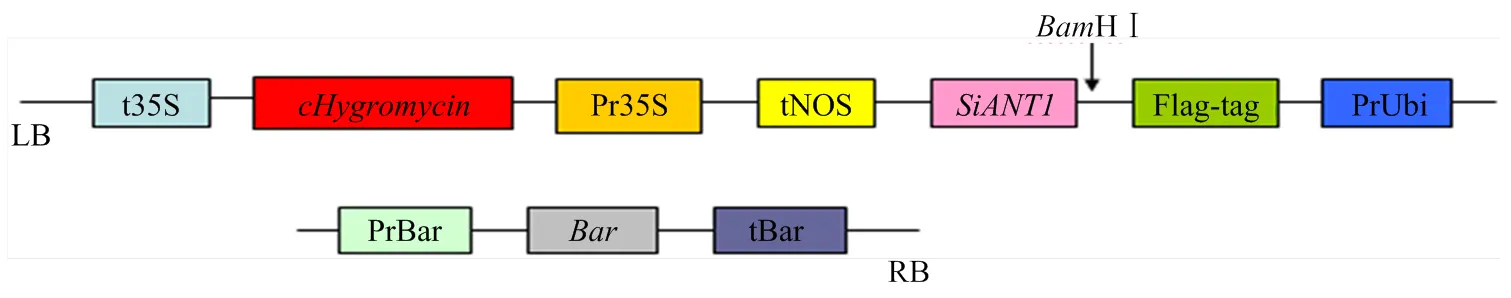
图3 LP047 1118-Bar-ubi-EDLL载体的T-DNA区段
2.4 过表达SiANT1水稻株系的鉴定与表现
2.4.1 过表达水稻各株系萌芽期表型及发芽率 通过对0.9%NaCl处理后的种子发芽率进行统计。0.9%NaCl处理66 h时,野生型Kitaake的发芽率(以露白为准)明显高于过表达水稻株系(图4-A)。0.9%NaCl处理120 h时,野生型Kitaake的发芽率与过表达水稻株系发芽率相差不大,但形态大小上差异明显(图4-C)。种子发芽120 h时,用自来水萌发的种子芽部大小整体大于相应的0.9%盐水处理(图4-D)。过表达水稻株系和野生型Kitaake用自来水发芽时无太大差异,各株系发芽率随时间变化几乎一致(图4-B和图4-F)。然而,用0.9%NaCl盐水萌发种子54 h时,野生型Kitaake发芽率明显高于此时自来水浸泡下的萌发率,说明高盐胁迫加快了野生型Kitaake的萌发;但是,过表达水稻株系的发芽率明显低于此时自来水浸泡下的萌发率,表明高盐胁迫抑制了过表达水稻株系的萌发,即对高盐胁迫是敏感的、有响应的。
2.4.2 过表达水稻各株系幼苗干重的变化过表达水稻各株系及受体苗期室内盐胁迫处理后,统计各株系地上部干重、根干重,并计算出单株干重(表1),重复3组,从表中可看出:过表达水稻各株系地上部干重较受体野生型增加0.49%—21.07%,其中M30103-15株系与受体野生型之间无显著差异;过表达水稻各株系根干重较受体野生型增加57.07%—100.51%(<0.01);过表达水稻各株系单株干重较受体野生型增加14.11%—37.42%(<0.01)。这说明过表达在一定程度上提高了水稻苗期的耐盐性,根部差异更为突出。
2.4.3 过表达水稻各株系的田间表现 由表2可看出,在田间土样离子浓度差异不显著的前提下,过表达水稻株系M30103-13、M30103-14的单株分蘖数增多,而M30103-15减少但与受体野生型差异不显著;3个株系株高较受体野生型增加3.33%—7.67%,与受体野生型株高差异不显著;过表达水稻株系单株籽粒重较受体野生型增加56.12%—76.58%,且呈显著水平。表明过表达水稻在田间相对于受体水稻耐盐性显著提高。
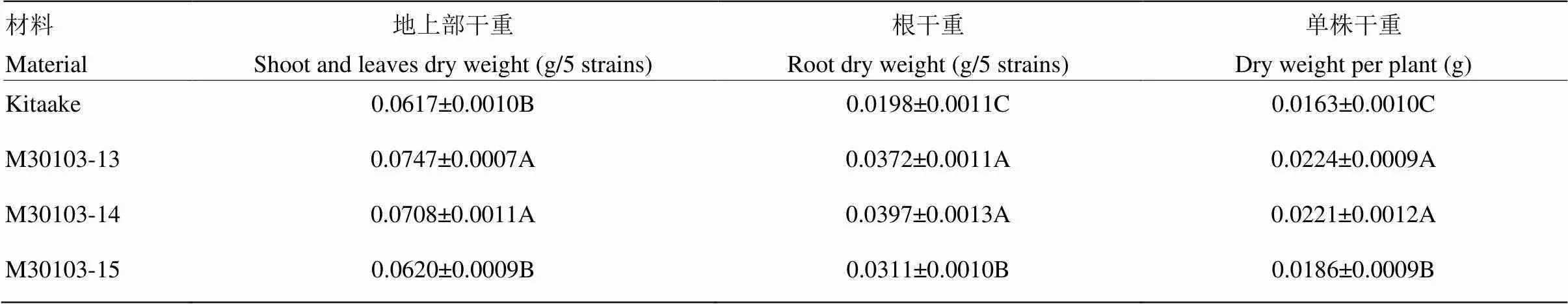
表1 培养室内过表达SiANT1水稻各株系及受体Kitaake苗期盐处理后数据统计
表中数据为均值±标准差,同列不同大写字母表示不同材料之间在0.01水平差异显著(n=3)
The data in the table are Means±SD, with different capital letters in the same column indicating significant differences (0.01) between the different materials (n=3)
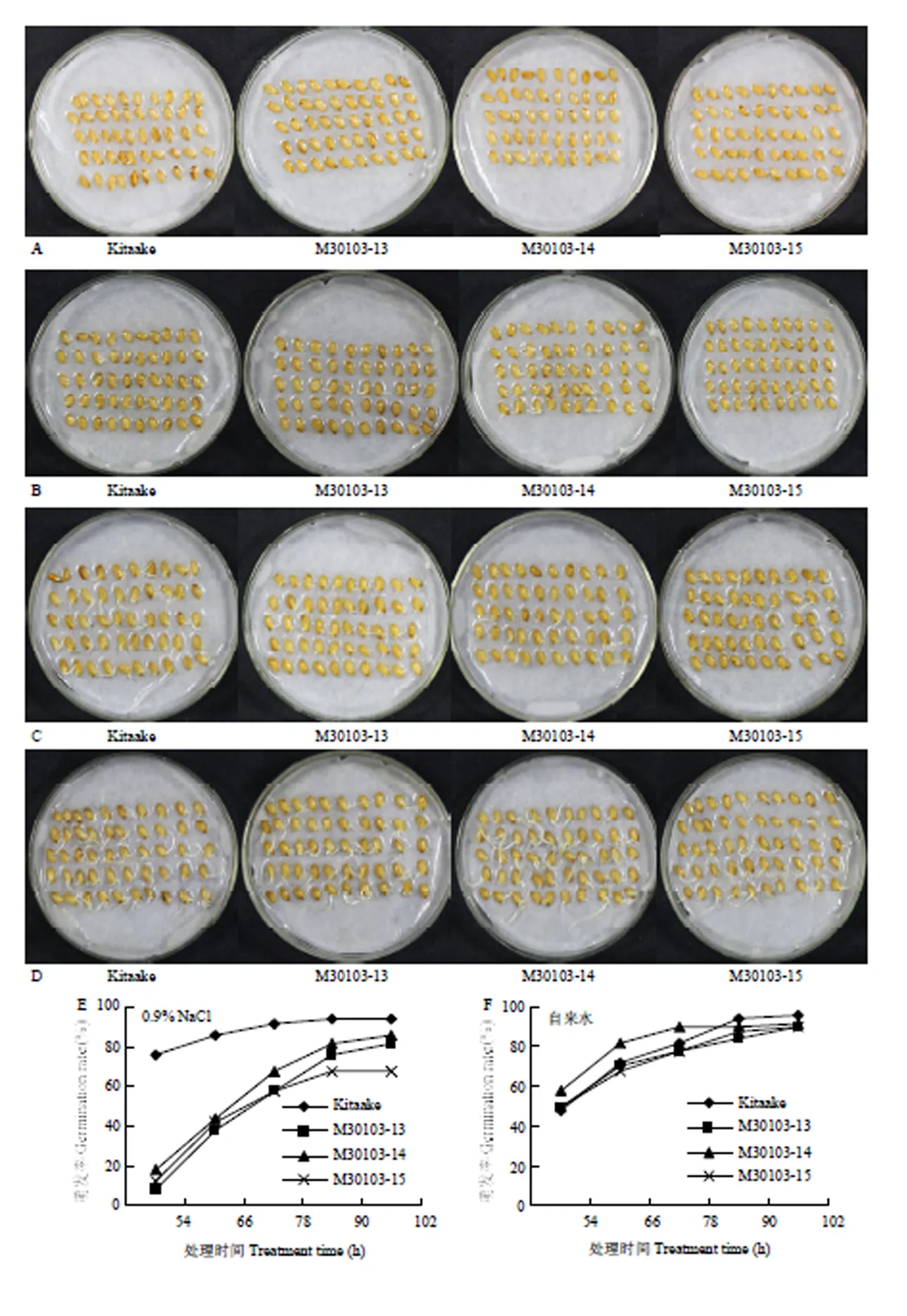
A:0.9%NaCl处理66 h;B:自来水处理66h;C:0.9%NaCl处理120h;D:自来水处理120 h;E、F:分别表示0.9%NaCl和自来水处理的发芽率
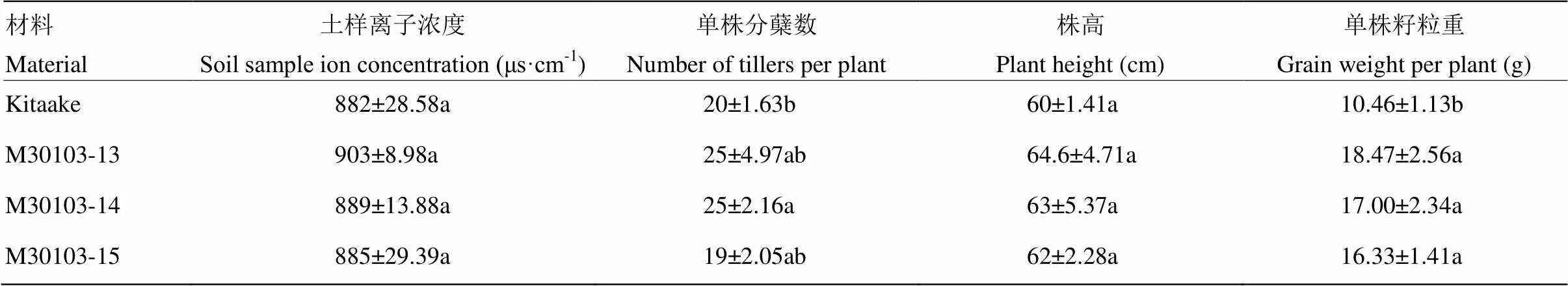
表2 田间全生育期农艺性状调查
表中数据为均值±标准差,同列不同小写字母表示不同材料之间在0.05水平差异显著(n=3)
The data in the table are Means±SD, with different normal letters in the same column indicating significant differences (0.05) between the different materials (n=3)
2.5 过表达SiANT1水稻的分子检测及相关基因表达模式分析
2.5.1 过表达水稻株系阳性植株检测 通过对过表达水稻株系和野生型水稻进行PCR检测(图5)。结果显示,过表达水稻3个株系都是阳性,即谷子已转入水稻中。

M:marker;1:阳性质粒对照;2:阴性H2O对照;3:Kitaake;4:M30103-13;5:M30103-14;6:M30103-15
2.5.2 过表达水稻的半定量和定量PCR分析 通过对转基因株系中进行半定量RT-PCR分析(图6-A),在亮度一致的前提下,过表达株系(M30103-13、M30103-14和M30103-15)均扩增出大小一致的条带,但受体野生型无带,说明野生型水稻中不存在谷子基因,并且这三个过表达水稻株系都在RNA水平上表达。进一步定量PCR检测(图6-B),过表达水稻中的相对表达量为受体的178—10 615倍,其中M30103-13株系的相对表达量最高,M30103-15次之,M30103-14最低,该结果与半定量结果一致。
利用农杆菌转化法将转入水稻中,但是,是否插入水稻DNA中并形成重组DNA,插入位点及插入个数均不确定。已证明水稻株系M30103-13、M30103-14、M30103-15的DNA内含有,并且都在RNA水平上表达,只是RNA相对表达量有所差异,因此,推测3个过表达水稻株系之间相对表达量的差异可能是由于拷贝数不同造成的。

A:转基因水稻的RT-PCR分析;B:转基因水稻的Real-time PCR分析。***与野生型水稻Kitaake相比,该过表达SiANT1水稻株系SiANT1的相对表达量在0.001水平上差异显著(n=3)
2.5.3 可能的下游基因的表达 根据前面的推测,进一步检测细胞分裂素等植物激素形成和耐盐性相关基因在转基因株系中的表达水平。通过对水稻和进行实时荧光定量PCR检测(图7-A和图7-B),与受体Kitaake比较,水稻的相对表达量在M30103-13、M30103-14提高1.5倍以上,M30103-15提高1.1倍(图7-A);在M30103-13、M30103-14和M30103-15中的相对表达量均提高1.6倍以上(图7-B),呈显著增加水平。结果表明,在过表达水稻株系和的相对表达量相对于受体野生型显著提高,结合水稻和[26-28]已被前人验证可以提高作物耐盐性,说明过表达水稻株系可能增强了内源和的表达,进而提高了耐盐性。
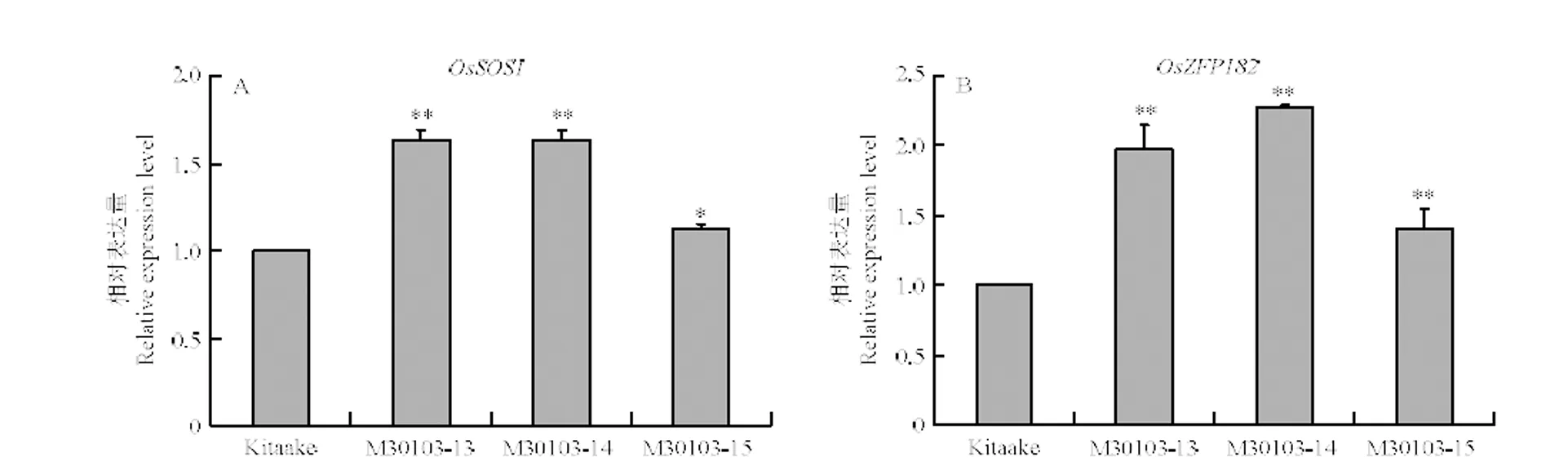
A:OsSOS1;B:OsZFP182。*在0.05水平上差异显著(n=3)* significantly different at 0.05 level (n = 3)
3 讨论
盐胁迫增加了土壤中Na+和Cl-浓度,使植物受到渗透胁迫和离子毒害,造成植物生长发育迟缓甚至死亡。形态方面:叶面积减小、叶片卷曲干枯、植株矮小;生理生化方面:单株干重降低,光合作用受抑制、营养物质摄取受阻、活性氧增多、质膜解体、代谢毒物积累等[29]。植物为了降低盐胁迫对自身的伤害,增加糖类、脯氨酸、甜菜碱等渗透调节物质的合成,这起到保护细胞膜表面的作用,提高了细胞的耐逆性;大量的膜蛋白被激活将Na+最终转移到液泡中,重建胞内、胞外、液泡的离子平衡[29-30]。植物盐应答的分子包括两类:一类是直接参与代谢变化等生化应答过程,产生耐盐效应的效应分子;另一类是位于效应分子上游,介导信号传递,包括转录因子和信号级联系统中各种激酶的调控分子[29,31]。转录因子是能够与真核基因启动子区域中的顺式作用元件发生特异相互作用的结合蛋白,通过它们之间的相互作用,激活或抑制转录,进而激活或抑制下游基因表达[32]。转录因子在植物对逆境的应答过程中起着重要的调控作用,通过它可以精细地调控下游功能基因适时适量地表达,从而实现自身生长发育和繁殖的需要以及对各种生物或非生物逆境的应答[32-33]。MYB、MYC、bZIP、AP2/ERF and zinc finger protein等转录因子都参与逆境胁迫相关基因表达[32]。
本文在水稻中过表达,SiANT1蛋白属于AP2/ERF转录因子里的AP2亚家族。目前关于AP2亚族的报道,大多与植物生长发育相关[7]。本文主要研究是否响应盐胁迫,是否可以提高作物耐盐性,如若可以,可能是通过怎样的方式调控。SiANT1是一个转录因子,将谷子利用农杆菌转化法转到水稻中,随着载体质粒的T-DNA区进入水稻,是否转入、插入位置、以及插入个数等都是不确定的,后续通过DNA检测筛选阳性植株,并筛选在RNA水平上表达的植株,本文未对表达的蛋白进行研究。本文在高盐胁迫条件下,过表达水稻种子萌芽期受抑制,然而,在自来水中过表达水稻种子正常发芽,说明谷子对高盐敏感。萌芽期的高盐胁迫试验结果,过表达水稻种子萌芽受抑制,这说明可能在萌芽期不具有耐盐性,但是,与野生型水稻种子相比,最终(0.9%NaCl处理120 h)两者发芽率相差不大,又说明可能过表达水稻种子露白推迟是一种耐盐胁迫应答反应。过表达水稻株系苗期干重和田间籽粒重相对于受体水稻显著增加,证明过表达可以提高转基因水稻苗期及成熟期耐盐性。苗期干重和田间籽粒重都属于生物量,较野生型而言,在0.9%NaCl处理下,过表达水稻株系生物量增加,可能是由于的过表达导致细胞分裂素增多、细胞数目增加,叶茎器官增大、侧根形成增加[10,34],在此基础上,由于表达的蛋白是转录因子,过表达可能也通过增强相关下游耐盐基因表达来提高水稻耐盐性。本文中可能的下游基因Os、Os的相对表达量在过表达水稻中较野生型增加。SOS1蛋白是一种质膜Na+/H+逆向转运蛋白,是一个重要的耐逆决定簇,参与Na+从细胞中排出[35-36]。Martínez-Atienza等[26]和SCHMIDT等[27]证明了OsSOS1水稻转运蛋白在酵母细胞的质膜囊泡中具有Na+/H+交换能力,降低了细胞中Na+净含量,减轻了Na+对细胞造成的离子毒害。Na+/H+逆向转运蛋白SOS1是迄今为止植物质膜上唯一的Na+流出蛋白[26]。缺少SOS1的拟南芥突变体对盐是极其敏感的,其Na+排出和将该离子从根部长距离运输到茎都有缺陷[36-37]。SOS1蛋白主要表达于植物根尖表皮和木质部共质体边界的木质部薄壁组织[36]。在根-土壤界面处,SOS1将从根表皮细胞排出过量的Na+离子。此外,突变体中Na+根冠分配的分析表明,在不同盐度条件下SOS1也以复杂的方式参与Na+在根和茎之间的重新分配[36]。在适度的盐(25 mmol·L-1NaCl)胁迫下,突变体植株在其地上部中积累的Na+比野生型低,这表明SOS1的功能是将Na+装载到木质部用于控制传递至茎。相反,在高盐(100 mmol·L-1NaCl)胁迫下,突变体植株的根和地上部分比野生型植物积累更多的Na+,这可能是由于根表皮Na+排出以及Na+穿过木质部-共质体边界的电化学梯度受到破坏[36,38]。锌指蛋白(zinc finger Protein)是一类具有“手指状”结构域的转录因子,负责调控基因的表达[32]。锌指蛋白的共同特征是通过结合Zn2+来稳定一种很短的可自我折叠成“手指”的蛋白结构[32]。ZFP182蛋白大小为18.2 kD,含有2个C2H2型锌指基序,一个核定位信号和一个富含Leu的结构域。大多数植物的C2H2型锌指蛋白中存在DLN-box/EAR-motif结构域,但是ZFP182中不存在[28]。在成年稻的叶,茎,根和穗中组成型表达,并且水稻幼苗分别在冷(4℃)、150 mmol·L-1NaCl和0.1 mmol·L-1ABA处理下,明显被诱导表达[28]。将水稻的的启动子区约1.4 kb融合到报告基因中并转化烟草,组织化学分析结果显示,转基因烟草幼苗在正常条件下不能检测到GUS的表达,但在用NaCl或KCl处理后的烟草叶片和根的维管组织中强烈观察到GUS的表达[28]。Huang等[28]研究表明水稻中过表达提高了盐胁迫的耐受性。目前关于ZFP182的作用机理仍不清楚。本研究证明过表达提高了水稻的耐盐性,可能是通过增强水稻中下游内源基因和Os的表达实现的,为证明谷子具有耐盐性进一步提供了依据。
4 结论
谷子受100 mmol·L-1NaCl诱导上调表达,在24 h时相对表达量最高。过表达提高水稻耐盐性,其通过增强可能的下游基因和的表达提高耐盐性。
[1] MO J B, LI D Y, ZHANG H J, SONG F M. Roles of ERF Transcription factors in biotic and abiotic stress response in plants., 2011, 47(12): 1145-1154.
[2] 黄锁. 谷子耐盐相关基因SiNF-YA5和SiATG4的功能分析及调控途径解析[D]. 北京: 中国农业科学院, 2016.
HUANG S. Functional analysis and regulatory pathway analysis of salt-tolerance related genesandChinese Academy of Agricultural Sciences, 2016. (in Chinese)
[3] OHME-TAKAGI M, SHINSHI H. Ethylene-inducible DNA binding proteins that interact with an ethylene-responsive element., 1995, 7: 173-182.
[4] SAKUMA Y, LIU Q, DUBOUZET J G, ABE H, SHINOZAKI K, YAMAGUCHI S K. DNA-binding specificity of the ERF/AP2 domain ofDREBs transcription factors involved in dehydration-and cold-inducible gene expression., 2002, 290(3): 998-1009.
[5] 张计育, 王庆菊, 郭忠仁. 植物AP2/ERF类转录因子研究进展. 遗传, 2012, 34(7): 835-847.
ZHANG J Y, WANG Q J, GUO Z R. Research progress on plant AP2 /ERF transcription factors., 2012, 34(7): 835-847. (in Chinese)
[6] OHTO M A, FISCHER R L, GOLDBERG R B, NAKAMURA K, HARADA J J. Control of seed mass by., 2005, 102(8): 3123-3128.
[7] KLUCHER K M, CHOW H, REISER L, FISCHER R L. Thegene of Arabidopsis required for ovule and female gametophyte development is related to the floral homeotic gene., 1996, 8(2): 137-153.
[8] ELLIOTT R, BETZNER A, HUTTNER E, OAKES M, TUCKER W, GERENTES D, PEREZ P, SMYTH D., an-like gene ofwith pleiotropic roles in ovule development and floral organ growth., 1996, 8: 155-168.
[9] KRIZEK B. Ectopic expression ofin Arabidopsis plants results in increased growth of floral organs., 1999, 25: 224-236.
[10] MIZUKAMI Y, FISCHER R. Plant organ size control:regulates growth and cell numbers during organogenesis., 2000, 97: 942-947.
[11] NOLEWILSON S, KRIZEK B.contributes to organ polarity and regulates growth of lateral organs in combination withgenes., 2006, 141: 977-987.
[12] KRIZEK B.andLIKE6 act redundantly to regulatefloral growth and patterning., 2009, 150: 1916-1929.
[13] KAGAYA Y, OHMIYA K, HATTORI T. RAV1, a novel DNA-binding protein, binds to bipartite recognition sequence through two distinct DNA-binding domains uniquely found in higher plants., 1999, 27(2): 470-478.
[14] SOHN K H, LEE S C, JUNG H W, HONG J K, HWANG B K. Expression and functional roles of the pepper pathogen-induced transcription factor RAV1 in bacterial disease resistance, and drought and salt stress tolerance., 2006, 61(6): 897-915.
[15] YAMAGUCHISHINOZAKI K, SHINOZAKI K. A novel cis-acting element in angene is involved in responsiveness to drought, low-temperature, or high-salt stress., 1994, 6(2): 251-264.
[16] THOMASHOW M F. PLANT COLD ACCLIMATION: freezing tolerance genes and regulatory mechanisms., 1999, 50: 571-599.
[17] ZHANG X X, TANG Y J, MA Q B, YANG C Y, MU Y H, SUO H C, LUO L H, NIAN H. OsDREB2A, a rice transcription factor, significantly affects salt tolerance in transgenic soybean., 2013, 8(12): e83011.
[18] HAO D Y, OHME-TAKAGI M, SARAI A. Unique mode of GCC box recognition by the DNA-binding domain of ethylene responsive element–binding factor (ERF domain) in plants., 1998, 273(41): 26857-26861.
[19] TANG Y H,QINS S, GUO Y L, CHEN Y B, WU P Z, CHEN Y P, LI M R, JIANG H W, WU G J. Genome-wide analysis of the AP2/ERF gene family in physic nut and overexpression of thegene in rice increased its sensitivity to salinity stress., 2016, 11(3): e0150879.
[20] LIU D F, CHEN X J, LIU J Q, YE J C, GUO Z J. The rice ERF transcription factornegatively regulates resistance toand salt tolerance., 2012, 63(10): 3899-3911.
[21] LI C, YUE J, WU X W, XU C, YU J J. An ABA-responsive DRE-binding protein gene from,, the target gene of SiAREB, plays a criticalrole under drought stress., 2014, 65(18):5415-5427.
[22] 窦祎凝, 秦玉海, 闵东红, 张小红, 王二辉, 刁现民, 贾冠清, 徐兆师, 李连城, 马有志, 陈明. 谷子转录因子通过ABA 信号途径正向调控干旱条件下的种子萌发. 中国农业科学, 2017, 50(16): 3071-3081.
DOU Y N, QIN Y H, MIN D H, ZHANG X H, WANG E H, DIAO X M, JIA G Q, XU Z S, LI L C, MA Y Z, CHEN M. Transcription factorpositively regulates seed germination under drought stress through ABA signaling pathway in foxtail millet (L.)., 2017, 50(16): 3071-3081. (in Chinese)
[23] 王智兰, 杜晓芬, 王军, 杨慧卿, 王兴春, 郭二虎, 王玉文, 袁峰,
田岗, 刘鑫, 王秋兰, 李会霞, 张林义, 彭书忠. 谷子的克隆、表达分析和功能标记开发. 中国农业科学, 2017, 50(22): 4266-4276.
WANG Z L, DU X F, WANG J, YANG H Q, WANG X C, GUO E H,WANG Y W, YUAN F, TIAN G, LIU X, WANG Q L, LI H X, ZHANG L Y, PENG S Z. Molecular cloning, expression analysis and development of functional markers forgene in foxtail millet., 2017, 50(22): 4266-4276. (in Chinese)
[24] 杨霞. 两个水稻金属离子转运体基因和两个水稻锌指蛋白基因的克隆与功能研究[D]. 南京: 南京农业大学, 2007.
YANG X. Cloning and functional analysis of two rice metal ion transporter genes and two rice zinc finger protein genes[D]. Nanjing: Nanjing Agricultural University, 2007. (in Chinese)
[25] KIM B R, NAM H Y, KIM S U, KIM S I , CHANG Y J. Normalization of reverse transcription quantitative-PCR with housekeeping genes in rice., 2003, 25: 1869-1872.
[26] MARTíNEZ-ATIENZA J, JIANG X, GARCIADEBLAS B, MENDOZA I, ZHU J K, PARDO J M, QUINTERO F J. Conservation of the salt overly sensitive pathway in rice., 2007, 143(2): 1001-1012.
[27] SCHMIDT R, MIEULET D, HUBBERTEN H M, OBATA T, HOEFGEN R, FERNIE A R, FISAHN J, SAN-SEGUNDO B, GUIDERDONI E, SCHIPPERS J H, MUELLER-ROEBER B. SALT-RESPONSIVE ERF1 regulates reactive oxygen species- dependent signaling during the initial response to salt stress in rice., 2013, 25(6): 2115-2131.
[28] HUANG J, YANG X,WANG M M,TANG H J, DING L Y, SHEN Y, ZhangH S. A novel rice C2H2-type zinc finger protein lacking DLN-box/EAR-motif plays a role in salt tolerance., 2007, 1769(4): 220-227.
[29] 沈义国, 陈受宜. 植物盐胁迫应答的分子机制. 遗传, 2001, 23(4): 365-369.
SHEN Y G, CHEN S Y. Molecular mechanism of salt stress response in plants., 2001, 23(4): 365-369. (in Chinese)
[30] BOHNERT H J, SHEN B. Transformation and compatible solutes., 1999, 78: 237-260.
[31] HASEGAWA P M, BRESSAN R A, ZHU J K, BOHNERT H J. Plant cellular and molecular responses to high salinity., 2000, 51: 463-499.
[32] 张宏. 水稻C2H2型锌指蛋白ZFP182和ZFP36在ABA诱导的抗氧化防护中的功能分析[D]. 南京: 南京农业大学, 2012.
ZHANG H. Functional analysis of rice C2H2 zinc finger proteins ZFP182 and ZFP36 in anti-oxidative protection induced by ABA[D]. Nanjing: Nanjing Agricultural University, 2012. (in Chinese)
[33] CALKHOVEN C F, GEERT A B. Multiple steps in the regulation of transcription-factor level and activity., 1996, 317(2): 329-342.
[34] RANDALL R S, SORNAY E, DEWITTE W, MURRAYJ A H.and the D-type cyclin CYCD3;1 independently contribute to petal size control in: evidence for organ size compensation being an emergent rather than a determined property., 2015, 66:3991-4000.
[35] DONGHA O, SANGYEOL L, BRESSAN R A, YUN D J, BOHNERT H J. Intracellular consequences of SOS1 deficiency during salt stress., 2010, 61(4): 1205-1213.
[36] SHI H, QUINTERO F J, PARDO J M, ZHU J K. The putative plasma membrane Na+/H+antiporter SOS1 controls long-distance Na+transport in plants., 2002, 14: 465-477.
[37] QIU Q S, GUO Y, DIETRICH M A, SCHUMAKER K S, ZHU J K. Regulation of SOS1, a plasma membrane Na+/H+exchanger in, by SOS2 and SOS3., 2002, 99(12): 8436-8441.
[38] PARDO J M, CUBERO B, LEIDI E O, QUINTERO F J. Alkali cation exchangers: roles in cellular homeostasis and stress tolerance., 2006,57(5):1181-1199.
(责任编辑 李莉)
Rice Overexpression of MilletGene Increases Salt Tolerance
NING Lei1, WANG ShuGuang1, JU PengJu1, BAI XingXuan1, GE LinHao2, QI Xin2, JIANG QiYan2, SUN XianJun2, CHEN Ming2, SUN DaiZhen1
(1College of Agronomy, Shanxi Agricultural University, Taigu 030800, Shanxi;2Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081)
【Objective】High salt stress is one of the main factors affecting crop yield.L. is characterized by its strong tolerance to stress. Therefore, screening salt tolerant related genes from millet is important for salt stress tolerant improvement of crops through genetic transformation.【Method】The expression profile of AP2/ERF like genewere analyzed in Yugu 1 at seedling stage under high salt, low nitrogen and PEG simulated drought conditions. Millet genewas transformed into rice Kitaake by agrobacterium-mediated transformation method. overexpressingrice seeds and wild type Kitaake were digested with 0.9% saline and tap water, respectively. The phenotypes of germinating stage and germination rate were observed. Rice seedlings were treated with 0.9% NaCl in Hogland nutrient solution for 10 days and then dried at 65℃ for 48 hours dry weights of all the rice overexpressingand wild type (WT) plants were analyzed. At the same time, phenotype of all plants was analyzed in the field and all plants were irrigated once with 0.9% saline before rice pregnancy. Under the similar salt treatment condition, single seed grain weigh plant height and tiller number per plant of the overexpressingrice lines and wild type were also analyzed.The real time-PCR method was used to verify the expression ofand its possible downstream genes in transgenic rice lines.【Result】L. SiANT1 protein (XP004985124.1, SiANT1) belongs to the AP2 subfamily and has a close genetic relationship with sorghum (XP021318293.1, SbANT1) and maize (XP008658933.1, ZmAP2).The gene ofwas induced by high salt stress (100 mmol·L-1NaCl) in Yugu 1. Thewas ligated to LP047 1118-Bar-ubi-EDLL vector, Agrobacterium transformation method was used to transferinto rice genome, positive T0plants were screened, thenbreeded two generations and got T3seeds. Under water immersion, the germination rate ofoverexpression rice was similar to that of wild type. Germination rate was more than 90%. Under 0.9% saline immersion, the overexpression ofwas significantly inhibited.Compared with the wild type, over-expression ofrice seeds dew white was delayed, but finally(treatmented for 120h) germination rate was more than 80% .After treatment with 0.9% NaCl at seedling stage in greenhouse, the dry weight of single plant overexpressingincreased 14.11%-37.42% more than that of WT, which had significant difference at the 1% level. the grain weight of transgenic rice overexpressionwhich had significant difference at the 5% level,the number of tillers per plant and plant height increased, but there was no significant difference with the wild-type. Semi-quantitative RT-PCR analysis showed that all three rice lines overexpressingexpressedat the RNA level. Real time-PCR results showed that the relative expression level ofwas different but significantly increased compared with that of the recipient, and the relative expression levels of endogenous salt-tolerance-related genesandwere 1.1-1.7 fold and 1.6-2.3 fold respectively higher than that of the recipient.【Conclusion】It has been confirmed thatandare salt tolerance related genes.gene has certain salt tolerance. Overexpression ofand.
millet (L.);gene; AP2 / ERF transcription factor; salt tolerance; salt tolerant related genes
10.3864/j.issn.0578-1752.2018.10.002
2017-12-01;
2018-02-02
国家转基因生物新品种培育科技重大专项(2016ZX08002005)、谷子氮素高效利用重要基因克隆及育种价值评估(2018ZX0800924B)
宁蕾,E-mail:ninglei02@163.com。通信作者孙黛珍,E-mail:sdz64@126.com。通信作者陈明,E-mail:chenming02@caas.cn
