一款轻量级的复杂生物网络图形化分析软件的设计与实现
2018-05-23赵琳琳赵亚莉张牧歌李文龙
赵琳琳 赵亚莉 张牧歌 李文龙

摘 要: 近年来,复杂网络理论受到广泛的关注和研究。本文将复杂网络的理论应用到生物数据分析领域,设计并实现一款智能化和可视化的生物网络特征分析软件。该软件可以实现数据输入或者批量数据导入功能,通过对数据的读取和结合相应的生物学背景知识,建立数据的网络视图,以及提供对该生物网络的特征系数的参考值及分析结果,能有效帮助用户发现节点之间存在的隐藏联系及功能模块之间的特征,是一款交互性较好的轻量级应用软件。
关键词: 复杂生物网络;可视化;生物信息节点;复杂网络
Abstract:In recent years complex network theory has received extensive attention and research. This paper applies the theory of complex network to the field of biological data analysis and designs and implements a kind of intelligent and visualized biological network feature analysis software.This software can achieve the data input or batch data import function through the data read and combined with the corresponding biological background knowledge it can establish a data network view and provide reference value of the coefficient of the characteristics of biological networks and the results of the analysis which could help users to find the hidden relationship between the nodes and characteristics between functional modules. Thus it can be taken as a good lightweight interactive application software.
Key words: complex biological network;visualization;biological information node;complex networks
引言
近年来,各个领域中对复杂网络的研究正日趋进步与深入,人们开始广泛关注网络结构复杂性及其与网络行为之间的关系。复杂网络的研究最早可以追溯到1736年Euler的七桥问题,长期发展演变至今,复杂网络的探究也已从早期的数学领域逐渐扩展到实际生活的其它多个方面,诸如从社交网络、到物理学网络、再到生物学网络。而且,复杂网络借助数学、统计学、计算机科学等现代分析工具[1],已经形成相对成熟的系统理论。例如,在复杂网络的理论研究方面主要集中在对实际复杂网络进行统计分析,建立复杂网络的模型进行特征性分析[2],以及复杂网络的混沌同步及控制研究上。就目前来看,未来复杂网络将推动研究学界着重设计采用一个简单方便的方法来应对处理,即这些将复杂的系统视作一个个网络图来展开理论阐释与研究,同时还要进一步去延伸探索复杂网络的结构以及结构与系统功能之间的联系。
不仅如此,复杂网络的现有理论结果同时也吸引了为数可观生物学者的兴趣与关注。对于节点众多、连接结构复杂的网络所设定的整体结构的研究过程中发现,可以将复杂网络与生物网络密切联系起来,通过借助复杂网络日趋完备的理论对复杂生物网络有针对性的探讨分析[3],即可更加便利地掌握各个生物信息之间的微小联系,比如复杂网络中的幂律分布,小世界效应运用在生物蛋白质领域,调用复杂网络特征提取展示的不同蛋白质之间的内在相关性。分析复杂生物网络图的内在理论特点,根据理论特征分析得到的直观结果,则能成功分析出相对应节点之间蛋白质是如何相互作用的,基因之间有什么相似之处与关联,生物细胞变异有什么原因等等这些问题的机理与内在奥秘。除此之外,还可将复杂网络应用在生物神经网络中来模拟探测人类大脑神经的结构同样具有现实重大的研究意义。
目前,对复杂网络应用在生物学的领域研究已成为业界热点,而且也已经成为一个系统综合的用来解释基因数据水平及其内在联系的有利工具。已有不少软件率先做出了功能范例,诸如Cytoscape、Vanted、Vis Ant等一系列用于分析复杂生物网络的软件已经可以实现预计功能,然而却也仍然存在着不足之处。正因为其偏近于专业化,使得研发面对的可行域是有限的,对于生物领域方面分析的可视化结构可堪称完美,能够完善地展示和处理已构建的复杂网络图,但是对于其它领域则呈现出设计欠缺。其中的关键点就是并未设定高端的数据处理分析,不能立足在复杂网络本质特征的角度去释疑与分析,并通過对复杂网络的特征分析做出相应的结果输出,使用户能够拥有更多功能选择以及收获数据的精确分析。本文拟将提出的研究内容就是建立在Cytoscape软件基础之上的,不同之处在于,本文将对于所有输入的节点信息,不仅具备其可视化的展示部分如图1所示,还包括对可视化展示后的复杂网络的特征分析及数据处理结果显示,并将最终结果提供给用户,帮助用户理解和分析研究内容。
1 复杂网络基本理论
1.1 复杂网络
复杂网络(Complex Network)是指具有自组织、自相似、吸引子、小世界、无标度中部分或全部性质的网络,换言之,即呈现高度复杂性的网络。复杂网络的基本特征包括小世界、集群即集聚程度的概念、幂律的度分布概念。其复杂性主要表现在如下方面,其内容可表述为[4]:
(1)结构复杂。表现在节点数目巨大。
(2)网络结构呈现多种不同特征。表现在节点或连接的产生与消失,例如World-Wide Network,网页或链接随时可能出现或断开,导致网络结构不断发生变化。
(3)连接多样性。体现在节点之间的连接权重存在差异,且可能存在方向性。
(4)动力学复杂性。体现在节点集可能属于非线性动力学系统,例如节点状态随时间发生复杂变化。
(5)节点多样性。体现在复杂网络中的节点可以代表任何事物。例如,人际关系构成的复杂网络中,节点代表单独个体;万维网组成的复杂网络中,节点可以表示不同网页。
(6)多重复杂性融合。即以上多重复杂性相互影响,导致更加难以预料的结果。例如,设计一个电力供应网络需要考虑此网络的进化过程,其进化过程将决定网络的拓扑结构。当2个节点之间频繁进行能量传输时,节点之间的连接权重会随即增加,通过不断地学习与记忆逐步改善网络性能。
复杂网络已经应用在很多领域,也在实际领域中得到了工程统计数据来实现后续的数据分析,部分示例数据可见表1。
1.2 复杂网络的基本概念
这里,关于图的基本概念术语以及相关特征系数将给出如下分析概述。
在本次研究中,将蛋白质作为简单的节点,即相互作用网络將表示成无向图,故而仅需聚焦在无向图的相关定义上。设无向图G=(V,E),其中,V是图中点的集合,E是图中边的结合,可得定义形式如下:
(1)对于边e∈E,若e=(u v),则称边e与顶点u v关联。
(2)对于顶点u,v∈V,如果存在边e=(u v),使得e=(u v),则称u与v邻接,或说u和v是相邻的顶点。
(3)对于边e1,e2∈E,若e1和e2关联共同的顶点,则称e1和e2邻接,或说e1和e2是相邻的边。
1.3 复杂网络的基本属性介绍
在对基本图的研究中,一般要探讨5个方面的网络属性,具体表述如下:
(1) 度分布: 一个节点有k条边的概率P(k)。
(2) 小世界效应(最短路径) :任意2个节点能通过最少几条边的路径建立连接。小世界效应的网络更有利于在网络节点之间的信息传播。
(3) 聚类系数:节点近邻之间联系的紧密程度,通常可以用网络中包含的三角形个数予以表征说明。
(4)平均路径长度:任意两节点之间距离的平均值,主要是为了分析复杂网络的网络拓扑结构[6]。
(5) 鲁棒性(弹性):网络的容错与抗攻击能力。
在生物复杂网络的研究中,也是从上述5个方面来考虑生物网络图的各种性质,通过探析各个节点之间相互作用网络的结构和机制,分析其在整张复杂网络里面所具备的特征。
1.3.1 复杂网络的聚类系数
在时下的社交网络中,经常会出现这样一种情况,某人的2个朋友之间也可能是朋友,关于这种特性就可称之为聚类特性[4]。一般条件下,假设网络中节点i有Ki个边,这个边将该节点与其它节点连在一起,那么这Ki个节点就是i的邻居。在这个Ki个节点之间最多有Ki(Ki+1)/2条边。用Ei表示这Ki个节点之间实际存在的边的个数。则节点i的聚类系数、也称局部聚类系数Ci的数学公式可表述如下[4]:
全局聚类系数就是整个网络的聚类系数C,表示了所有节点i的聚类系数Ci的平均值。聚类系数的值是在0和1之间。对于全局聚类系数C,当C=0时,当且仅当复杂网络中所有的节点均为孤立节点,所有节点之间没有任何相连的边。对于一个含有N个节点的完全随机网络,当N很大时,C=O(1/N)。在实际生活中,已有的许多大规模网络都具有明显的聚类效应,在某种程度上和社交关系网络中的“物以类聚,人以群分”之间表现出颇高的相似性,这些网络的聚类系数远小于1但是又比O(1/N)大得多。在人们的社交网络中,某人朋友的朋友与此同时也是其本人朋友的概率会随着网络规模的增加趋向于一个非零的常数。另外,还需指出,在日常接触的复杂网络中,大多数往往都不是完全随机的网络。
1.3.2 平均路径长度
设连接网络中节点i和j之间最短路径上的边数为i和j之间的距离dij。在复杂网络中任意两点之间距离的最大值为复杂网络的直径,即:
复杂网络的平均路径长度也称为复杂网络的特征路径长度(Characteristic path length),当不包含节点到自身的距离时,研究推得L的运算公式可见如下:
在社交网络中,L的含义是连接2个人之间最短关系链中朋友的平均个数。在复杂生物网络中,L的含义是2个蛋白质之间最短关系链中蛋白质的个数。
在很多复杂网络中,尽管节点个数巨大,但是平均路径却很小。如果对于固定的网络节点平均度
1.3.3 度与度的分布
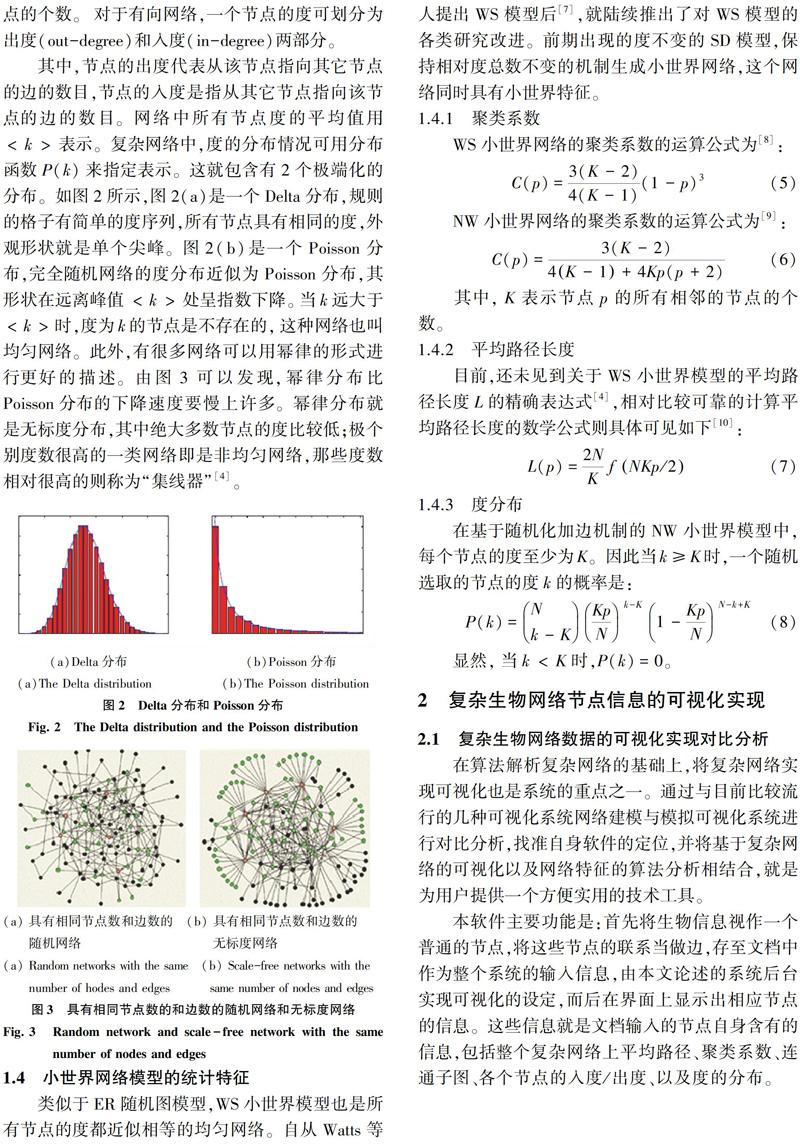
在复杂网络中,用ki表示节点i连接的其它节点的个数。对于有向网络,一个节点的度可划分为出度(out-degree)和入度(in-degree)两部分。
其中,节点的出度代表从该节点指向其它节点的边的数目,节点的入度是指从其它节点指向该节点的边的数目。网络中所有节点度的平均值用
1.4 小世界网络模型的统计特征
类似于ER随机图模型,WS小世界模型也是所有节点的度都近似相等的均匀网络。自从Watts等人提出WS模型后[7],就陆续推出了对WS模型的各类研究改进。前期出现的度不变的SD模型,保持相对度总数不变的机制生成小世界网络,这个网络同时具有小世界特征。
1.4.1 聚类系数
1.4.2 平均路径长度
目前,还未见到关于WS小世界模型的平均路径长度L的精确表达式[4] ,相对比较可靠的计算平均路径长度的数学公式则具体可见如下[10]:
1.4.3 度分布
2 复杂生物网络节点信息的可视化实现
2.1 复杂生物网络数据的可视化实现对比分析
在算法解析复杂网络的基础上,将复杂网络实现可视化也是系统的重点之一。通过与目前比较流行的几种可视化系统网络建模与模拟可视化系统进行对比分析,找准自身软件的定位,并将基于复杂网络的可视化以及网络特征的算法分析相结合,就是为用户提供一个方便实用的技术工具。
本软件主要功能是:首先将生物信息视作一个普通的节点,将这些节点的联系当做边,存至文档中作为整个系统的输入信息,由本文論述的系统后台实现可视化的设定,而后在界面上显示出相应节点的信息。这些信息就是文档输入的节点自身含有的信息,包括整个复杂网络上平均路径、聚类系数、连通子图、各个节点的入度/出度、以及度的分布。当然,这些输入可以是生物领域的蛋白质内在联系分析,也可以是基因组与人类疾病联系的相关输入,甚至可以是人的社交网络,节点代表人等等。最后,可利用聚类系数对整个复杂网络进行区域划分,将关系相近的节点划分在一个区域,用户可以利用软件分析的结果并依据需要展开相应的研究。同时,本文定制提出的软件还可以实现界面节点注释信息的展示。
2.1.1 Cytoscape
Cytoscape就是一个开源的网络可视化和分析软件系统。目前可以说在复杂网络的可视化方面已呈现出高端技术优势[11]。该软件可以通过生物分子相互作用网络可视化的形式将高通量基因表达数据、分子状态信息有机整合在一起。而且,其最重要的设计应用就在于可视化大规模蛋白质-蛋白质相互作用、蛋白质-DNA和遗传相互作用等复杂生物网络的分析,并能将这些网络与功能注释等数据库进行有效链接。另外,由于软件内嵌有良好与成熟的插件开发,用户还可以方便地将自身的数据转换为复杂网络的可视化。
2.1.2 VisAnt
VisAnt是一个在线可视化建模系统,用户在线即可连接数据库,并且将数据信息借助这一工具,以图形模式绘制并显示出来。该软件能够在线可视化GO(geneontology)以及诸如提供与KEGG和Swiss rot等数据库的在线连接及可视化。目前,VisAnt主要生成的布局算法是Spring-embedded算法。
2.1.3 VANTED
VANTED是一个在综合分析实验数据基础上能够输出生物网络可视化的功能软件,适用于网络中每个节点(如蛋白质、酶、代谢物)均集结有大量的实验数据(Excel表格方式提供)的可视化分析。其中,主要包括了力导向算法、环布局等算法。
2.2 信息节点可视化显示
通过参考cytoscape.Js库中的常用函数,在可视化界面将节点以及节点之间的联系终端页面显示出来,不同节点之间将采用不同的色彩加以区分,节点自身包含着共享的细节信息,点击相应的节点即可智能显示节点内部信息。在复杂生物网络的可视化页面,用户可以随时获知当前的节点是何种蛋白质、代表什么基因等等。并且还可以根据用户需要引入一定的位置调整,与此同时当前页面的节点坐标也将随即发生变动,同时还将结合用户可操作控制的页面按钮,用户就可以选择全部输出,或者只输出局部的统计特征。通过点击按钮,借助Javascripe函数的运行,调用深层机制,灵活选择指定函数以及从数据库中读取目标数据。在得到相应的输出结果之后,再基于可实时交互的开放页面,用户可以在终端页面根据相应的结果信息,进行一定的增、删调整,还可以根据需要重新设计结果的输出,数据库的相应信息也会随着交互页面的变动发生联动调整,从而输出最终更新的数据,并提供给客户用于后续数据分析和相关研究。
2.3 后台数据库处理及运行机制的分析
数据库的数据录入部分,主要是借助DIP数据库目录通过实验确定蛋白质之间的相互作用。过程中结合了各种来源的信息,并创建了一整套一致的蛋白质 - 蛋白质相互作用。在DIP网站中寻找到完整的DIP数据集再以“PSI-MI TAB”格式下载了最新数据。此后可以将以TAB为分隔符的txt文件导入到Excel文件中。对完整的DIP数据集进行符合本次项目数据要求的修改,得到两两蛋白质之间的对应关系,以及对应关系出现的方法或文献信息。后台数据库用于支持维护数据的录入和读取,并且作为整个软件机制的数据源,可为软件前端界面的可视化显示派做信息依据,将用户信息以表格形式存储在相应的系统之中,供中央处理器调用和计算处理,以及前端可视化展示。
2.4 将节点信息进行公式理论化处理
将复杂网络的算法以Java的形式存储起来,将每个算法作为独立的函数,前端页面设置对应函数的标志性按钮,若触发该按钮,系统会读取当前的可视化信息,并在系统数据库后台调用节点信息,作为输入传送至函数中,再启用函数执行相关的数据计算,得到优化处理输出,进而反馈给Jsp页面显示相应的结果。在最终可视化页面,设置各类主题按钮供用户根据自身需要选择相应的输出。作用机制就是利用框架原理,将函数计算部分作为javabean,与可视化部分联系在一起,同时与数据库创建关联,从数据库中读取要计算的输入数据。
3 结束语
在本次研究过程中,初步实现的可视化软件是将用户输入的文档信息转化为可视化的复杂网络,为用户实现了预计的特征分析功能。在可视化页面,节点按钮存储基本的节点信息,帮助用户进一步了解研究指定分析的节点与节点之间的内在特征及在网络中的价值,满足用户分析数据的需要,并在可[CM(22]视化界面研究嵌入了良好人机交互,用户可以根据已有的结果信息将原初数据进行可控性的调整和更改,重新启动页面按钮得到新的可视化显示和数据分析的关联结果。同时,本文设计论述的软件还可以实现界面节点注释信息的展示,方便用户及时探查和使用信息。但是,目前本软件涉及分析的特征数据信息仍然较为有限,并未达到完善和全面,复杂网络的其它重要的特征数据,并没有完全配备各类规范运算,且对于部分用户数据分析结果也僅限于提供辅助参考作用。
参考文献
[1] 陆锁军. 生物复杂网络抉择行为与混沌同步研究[D]. 上海:东华大学,2008.
[2] 江珊,蒋勃,徐桂珍,等. 使用Cytoscape对生物网络数据的建模和分析[J]. 农业网络信息,2017(6):32-37.
[3] 周洪伟. 复杂网络理论及其在生物网中的应用[D]. 南京:南京航空航天大学,2007.
[4] 汪小帆,李翔,陈关荣. 复杂网络理论及其应用[M]. 北京:清华大学出版社 2006.
[5] ALBERT R BARABASI A L. Statistical mechanics of complex network[J]. arXiv preprint arXiv: cond-mat/0106096 2001.
[6] 周云龙. 复杂网络平均路径长度的研究[D]. 合肥:合肥工业大学,2013.
[7] NEWMAN M E J.The structure and function of complex networks[J]. SIAM Review 2003,45(2):167-256.
[8] ERDS P RNYI A.On the evolution of random graphs[J]. Transactions of the American Mathematical Society,1960(5):17-61.
[9] NEWMAN M E J. The structure and function of networks[J]. Computer Physics Communications,2002,147(1):40-45.
[10]NEWMAN M E J WATTS D J. Renormalization group analysis of the small-world network model[J]. Physics Letters A,1999,263(4-6):341-346.
[11]孙玺箐,司守奎. 复杂网络算法与应用[M]. 北京:国防工业出版社,2015.
[12]王林,戴冠中. 复杂网络的度分布研究[J]. 西北工业大学学报,2006,24(4):405-409.
[13]刘涛,陈忠,陈晓荣. 复杂网络理论及其应用研究概述[J]. 系统工程,2005,23(6):1-7.
[14]刘强,方锦清,毕桥,等. 不同机制的小世界模型的同步能力比较[J]. 中国原子能科学研究院年报,2006:183-185.
