岩生树花地衣型真菌NRPS基因的生物信息学分析
2018-05-16原晓龙陈剑华梅李云琴王娟杨宇明王毅
原晓龙,陈剑,华梅,李云琴,王娟,杨宇明,王毅
(1.云南省林业科学院,云南 昆明 650201;2.云南省森林植物培育与开发利用重点实验室/国家林业局云南珍稀濒特森林植物保护和繁育重点实验室,云南 昆明 650201)
非核糖体肽(Nonribosomal peptides, NRPs)中含有许多具有医药价值的抗生素类药物,是一种结构丰富,由微生物经非核糖体多肽合成酶(Nonribosomal peptides synthetases,NRPSs)超酶家族合成[1]。非核糖体多肽合成酶运输氨基酸和肽链中间体到含有泛酰巯基乙胺辅因子的肽酰基载体结构域及其他不同的催化结构域,促使初生肽链的延伸、修饰直至最终的释放[2]。NRPSs由A(腺苷酰化结构域,A结构域)、C(缩合结构域,C结构域)、T(肽酰基载体蛋白结构域,或巯基化结构域,T结构域) 3个相互独立及其他一些结构域按一定的时空顺序排列,形成具不同催化活性功能的酶[2]。NRPs被用于抗生素、抗真菌药物、抗癌药物、抗病毒药物及免疫抑制剂等[3],其在细菌、放线菌及真菌中广泛存在[1,4-6]。因此,非核糖体多肽合成酶编码基因的可挖掘潜力巨大,通过基因挖掘技术、微生物发酵等技术合成药品的类似物,甚至有较大的可能合成全新的药品。随着测序技术的不断发展,基因操作手段持续成熟,基因挖掘技术已成为从基因组和转录组数据中挖掘新的天然产物的重要手段[7]。基因挖掘技术除能够发现传统方法不能发现的新化合物外,还能够避免传统方法重复发现的困境。对于在通常培养条件下不产生的化合物,利用基因挖掘技术,从基因组或转录组数据出发,分析可能的基因簇,再进行不同方式培养,敲除负调控基因及异源表达等方式能获得大量新的多肽、聚酮类、萜类化合物等[7]。
岩生树花(Ramalinaintermedia)属子囊菌门(Ascomycetes)子囊菌纲(Ascomycetes)茶渍目(Lecanorales)树花科(Ramalinaceae)树花属(Ramalina)[8],是一种广泛分布,多生长于岩石上的树状地衣[9]。岩生地衣含有石花酸、松萝酸、荔枝素、扁枝衣二酸、树花地衣酸等次生代谢产物[9-10],可作为天然药物来源之一。随着测序技术的不断发展,以基因组和转录组数据为基础,结合生物信息学分析手段,挖掘查找新基因,已被广泛应用于天然次生代谢产物研究[11]。Lautru等[12]利用基因组挖掘技术从Streptomycescoelicolor中得到新的多肽化合物coelichelin;Wu等[13]通过分析西洋参(Panaxquinquefolius)的3个不同组织器官的转录组信息,从中克隆并鉴定了MVA途径中的限速酶4-羟基-4-甲基戊二酰辅酶A还原酶(HMGR)基因。在真菌中模块化的聚酮合酶(Polyketide synthase,PKS)的挖掘中,效果尤其显著,近年来通过生物信息学分析不同的真菌转录组,从单一真菌菌株中发现的基因簇数量可达20-100个,这些基因簇中有接近一半数量的PKS[14-15];Abdel等[16]利用antiSMASH、NaPDoS等生物信息学分析软件从Cladoniauncialis中发现并鉴定出了6-Hydroxymellein合成酶。利用生物信息学分析从转录组数据挖掘基因,其可靠性较高。本研究以岩生树花的转录数据为分析基础,分离得到RiNRPS基因,通过一系列生物信息学分析软件和工具,对其产物进行了预测分析,预测得到RiNRPS基因所编码的次生代谢产物。通过对RiNRPS基因及其蛋白氨基酸序列的BLAST比对,结合antiSMASH、NRPSpredictor、NaDPoS(Natural Products Domain Seeker)及Norine等分析NRPS的在线工具,挖掘岩生树花的RiNRPS基因,为岩生树花的非核糖体多肽的研究提供前期基础。
1 材料与方法
1.1 材料
岩生树花于2015年5月从云南省昆明市西山(24.94278°N、102.633192°E)采集。
1.2 方法
在实验室用藻菌分离技术分离出岩生树花的地衣型真菌。将地衣型真菌在恒温培养箱25℃条件MY培养基上培养60d后,转录组取样方法:将培养60d的菌丝体收获后,立即放入液氮中,用干冰将样品送测序公司进行测序,测序平台采用Illumina NextSeq,测序读长为2×150bp,采用 FastQC(http://www.bioinformatics.babraham.ac.uk/projects/fastqc)对下机数据进行质量控制,基于DBG(De Bruijn Grahp)拼接原理,利用Trinity (版本r20140717,k-mer 25bp)软件对获得的序列进行拼接。最后利用本地BLAST对转录组数据进行比对,并结合在线数据库比对获得岩石树花地衣型真菌NRPS基因序列。
将获得的NRPS基因序列在NCBI上进行BLAST比对后,初步确定RiNRPS基因,并查找其开放阅读框。选择结构域相似度较高的蛋白序列和外加蛋白序列,多序列比对用MEGA 6.0软件中的Cluster程序完成,采用默认参数;系统进化树采用邻位相接法(Neighbor-Joining),自展值设为1 000进行绘制;理化性质的预测借助于ProParam在线工具;信号肽预测利用SignalP 4.1 server;用Target P预测其亚细胞定位,用NCBI中的Conversed Domain Database数据库搜索RiNRPS蛋白的结构功能域;再利用分析NRPS的在线工具确定其可能的化合物及其组成。
通过NRPS和PKS的软件分析RiNRPS基因,通过antiSMASH从岩生树花转录组中挖掘出RiNRPS基因并对其化合物进行识别和分析,获得其基本结构域和具体的母核结构;用NRPS predictor推断该基因各个A-domain负责转运和活化的氨基酸;采用NaPDoS对该基因C-domain进行识别和分类;最后通过Norine对该基因所产生多肽的具体结构、活性等进行推测生物信息学所用在线工具及其网址见表1。
2 结果与分析
2.1 RiNRPS基因理化性质分析
用在线软件ProtParam预测RiNRPS蛋白氨基酸残基序列的理化性质,相对分子质量为702 699.9,结构式C31099H49135N8579O9469S249,等电点5.83,哺乳动物体外培养半衰期30h,酵母体内的半衰期大于20h,大肠杆菌(Escherichiacoli)体内的半衰期大于10h,脂肪系数87.59,不稳定系数41.01,为不稳定蛋白。ORF Finder分析结果显示,其开放阅读框总长19 098bp,编码6 362个氨基酸;SignalP分析显示RiNRPS蛋白不存在信号肽,为非分泌蛋白;采用Target P软件预测其亚细胞定位,结果表明其蛋白位于细胞质基质中。
2.2 构建RiNRPS蛋白的分子系统进化树
通过对岩生树花RiNRPS蛋白序列进行BLAST比对,选择与其同源性较高且结构域基本相似的其他蛋白序列,并添加与其结构域具明显差别的17条蛋白序列构建分子系统进化树(图1)。
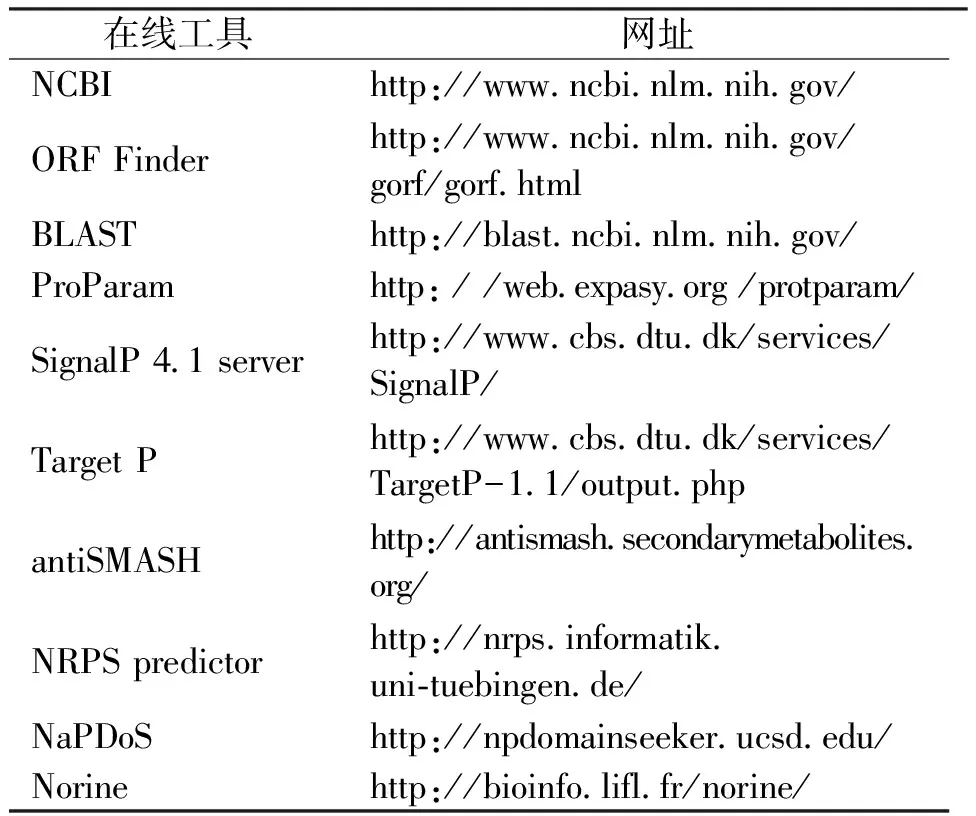
表1 生物信息学在线工具及其网址Tab.1 The softwrae online of Bioinformatics and its websites
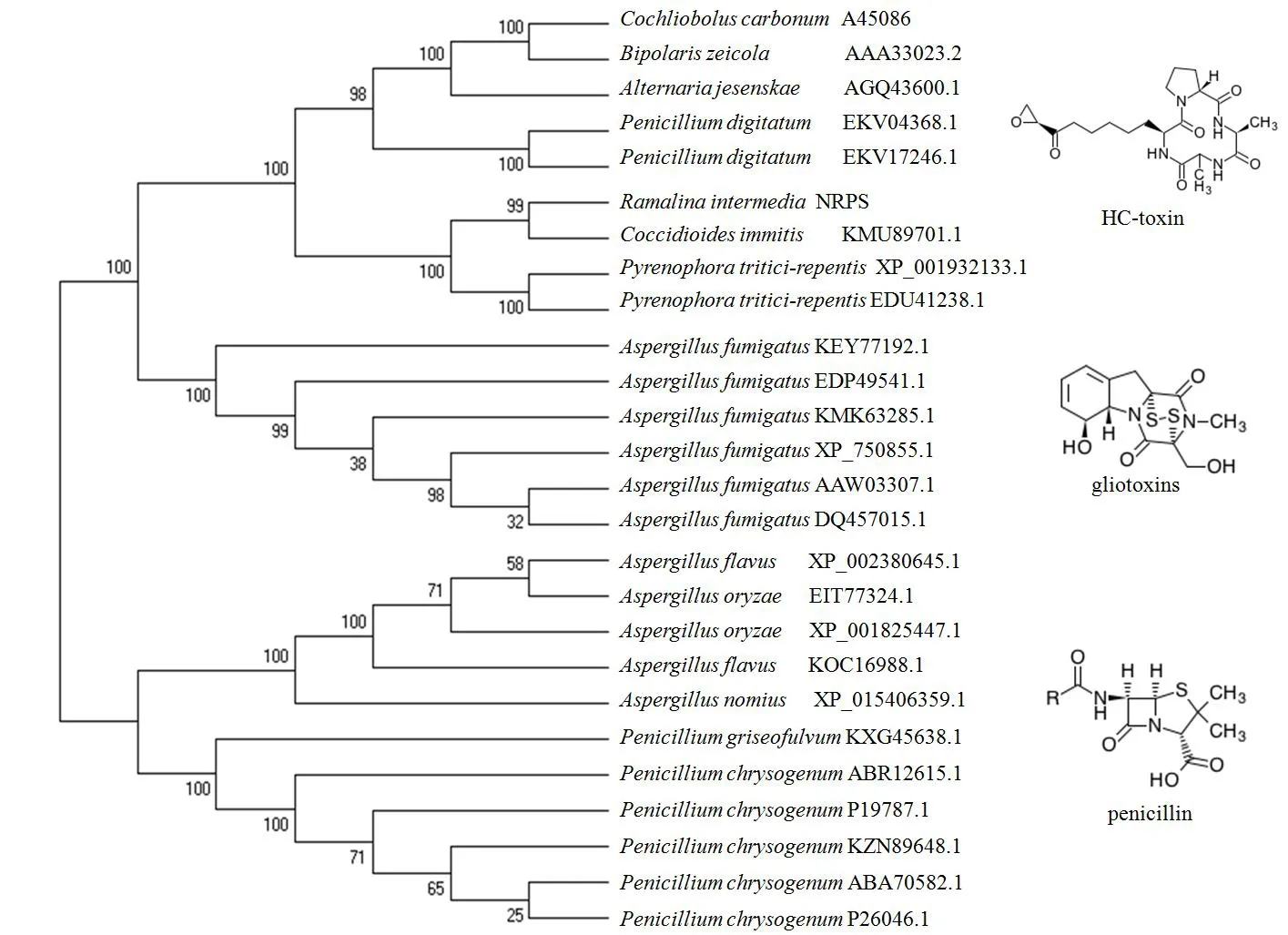
图1 RiNRPS蛋白序列与其他25条蛋白序列的分子系统进化分析Fig.1 Phylogenetic analysis of RiNRPS and other 25 protein sequences
由图1可以看出,岩生树花的RiNRPS蛋白与玉米(Zeamays)圆斑病病菌(Cochlioboluscarbonum,A45086)、玉米生离蠕孢菌(Bipolariszeicola, AAA33023.2)、互隔交链孢霉菌(Alternariajesenskae,AGQ43600.1)、指状青霉菌(Penicilliumdigitatum,EKV04368.1,EKV17246.1)、粗球孢子菌(Coccidioidesimmitis,KMU89701.1)和小麦黄斑叶枯病菌(Pyrenophoratritici-repentis,XP001932133.1、EDU41238.1)聚为一类,其产物均为HC-toxin母核,而与产生胶霉毒素(Glitoxin)、青霉素母核(Penicillin)的距离较远,未聚为一类,二者各自聚为一类。
2.3 RiNRPS基因所产生酶及化合物的分析
antiSMASH分析结构显示该基因为NRPS,其结构域顺序为A-T-C-A-T-E-C-A-T-C-A-T-E-C-T-C(A为腺苷酰化结构域、T为PCP结构域,即肽酰基载体蛋白结构域、E为差向异构结构域、C为缩合结构域)(图2A)。NRPS predictor 2能够预测细菌和真菌A domain识别的氨基酸种类,推断出该基因的A domain转运的氨基酸依次为Orn(鸟氨酸)、Gln(谷氨酰胺)、Gln(谷氨酰胺)、Cys(半胱氨酸)。NaPDoS(Natural Products Domain Seeker)通过对RiNRPS基因的2个C domain聚类分析,结果显示RiNRPS基因的2个C domain均与HC-toxin 聚为一类,表明该基因的可能目的化合物为HC-toxin。综上所述,通过antiSMASH、NRPS predictor、NaPDoS分析,得出RiNRPS基因可能编码的蛋白为HC-toxin 合成酶,其潜在化合物由Orn(鸟氨酸)、Gln(谷氨酰胺)、Gln(谷氨酰胺)、Cys(半胱氨酸)4个氨基酸单体合成,是一种三轮状结构的环状四肽,具毒素活性的多肽,其可能结构式为C21H32N4O6。

图2 antiSMASH在线软件分析的结构域顺序Fig.2 The domain’s sequence by the online software antiSMASH
3 讨论
转录组测序技术结合代谢调控、生物信息学分析等手段,可以通过不同次生代谢产物的变化查找新基因、推测代谢途径和阐释基因功能,在天然次生代谢产物生物合成途径及相关基因功能注释的研究中被广泛应用,已成为基因挖掘不可或缺的技术[11]。真菌的非核糖体多肽(NRPs)具有广泛的生物活性,可作为抗生素、铁载体、免疫抑制剂、抗癌药物等[17-19]。NRPs是由NRPSs催化合成的,NRPSs因其结构的多样性,从微生物中发现的新NRPS基因其潜在化合物成药性较高,如Pullen等[20]从卫矛科(Celastraceae)植物中分离得到的内生链霉菌产生的Chloropyrrol抗生素可抑制多种耐药性细菌和分歧杆菌;Combs等[21]在蛇藤(ColubrinaasiaticaBrongn)中分离得到的新链霉菌StreptomycesNRRL30562,产生了4种新的广谱抗生素Munumbicins A、B、C和D,能抑制杀灭多种植物的致病性霉菌、细菌。NRPS在生物合成途径中因其结构域的多样性及其组合方式的不同变化,可形成的化合物数量及其庞大,结构的多样性形成了生物活性丰富的天然次生代谢产物[22-23]。
antiSMASH是一个分析次生代谢产物中抗生素和其他医药类的在线软件,运用其可加速对天然此生代谢产物的识别和挖掘[24]。NaPDoS是一种能快速检测和分析天然次生代谢产物的生物信息学分析工具,通过对DNA或蛋白质序列中的C domain进行快速检测和聚类分析能够对NRPS进行有效地识别和分类,亦可有效地预测其化合物[25]。NRPS predictor 2是一个通过对能够产生次生代谢产物的新NRPS基因簇进行识别和注释,并对其A domain的底物专一性进行预测和分析的在线软件[26]。本文利用生物信息学分析从岩生树花地衣型真菌中挖掘出RiNRPS基因,推断其蛋白可能为HC-toxin合成酶,潜在合成产物为HC-toxin的一种。HC-toxin是一种三轮状结构的环状四肽,是黑粉病菌(Thecaphorasolani)侵染玉米过程中的特异性和毒力的决定性因素,是微生物中不常见的次生代谢产物,是植物、昆虫和哺乳动物中组蛋白脱乙酰基酶的抑制因子[27-30];Orn是一种碱性非蛋白氨基酸,但经常存在于具生理活性的环状多肽中,如短杆菌肽S(Gramicidin S)含有两个Orn,因Orn具α-和δ-两个氨基,其中δ-氨基对短杆菌肽S在生物膜上的吸附和抗微生物活性方面尤其重要,Kondejewski等证明了这一点[31-32]。各种生物信息学分析方法结果一致,同HC毒素合成酶的同源序列比对、分子系统进化分析及antiSMASH、NaPDoS等的分析均显示该基因的转录产物可能为HC-toxin合成酶。生物信息学分析的手段,是现代生物学中发现新基因的重要技术手段,与其他生物学技术相结合可作为发现NRPS、PKS及其他一些生物合成基因研究的必要途径。本文通过对岩生树花地衣型真菌转录组数据的分析,挖掘出岩生树花地衣型真菌的RiNRPS基因,对其转录产物及其潜在的化合物做出了预测,为今后岩生树花地衣型真菌的非核糖体多肽的研究提供前期的研究基础。
参考文献:
[1]Joleen M,Wesley M,Lingjie G,etal.A PKS/NRPS/FAS hybrid gene cluster fromSerratiaplymuthicaRVH1 encoding the biosynthesis of three broad spectrum,zeamine-related antibiotics[J].PLoS one,2013,8(1):350-352.
[2]Evans B S.Nonribosomal peptide and polyketide biosynthesis methods and protocols[M].Human press,London,UK,2016:53-62.
[3]Gregory L C,James H N.Structure aspects of non-ribosomal peptide biosynthesis[J].Current Opinion in Structural Biology,2004,14(6):748-756.
[4]Madeleine P,Kristina H r,Clara B,etal.Regulation of the P450 oxygenation cascade Involved in glycopeptide antibiotic biosynthesis[J].Journal of the American Chemical Society,2016,138:6746-6753.
[5]任丹,张波,张小平,等.川楝内生真菌的遗传及PKS、NRPS基因的多样性[J].中草药,2014,45(10):1461-1467.
[6]马艳玲,邓海,魏菁菁,等.稀有海洋放线菌Salinisporaarenicola非核糖体肽合成酶和卤代酶生物合成基因簇核心区的克隆及序列分析[J].生物技术通报,2011(2):157-162.
[7]Kim S K,Wijesekara I.Development and biological activities of marine-derived bioactive peptides: A review[J].Journal of Functional Foods,2010,2(1):1-9.
[8]Kirk P M,Cannon P F,David J C,etal.Ainsworth &Bisby’s dictionary of the fungi:9thedition [M].CAB International,Wallingford Oxon,2001:1-655.
[9]文雪梅,阿迪力·阿不都拉,阿不都拉·阿巴斯,等.新疆北部树华属(RamalinaAch.)地衣生态分布与地理区系成分的初步分析[J].新疆农业科学,2009,46(3):626-629.
[10]Zrnzevic I,Stankovic M,Jovanovic V S.Ramalinacapitata(Ach.) Nyl.acetone extract: HPLC analysis,genotoxicity,cholinesterase,antioxidant and antibacterial activity[J].Excli Journal,2017,16:679-687.
[11]Liu T,Lv C G,WANG S,etal.Transcriptome-based gene mining and bioinformatics analysis of p-hydroxybenzoate geranyltransferase genes inArnebiaeuchroma[J].China Journal of Chinese Materia Medica,2016,41(8):56-63.
[12]Lautru S,Deeth R J,Bailey L M,etal.Discovery of a new peptide natural product byStreptomycescoelicolorgenome mining[J].Nature Chemical Biology,2005,1(5):265-269.
[13]Wu J,Zhong J J.Production of ginseng and its bioactive components in plant cell culture: current technological and applied aspects[J].Journal of Biotechnology,1999,68(2-3):89-99.
[14]Amselem J,Cuomo C A,van Kan J A,etal.Genomic analysis of the ecrotrophic fungal pathogensSclerotiniasclerotiorumandBotrytiscinerea[J].Plos Genetics,2011,7(8):1399-1400.
[15]O’Connell R J,Thon M R,Hacquard S,etal.Lifestyle transitions in plant pathogenicColletotrichumfungi deciphered by genome and transcriptome analyses[J].Nature Genetics,2012,44(9):1060-1065.
[16]Abdel H M,Bertrand R L,Piercey N M,etal.Identification of 6-Hydroxymellein Synthase and Accessory Genes in the LichenCladoniauncialis[J].Journal of Natural Products,2016,79(6):1645-1650.
[17]Cramer R A,Stajich J E,Yamanaka Y,etal.Phylogenomic analysis of non-ribosomal peptide synthetases in the genusAspergillus[J].Gene,2006,383: 24-32.
[18]Roongsawang N,Washio K,Morikawa M.Diversity of nonribosomal peptide synthetases involved in the biosynthesis of lipopeptide biosurfactants[J].International Journal of Molecular Sciences,2010,12(1):141-172.
[19]Meyer S,Kehr J C,Mainz A,etal.Biochemical dissection of the natural diversification of microcystin provides lessons for synthetic biology of NRPS[J].Cell Chemical Biology,2016,23(4):462-471.
[20]Pullen C,Schmitz P,Meurer K,etal.New and bioactive compounds fromStreptomycesstrains residing in the wood of Celasrraceae[J].Planta,2002,216(1):162-167.
[21]Combs J T,Franco C M,Loria R D,etal.Complete sequencing and analysis of pEN2701,a novel 13-kb plasmid from an endophyticStreptomycessp.[J].Plasmid,2003,49(1):86-92.
[][]
[22]朱鹏,郑立,林晶,等.抗菌和细胞毒活性海洋细菌的筛选及其次生代谢基因证据[J].微生物学报,2007,47(2):228-234.
[23]方剑,朱鹏,严小军.非核糖体肽合成酶的末端硫酯酶与多肽环化[J].中国生物化学与分子生物学报,2013,29(7):612-618.
[24]Kai B,Medema M H,Kazempour D,etal.antiSMASH 2.0-a versatile platform for genome mining of secondary metabolite producers [J].Nucleic Acids Research,2013,41(W1):W204-W212.
[25]Leclère V,Weber T,Jacques P,etal.Bioinformatics tools for the discovery of new nonribosomal peptides [J].Methods in Molecular Biology,2016,1401(14):209-232.
[27]Horbach R,Navarro-Quesada A R,Knogge W,etal.When and how to kill a plant cell: infection strategies of plant pathogenic fungi [J].Journal of Plant Physiology,2011,168(1):51-62.
[28]Walton J D.HC-toxin [J].Phytochemistry,2006,67(14):1406-1413.
[29]张杰,阮先乐,侯小歌,等.绿僵菌HC毒素合成酶基因的生物信息学分析[J].江苏农业科学,2015,43(9):71-74.
[30]Yamane H,Konno K,Sabelis M,etal.Chemical defence and toxins of plants [J].Chemical Ecology,2010,4:339-385.
[31]Berditsch M,Trapp M,Afonin S,etal.Antimicrobial peptide gramicidin S is accumulated in granules of producer cells for storage of bacterial phosphagens [J].Scientific Reports,2017,7:44324.
[32] Kondejewski L H,Farmer S W,Wishart D S,etal.Modulation of structure and antibacterial and hemolytic activity by ring size in cyclic gramicidin S analogs [J].Journal of Biological Chemistry,1996,271(41):25261-25268.
