植物MADS-box转录因子参与调控非生物胁迫的研究进展
2018-05-16姚琦园李纷芬张林成周升恩
姚琦园,李纷芬,张林成,周升恩
(重庆大学 生物工程学院,重庆 400044)
高等植物在生长发育过程中经常会受到环境的胁迫,如最常见的非生物胁迫干旱、低温、高盐会引起植物体内发生一系列的生理代谢反应,抑制植物的代谢和生长,严重时引起植株不可逆的伤害甚至死亡。另外,低温胁迫还会严重限制早春作物的栽种以及晚秋植物的收获。MADS-box转录因子作为重要的调控因子参与调控植物生长和发育的许多方面,包括调控植物的非生物胁迫、花器官的生长发育、根的发育、胚胎以及果实的发育等。迄今,许多学者已经对MADS-box转录因子的研究取得了很大进展,在胁迫方面的研究也取得了一定的进展,如Khong等发现水稻OsMADS26基因在植物抗旱过程中起负调控作用[1]。然而,目前还有很多MADS-box基因的功能尚不清楚,非生物胁迫方面的研究也较少。本文综述了高等植物中MADS-box转录因子的结构、分类、功能及其在非生物胁迫方面的研究进展,通过系统进化树分析了水稻MEF2-like型MADS-box家族基因,以期为进一步鉴定MADS-box转录因子的生物学功能以及培育抗逆境胁迫植株提供参考。
1 MADS-box转录因子
1.1 MADS-box转录因子的命名
MADS的名称是由最先发现的MINICHROMOS MEMAINTENANCE1 (MCM1)、AGAMOUS (AG)、DEFICIENS (DEF)及SERUMRESPONSE FACTOR (SRF)这4个转录因子的首字母组成的。MCM1来源于酵母; AG调控拟南芥的花器官; DEF是调控金鱼草一个花器官形成的决定因子; SRF是人体中调控血清应答及亚UN癌基因转录的因子。以上蛋白质的N端都含有由50~60个氨基酸组成的MADS-box保守结构域,故将含有此结构域的蛋白称为MADS-box蛋白,即由MADS-box基因编码[2]。
1.2 MADS-box转录因子的分布
MADS-box转录因子广泛存在于植物体内,从苔藓类到开花类陆生植物中均有发现[3]。在苔藓类植物中发现了共29个MADS-box基因[4]。在被子植物中发现的MADS-box基因也很多,小麦中有180个[5];基因组数据统计显示拟南芥中有107个,水稻中有约71个[6]。
1.3 MADS-box转录因子的分类及结构
MADS-box基因分为Ⅰ型(TypeⅠ)和Ⅱ型(TypeⅡ)。Ⅰ型主要分为SRF-like型、ARG80-like型和 M型三类,其中M型主要在植物中存在。Ⅱ型主要分为两类MADS-box基因,即MEF2-like型和MIKC型,其中MIKC型主要存在于植物中。MIKC型又分为MIKCc和MIKC*[7]。植物中的MIKC型MADS-box转录因子由M (MADS-box)结构域、I (Intervening)结构域、K (Keratin-like)结构域和C (C-terminal)结构域(图1)组成[8]。位于N端的MADS-box结构域是最保守的;I结构域主要与MADS-box结构域共同行使蛋白质二聚化的功能;K结构域是含有3个α-螺旋结构的次级保守结构域,是植物MADS-box蛋白特有的结构域,主要参与蛋白间的相互作用;位于C末端的C结构域可以介导不同MADS-box蛋白发挥不同的功能[9]。

图1 植物MIKC类MADS-box蛋白的结构[10]
2 MADS-box转录因子的功能
MADS-box转录因子在植物的所有器官的形态发生及植物生长发育的整个过程都有很重要的作用,例如根和叶片的生长[7]、花分生组织发育和控制开花时间[11]、花器官的发育[12]、果实成熟[13]、种子色素积累和胚胎发育[14]以及对植物非生物胁迫的调控[1]。本文就MADS-box转录因子对植物非生物胁迫调控(表1)而言,概述了MADS-box转录因子在干旱、高盐、高温、低温条件下影响植物生长的情况。
2.1 MADS-box转录因子参与干旱胁迫的响应
Khong等下调了水稻OsMADS26基因的表达,发现植株的抗旱能力较野生型的增强,而超表达该基因后植株的抗旱能力没有明显的变化[1]。这与Lee等[15]的研究结果不太一致,可能是因为品种差异,且基因芯片分析结果也有一定的差异。Khong等将转基因植株进行干旱处理后,检测了植株叶片的相对含水量(Relative Water Content, RWC)、叶绿素含量和胁迫相关基因,发现OsMADS26基因下调植株的RWC明显提高,叶绿素含量降低趋势变缓,且胁迫相关基因的表达量上调。另外,田间试验发现在干旱条件下,OsMADS26基因下调植株的产量较野生型的显著增高。Puig等发现OsMADS23和OsMADS25的表达被甘露醇强烈诱导,推测这两个基因可能参与干旱胁迫的响应[16]。Arora等指出:4个水稻MADS-box转录因子(OsMADS18、22、26和27)在响应干旱胁迫处理时,基因表达水平上调超过本底表达水平的2倍多;3个水稻MADS-box转录因子(OsMADS2、30和55)在干旱处理后,基因表达水平较未处理前下调了2倍多;其中OsMADS18可能会在胁迫条件下与OsMADS6、8、7和47相互影响[17]。Zhang等分析了玉米ZMM7-L基因,发现玉米幼苗经过甘露醇处理后ZMM7-L基因的表达量明显上升[18],说明玉米ZMM7-L基因可能参与干旱胁迫响应。张中保发现玉米ZmMADS4、ZmMADS6、ZmMADS9和ZmMADS10基因在干旱处理后被诱导表达且表达出现差异[19],预测这4个基因可能与干旱胁迫相关。Yin等用RNA干扰(RNA interference, RNAi)法沉默MADS-box基因SlMBP8,得到了转基因番茄,发现转基因番茄植株的抗旱能力明显增强;进一步检测了经干旱处理后的转基因番茄植株叶片的叶绿素含量、相对含水量、失水速率、丙二醛(MDA, Malondialdehyde)含量,以及胁迫相关基因的表达量,结果显示SlMBP8基因在抗旱的信号通路中起负调控因子的作用[20]。
2.2 MADS-box转录因子参与盐胁迫的响应
在番茄中,MADS-box基因SlMBP11在抗盐胁迫反应中起正调控作用[21]。Guo等为探究SlMBP11在番茄中的功能,构建了沉默及超表达载体,转染野生型番茄,获得了沉默及超表达转基因植株;将沉默植株用盐处理后,发现与野生型相比,沉默株系对盐的耐受性降低,其植株叶片的RWC及叶绿素含量降低,相对电导率及MDA含量均升高;除此之外,超表达植株的表型则与沉默植株相反,对盐的耐受力增强。因此,MADS-box基因SlMBP11是抗盐胁迫的正调控因子。除此之外,有研究表明番茄MADS-box基因SlMBP8作为负调控因子响应盐胁迫[20];沉默番茄MADS-box基因SlMADS23-like后,用200 mmol/L的NaCl处理转基因番茄幼苗,发现转基因幼苗的生长受抑制情况较野生型的轻,沉默SlMADS23-like基因能够增强番茄的抗盐能力[22]。水稻MADS-box基因OsMADS2、OsMADS30和OsMADS55在盐胁迫处理后,基因表达水平较未处理前下调了2倍多[17],盐处理明显地抑制了这3个基因的表达,推测这3个基因的生物学功能可能与盐胁迫相关。另外,OsMADS23和OsMADS25的表达被NaCl强烈诱导[16],推测这两个基因可能参与盐胁迫的响应。在玉米中已经发现NaCl能够诱导MADS-box基因ZMM7-L的表达,将ZMM7-L基因在拟南芥中超表达之后,超表达植株的种子在含有NaCl培养基上的发芽率比野生型的低,说明ZMM7-L基因可能是通过加速生命循环和抑制种子发芽来响应盐胁迫的负调控因子[18]。张中保对野生型玉米幼苗进行高盐处理后,发现ZmMADS4 和ZmMADS6被诱导表达且表达差异显著[19]。经过基因序列同源性比对,发现ZMM7-L和ZmMADS4是同一个基因,充分说明玉米MADS-box基因ZMM7-L在响应盐胁迫的过程中起重要作用。
2.3 MADS-box转录因子参与冷胁迫的响应
冯德党等对野生型水稻进行冷胁迫处理后,发现5个MADS-box基因(OsMADS2、OsMADS3、OsMADS18、OsMADS26和OsMADS58)出现表达异常,进而结合花粉育性和结实率推断这5个MADS-box基因可能参与植物的冷胁迫反应[23]。在玉米中,MADS-box基因ZmMADS4、ZmMADS6和ZmMADS10在低温处理下表达出现差异[19]。在番茄中,SlMADS23-like基因的沉默降低了植株的抗冷胁迫能力,因而SlMADS23-like基因在抗冷胁迫中起正调控的作用。此外, Lozano等对番茄进行冷处理后,发现MADS-box基因TM4、TM5、TM6和TAG1的表达量均有很大程度的提高[24],说明这4个基因可能均参与番茄冷胁迫的响应。
2.4 MADS-box转录因子参与其它胁迫的响应
在水稻中,OsMADS26基因超表达后,发现转基因植株出现矮化、黄化以及根卷曲等胁迫相关的表型[15];进而又获得了携带OsMADS26-GR(糖皮质激素受体)融合载体的转基因植物,通过基因芯片分析,发现转基因植株中茉莉酸等的生物合成基因和下游涉及胁迫过程的靶基因的表达上调,说明OsMADS26基因诱导了胁迫相关的多重响应[15]。除此之外,转基因植株的病原菌侵染实验发现OsMADS26基因下调的植株对病原菌的敏感性降低,表明OsMADS26基因在水稻的抗病过程中起负调控作用,该基因可能是调控和激活防御机制的中心[1]。水稻OsMADS87基因被沉默后,得到的沉默植株种子对热激的敏感性比野生型和超表达该基因的种子低,表明OsMADS87可以作为提高水稻生殖发育过程中热弹性的潜在目标基因[25]。水稻胚乳中OsMADS7的特异性抑制,能提高高温条件下直链淀粉的稳定性,因此水稻胚乳中OsMADS7的特异性下调是未来培育抗高温且高质量水稻的有效途径[26]。玉米MADS-box基因ZmMADS4、ZmMADS6和ZmMADS10能够被ABA有效地诱导表达[19]。
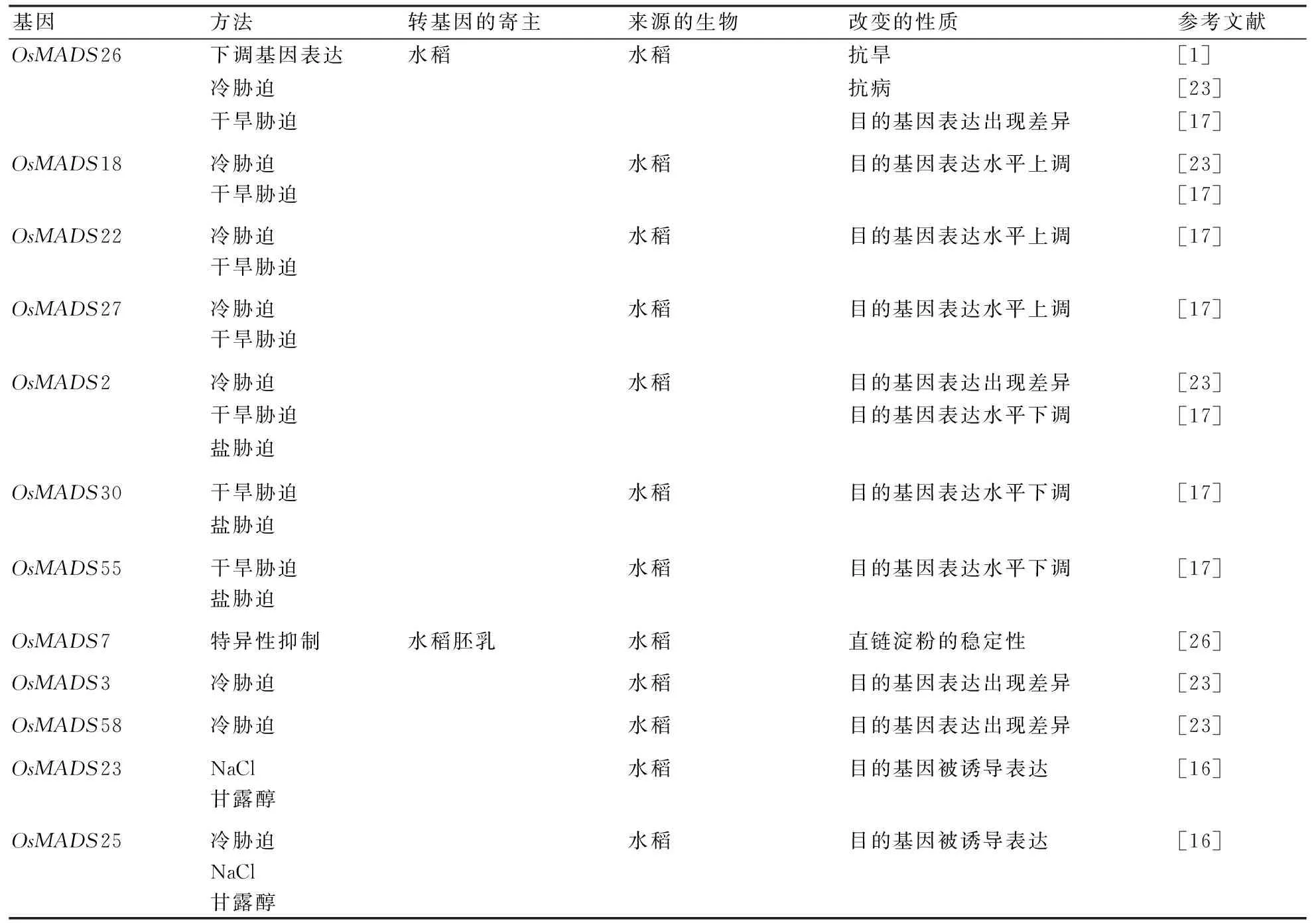
表1 已知与非生物胁迫相关的MADS-box基因及其功能
续表1:

基因方法转基因的寄主来源的生物改变的性质参考文献OsMADS87超表达水稻水稻抗热[25]ZMM7-L超表达拟南芥玉米负调控干旱抗性[18]高盐胁迫目的基因被诱导表达[19]低温胁迫ABA处理ZmMADS6干旱胁迫玉米目的基因被诱导表达[19]高盐胁迫低温胁迫ABA处理ZmMADS9干旱胁迫玉米目的基因表达出现差异[19]ZmMADS10干旱胁迫玉米目的基因表达出现差异[19]低温胁迫ABA处理SlMBP8RNAi番茄番茄负调控干旱和盐的抗性[20]SlMBP11RNAi番茄番茄不抗盐[21]超表达抗盐SlMADS23-likeRNAi番茄番茄抗盐、不耐冷[22]TM4冷胁迫番茄目的基因表达量上升[24]TM5冷胁迫番茄目的基因表达量上升[24]TM6冷胁迫番茄目的基因表达量上升[24]TAG1冷胁迫番茄目的基因表达量上升[24]
3 MADS-box蛋白的进化树分析
本文将水稻MIKC型MADS-box蛋白(从NCBI查找)和已知与胁迫相关的MADS-box蛋白共51个作了进化树分析(图2),图2中用菱形符号标出来的是已知参与胁迫调控过程的蛋白。Zhang等初步分析认为玉米中ZMM7-L基因(即ZmMADS4)可能与干旱相关[18];张中保初步分析认为玉米中ZmMADS6、ZmMADS9、ZmMADS10与干旱、低温或者激素相关[19]。在番茄中也发现了MADS-box基因SlMBP8、SlMBP11和SlMADS23-like参与非生物胁迫的调控[20-22]。另外,番茄MADS-box基因TM4和TAG1的生物学功能主要是参与果实发育调控[27-28],TM5和TM6主要参与花发育的调控[29-30],但是,这4个基因在非生物胁迫中的功能还不是很清楚。已经证实水稻OsMADS26和OsMADS87基因在抗旱和抗热的非生物胁迫中起正调控作用[1,15,25];OsMADS2等基因参与高温、低温、干旱或者盐的响应[16-17,23,25-26],但是对它们具体的生物学功能并不清楚。
图2中MADS-box蛋白的NCBI登录号如下: SlMBP8 (XP_004252712)、SlMBP11 (XP_010324817)、SlMADS23-like (XP_004252725)、TM4 (NP_001234173)、TM5 (NP_001234384)、TM6 (NP_001311309)、TAG1 (NP_001266181)、OsMADS1 (XP_015628585)、OsMADS2 (XP_015623995)、OsMADS3 (XP_015632498)、OsMADS4 (XP_015640710)、OsMADS5 (XP_015641896)、OsMADS6 (XP_015623947)、OsMADS7 (XP_015648761)、OsMADS8 (XP_015610824)、OsMADS13 (XP_015618483)、OsMADS14 (XP_015631033)、OsMADS15 (XP_015645259)、OsMADS16 (XP_015641661)、OsMADS17 (XP_015634419)、OsMADS18 (XP_015647484)、OsMADS20 (XP_015619865)、OsMADS21 (XP_015621182)、OsMADS22 (XP_015624915)、OsMADS23 (XP_015650847)、OsMADS23-like (XP_015641671)、OsMADS25 (XP_015635120)、OsMADS26 (XP_015650791)、OsMADS27 (XP_015626695)、OsMADS27-like (XP_015636306)、OsMADS29 (XP_015624838)、OsMADS30 (XP_015644346)、OsMADS31-like (XP_015635704)、OsMADS32 (XP_015642650)、OsMADS33 (XP_015618440)、OsMADS34 (XP_015631035)、OsMADS47 (XP_015630610)、OsMADS50 (XP_015630979)、OsMADS50-like (XP_015624326)、OsMADS55 (XP_015641678)、OsMADS56 (XP_015614799)、OsMADS57 (XP_015626794)、OsMADS58 (XP_015638060)、OsMADS58-like (XP_015638533)、OsMADS87 (ABF97316)、OsCAULIFLOWER- D (XP_015637117)、OsMADS SOC1-like (XP_015625202)、ZmMADS4 (NP_001171777)、ZmMADS6 (NP_001105153)、ZmMADS9 (NP_001171336)、ZmMADS10 (NP_001105333)。

图2 MADS-box蛋白的进化树分析结果
4 结论及展望
MADS-box转录因子的结构保守,在动植物和真菌中均具有1个由58个氨基酸组成的MADS-box盒[7]。MADS-box家族基因广泛调控植物生长发育过程,其中在模式植物拟南芥中MADS-box转录因子的研究较为广泛,如拟南芥中AGL6调控侧生器官发育和开花时间[31];XAANTAL1增加了拟南芥根和茎干细胞的增殖[32]。然而在水稻、玉米、番茄和小麦中MADS-box转录因子胁迫相关的研究相对匮乏(表1)。目前,在小麦中还没有关于MADS-box家族基因参与非生物胁迫的研究;在大白菜中发现有13个MADS-box转录因子响应冷胁迫,有8个响应干旱胁迫,有6个响应盐胁迫[33]。本文综述了MADS-box转录因子在胁迫方面的研究进展,发现激素对植物的影响在一定程度上能够表明非生物胁迫对植物的作用,可能激素在植物抗逆境胁迫的调控网中起重要的调控作用;也提到了许多参与胁迫反应的MADS-box转录因子,推断转录因子是通过控制抗逆境胁迫过程中的关键酶类起到调控抗逆境的作用,但是仍然有很多MADS-box转录因子具体的生物学功能还未被证实。因此今后对MADS-box转录因子在非生物胁迫方面的作用还需要进行更广泛更深入的研究。本文通过进化树分析胁迫的相关基因与水稻MIKC型MADS-box基因的同源性,来进一步解释可能参与调控非生物胁迫的基因,也为下一步的MIKC型MADS-box家族基因生物学功能研究提供了一定的参考。
近几十年来,植物中非生物胁迫方面的研究有很大的进展,包括MADS-box转录因子在其中的作用,但是仍然有很多基因的功能还未被证实。另外如何将这些胁迫相关的研究成果应用到植物抗旱、抗盐、抗低温等能力的提高上,是一个很有意义、很大的挑战[34]。除此之外,这些研究主要集中在分子水平和基因功能的研究上,植物应对非生物胁迫具体的调控机制与信号通路并不清楚,对各个胁迫相关基因间的关联性未做分析,也需要进行更深入的研究,从而更好地把握各基因的功能,为抗性植株的培育奠定基础。
参考文献:
[1] Khong G N, Pati P K, Richaud F, et al.OsMADS26 negatively regulates resistance to pathogens and drought tolerance in rice [J]. Plant Physiol, 2015, 169(4): 2935-2949.
[2] Alvarez-Buylla E R, Liljegren S J, Pelaz S, et al. MADS-box gene evolution beyond flowers: expression in pollen, endosperm, guard cells, roots and trichomes [J]. The Plant Journal, 2000, 24(4): 457-466.
[3] Gramzow L, Theissen G. A hitchhiker’s guide to the MADS world of plants [J]. Genome Biology, 2010, 11(6): 214.
[4] 易吉明,黄婷,黄勇,等.小立碗藓MADS-box基因家族的系统进化分析[J].植物生理学报,2015(2):197-206.
[5] Ma J, Yang Y J, Luo W, et al. Genome-wide identification and analysis of the MADS-box gene family in bread wheat (TriticumaestivumL.) [J]. Plos One, 2017, 12(7).
[6] Nam J, Kim J, Lee S, et al. Type I MADS-box genes have experienced faster birth-and-death evolution than type II MADS-box genes in angiosperms [J]. Proc Natl Acad Sci USA, 2004, 101(7): 1910-1915.
[7] Alvarez-Buylla E R, Pelaz S, Liljegren S J, et al. An ancestral MADS-box gene duplication occurred before the divergence of plants and animals [J]. Proc Natl Acad Sci USA, 2000, 97(10): 5328-5333.
[8] Ma H, Yanofsky M F, Meyerowitz E M.AGL1-AGL6, anArabidopsisgene family with similarity to floral homeotic and transcription factor genes [J]. Genes Dev, 1991, 5(3): 484-495.
[9] Lamb R S, Irish V F. Functional divergence within theAPETALA3/PISTILLATAfloral homeotic gene lineages [J]. Proc Natl Acad Sci USA, 2003, 100(11): 6558-6563.
[10] Kaufmann K, Melzer R, Theissen G. MIKC-type MADS-domain proteins: structural modularity, protein interactions and network evolution in land plants [J]. Gene, 2005, 347(2): 183-198.
[11] Dreni L, Zhang D B. Flower development: the evolutionary history and functions of theAGL6 subfamily MADS-box genes [J]. J Exp Bot, 2016, 67(6): 1625-1638.
[12] Smaczniak C, Immink R G H, Muino J M, et al. Characterization of MADS-domain transcription factor complexes inArabidopsisflower development [J]. Proc Natl Acad Sci USA, 2012, 109(5): 1560-1565.
[13] Vrebalov J, Ruezinsky D, Padmanabhan V, et al. A MADS-box gene necessary for fruit ripening at the tomato ripening-inhibitor(Rin) locus [J]. Science, 2002, 296(5566): 343-346.
[14] Nesi N, Debeaujon I, Jond C, et al. The TRANSPARENT TESTA16 locus encodes the ARABIDOPSIS BSISTER MADS domain protein and is required for proper development and pigmentation of the seed coat [J]. Plant Cell, 2002, 14(10): 2463-2479.
[15] Lee S, Woo Y M, Ryu S I, et al. Further characterization of a rice AGL12 group MADS-box gene,OsMADS26 [J]. Plant Physiol, 2008, 147(1): 156-168.
[16] Puig J, Meynard D, Khong G N, et al. Analysis of the expression of the AGL17-like clade of MADS-box transcription factors in rice [J]. Gene Expression Patterns, 2013, 13(5-6): 160-170.
[17] Arora R, Agarwal P, Ray S, et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress [J]. BMC Genomics, 2007, 8(1): 242.
[18] Zhang Z B, Li H Y, Zhang D F, et al. Characterization and expression analysis of six MADS-box genes in maize (ZeamaysL.) [J]. J Plant Physiol, 2012, 169(8): 797-806.
[19] 张中保.玉米干旱胁迫相关基因ZmMADS4的克隆与分析[D].北京:中国农业科学院,2007.
[20] Yin W C, Hu Z L, Hu J T, et al. Tomato (Solanumlycopersicum) MADS-box transcription factorSlMBP8 regulates drought, salt tolerance and stress-related genes [J]. Plant Growth Regulation, 2017, 83(1): 55-68.
[21] Guo X H, Chen G P, Cui B L, et al.Solanumlycopersicumagamous-like MADS-box protein AGL15-like gene,SlMBP11, confers salt stress tolerance [J]. Mol Breed, 2016, 36(9).
[22] 封叶.番茄SlMADS23-like基因的克隆及功能研究[D].重庆:重庆大学,2016.
[23] 冯德党,吕永刚,王国斌,等.水稻品种生殖生长期耐冷性及其低温胁迫下MADS-box基因表达差异分析[J].分子植物育种,2012,10(5):501-511.
[24] Lozano R, Angosto T, Gomez P, et al. Tomato flower abnormalities induced by low temperatures are associated with changes of expression of MADS-box genes [J]. Plant Physiol, 1998, 117(1): 91-100.
[25] Chen C, Begcy K, Liu K, et al. Heat stress yields a unique MADS box transcription factor in determining seed size and thermal sensitivity [J]. Plant Physiol, 2016, 171(1): 606-622.
[26] Zhang H, Xu H, Feng M, et al. Suppression ofOsMADS7 in rice endosperm stabilizes amylose content under high temperature stress [J]. Plant Biotechnol J, 2017.
[27] Bemer M, Karlova R, Ballester A R, et al. The tomatoFRUITFULLhomologsTDR4/FUL1 andMBP7/FUL2 regulate ethylene-independent aspects of fruit ripening [J]. Plant Cell, 2012, 24(11): 4437-4451.
[28] Gimenez E, Castaneda L, Pineda B, et al.TOMATOAGAMOUS1 andARLEQUIN/TOMATOAGAMOUS-LIKE1 MADS-box genes have redundant and divergent functions required for tomato reproductive development [J]. Plant Mol Biol, 2016, 91(4-5): 513-531.
[29] de Martino G, Pan I, Emmanuel E, et al. Functional analyses of two tomatoAPETALA3 genes demonstrate diversification in their roles in regulating floral development [J]. Plant Cell, 2006, 18(8): 1833-1845.
[30] Litt A, Irish V F. Duplication and diversification in theAPETALA1/FRUITFULLfloral homeotic gene lineage: implications for the evolution of floral development [J]. Genetics, 2003, 165(2): 821-833.
[31] Koo S C, Bracko O, Park M S, et al. Control of lateral organ development and flowering time by theArabidopsisthalianaMADS-box geneAGAMOUS-LIKE6 [J]. Plant J, 2010, 62(5): 807-816.
[32] Garcia C K V, Garcia P B, Garay A A, et al. The MADS-boxXAANTAL1 increases proliferation at theArabidopsisroot stem-cell niche and participates in transition to differentiation by regulating cell-cycle components [J]. Ann Bot, 2016, 118(4): 787-796.
[33] Saha G, Park J I, Jung H J, et al. Genome-wide identification and characterization of MADS-box family genes related to organ development and stress resistance inBrassicarapa[J]. BMC Genomics, 2015, 16: 178.
[34] Qin F, Shinozaki K, Yamaguchi S K. Achievements and challenges in understanding plant abiotic stress responses and tolerance [J]. Plant Cell Physiol, 2011, 52(9): 1569-1582.
