血清miR-21、miR-92联合检测对结直肠癌诊断效能分析及术后复发的预测价值
2018-05-15华红郝贵亮韩彩惠解童玲姚鲁璐
华红,郝贵亮,韩彩惠,解童玲,姚鲁璐
(1 青岛市海慈医疗集团,山东青岛266033;2 青岛市妇女儿童医院)
癌胚抗原(CEA)、糖类抗原CA199是目前临床上结直肠癌早期筛查及术后复发或转移监测的常用肿瘤标志物,但其诊断结直肠癌的敏感性仅为50%~65%,且预测术后复发的效果亦不理想,远不能满足临床需求[1,2]。微小RNA(miRNA)是一组高度保守的单链非编码小分子RNA,可通过结合到mRNA的3′非翻译区来调节其转录或降解,从而影响细胞增殖、分化、凋亡等生物学行为[3]。miRNA不仅存在于细胞中,还可以cell-free miRNA形式存在于血液中,被称为循环miRNA[4~6]。循环miRNA在反复冻融条件下仍能保持稳定,有作为肿瘤标志物的潜能[7~11]。miR-21、miR-92是miRNA家族中的成员。miR-21的编码基因定位于染色体17q23.2[12]。miRNA-92是近年新发现的一种miRNA,其编码基因定位于染色体13q31.3[13]。已有研究证实,结直肠癌患者外周血miR-21、miR-92水平上调[14]。但二者能否作为结直肠癌筛查的肿瘤标志物尚不清楚。本研究探讨血清miR-21、miR-92联合检测对结直肠癌诊断效能及术后复发的预测价值。
1 临床资料
1.2 血清miR-21、miR-92水平检测及结果分析 结直肠癌组于术前及术后1个月,对照组于体检日,采集清晨空腹外周静脉血5 mL,静置30 min,室温3 000×g离心10 min,分离血清,-80 ℃冰箱保存。采用miRNeasy血清/血浆试剂盒提取血清总RNA,采用NanoDrop Lite分光光度仪鉴定总RNA纯度,A260/A280为1.9~2.1,说明提取的总RNA纯度合格,符合实验要求。取总RNA 1 μL,严格按miscriptⅡRT试剂盒说明将总RNA逆转录为cDNA,以cDNA为模板,按miscript SYBR Green PCR试剂盒说明进行PCR扩增。所有引物由上海生工生物工程股份有限公司设计合成。引物序列:miR-21上游引物5′-GCGGCGGTAGCTTATCAGACTG-3′,下游引物5′-ATCCAGTGCAGGGTCCGAGG-3′;miR-92上游引物5′-GCGGTAGCTTATCAGACTGA-3′,下游引物5′-TGCGTGTCGTGGAGTC-3′;内参U6上游引物:5′-CTCGCTTCGGCAGCACA-3′,下游引物5′-AACGCTTCACGAATTTGCGT-3′。PCR反应体系共20 μL:cDNA 10 μL,5×miscript逆转录混合液4 μL,上下游引物各2 μL,无RNA酶水2 μL;反应条件:95 ℃ 15 min,94 ℃ 15 s、55 ℃ 30 s共40个循环,最后70 ℃ 30 s。以U6为内参,采用2-ΔΔCt法计算miR-21、miR-92相对水平。实验重复3次,取平均值。结果显示,对照组血清miR-21、miR-92相对水平分别为1.00±0.25、1.00±0.19,结直肠癌组术前血清miR-21、miR-92相对水平分别为9.63±3.12、6.50±2.59,两组比较P均<0.01。结直肠癌组术后复发者与未复发者术后1个月血清miR-21相对水平分别3.94±0.59、1.57±0.21,血清miR-92相对水平分别为2.77±0.42、1.24±0.37,二者比较P均<0.05。
1.3 血清miR-21、miR-92单独或联合检测诊断结直肠癌的效能分析 绘制血清miR-21、miR-92单独或联合检测诊断结直肠癌的ROC曲线,计算曲线下面积。结果显示,血清miR-21、miR-92单独检测诊断结直肠癌的ROC曲线下面积分别为0.892(95%CI:0.819~0.918,P<0.01)、0.905(95%CI:0.827~0.935,P<0.01),其cut off值分别为4.51、2.25,此时其诊断敏感性分别为85.4%、65.3%,特异性分别为80.3%、90.1%。二者联合检测诊断结直肠癌的ROC曲线下面积为0.973(95%CI:0.927~0.991,P<0.01),敏感性为81.3%,特异性为96.1%。见图1。
1.4 血清miR-21、miR-92单独或联合检测预测结直肠癌患者术后复发的效能分析 绘制血清miR-21、miR-92单独或联合检测预测结直肠癌患者术后复发的ROC曲线,计算曲线下面积。结果显示,血清miR-21、miR-92单独检测预测结直肠癌患者术后复发的ROC曲线下面积分别为0.802(95%CI:0.769~0.837,P<0.01)、0.855(95%CI:0.805~0.915,P<0.01),其cut off值分别为2.97、2.08,此时其预测结直肠癌患者术后复发的敏感性分别为79.9%、61.7%,特异性分别为76.2%、85.3%。二者联合检测预测结直肠癌患者术后复发的ROC曲线下面积为0.957(95%CI:0.917~0.981,P<0.01),敏感性为80.6%,特异性为95.3%。见图2。
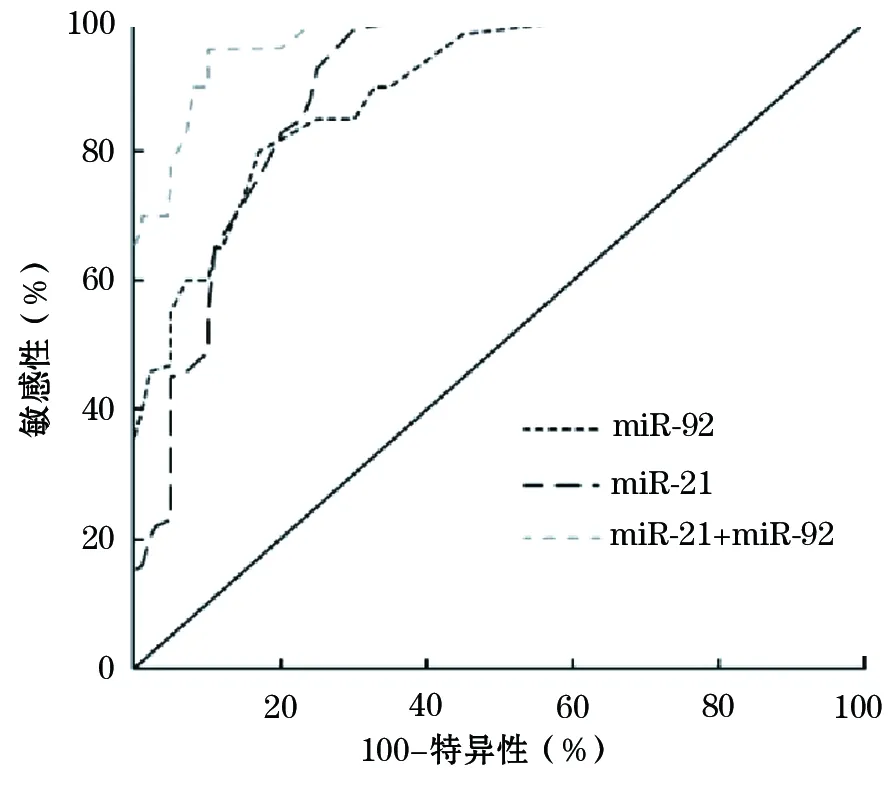
图1 血清miR-21、miR-92单独和联合检测诊断结直肠癌的ROC曲线
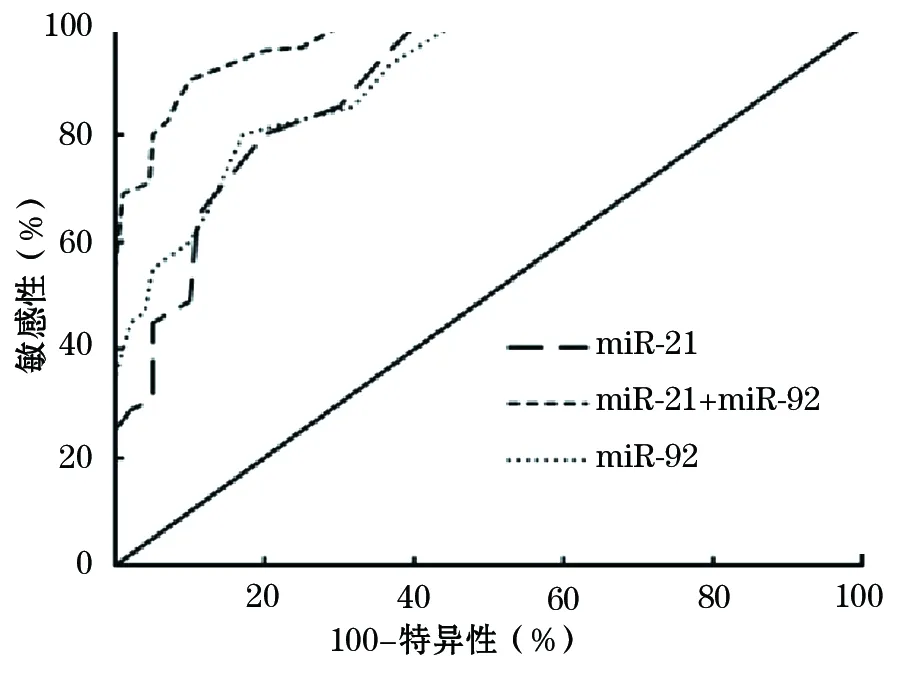
图2 血清miR-21、miR-92单独和联合预测结直肠癌患者术后复发的ROC曲线
2 讨论
miRNA是一种分子进化保守的非编码小分子RNA,长度为17~25个核苷酸,在细胞核中合成,经过Drosha酶复合体加工后,由输出蛋白5转运出细胞核,在胞质中进一步经过Dicer酶复合体的加工成为成熟的miRNA[15~18]。目前已发现1 000多种miRNA,一种miRNA可与数百甚至上千个mRNA互补结合,调控mRNA降解或沉默蛋白翻译过程,从而影响细胞增殖、凋亡等生物学行为[19,20]。研究表明,miRNA可作为癌基因或抑癌基因参与肿瘤细胞增殖、凋亡等过程[21],在肿瘤发生、发展过程中发挥重要作用。不同类型肿瘤有特异的miRNA表达谱[22],目前miRNA作为肿瘤标志物的价值备受关注。miRNA亦能在血液、尿液、乳汁、脑脊液等体液中被检出[23],而且循环miRNA在反复冻融的条件下仍能保持稳定,有作为肿瘤标志物的潜能。
结直肠癌患者外周血miR-21、miR-92水平上调。但二者能否作为结直肠癌早期筛查和术后复发预测的分子标志物尚不清楚。本研究结果显示,结直肠癌组术前血清miR-21、miR-92水平明显高于对照组,术后复发者术后1个月血清miR-21、miR-92水平高于未复发者。说明循环miR-21、miR-92有可能作为筛查结直肠癌和预测术后复发的肿瘤标志物。本研究结果显示,血清miR-21、miR-92单独诊断结直肠癌的ROC曲线下面积、诊断敏感性均高于CEA、CA199[1,2]。研究发现,不同肿瘤的特异性miRNA存在交叉性和多样性,故单项miRNA检测对结直肠癌的诊断可能无法达到理想的敏感性和特异性。本研究结果显示,血清miR-21、miR-92联合检测诊断结直肠癌的ROC曲线下面积、敏感性及特异性均明显高于二者单独检测;二者联合检测预测结直肠癌患者术后复发的ROC曲线下面积、敏感性及特异性亦均高于单独检测。说明二者联合检测对结直肠癌筛查和患者术后复发预测的效能高于二者单独检测。
综上所述,血清miR-21、miR-92可作为筛查结直肠癌和预测患者术后复发的肿瘤标志物,二者联合检测效能更佳。
参考文献:
[1] 朱珊玲,王浩.CA724、CA125、CA199、CEA在结直肠癌诊断中的价值[J].海南医学院学报,2014,20(1):87-89.
[2] 郄燕.血清中CEA、CA199及CA724联合检测在结直肠癌诊断中的意义[J].国际检验医学杂志,2012,33(14):1764-1766.
[3] Shen J, Stass SA, Jiang F. MicroRNAs as potential biomarkers in human solid tumors[J]. Cancer Lett, 2013,329(2):125-136.
[4] Savelyeva AV, Kuligina EV, Bariakin DN. Variety of RNAs in peripheral blood cells, plasma, and plasma fractions[J]. Biomed Res Int, 2017,2017:7404912.
[5] Yeri A, Courtright A, Reiman R. Total extracellular small RNA profiles from plasma, saliva, and urine of healthy subjects[J]. Sci Rep, 2017(7):44061.
[6] Weber JA, Baxter DH, Zhang S, et al. The microRNA spectrum in 12 body fluids[J]. Clin Chem, 2010,56(11):1733-1741.
[7] Yamamoto Y, Kosaka N, Tanaka M, et al. MicroRNA-500 as a potential diagnostic marker for hepatocellular carcinoma[J]. Biomarkers, 2009,14(7):529-538.
[8] Zeng W, Tu Y, Zhu Y, et al. Predictive power of circulating miRNAs in detecting colorectal cancer[J]. Tumour Biol, 2015,36(4):2559-2567.
[9] Dong Y, Yu J, Ng SS, et al. MicroRNA dysregulation as a prognostic biomarker in colorectal cancer[J]. Cancer Manag Res, 2014(6):405-422.
[10] Boisen MK, Dehlendorff C, Linnemann D, et al. Tissue microRNAs as predictors of outcome in patients with metastatic colorectal cancer treated with first line capecitabine and oxaliplatin with or without bevacizumab[J]. PLoS One, 2014,9(10):e109430.
[11] Mitchell PS, Parkin RK, Kroh EM, et al. Circulating microRNAs as stable blood-based markers for cancer detection[J]. Proc Natl Acad Sci U S A, 2008,105(30):10513-10518.
[12] Qian B, Katsaros D, Lu L, et al. High miR-21 expression in breast cancer associated with poor disease-free survival in early stage disease and high TGF-beta1[J]. Breast Cancer Res Treat, 2009,117(1):131-140.
[13] Kinose Y, Sawada K, Nakamura K, et al. The role of microRNAs in ovarian cancer[J]. Biomed Res Int, 2014,2014:249393.
[14] 刘维薇,胡婷婷,关明,等.外周血microRNAs在结直肠癌诊疗中的应用及检测方法进展[J].中华检验医学杂志,2015,10(38):653-656.
[15] Lagos-Quintana M, Rauhut R, Lendeckel W, et al. Identifcation of novel genes coding for small expressed RNAs[J]. Science, 2001,294(5543):853-858.
[16] Wang J, Zhang KY, Liu SM, et al. Tumor-associated circulating microRNAs as biomarkers of cancer[J]. Molecules, 2014,19 (2):1912-1938.
[17] Lin S, Gregory RI. MicroRNA biogenesis pathways in cancer[J]. Nat Rev Cancer, 2015,15(6):321-333.
[18] Olive V, Minella AC, He L. Outside the coding genome, mammalian microRNAs confer structural and functional complexity[J]. Sci Signal, 2015,8(368):re2.
[19] Carthew RW, Sontheimer EJ. Origins and mechanisms of miRNAs and siRNAs[J]. Cell, 2009,136(4):642-655.
[20] Yoruker EE, Aydogan F, Gezer U, et al. Analysis of circulating microRNAs during adjuvant chemotherapy in patients with luminal a breast cancer[J]. Mol Clin Oncol, 2015,3(4):954-958.
[21] Huang Y, Yang YB, Zhang XH, et al. MicroRNA-21 gene and cancer[J]. Med Oncol, 2013,30(1):376.
[22] Yates LA, Norbury CJ, Gilbert RJ. The long and short of microRNA[J]. Cell, 2013,153(3):516-519.
[23] Saikumar J, Ramachandran K, Vaidya VS. Noninvasive micromarkers[J]. Clin Chem, 2014,60(9):1158-1173.
