散发性结直肠癌组织MMR蛋白表达变化及其与患者临床病理参数和预后的关系
2018-05-15宋蒨石红建任景丽黄优华徐强孙钟武张中平
宋蒨,石红建,任景丽,黄优华,徐强,孙钟武,张中平
(1 江苏大学附属武进医院,江苏常州213002;2 郑州大学第二附属医院)
据统计,约95%结直肠癌为散发性结直肠癌(SCC)[1,2],而15% SCC是由错配修复基因(MMR)蛋白表达缺失引起的[3]。目前在人体内发现的MMR有9种,即hMLH1、hMLH3、hPMS1、hPMS2、hMSH2、hMSH3、hMSH4、hMSH5、hMSH6,其作用主要是识别错配位点并予以纠正,以保证DNA复制的准确性。如MMR发生突变,将引起机体错配修复功能缺失,导致整个基因组不稳定,从而使某些癌基因或突变的抑癌基因快速聚集,继而形成肿瘤[4]。在MMR中,hMLH1、hMSH2、hMSH6突变约占MMR突变的90%以上[5,6]。本研究观察了SCC组织MMR蛋白表达变化,现分析其表达变化与患者临床病理参数和预后的关系。
1 临床资料
1.1 基本资料 研究对象为2009年6月~2012年6月在江苏大学附属武进医院行手术治疗的SCC患者180例,均经术后组织病理检查明确诊断。患者男98例、女82例,年龄60~86(70.89±8.13)岁;肿瘤直径:≥5 cm 76例,<5 cm 104例;肿瘤形态:溃疡型95例,隆起型47例,浸润型38例;肿瘤部位:右半结肠106例,左半结肠或直肠74例;病理类型:腺癌136例,黏液腺癌或其他类型44例;TNM分期[7]:Ⅱ期86例,Ⅲ期94例;组织分化程度:高分化68例,中低分化112例;有淋巴结转移94例,无淋巴结转移86例。术后每3个月随访1次,随访方式包括门诊及住院复查、电话随访等。随访截至2017年6月,中位随访时间60个月,死亡68例。
1.2 统计学方法 采用SPSS21.0统计软件。计数资料比较采用χ2检验。生存分析采用Kaplan-Meier法,生存率比较采用Log-rank检验。P<0.05为差异有统计学意义。
1.3 SCC组织hMLH1、hMSH2、hMSH6蛋白表达检测 取手术切除的SCC组织,采用免疫组化法检测hMLH1、hMSH2、hMSH6蛋白表达。以正常结直肠黏膜上皮作为阳性对照,用PBS代替一抗作为阴性对照。结果判断:MMR蛋白定位于细胞核,以细胞核出现棕黄色或黄褐色颗粒为阳性细胞。随机选取5个200倍视野,每个视野计数100个细胞,计算阳性细胞所占比例。根据染色强度和阳性细胞所占比例综合判断MMR蛋白表达。染色强度:无着色为0分,淡黄色为1分,棕黄色为2分,棕褐色为3分。阳性细胞所占比例:无阳性细胞为0分,≤10%为1分,>10%~≤50%为2分,>50%~≤75%为3分,>75%为4分。两项评分的乘积>3分为MMR蛋白表达正常,≤3分为MMR蛋白表达缺失。hMLH1、hMSH2、hMSH6蛋白均正常表达为正常表达(pMMR),至少一项蛋白表达缺失即为阴性表达(dMMR)。本组MMR蛋白表达缺失29例,表达缺失率为16.1%。其中hMLH1蛋白表达缺失19例,hMSH2蛋白表达缺失13例,hMSH6蛋白表达缺失10例;仅hMLH1蛋白表达缺失11例,仅hMSH2蛋白表达缺失4例,仅hMSH6蛋白表达缺失3例,hMLH1、hMSH2蛋白表达均缺失4例,hMSH2、hMSH6蛋白表达均缺失3例,hMLH1、hMSH6蛋白表达均缺失2例,hMLH1、hMSH2、hMSH6蛋白表达均缺失2例。本组pMMR 29例,dMMR151例。
1.4 SCC组织MMR蛋白表达与患者临床病理参数的关系 见表1。
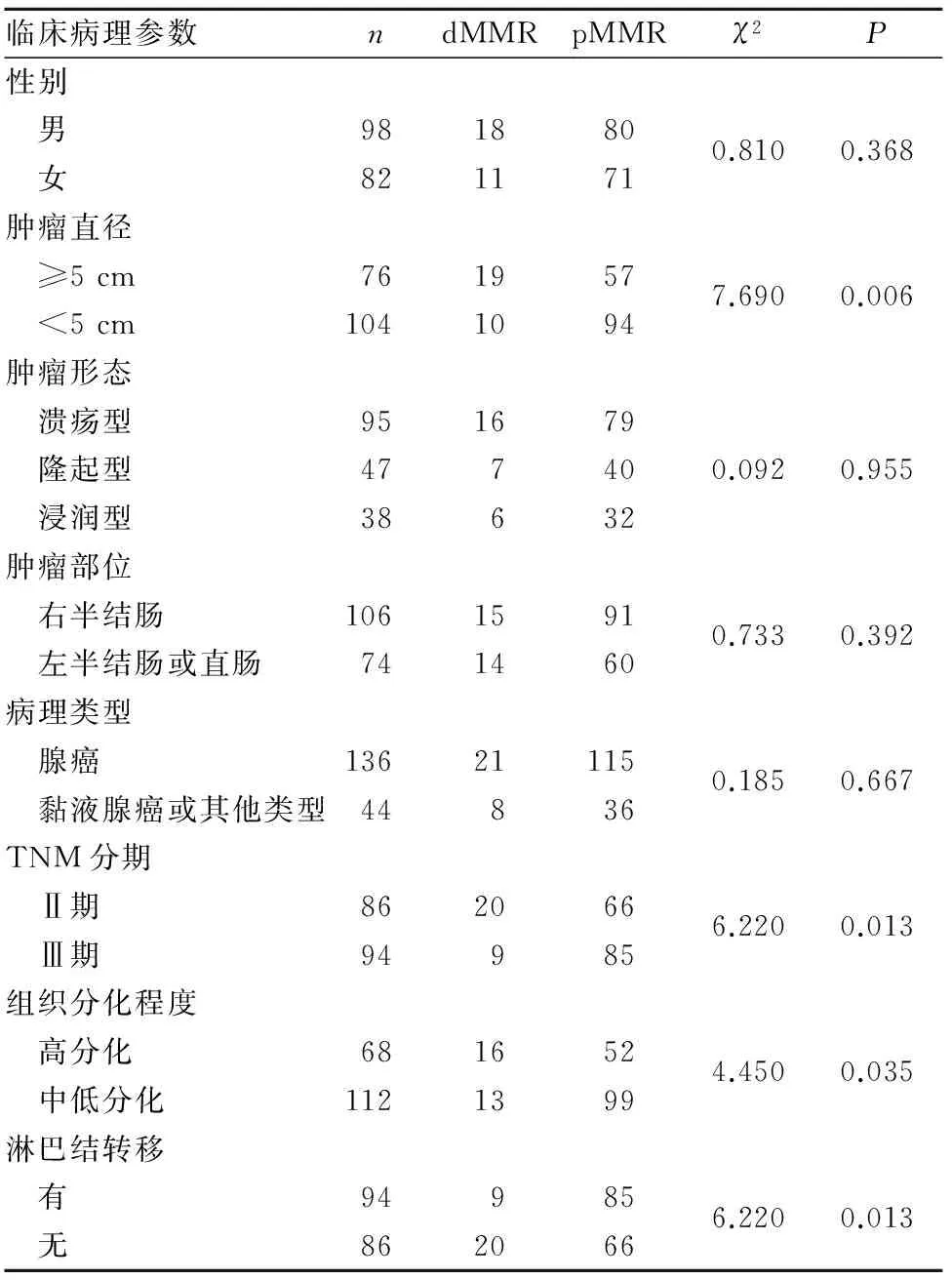
表1 SCC组织MMR蛋白表达与患者临床病理参数的关系(例)
1.5 不同MMR表达者5年无病生存率及5年总生存率比较 采用Kaplan-Meier法绘制生存时间曲线,经Log-rank检验,dMMR者与pMMR者5年无病生存率分别为75.9%(22/29)、43.7%(66/151),5年总生存率分别为82.8%(24/29)、58.3%(88/151),两者比较差异均有统计学意义(χ2分别为10.872、6.044,P均<0.01)。见图1、2。
2 讨论
1993年Lothe等[8]发现了一条新的可导致SCC途径——MMR途径。MMR蛋白的主要功能是保证基因复制的精确性,在DNA复制过程中出现碱基错配时,MMR识别并予以修复,以保证基因组稳定性。如MMR发生突变,将引起错配修复功能缺失,导致整个基因组不稳定,从而使某些癌基因或突变的抑癌基因快速聚集,继而形成肿瘤[4]。
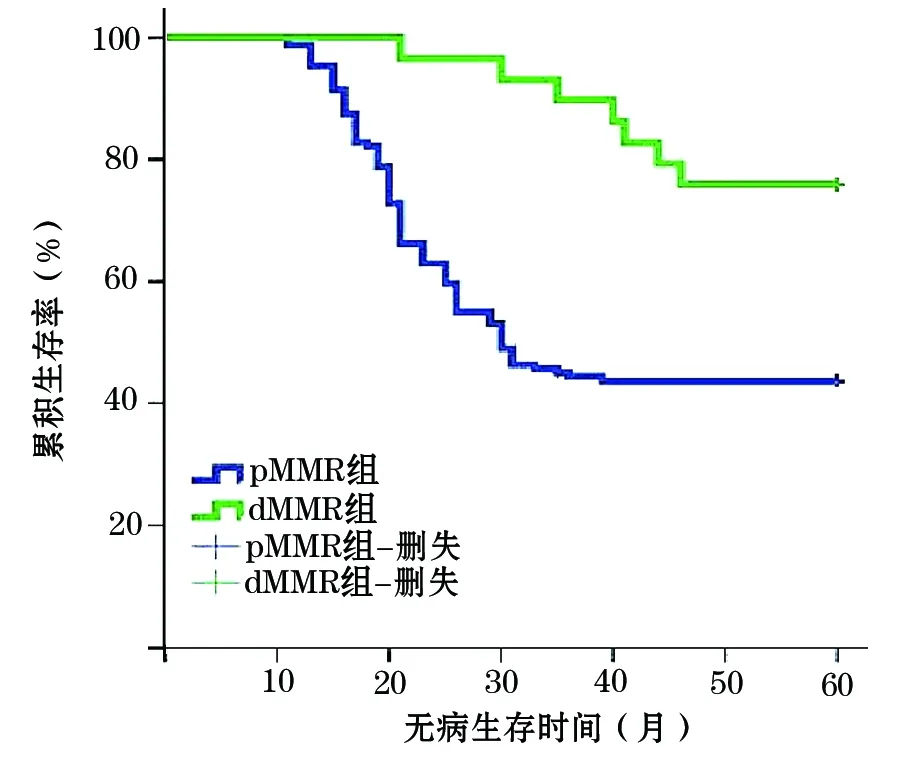
图1 不同MMR表达SCC患者无病生存时间曲线
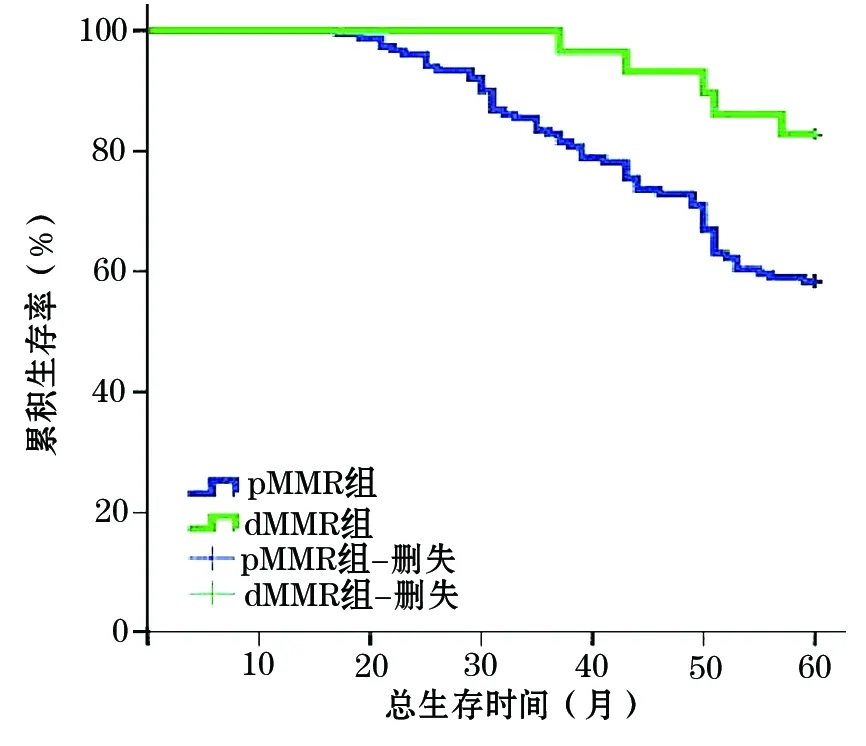
图2 不同MMR表达SCC患者总生存时间曲线
目前已在人体内发现了9种MMR蛋白,任意一个MMR蛋白表达缺失,均会导致MMR系统功能缺失。在MMR突变中,hMLH1、hMSH2、hMSH6突变超过90%。hMLH1、hMSH2、hMSH6多形成异源二聚体发挥作用,可特异性识别核苷酸链上的错配位点,并进行切除、修复。hMLH1蛋白在错配修复过程中主要参与核酸内切酶激活,在切开碱基及解除双螺旋过程中发挥重要作用,并参与合成新的核苷酸链;还可与hPMS1形成Mutlα,纠正错误插入的碱基。hMSH2是最早发现的MMR蛋白,其表达于所有正常结直肠黏膜,通过与hMLH1蛋白结合形成异源二聚体hMutLα,hMutLα识别并结合错配修复的碱基,继而激活核酸内切酶,切除错配的碱基。hMSH6具有ATP酶活性,在错配修复过程中发挥调节作用,还可与hMSH2蛋白形成二聚体,接受ATP供能,与相应的错配碱基结合并将其游离。在DNA复制过程发生碱基错配后,MMR系统启动修复。修复过程包括错配碱基识别、MMR蛋白聚集、错配碱基修复。相应的MMR蛋白在形成异二聚体后,切除包含错配碱基的DNA片段,然后补充新合成的正确DNA片段,以确保基因信息准确。当MMR基因发生突变或修饰后,MMR蛋白表达缺失,继而MMR功能缺失,最终导致肿瘤发生[9]。MMR蛋白低表达可降低基因错配修复功能,增加肿瘤发生的易感性。其引起肿瘤发生的机制:①MMR功能缺失可引起基因组变化,造成简单重复序列的累积,发生微卫星不稳定,而微卫星不稳定可造成某些癌基因的激活,进一步诱发肿瘤[10];②MMR系统缺陷会直接造成某些原癌基因和抑癌基因突变,如无法及时纠正,将导致癌基因异常表达、累积,最终导致肿瘤发生。
本研究结果显示,SCC组织MMR蛋白表达缺失率达16.1%,与Ghanipour等[11]研究结果基本一致。既往研究发现,MMR蛋白表达缺失与肿瘤某些临床病理特征有关[12,13]。本研究发现,MMR蛋白表达缺失与肿瘤直径、TNM分期、组织分化程度、淋巴结转移有关,与既往研究[14,15]结果基本一致。有研究发现,MMR蛋白表达变化与肿瘤患者预后密切相关[14,16];以5-Fu为基础的化疗可延长结直肠癌患者生存时间,提高患者5年生存率[17]。但也有研究认为,dMMR者无法从以5-Fu为基础的化疗方案中获益,对TNM分期为Ⅱ期的dMMR者甚至有害[18]。本研究发现,dMMR者5年无病生存率及总生存率均高于pMMR者。提示dMMR者预后相对较好,可能与MMR可增强患者免疫监视功能有关[19],也可能与肿瘤组织胸苷合成酶和二氢嘧啶脱氢酶过表达有关[13]。
综上所述,SCC组织可出现MMR蛋白表达缺失,其表达变化与肿瘤的发生、发展及患者预后有关。
参考文献:
[1] Siegel R, Desantis C, Jemal A. Colorectal cancer statistics, 2014[J]. CA Cancer J Clin, 2014,64(2):104-117.
[2] Hashmi AA, Ali R, Hussain ZF, et al. Mismatch repair deficiency screening in colorectal carcinoma by a four-antibody immunohistochemical panel in Pakistani population and its correlation with histopathological parameters[J]. World J Surg Oncol, 2017,15(1):116.
[3] Farchoukh L, Kuan SF, Dudley B, et al. MLH1-deficient colorectal carcinoma with wild-type BRAF and MLH1 promoter hypermethylation harbor KRAS mutations and arise from conventional adenomas[J]. Am J Surg Pathol, 2016,40(10):1390-1399.
[4] Lothe RA, Peltomki P, Meling GI, et al. Genomic instability in colorectal cancer: relationship to clinicopathological variables and family history[J]. Cancer Res, 1993,53(24):5849-5852.
[5] Niessen RC, Berends MJ, Wu Y, et al. Identification of mismatch repair gene mutations in young patients with colorectal cancer and in patients with multiple tumours associated with hereditary non-polyposis colorectal cancer[J]. Gut, 2006,55(12):1781-1788.
[6] Day LW, Velayos F. Colorectal cancer of the elderly[J]. Curr Treat Options Gastroenterol, 2014,12(3):269-282.
[7] Edge SB, Compton CC. The American Joint Committee on Cancer: the 7th edition of the AJCC cancer staging manual and the future of TNM[J]. Ann Surg Oncol, 2010,17(6):1471-1474.
[8] Lothe RA, Peltomäki P, Meling GI, et al. Genomic instability in colorectal cancer: relationship to clinicopathological variables and family history[J]. Cancer Res, 1993,53(24):5849-5852.
[9] Vlaykova T, Mitkova A, Stancheva G, et al. Microsatellite instability and promoter hypermethylation of MLH1 and MSH2 in patients with sporadic colorectal cancer.[J]. J BUON, 2011,16(2):265-273.
[10] Shia J. Immunohistochemistry versus microsatellite instability testing for screening colorectal cancer patients at risk for hereditary nonpolyposis colorectal cancer syndrome : Part I. The Utility of Immunohistochemistry[J]. J Mol Diagn, 2008,10(4):293-300.
[11] Ghanipour L, Jirström K, Sundström M, et al. Associations of defect mismatch repair genes with prognosis and heredity in sporadic colorectal cancer[J]. Eur J Surg Oncol, 2017,43(2):311-321.
[12] Korphaisarn K, Pongpaibul A, Limwongse C, et al. Deficient DNA mismatch repair is associated with favorable prognosis in Thai patients with sporadic colorectal cancer[J]. World J Gastroenterol, 2015,21(3):926-934.
[13] Etienne-Grimaldi MC, Mahamat A, Chazal M, et al. Molecular patterns in deficient mismatch repair colorectal tumours: results from a French prospective multicentric biological and genetic study[J]. Br J Cancer, 2014,110(11):2728-2737.
[14] Park JW, Chang HJ, Park S, et al. Absence of hMLH1 or hMSH2 expression as a stage-dependent prognostic factor in sporadic colorectal cancers[J]. Ann Surg Oncol, 2010,17(11):2839-2846.
[15] Andersen HS, Bertelsen CA, Henriksen R, et al. The pathological phenotype of colon cancer with microsatellite instability[J]. Dan Med J, 2016,63(2). pii:A5198.
[16] Lanza G, Gafà R, Santini A, et al. Immunohistochemical test for MLH1 and MSH2 expression predicts clinical outcome in stage Ⅱ and Ⅲ colorectal cancer patients[J]. J Clin Oncol, 2006,24(15):2359-2367.
[17] Sinicrope FA, Mahoney MR, Smyrk TC, et al. Prognostic impact of deficient DNA mismatch repair in patients with stage Ⅲ colon cancer from a randomized trial of FOLFOX-based adjuvant chemotherapy[J]. J Clin Oncol, 2013,31(29):3664-3672.
[18] Sargent DJ, Marsoni S, Monges G, et al. Defective mismatch repair as a predictive marker for lack of efficacy of fluorouracil-based adjuvant therapy in colon cancer[J]. J Clin Oncol, 2010,28(20):3219-3226.
[19] Scarpa M, Ruffolo C, Canal F, et al. Mismatch repair gene defects in sporadic colorectal cancer enhance immune surveillance[J]. Oncotarget, 2015,6(41):43472-43482.
