木薯sRNA测序分析及其开花相关microRNA的挖掘
2018-05-14丛汉卿龙娅丽王荣香孙化鹏
丛汉卿 龙娅丽 王荣香 孙化鹏
摘 要 为研究木薯花序与叶片中sRNA表达特性,探索microRNA对其开花过程的调控。本研究以木薯品种“华南八号”花序和幼叶为材料,利用RNA-seq技术进行sRNA测序分析;并筛选开花相关microRNAs进行表达特性分析。从花序和幼叶测序结果中分别获得12 092 109条和11 499 655条sRNA序列。花序中筛选出139条已知microRNAs和253条新预测microRNA,分别预测得到992和3 736条靶基因;幼叶中筛选出134条已知microRNA和191条新预测microRNAs,分别预测得到979和2 603条靶基因。最终筛选出8种与开花调控相关和3种花色调控相关的microRNAs。表达分析显示,miRNAs在两样品间呈现出较大的整体性差异,并且在功能与代谢通路上也不尽相同;开花相关microRNAs中表达差异较大的为miR156、miR172和miR169,其中miR156表达量在花序中高于幼叶,而miR172表达量在花序中低于幼叶,推测其功能为抑制后续的开花过程,避免过度开花。本研究分析了木薯花序与幼叶中sRNA的整体特性,通过表达差异分析初步明确了microRNAs对木薯开花的调控,为进一步深入探索奠定了基础。
关键词 木薯;sRNA;microRNA;开花;表达特性中图分类号 S533 文献标识码 A
DOI10.3969/j.issn.1000-2561.2018.12.015
木薯(Manihot esculentaCrantz)是世界三大薯类作物之一,也是第四大粮食作物,原产地为南美洲,属于大戟科(Euphorbiaceae)木薯属(ManihotP. Miller)[1-2]。木薯的主产区位于热带和亚热带地区[3]。木薯在中国广东、广西、云南等主产区不能正常开花结果以获得成熟的木薯种子,从而无法在当地实现木薯杂交育种工作,其原因在于当地的气候条件,因此目前中国木薯杂交育种工作仅限在海南岛开展,而如广西地区种植的品种主要依赖从国外或海南等地引进,且品种单一、部分品种已开始退化[4]。因此,海南以外木薯主产区无法进行杂交育种的问题,已成为中国木薯产业发展的重大课题之一,木薯开花问题的研究就显得尤为关键。研究和利用木薯开花信号途径中关键调控因子的调控特性,是利用生物技术解决这一问题的有力支撑。
MicroRNAs,又称为miRNAs,长度为20~24个碱基的非编码单链小RNA。通过碱基互补配对原则特异地结合在靶基因mRNA上,在转录后水平抑制或降解基因的表达,是一种负调控因子[5]。miRNAs广泛参与植物发育过程,例如植物器官的形态建成、激素应答、逆境胁迫与营养代谢、phasiRNAs和自身代谢的反馈调节等功能[6]。开花作为植物从营养生长向生殖生长转变的重要事件[7],受一系列复杂的内源和外源信号途径所组成的网络所调控。miRNAs在这一网络中,处于相当关键的位置,参与了从时期转变到花芽形成再到花器官分化的整个过程。
对木薯sRNA的研究,前人已有很多相关报道,涉及miRNAs的挖掘[8]、ta-siRNAs和顺式- siRNAs对白叶枯病的响应[9]、非生物胁迫下miRNA的表达[10]、miRNA对木薯淀粉合成的影响[11]以及在木薯不同组织中miRNA的筛选与表达[12],然而至今尚没有对于木薯开花相关miRNAs的针对性研究报道。本研究通过对木薯品种“华南八号”的花序和幼叶sRNA测序数据的对比分析,对木薯的sRNAs序列进行了分类注释;获得了大量的miRNA及其靶基因;并挖掘了开花相关miRNA,根据其在两样品间的表达特性差异,初步明确了其对木薯开花的调控过程。本研究旨在通过木薯开花相关miRNAs的挖掘和表达分析,明确有哪些miRNAs参与了开花过程,并初步确定其表达特性。为进一步探索木薯的开花调控过程,及miRNAs如何在其中发挥相应的调控功能奠定基础,并对后续利用分子手段研究和解决除海南外其他中国木薯主产区的开花问题提供理论支持。
1 材料与方法
1.1 材料及预处理
实验材料为木薯品种“华南八号”,2014年9月栽培于中国热带农业科学院热带作物品种资源研究所木薯中心实验基地。挑选生长发育良好、生长趋势均一、花序发育初期的植株。分别采集完整花序与幼叶,液氮速冻后于-80 ℃冰箱保存。
1.2 方法
1.2.1 总RNA提取 分别将两组样品液氮研磨后,使用Axygen总RNA小量制备试剂盒(Axygen)分别提取幼葉与花序样品的总RNA,操作步骤参照试剂盒说明书。通过琼脂糖凝胶电泳检测完整性,并用核酸蛋白分析仪DU800(Beckman Coulter)检测浓度和纯度。
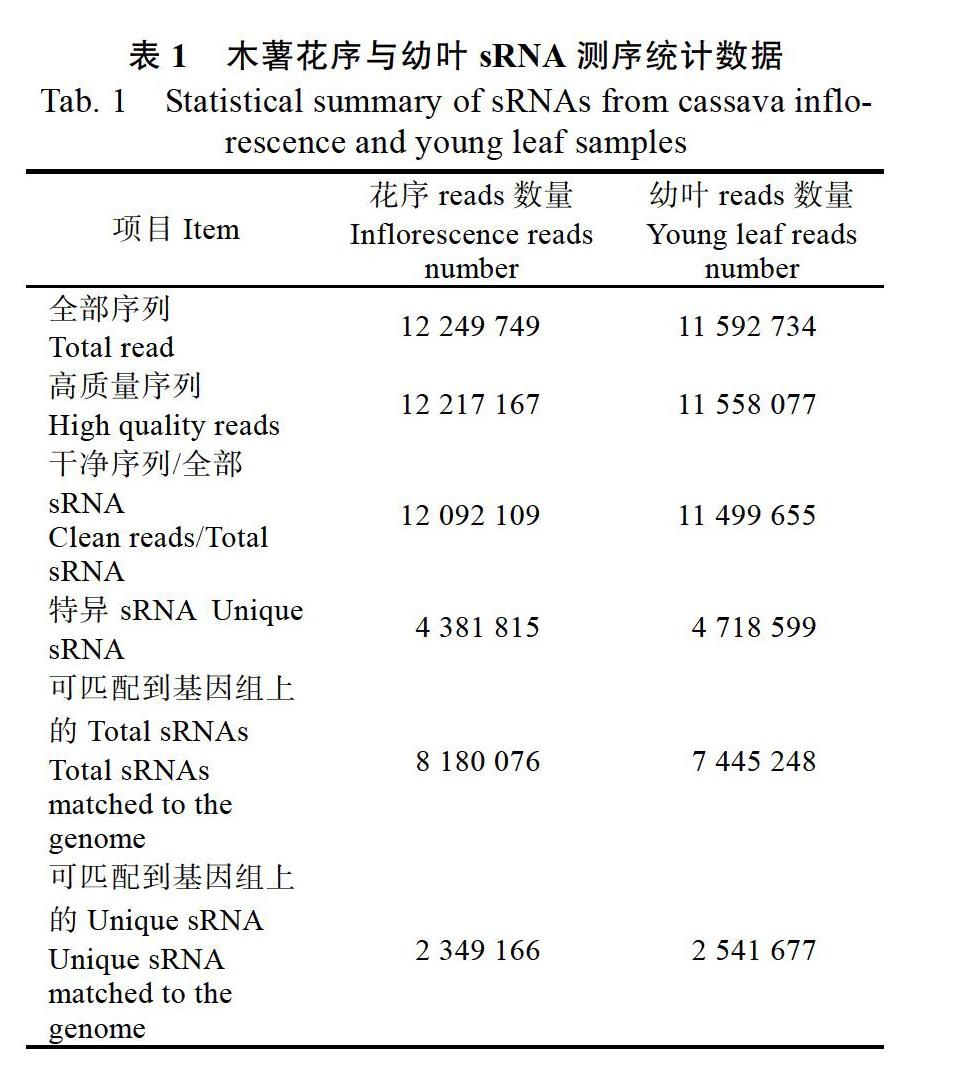
1.2.2 sRNA测序 花序和幼叶样品的总RNA,送至深圳华大基因(BGI)分别进行sRNA测序,参考基因组为JGI的木薯基因组数据库(http:// phytozome.jgi.doe.gov/pz/portal.html#!info?alias= Org_Mesculenta_er)[13]。

2.5 靶基因预测
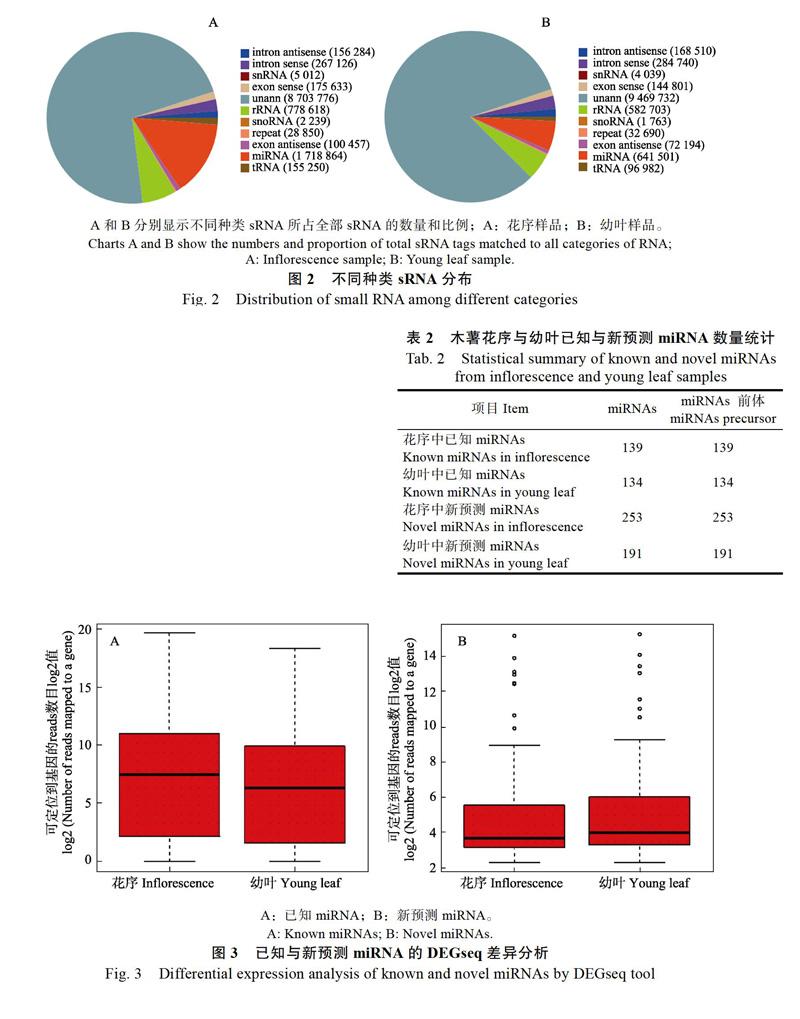
利用psRobot和TargetFinder工作对已知和新预测miRNAs进行了靶基因预测,我们发现,虽然已知miRNAs和新预测miRNAs的数目相差不大,但二者靶基因数目却有较大的差距(表3)。新预测miRNAs的靶基因数远多于已知miRNAs,因此新预测miRNAs有更多的未知功能值得探究。
2.6 开花相关miRNAs的筛选与分析
通过参考前期文献的报道,筛选出9种与开花相关的miRNAs(表4)。其中8种为开花调控相关,分别为miR156/157、miR159、miR164、miR165/166、miR167、miR169、miR172和miR319a,涉及调节生物生长时期转变、开花时间及花器官发育等功能;3类与花色相关,可调控花青素合成相关基因,分别为miR156/157、miR165/166、miR828。其中156家族与165/166家族具有双重功能,既可以调控花的生长发育,又可调控花青素的合成,从而进一步影响花色。从靶基因预测结果显示这些miRNA都具有不同数目的靶基因位点,miR156/157较多,具有76个,而miR165/166较少,仅为8个。
从上述开花相关的miRNAs中挑选出较为典型的几个成员进行表达差异分析(图5),表达差异较大(≥2倍)的为miR156、miR172和miR169,其余成员差异并不显著。
花序中miR156表达量远远大于幼叶,约为34倍。高表达的miR156可抑制SPL家族转录因子,抑制了植物从营养生长转向生殖生长期。miR172幼叶中的表达量约为花序中的3.7倍。花发育之前,miR172的靶基因——AP2基因家族成员可以抑制开花,花发育开始后,其又会参与调节分生组织的形成和器官生成。miR169在花序中表达量明显大于幼叶中,约为5.6倍。
表达差异并不显著的miRNAs中,miR159在两样品中表达水平基本一致;miR164在花序中略大于幼叶,约1.86倍;miR166在两者中都具有较高的表达水平,reads数皆大于30×104,花序略大于幼叶,约1.35倍,因为miR166除可影响花器官极性外,还可调控叶片的极性、茎尖和侧面的分生组织的形成、导管的发育等,所以在2个样品中都有较高表达量;miR167的2个成员部分基本一致,叶片中含量约为花序中的1.29倍,其靶基因ARF家族作为生长素响应因子,在植物体内广有分布;miR319的reads数,在花序中50条,幼叶中31条,表达量较低,差异不够显著。miR319家族的序列与miR159很类似,但其靶基因除了TCP外,还可调控相关MYB转录因子,木薯中较低的表达量,是否说明其功能与159有冗余,还需进一步验证。此外,可參与花青素合成调控的miR828,因测序所获得的reads数极少,花序中3条,幼叶中1条。
3 讨论
sRNA测序数据中,仅有少量低质量序列,说明数据质量较高,可为具体分析提供基础。另有部分测序reads无法匹配到基因组数据上,可能因为所使用的参考基因组数据不够完整。sRNA的组成非常复杂,包含有多种非编码RNA(ncRNA),已知的如miRNA、snRNA、snoRNA和重复序列(repeat)等都具有其独特的生物学功能。本研究中木薯花序和幼叶中分别有超过70%和80%的sRNAs无法确定其种类和功能,具有进一步挖掘和研究的价值。
本研究发现,木薯花序样品中miRNA的整体表达数量约为幼叶中的3倍,说明花序中含有更为丰富的miRNA参与开花的调控,较多的数量能够增强其调控能力,从侧面印证了miRNAs在开花调控过程中的作用。两样品中新预测的miRNAs共有257个成员,其中部分成员前体具有较为完美的发卡结构,预示其有极大可能为真正的miRNA,结合其预测的靶基因信息,具有一定的后续研究价值。在miRNAs的表达差异方面,根据DEGseq、GO、KEGG分析结果可以看出已知两样品的miRNAs和新预测miRNAs在总体表达特征、功能、代谢通路上都具有一定的差异,说明了其器官分布和调控功能的特性。
木薯中具有全部的已有报道的花发育相关miRNAs[39],但在可能起调控花色的miRNAs中,缺少miR858[40]和miR778[41],其中miR858调控MYB12,miR778的研究发现过表达会增加花青素含量,沉默表达则基本无影响,说明miR778对花色调控并非起关键作用。在金鱼草和矮牵牛中,miR169能够抑制C类基因[42],推测木薯花序中的相对高表达的miR169可以维持花的正常发育。差异表达的miRNAs中较为特别的是对植物开花进行负调控的miR156和正调控的miR172,花序中有更高的miR156和更低的miR172含量。已有研究表明,mi156在营养生长时期的表达量显著高于生殖生长时期[43]。猜测因花序已经完成了从营养生长向生殖生长的转变,处于正常的开花进程,高表达的miR156可以抑制植物后续过度开花。花序中相对低的miR172表达量说明AP2类转录因子的抑制作用得到了一定的解除,鉴于花的发育已经开始,其主要作用可能为调控花器官的生成,同时也抑制植株过度开花,影响植株正常的生理过程。鉴于miR156和miR172相对较高的表达数量及较为悬殊的表达差异,说明这两类miRNAs在木薯开花过程中较为活跃,并起着相对重要的作用。
本研究中的花序样品为处于不同发育时期的雌雄花多种花器官的混合样品,只能在整体上体现开花相关miRNAs的表达特点,无法特异反映其在不同时期和不同器官中的表达特性。要明确其具体调控过程,需要在开花过程中选择更密集的时间点,取用更特异的花器官为材料进行测序,并结合相应转录组测序中靶基因表达量数据进行关联分析。本文初步研究了miRNAs在花序和幼叶中的表达差异,为进一步探索miRNAs如何调控木薯的开花奠定了基础,通过后续研究,可为解决中国除海南以外主产区木薯无法正常开花并进行杂交育种的问题提供思路。
参考文献
- 韦本辉. 中国木薯栽培技术与产业发展[M]. 北京: 中国农业出版社, 2008.
- 周 超,王 晋. 木薯华南7号在普洱引种的表现及高产栽培技术[J]. 云南农业, 2008(6): 11-12.
- 劳天源. 广西木薯生产的展望[J]. 广西农学院学报, 1987(1): 1-8.
- 韦本辉, 甘秀芹, 陆柳英, 等. 广西木薯诱导开花结实及发芽试验研究初报[J]. 广西农业科学, 2009, 40(8): 982- 986.
- Zhang B H, Wang Q L, Pan X P. MicroRNAs and their regulatory roles in animals and plants[J]. Journal of Cellular Physiology, 2007, 210(2): 279-289.
- 鲁玉柱, 封 振, 边黎颖, 等. 植物发育与microRNA[J]. 西北植物学报, 2009, 29(5): 1 066-1 072.
- Poethig R S. Phase change and the regulation of developmental timing in plants[J]. Science, 2003, 301(5631): 334-336.
- Patanun O, Lertpanyasampatha M, Sojikul P, et al. Computational identification of MicroRNAs and their targets in cassava (Manihot esculenta Crantz.)[J]. Molecular Biotechnology, 2013, 53(3): 257-269.
- Quintero A, Pérez-Quintero A L, López C. Identification of ta-siRNAs and cis-nat-siRNAs in cassava and their roles in response to cassava bacterial blight[J]. Genomics, Proteomics & Bioinformatics, 2013, 11(3): 172-181.
- Ballén-Taborda C, Plata G, Ayling S, et al. Identification of cassava MicroRNAs under abiotic stress[J]. International Journal of Genomics, 2013, 2013: 1-10.
- Chen X, Xia J, Xia Z Q, et al. Potential functions of microRNAs in starch metabolism and development revealed by miRNA transcriptome profiling of cassava cultivars and their wild progenitor[J]. BMC Plant Biology, 2015, 15(1): 33.
- Khatabi B, Arikit S, Xia R, et al. High-resolution identification and abundance profiling of cassava (Manihot esculenta Crantz) microRNAs[J]. BMC Genomics, 2016, 17: 85.
- Prochnik S, Marri P R, Desany B, et al. The cassava genome: current progress, future directions[J]. Tropical Plant Biology, 2012, 5(1): 88-94.
- Li R, Yu C, Li Y, et al. SOAP2: an improved ultrafast tool for short read alignment[J]. Bioinformatics, 2009, 25(15): 1 966-1 967.
- Kalvari I, Argasinska J, Quinonesolvera N, et al. Rfam 13.0: shifting to a genome-centric resource for non-coding RNA families[J]. Nucleic Acids Research, 2018, 46(D1): D335-D342.
- Kalvari I, Nawrocki E P, Argasinska J, et al. Non-coding RNA analysis using the rfam database[J]. Current Protocols in Bioinformatics, 2018, 62(1): e51.
- Kozomara A, Griffiths-Jones S. miRBase: annotating high confidence microRNAs using deep sequencing data[J]. Nucleic Acids Research, 2014, 42(D1): D68-D73.
- Wu H J, Ma Y K, Chen T, et al. PsRobot: a web-based plant small RNA meta-analysis toolbox[J]. Nucleic Acids Research, 2012, 40(W1): W22-W28.
- Allen E, Xie Z, Gustafson A M, et al. microRNA-directed phasing during trans-acting siRNA biogenesis in plants[J]. Cell, 2005, 121(2): 207-221.
- Fahlgren N, Howell M D, Kasschau K D, et al. High- throughput sequencing of arabidopsis microRNAs: evidence for frequent birth and death of MIRNA genes[J]. PLoS ONE, 2007, 2(2): e219.
- Wang L K, Feng Z X, Wang X, et al. DEGseq: an R package for identifying differentially expressed genes from RNA-seq data[J]. Bioinformatics, 2010, 26(1): 136-138.
- Poethig R S. Small RNAs and developmental timing in plants[J]. Current Opinion in Genetics & Development, 2009, 19(4): 374-378.
- Gou J Y, Felippes F F, Liu C J, et al. Negative regulation of anthocyanin biosynthesis inarabidopsisby a mir156-targeted SPL transcription factor[J]. The Plant Cell, 2011, 23(4): 1 512-1 522.
- Millar A A, Gubler F. The arabidopsis GAMYB-like genes, MYB33 and MYB65, are MicroRNA-regulated genes that redundantly facilitate anther development[J]. The Plant Cell, 2005, 17(3): 705-721.
- Huang T, López-Giráldez F, Townsend J P, et al. RBE controls microRNA164 expression to effect floral organogenesis[J]. Development, 2012, 139(12): 2 161-2 169.
- Husbands A Y, Chitwood D H, Plavskin Y, et al. Signals and prepatterns: new insights into organ polarity in plants[J]. Genes & Development, 2009, 23(17): 1 986-1 997.
- Mallory A C, Reinhart B J, Jones-Rhoades M W, et al. MicroRNA control of PHABULOSA in leaf development: importance of pairing to the microRNA 5′ region[J]. The EMBO Journal, 2004, 23(16): 3 356-3 364.
- Kim J, Jung J H, Reyes J L, et al. microRNA-directed cleavage of ATHB15 mRNA regulates vascular development in Arabidopsis in florescence stems[J]. The Plant Journal, 2005, 42(1): 84-94.
- Yan J, Gu Y, Jia X, et al. Effective small RNA destruction by the expression of a short tandem target mimic in arabidopsis[J]. The Plant Cell, 2012, 24(2): 415-427.
- Wu M F, Tian Q, Reed J W. Arabidopsis microRNA167 controls patterns of ARF6 and ARF8 expression, and regulates both female and male reproduction[J]. Development, 2006, 133(21): 4 211-4 218.
- Cartolano M, Castillo R, Efremova N, et al. A conserved microRNA module exerts homeotic control over Petunia hybrida and Antirrhinum majus floral organ identity[J]. Nature Genetics, 2007, 39(7): 901-905.
- Lauter N, Kampani A, Carlson S, et al. MicroRNA172 down-regulates glossy15 to promote vegetative phase change in maize[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(26): 9 412-9 417.
- Wu G, Park M Y, Conway S R, et al. The sequential action of mir156 and mir172 regulates developmental timing in arabidopsis[J]. Cell, 2009, 138(4): 750-759.
- Crawford B C W, Nath U, Carpenter R, et al. CINCINNATA controls both cell differentiation and growth in petal lobes and leaves of antirrhinum[J]. Plant Physiology, 2004, 135(1): 244-253.
- Efroni I, Blum E, Goldshmidt A, et al. A protracted and dynamic maturation schedule underlies Arabidopsis leaf development[J]. The Plant Cell, 2008, 20(9): 2 293-2 306.
- Luo D, Carpenter R, Copsey L, et al. Control of organ asymmetry in flowers of antirrhinum[J]. Cell, 1999, 99(4): 367-376.
- Hsieh L C, Lin S I, Shih A C C, et al. Uncovering small RNA-mediated responses to phosphate deficiency in arabidopsis by deep sequencing[J]. Plant Physiology, 2009, 151(4): 2 120-2 132.
- Chen H M, Chen L T, Patel K, et al. 22-nucleotide RNAs trigger secondary siRNA biogenesis in plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(34): 15 269-15 274.
- 王 榮, 魏梦苒, 赵大球. miRNAs在植物色泽调控中的研究进展[J]. 湖北农业科学, 2017, 56(6): 1 001-1 005.
- 沈 洁. 番茄miR858的克隆和干旱应答及其对花青素生物合成的负调控机制研究[D]. 晋中: 山西农业大学, 2015.
- Wang L, Zeng H Q, Song J, et al. miRNA778 and SUVH6 are involved in phosphate homeostasis in Arabidopsis[J]. Plant Science, 2015, 238: 273-285.
- Cartolano M, Castillo R, Efremova N, et al. A conserved microRNA module exerts homeotic control over Petunia hybrida and Antirrhinum majus floral organ identity[J]. Nature Genetics, 2007, 39(7): 901-905.
- Yang L, Xu M, Koo Y, et al. Sugar promotes vegetative phase change in Arabidopsis thaliana by repressing the expression of MIR156A and MIR156C[J]. eLife, 2013, 2(2): e00260.
