土施维生素B6对苹果园土壤微生物多样性的影响
2018-04-25毛云飞于文章王增辉杨恒峰张佳腾高付凤陈学森毛志泉
毛云飞,于文章,王增辉,杨恒峰,张佳腾,高付凤,陈学森,毛志泉,沈 向
(山东农业大学园艺科学与工程学院/作物学国家重点实验室/山东省高等学校协同创新中心,山东泰安 271018)
苹果作为我国果品产业的支柱产业,其种植面积和产量均位于其它果品前列,改善其营养条件、增产提质一直是研究热点。夏燕飞等[1]对红富士苹果园土壤成分分析认为,土壤中维生素B2、维生素B6和烟酸与富士苹果中可溶性糖含量具有显著的正相关性,烟酸和维生素B12与果实花色苷具有显著正相关性,而且这些B族维生素与富士苹果维生素C含量都具有正相关性。王安然等[2]研究表明,在不同月份向土壤中施加维生素B6,4月份施用的维生素B6可显著提高富士单果重和果实的营养品质,6月份施加的维生素B6对富士果实香气品质具有显著的促进作用。维生素B6是土壤有机营养的一种[3],张宏伟等[4]研究发现有机营养与矿质元素结合生成有机无机胶体,为形成土壤团聚体创造条件,从而使土壤孔隙增加,水分、空气、肥料以及温度相互协调,改善土壤环境,土壤环境中包含C、H、O、N等元素,可以为土壤微生物提供大量的碳源和氮源,进而改善土壤微生物环境。
物种多样性是分析土壤微生物的重要参数,群落丰富度指数 (abundance indices) 和多样性指数(diversity indices) 是用来描述物种多样性的两个常用指标[5]。在土壤微生物的传统研究方法中,常采用微生物平板培养法、Biolog鉴定法、核酸分析法等来识别鉴定土壤中的微生物,但这些方法往往会低估土壤中微生物的组成,对目前尚无法培养的微生物无能为力,且无法描绘出微生物群落中的组成差异[6]。近年来,高通量测序技术不断发展,其无需培养即能客观准确地还原微生物群落组成,且受主观因素干扰小,为深入分析微生物的多样性提供技术支持[7]。根据本研究前期试验基础,即土施7 g/株的维生素B6对提升富士苹果果实营养品质和香气品质效果最佳[2],推测土施维生素B6的提质作用在很大程度上可能是由于影响了土壤微生物环境,故本研究采用高通量测序技术,在前期最佳浓度的基础上,选用5 g/株和10 g/株的浓度,分析施加维生素B6后,果树土壤中的微生物多样性变化,从而揭示土施维生素B6提质功效的机理。
1 材料与方法
1.1 试验设计
于2015年5月23日在蓬莱市大辛庄镇三十里堡村,选用4年生生长势一致、产量均等的以八棱海棠为砧木的富士苹果树 (株行距3 m × 4 m) 作为试验材料,在行间距离苹果树体主干1 m处,在每株树两侧挖2条深25 cm、宽15 cm、长1.5 m的沟,用维生素B6进行树体沟施,撒入维生素B6。该处理设置2个浓度梯度:5 g/株和10 g/株,每个浓度梯度设3次重复,以不施加维生素B6处理的树体作对照,按常规管理方式进行管理。试验地土壤中全氮含量为1.53 g/kg,有效磷含量为31.96 mg/kg,速效钾含量为154.45 mg/kg,有效镁含量为2.60 mg/kg,有效铁含量为7.90 mg/kg,有效铜含量为0.14 mg/kg,有效锌含量为1.74 mg/kg。
1.2 试验样品采集
于果实成熟期 (2015年10月中旬) 进行土壤样品采集。采用“X”形土壤样品采集方法,分别在每个处理的3次重复的每株树东、南、西、北4个方位距树干60 cm处采集0—40 cm土层2000 g,在去除杂质后进行混合,装进封口袋进行微生物测定。
1.3 测序试验方法
1.3.1 基因组DNA的提取和PCR扩增 采用CTAB方法对样本的基因组DNA进行提取,之后利用琼脂糖凝胶电泳检测DNA的纯度和浓度,取适量的样品于离心管中,使用无菌水稀释样品至1 ng/μL。以稀释后的基因组DNA为模板,根据测序区域的选择,使用带Barcode的特异引物,New England Biolabs公司的Phusion® High-Fidelity PCR Master Mix with GC Buffer,和Thermo公司的高效高保真酶进行PCR扩增,确保扩增效率和准确性。引物对应区域:16S V4+V5区引物 (515F和806R),鉴定细菌多样性;ITS1+ITS2区引物 (ITS5-1737F和ITS2-2043R),鉴定真菌多样性。
1.3.2 PCR产物的混样和纯化 PCR产物使用2%浓度的琼脂糖凝胶进行电泳检测,根据PCR产物浓度,将各样品进行等量混样,充分混匀后使用2%的琼脂糖凝胶电泳检测PCR产物,对目的条带使用Qiagen公司提供的胶回收试剂盒回收产物,并用纯化试剂盒对其进行纯化。
1.3.3 文库构建和上机测序 使用TruSeq® DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库经过Qubit和Q-PCR定量,文库合格后,使用HiSeq2500 PE250进行上机测序。
1.3.4 数据处理 根据Barcode序列和PCR扩增引物序列从下机数据中拆分出各样品数据,截去Barcode和引物序列后使用FLASH[8]对每个样品的reads进行拼接,得到的拼接序列为原始Tags数据(Raw Tags);拼接得到的Raw Tags,需要经过严格的过滤处理[9]得到高质量的Tags数据 (Clean Tags),并参照Qiime[10]的Tags质量控制流程。经处理后得到的Tags需要进行去除嵌合体序列的处理,Tags序列[11]通过与数据库 (Gold database,http://drive5.com/uchime/uchime_download.html) 进行比对检测嵌合体序列,并最终去除其中的嵌合体序列[12],得到最终的有效数据 (Effective Tags)。
利用Uparse软件 (Uparse v7.0.1001,http://drive5.com/uparse/)[13]对所有样品的全部Effective Tags进行聚类,默认以97%的一致性将序列聚类成为OTUs(operational taxonomic units),同时会选取OTUs的代表性序列,依据其算法原则,筛选OTUs中出现频数最高的序列作为OTUs的代表序列。
使用Qiime软件 (Version 1.7.0) 计算Chao、Shannon、Simpson、ACE指数,使用R软件(Version 2.15.3) 绘制稀释曲线,并进行Alpha多样性指数组间差异分析和Beat多样性指数组间差异分析,使用R软件绘制花瓣图、热图和主成分分析图,使用Excel和SPSS进行数据处理。
2 结果与分析
2.1 测序结果质量分析
为了研究样品的菌种组成多样性,对所有样品的有效序列数量进行聚类,通过对9个样品 (3个处理3次重复) 细菌多样性的测序,共测得原始序列条数为548289,过滤掉低质量的序列后,有效序列的总数为530714,根据Barcode标签进行样品序列拆分,再对初始序列进行去冗余处理后,获得16S rRNA基因Unique reads,有效序列比例均大于96%。真菌多样性检测共测得原始序列条数为549931,过滤掉低质量的序列后,有效序列总数为434886,有效序列比例均大于62%,各样品的相关数据如表1所示。
稀释曲线 (rarefaction curve) 反映了样品的取样
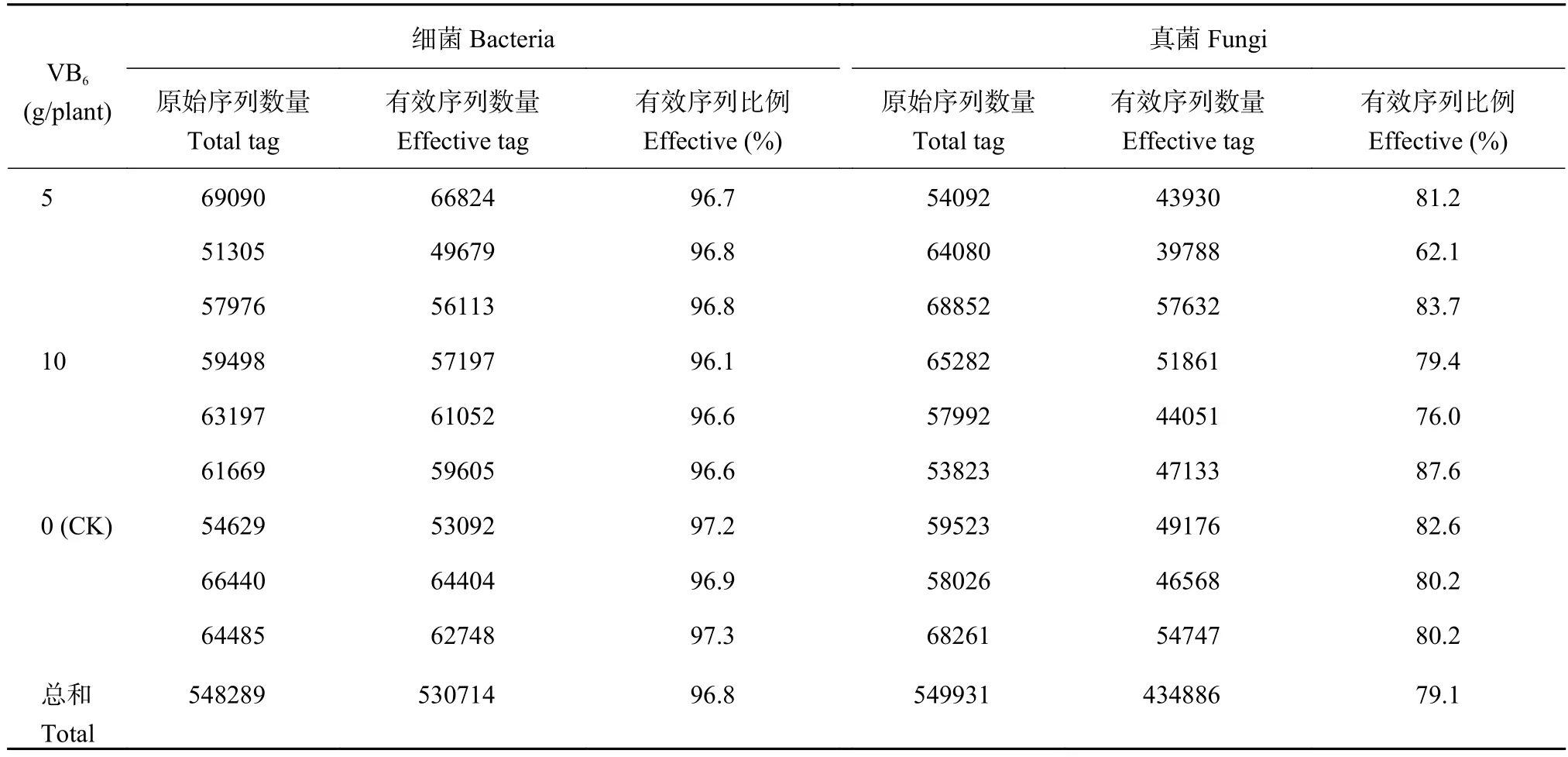
表 1 土壤样本细菌和真菌测序结果统计Table 1 Sequencing data statistics among soil samples of bacteria and fungi
深度,可以用来评价测序量是否足以覆盖所有类群。从稀释曲线 (图1)可知,序列数量到5000时各样品稀释曲线均基本趋于平缓,说明取样基本合理,真实环境中细菌和真菌群落结构的置信度较高,能够比较真实地反映土壤样本的细菌和真菌群落。
2.2 各处理群落丰富度和多样性分析
Ace和Chao指数可反映群落物种丰富度,Simpson和Shannon指数反映微生物多样性[14–15]。由表2得知,在细菌数量中,维生素B65 g/株处理显著提高了Ace、Chao和Shannon指数,与CK相比,分别提高了62.6%、71.6%和22.3%,Simpson指数与CK相比无显著差异,维生素B610 g/株处理4种指数与CK相比均无显著差异;在真菌数量中,与CK相比,维生素B65 g/株处理显著降低了Chao、Simpson和Shannon指数,分别降低了18.6%、29.2%和34.4%,Ace指数与CK无显著差异,维生素B610 g/株处理的4种指数与CK均无显著差异。由此可知,维生素B65 g/株处理提高了细菌群落的丰富度和细菌的多样性,降低了真菌群落的丰富度和多样性;维生素B610 g/株处理对细菌和真菌的多样性和丰富度无影响。
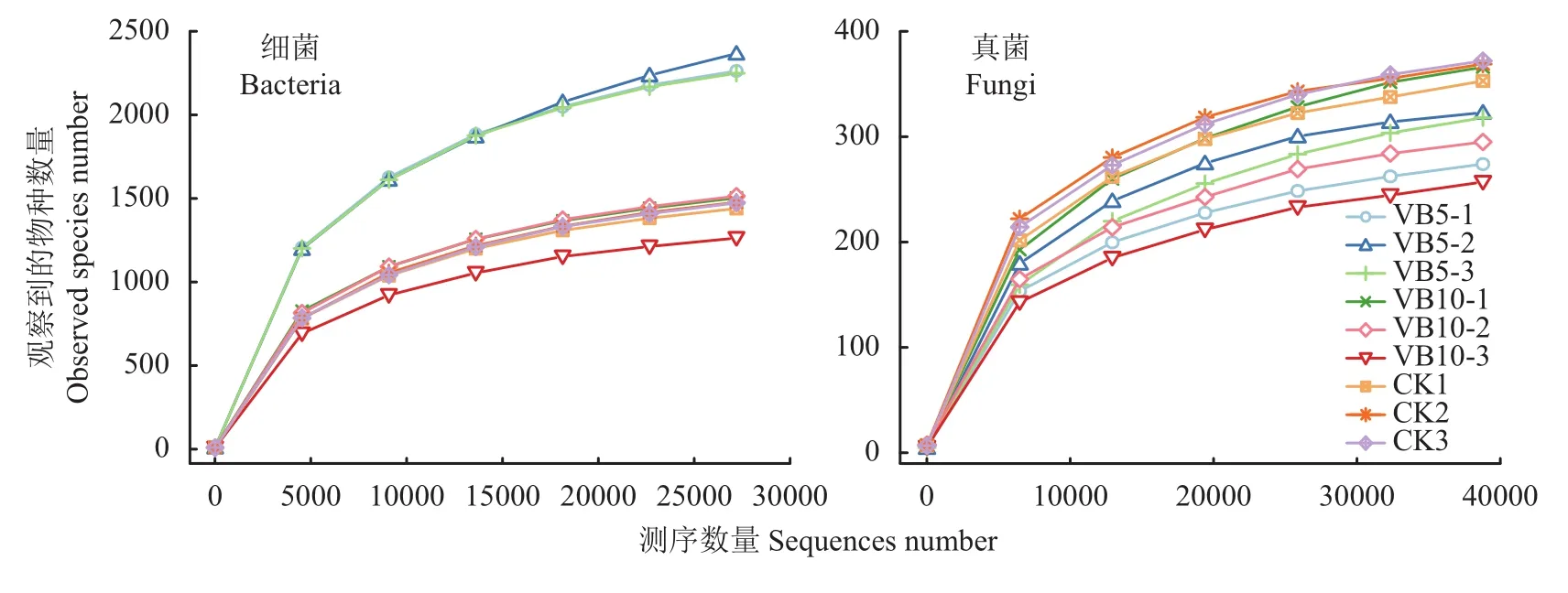
图 1 土壤样本细菌和真菌稀释曲线Fig. 1 Rarefaction curves of bacteria and fungi among soil samples
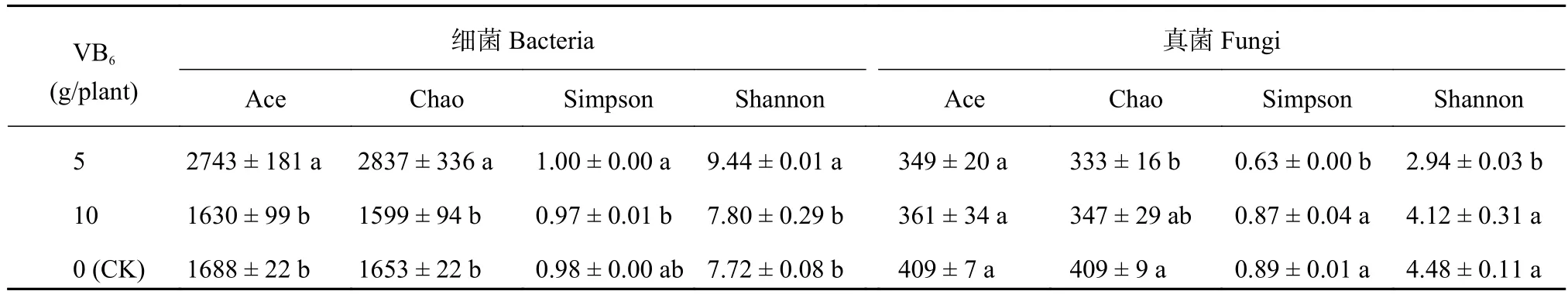
表 2 施用VB6土壤样本群落丰富度和多样性指数Table 2 The abundance and diversity indices of microbial community in soil samples
2.3 各处理群落Alpha群落类群分析
为了研究各群落类群组成,在对有效序列分类基础上,以97%的一致性将序列聚类成为OTUs(operational taxonomic units),然后对OTUs的代表序列进行物种注释。在0.97的相似度下,得到了每个样品的OTU个数,如图2所示,并同时绘出花瓣图,结果可以展示多样品相同和各自特有的OTU数目,直观展示样品间OTU的重叠情况(图3)。Alpha群落类群主要用于种内种间比较,结合OTU所代表的物种,可以找出不同环境中的核心微生物。
细菌水平上,结合图2(左) 与图3(左),高通量分析得知,9个样品测序共可获得1441~2535种水平分类的细菌 (OTU),其中,共有的OTU数量为566,特异性OTU数量为13~197个,以维生素B65 g/株处理效果最突出,与CK相比,维生素B65 g/株 处理不仅增加了土壤中的细菌微生物OTU总量,也增加了其中的某些特异性OTU数目,其中,CK处理第1组中,其特异性细菌微生物OTU有22个,样品总细菌微生物OTU数有1611个,维生素B65 g/株处理第1组中,其特异性细菌微生物OTU有197个,样品总细菌微生物OTU数有2 532个,与CK相比,分别增加了795.45%和57.17%。维生素B610 g/株 处理与CK在OTU数量上并无显著差异。
真菌水平上,由图2 (右) 与图3 (右) 分析得知,9个样品测序共可获得315~431种水平分类的真菌(OTU),其中,共有的OTU数量为73,特异性OTU数量为11~55个,与CK相比,维生素B65 g/株处理不仅降低了土壤中真菌微生物OTU总量,也降低了其中的某些特异性OTU数目,维生素B610 g/株处理中,除第1组重复外,其他2次重复均降低了真菌微生物OTU总量及某些特异性OTU数目,其中,CK处理第1组中,其特异性真菌微生物OTU有19个,样品总真菌微生物OTU数有417个,维生素B65 g/株处理第1组中,其特异性真菌微生物OTU有11个,样品总真菌微生物OTU数有315个,与CK相比,分别降低了42.1%和24.5%。
2.4 各处理群落Beta多样相关性研究
Beta多样性研究中,选用Weighted Unifrac距离和Unweighted Unifrac距离2个指标来衡量2个样品间的相异系数,其值越小,表示这2个样品在物种多样性方面存在的差异越小。以Weighted Unifrac和Unweighted Unifrac距离绘制的热图 (Heatmap) 展示结果。主成分分析 (PCA,Principal Component Analysis) 是一种应用方差分解对多维数据进行降维,从而提取出数据中最主要的元素和结构的方法[16]。应用PCA分析,能够提取出最大程度反映样品间差异的两个坐标轴,从而将多维数据的差异反映在二维坐标图上,进而揭示复杂数据背景下的简单规律。如果样品的群落组成越相似,则它们在PCA图中的距离越接近。
由图4 (左) 分析得知,对细菌而言,维生素B65 g/株处理与CK相比,组间相异系数达到0.247,维生素B610 g/株处理与CK相比,组间相异系数达到0.222,维生素B65 g/株处理与维生素B610 g/株处理相比,组间相异系数达到0.236。
由图4 (右) 分析得知,对真菌而言,维生素B65g/株处理与CK相比,组间相异系数达到1.238,维生素B610 g/株处理与CK相比,组间相异系数达到1.233,维生素B65 g/株处理与维生素B610g/株处理相比,组间相异系数达到1.350。
由图5 PCA分析可知,各组间距离大,组内之间距离小,表示各组间差异性大,组内差异性小,即在细菌和真菌水平上,施加维生素B65 g/株和维生素B610 g/株两组处理以及CK,三组间均具有显著的差异性,表明施入维生素B6后,土壤微生物发生了显著变化。
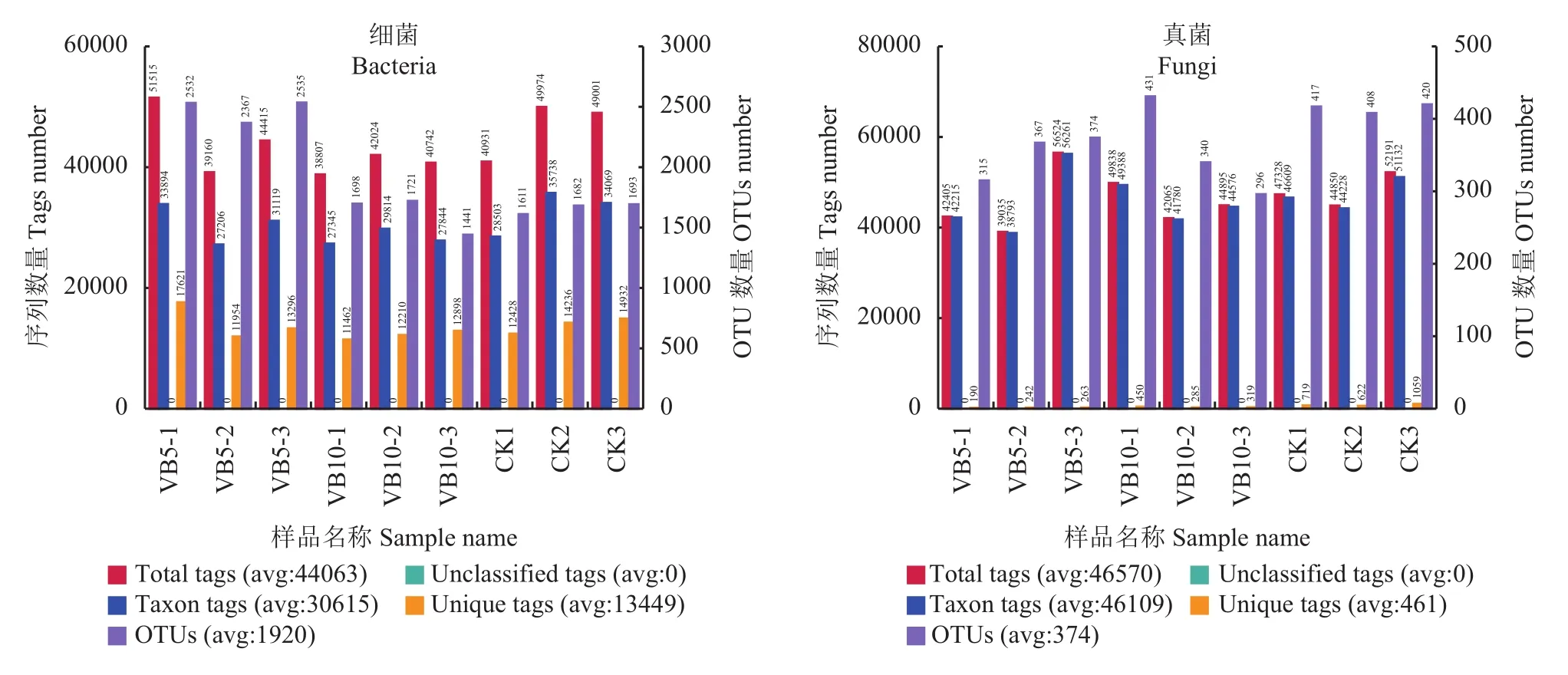
图 2 土壤样品细菌 (左)、真菌 (右)OTUs聚类分析Fig. 2 The analysis of bacteria (left) and fungi (right) OTUs among soil samples
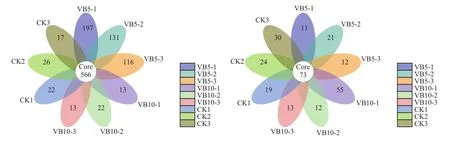
图 3 土壤样品细菌 (左)、真菌 (右) 多样性的相关性分析Fig. 3 Similarity analysis of bacteria (left) and fungi (right) diversity among soil samples
2.5 各处理样品菌群相对丰度组成分析
根据物种注释结果,选取每个样品或分组在各分类水平 (Phylum、Class、Order、Family、Genus)上最大丰度排名前10的物种,生成物种相对丰度柱形累加图,以便直观查看各样品在不同分类水平上相对丰度较高的物种及其比例。以门水平物种相对丰度柱形图为例展示,如图6所示。
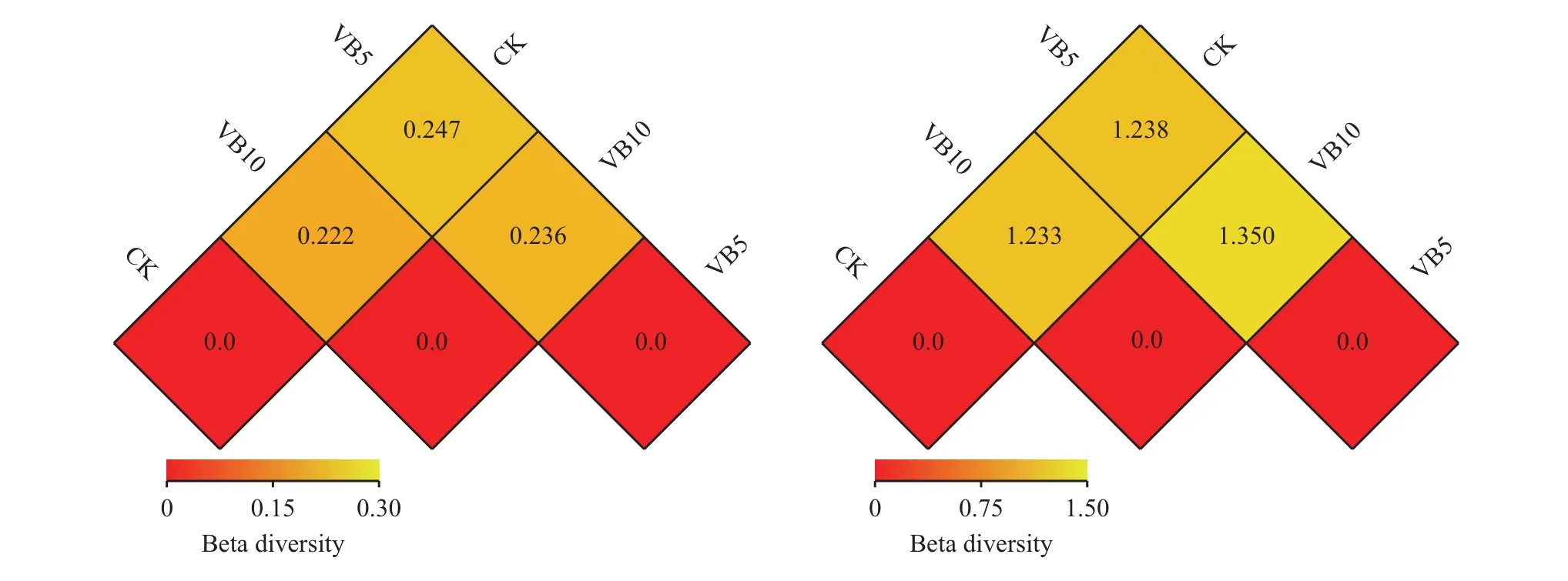
图 4 土壤样品细菌 (左)、真菌 (右) 组间差异性相关分析Fig. 4 Similarity analysis of bacteria (left) and fungi (right) difference in groups among soil samples
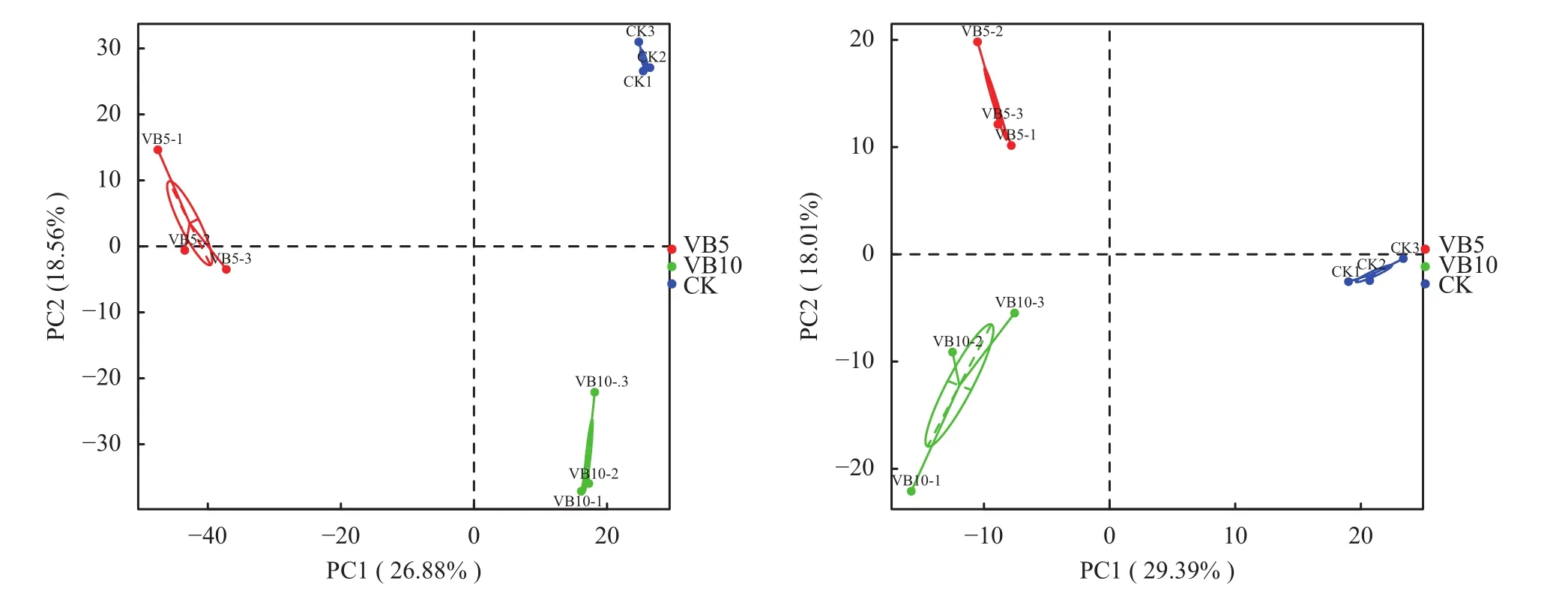
图 5 土壤样品细菌 (左)、真菌 (右) 主成分分析Fig. 5 Principle component analyses of bacteria (left) and fungi (right)
在细菌水平,由图6 (左) 分析可知,丰富度较高的前10个门分别为变形菌门 (Proteobacteria)、放线菌门 (Actinobacteria)、拟杆菌门 (Bacteroidetes)、酸杆菌门 (Acidobacteria)、绿弯菌门 (Chloroflexi)、芽单胞菌门 (Gemmatimonadetes)、浮霉菌门(Planctomycetes)、厚壁菌门 (Firmicutes)、疵微菌门(Verrucomicrobia)、蓝藻菌门 (Cyanobacteria),由图中各细菌相对含量分析可知,在三组处理中,优势类群分别为变形菌门、放线菌门、拟杆菌门及酸杆菌门,这4种优势类群在各自处理中所占比例均超过80%,在这4个优势群中,维生素B610 g/株处理提高了土壤中变形菌门、放线菌门的相对丰度,与CK相比,分别提高了12.6%和56.7%,降低了拟杆菌门、酸杆菌门相对丰度,与CK相比,分别降低了59.9%和41.5%,维生素B65 g/株处理则降低了拟杆菌门的相对丰度,与CK相比,降低了34.8%,对其他3种优势菌门无影响。
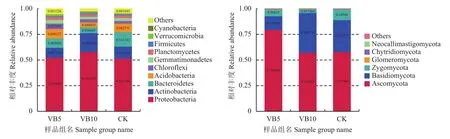
图 6 土壤样品在细菌 (左)、真菌 (右) 门分类水平上相对丰度变化Fig. 6 Changes in relative abundance of soil bacteria (left) and fungi (right) samples at phylum level
在真菌水平,由图6 (右) 分析得知,丰富度较高的前6个门分别为子囊菌门 (Ascomycota)、担子菌门 (Basidiomycota)、接合菌门 (Zygomycota)、球囊菌门 (Glomeromycota)、壶菌门 (Chytridiomycota)、新美鞭菌门 (Neocallimastigomycota),结合图中各真菌相对含量,在三组处理中,优势类群分别为子囊菌门、担子菌门、接合菌门,这3种优势类群在各自处理中所占比例均超过98%,在这3个优势群中,与CK比较,维生素B65 g/株处理将土壤中子囊菌门相对丰度提高了36.9%,分别将担子菌门和接合菌门的相对丰度降低了57.01%和37.18%,维生素B610g/株处理将土壤中担子菌门相对丰度提高了24.8%,将接合菌门相对丰度降低了64.5%,而对子囊菌门的相对丰度无影响。
3 讨论与结论
土壤微生物作为陆地生态系统植物多样性和生产力的重要驱动者,与植物生长发育密切相关[17–18],一方面植物作为生产者,为微生物提供碳源和氮源,经过微生物的分解为植物提供营养,从而达到互惠作用。本试验利用高通量测序技术,施加维生素B65 g/株处理,提高了细菌总OTU及特异性OTU数量,也提高了细菌的丰富度和多样性,同时降低真菌总OTU、特异性OTU数量,降低了真菌的丰富度和多样性,经Alpha多样性分析得知,CK组间平均OTU数为1662,维生素B65 g/株处理组间平均OTU数为2478,与CK相比,提高了49.1%,特异性OTU数,CK为22个,维生素B65 g/株处理为148个,这与维生素B65g/株 处理与CK的Beta多样性分析结果相符,其组间多样性差异大。有研究报道,当土壤微生物由细菌主导型向真菌主导型转变时,植物更容易受到病原菌的侵染[19]。而施加维生素B65g/株 以后,细菌数量变多,真菌数量变少,在一定程度上防止了植物遭受病原菌侵害的几率。施加维生素B610 g/株,尽管对细菌和真菌的丰富度和多样性无显著影响,但降低了真菌的OTU,对植物仍有一定的保护作用。而维生素B6施用剂量增加,对细菌和真菌丰富度和多样性指数无显著影响,经Alpha多样性分析,维生素B610 g/株 处理与CK在总OTU及特异性OTU上无太大差别,而经Beta多样性分析得知,维生素B610 g/株处理与CK在组间多样性上仍存在差异[20],2种剂量处理的差异原因还需要进一步试验验证。
根据物种注释结果,9个土壤样品中的细菌菌类按优势类群分类,优势菌群分别为变形菌门、放线菌门、拟杆菌门及酸杆菌门,这四种优势类群在各自处理中所占比例之和均超过80%,其中,变形菌门的比例超过50%。本试验中,维生素B65 g/株处理降低了拟杆菌门的相对丰度,对其他3种优势菌种相对丰度无影响,维生素B610 g/株处理提高了变形菌门和放线菌门的相对丰度,降低了拟杆菌门和酸杆菌门的相对丰度。变形菌门和放线菌门等为土壤中的有益菌,变形菌门是革兰氏阴性菌,其数量大、多样性高,有研究称,变形菌门可以参与各种有机物的碳氮循环,代谢类型多样,还可以作为生物治理因子代谢环境中的化学污染,如甲苯等[21]。放线菌门大多数微生物主要降解纤维素甚至许多难降解的芳香族化合物,对于土壤的矿化作用起到十分重要的作用[22]。土壤中酸杆菌门大多是嗜酸菌,数量变化受多种因素影响,其丰富度也与土壤pH有关[23],施加不同浓度维生素后,其数量变化,可能与土壤pH变化有关。拟杆菌门与DNA、蛋白质和脂类等有机物质的转化紧密相关,这些有机物质的吸收和利用是碳循环的重要组成部分[24],尽管不同浓度维生素B6处理均降低了土壤中拟杆菌门的相对丰度,但由于细菌总OTU均升高,其具体数量变化还有待后续进一步研究。
将真菌优势群种进行分类,优势群种分别为子囊菌门、担子菌门和接合菌门,3种菌门所占比例之和超过98%,其中,子囊菌门在3种处理中的比例均超过50%,是绝对优势菌门,尽管有研究表明,子囊菌门中的2个变种,即Valsa malivar.mali和V.malivar.pyri,是苹果腐烂病的主要病原菌之一[25],但也有研究表明,子囊菌门中的Incertae sedis27也可促进生根[26]。担子菌门数量繁多,分布广泛,其中有些可食用,也有的可药用,而也有一些能引起植物和园林病害[27]。本试验中,维生素B65 g/株 处理提高了子囊菌门相对丰度,降低了担子菌门相对丰度,维生素B610 g/株 处理未降低子囊菌门相对丰度,但提高了担子菌门的相对丰度,其具体变化还有待研究。施加不同浓度维生素B6后,土壤中的接合菌门均会显著降低,而有研究表明,随着连作年限的增加,土壤中的接合菌门的比例会逐年增加[28],因此,施加维生素B6对防治重茬具有良好的效果。
综合以上研究结果,维生素B6可改变土壤中细菌和真菌的丰富度和多样性,改变土壤中门水平的细菌和真菌的相对丰度,维生素B65 g/株处理可提高土壤中细菌丰富度和多样性,降低真菌丰富度和多样性,降低了细菌中拟杆菌门 (Bacteroidetes) 的相对丰度,提高了真菌中子囊菌门 (Ascomycota) 的相对丰度,降低了担子菌门 (Basidiomycota) 和接合菌门 (Zygomycota) 的相对丰度,维生素B610 g/株处理提高了细菌中的变形菌门 (Proteobacteria) 和放线菌门 (Actinobacteria) 的相对丰度,降低了拟杆菌门(Bacteroidetes) 和酸杆菌门 (Acidobacteria) 的相对丰度,提高了真菌中担子菌门 (Basidiomycota) 的相对丰度,降低了接合菌门 (Zygomycota) 的相对丰度。维生素B6作为一种良好的根系生态调节途径,具有极佳的推广价值。
参 考 文 献:
[ 1 ]夏燕飞, 张文会, 王荣, 等. 土壤有机营养对‘红富士’苹果果实产量和品质的影响[J]. 植物营养与肥料学报, 2013, 19(4): 868–877.Xia Y F, Zhang W H, Wang R,et al. Effect of soil organic nutrition matter on yield and quality of Fuji apple[J]. Journal of Plant Nutrition and Fertilizer, 2013, 19(4): 868–877.
[ 2 ]王安然, 倪蔚茹, 贺锡燕, 等. 维生素B6施用时间和剂量对富士苹果品质的影响[J]. 植物营养与肥料学, 2016, 22(5): 1348–1355.Wang A R, Ni W R, He X Y,et al. Effects of vitamin B6application time and doses on fruit quality of Fuji[J]. Journal of Plant Nutrition and Fertilizer, 2016, 22(5): 1348–1355.
[ 3 ]Obrador A, Gonzalez D, Jose M A. Effect of inorganic and organic copper fertilizers on copper nutrition inSpinacia oleraceaand on labile copper in soil[J]. Agricultural and Food Chemistry, 2013,61(20): 4692–4701.
[ 4 ]张宏伟, 龙明杰, 曾繁森. 腐植酸接枝共聚物对赤红壤改良的研究[J]. 水土保持研究, 2001, 8(2): 115–117.Zhang H W, Long M J, Zeng F S. Study of amelioration to lateritic red soil by copolymer of humic acids grafted vinly monomer[J].Research of Soil and Water Conservation, 2001, 8(2): 115–117.
[ 5 ]唐玉妹, 魏朝富, 颜廷梅, 等. 土壤质量生物学指标研究进展[J]. 土壤, 2007, 39(2): 157–163.Tang Y M, Wei C F, Yan T M,et al. Biological indicator of soil quality: A review[J]. Soils, 2007, 39(2): 157–163.
[ 6 ]Schirmer M, Ijaz U Z, D'Amore R,et al. Insight into biases and sequencing errors for amplicon sequencing with the Illumina MiSeq platform[J]. Nucleic Acids Research, 2015, 43(6): e37.
[ 7 ]Cummings P J, Ahmed R, Durocher J A,et al. Pyrosequencing for microbial identification and characterization[J]. Journal of Visualized Experiments, 2013, 78: e50405.
[ 8 ]Magoč T, Salzberg S L. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21):2957–2963.
[ 9 ]Nicholas A B, Sathish S, Jeremiah J F,et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J].Nature Methods, 2013, 10(1): 57–59.
[10]Caporaso J G, Kuczynski J, Stombaugh J,et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335–336.
[11]Edgar R C, Haas B J, Clemente J C,et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011,27(16): 2194–2200.
[12]Haas B J, Gevers D, Earl A M,et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494–504.
[13]Edgar R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10):996–998.
[14]Pitta D W, Parmar N, Patel A K,et al. Bacterial diversity dynamics associated with different diets and different primer pairs in the rumen of Kankrej cattle[J]. PLoS One, 2014, 9(11): e111710.
[15]Sun R B, Zhang X X, Guo X S,et al. Bacterial diversity in soils subjected to long-term chemical fertilization can be more stably maintained with the addition of livestock manure than wheat straw[J].Soil Biology & Biochemistry, 2015, 88(4): 9–18.
[16]Avershina E, Ekaterina T, Rudi K.Denovo semi-alignment of 16S rRNA gene sequences for deep phylogenetic characterization of next generation sequencing data[J]. Microbes and Environments, 2013,28(2): 211–216.
[17]Van D, Heijden M G A, Bardgett R D,et al. The unseen majority:soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems[J]. Ecology Letters, 2008, 11(3): 296–310.
[18]Wardle D A, Bardgett R D, Klironomos J N,et al. Ecological linkages between above ground and below ground biota[J]. Science,2004, 304(5677): 1629–1633.
[19]Marschner P, Yang C H, Lieberei R, Crowley D E. Soil and plant specific effects on bacterial community composition in the rhizosphere[J]. Soil Biology & Biochemistry, 2001, 33(11):1437–1445.
[20]庄林杰, 夏超, 田晴, 等. 高通量测序技术研究典型湖泊岸边陆向深层土壤中厌氧氨氧化细菌的群落结构[J]. 环境科学学报, 2017,37(1): 261–271.Zhuang L J, Xia C, Tian Q,et al. Community structure of anaerobic ammonia oxidation bacteria in subsurface soil from riparian zone to land in Baiyangdian Lake using high-throughput sequencing technology[J]. Acta Scientiae Circumstantiae, 2017, 37(1): 261–271.
[21]O'Mahony M M, Dobson A D, Barnes J D,et al. The use of ozone in the remediation of polycyclic aromatic hydrocarbon contaminated soil[J]. Chemosphere, 2006, 63(2): 307–314.
[22]余悦. 黄河三角洲原生演替中土壤微生物多样性及其与土壤理化性质关系[D]. 山东济南: 山东大学博士学位论文, 2012.Yu Y. Relationships between soil microbial diversity and soil physicochemical properties along the primary succession of Yellow River Delta [D]. Jinan, Shandong: PhD Dissertation of Shandong University, 2012.
[23]Jones R T, Robeson M S, Lauber C L,et al. A comprehensive survey of soil acidobacterial diversity using pyrosequencing and clone library analyses[J]. International Society for Microbial Ecology Journal, 2009, 3(4): 442–453.
[24]Michand L, Lo G A, Troussellier M,et al. Phylogenetic characterization of the heterotrophic bacterial communities inhabiting a marine recirculating aquaculture system[J]. Journal of Applied Microbiology, 2009, 107(6): 1935–1946.
[25]周玉霞, 张美鑫, 翟立峰, 等. 梨和苹果腐烂病菌不同培养表型菌株的致病性分析[J]. 植物病理学报, 2014, 44(2): 217–220.Zhou Y X, Zhang M X, Zhai L F,et al. Comparison of pathogenicity ofValsa malistrains showing different culturing phenotypes[J]. Acta Phytopathologica Sinica, 2014, 44(2): 217–220.
[26]李发虎, 李明, 刘金泉, 等. 生物炭对温室黄瓜根际土壤真菌丰度和根系生长的影响[J]. 农业机械学报, 2017, 48(4): 265–270, 341.Li F H, Li M, Liu J Q,et al. Effect of biochar on fungal abundance of rhizosphere soil and cucumber root growth in greenhouse[J].Transactions of the Chinese Society of Agricultural Machinery, 2017,48(4): 265–270, 341.
[27]戴玉成. 中国木本植物病原木材腐朽菌研究[J]. 菌物学报, 2012,31(4): 493–509.Dai Y C. Pathogenic wood-decaying fungi on woody plants in China[J]. Mycosystema, 2012, 31(4): 493–509.
[28]赵帆, 赵密珍, 王钰, 庞夫花. 不同连作年限草莓根际细菌和真菌多样性分析[J]. 微生物学通报, 2017, 44(6): 1377–1386.Zhao F, Zhao M Z, Wang Y, Pang F H. Biodiversity of bacteria and fungi in rhizosphere of strawberry with different continuous cropping years[J]. Microbiology China, 2017, 44(6): 1377–1386.
