基于交叉预测的蛋白质交互识别
2018-04-13闵庆凯蔡松成
闵庆凯,蔡松成
(南京航空航天大学 计算机科学与技术学院,江苏 南京 211106)
1 概 述
蛋白质是生物细胞最重要的组成成分,通过交互作用执行着细胞内多数重要的分子过程。蛋白质交互作用(protein-protein interaction,PPI)的研究以及蛋白质交互网络的建立是生物信息学研究的重要内容。目前,已有的交互关系数据库,例如HPRD[1]、BIND[2]、DIP[3]、InAct[4]和MINT[5],均由生物医学专家通过人工识别的方法从医学文献中搜集得到。然而,随着生物医学文献的急剧增加,人工抽取的方法变得越来越不切实际,因此利用计算机信息抽取技术自动地从自然语言文本中抽取PPI成为一项重要的研究内容。
目前,用于PPI抽取的技术主要包括基于词共现的方法[6]、基于规则的方法[7]和基于统计机器学习的方法[8-9]。基于词共现的方法通过统计两个蛋白质名称在句子中共同出现的概率来判断是否存在交互关系,这种方法召回率高但很难发现词典外的PPI[10];基于规则的方法利用模式匹配的思想,可以取得较高的精确率,但泛化能力差,而且通过手动建立规则的方法需要大量的人力物力[11];基于统计机器学习的方法通过将关系抽取问题转换为分类问题,同时结合自然语言处理方法,较好地解决了上述两种方法存在的问题,目前广泛用于PPI的抽取。这类方法又可分为基于特征的方法和基于核函数的方法。其中,基于特征的方法从句子中提取大量的语言学特征,包括词法、语法和语义等特征来表示关系实例[12],能够简单有效地完成关系抽取任务;而基于核函数的方法通过设计核函数代替特征向量内积运算计算PPI间的相似度,具有良好的复合特性,在关系抽取领域也取得了不错的效果[13]。
上述机器学习方法均基于有监督的思想,语料库中的句子所包含的实体对及其关系都由人工标注完成,其性能非常依赖于训练样本的数量,当训练语料不足时,关系抽取效果就会大打折扣。但人工标注大规模文本需要耗费大量的人力物力,因此出现了基于远监督的方法:假设关系知识库中的一对实体存在某种关系,那么包含这对实体的句子则表达了实体对的这种关系,通过将知识库中的实体对与文本中的实体进行匹配,启发式地产生大量的标记数据[14]。远监督很好地解决了标注数据不足的问题,利用远监督得到大规模标注文本结合基于特征的方法在PPI抽取上也取得了很好的效果[15-16]。然而,与有监督下人工精确标注的方法相比,远监督采取的是相对粗糙的匹配方式,得到的标注数据并不总是正确的。如图1所示,第一个句子确实表达了Michael Jackson和Gary之间的place_of_birth关系,而第二个句子并不能表达这种关系,这种实际上被错误标记的句子即被视为标注语料中的噪音,这种噪音会对最终的关系抽取效果造成很大的影响。针对训练数据中存在的噪音,提出一种交叉预测的方法,并通过人工标注数据进行验证。

图1 远监督的自动标注
2 交叉预测去噪音
该方法以远监督为基础,首先搜索大规模医学文献获取包含目标蛋白质对的句子作为原始训练数据,从中提取特征,构建向量空间模型(vector space model,VSM),将每个句子映射为一个n维的特征向量;然后采用交叉预测识别出训练数据中的噪音,消除噪音并重新形成训练数据;最后利用训练得到的分类器对另一有人工标注的数据进行预测。
2.1 构建特征向量
文中采用向量空间模型来表示文本,每一个句子表示向量空间中的一个向量,选取句子中重要的一元词作为特征项,具体处理方法为:
(1)对句子进行分词,去除无意义的标点符号以及停止词;
(2)选取句子中两个蛋白质之间的单词作为特征项;
(3)选取第一个蛋白质左边2个单词和第二个蛋白质右边2个单词,作为特征项。
所得到每一个不同的特征项对应于向量空间中的一个维度,若句子中出现了该特征项,那么句子向量的对应维设为1,否则为0。
2.2 远监督
远监督:如果两个实体之间存在某种关系,那么包含这两个实体的句子就表达了这种关系。文中采用的知识库分为两部分:有交互关系的蛋白质对和无交互关系的蛋白质对,基于远监督得到训练数据的步骤如下:
(1)将知识库中的蛋白质对与大规模医学文本中的蛋白质进行匹配,筛选出所有包含知识库中蛋白质对的句子;
(2)所有包含有交互关系的蛋白质对的句子标注为训练数据中的正例即有交互关系;
(3)所有包含无交互关系的蛋白质对的句子标注为训练数据中的负例即无交互关系。
将得到的训练数据中的句子通过向量空间模型构建为特征向量,训练分类器,然后对人工标注的测试集进行测试。
2.3 交叉预测去噪音
交叉预测的方法是在远监督的基础上,如图2所示,将远监督得到的训练数据随机分为k组,取1组数据作为预测集,其余k-1组数据作为训练集进行训练,依次轮换训练集和预测集k次,对每组数据进行预测并去噪,具体步骤如下:
(1)随机将远监督得到的训练数据S划分为k个不相交的子集,假设S中句子个数为m,那么每个子集中有m/k个句子,相应的子集为{S1,S2,…,Sk};

图2 交叉预测去噪音
将去噪后的训练数据S'中的句子通过向量空间模型构建为特征向量,训练分类器,然后对人工标注的测试集进行测试。
3 实验及结果分析
3.1 实验数据及设置
文中采用的知识库中包含578对有交互关系的蛋白质对和576对无交互关系的蛋白质对。有交互关系的蛋白质对均直接来源于专业PPI数据库HPRD,HPRD是现有国际上最大的人类PPI数据库,数据可靠性高;而对于无交互关系的蛋白质对,采用生物医学领域常用方法,将HPRD中的蛋白质进行随机组合,去除其中已经包含在HPRD中的蛋白质对组合,剩余蛋白质对作为知识库中的无交互关系的蛋白质对。
提取的大规模医学文本来自PubMed数据库,PubMed是生物医学领域最具影响力的文献检索系统,内容丰富。通过将知识库中的蛋白质对与PubMed数据库中的文本进行匹配,可得到去噪前训练数据共11 147个句子,其中有交互的句子5 477个,无交互的句子5 670个。
通过对实验结果进行调整,采用五组交叉预测,即k=5,每组数据有2 229个句子,包括1 095个有交互的句子和1 134个无交互的句子。文中采用逻辑回归分类器对每组训练数据中的句子进行预测分类,并对人工标注的测试集进行测试,逻辑回归模型简单高效,易于实现,计算代价不高,在进行大规模线性分类时较为方便。
文中选取了AIMed语料中的1 000个标注作为测试数据。AIMed语料来自于PubMed摘要,是PPI实验中最具代表性的专家标注语料。实验采用的性能评价指标是当前PPI抽取系统主要使用的三个指标:精确度(Precision)、召回率(Recall)和F值。
Precision=TP/TP+FP
(1)
Recall=TP/(TP+FN)
(2)
F-Score=2×P×R/(P+R)
(3)
3.2 实验结果及讨论
远监督与交叉预测去噪后得到的训练数据如表1所示。

表1 训练数据
从表1可以看出,相比于远监督得到的训练数据,经过五折交叉预测去噪后,训练数据中有交互的句子数量减少了38%,无交互的句子数量减少了34%,句子总数减少了36%。由此可得,交叉预测较好地识别出了训练数据中的噪音,且对于有交互和无交互的句子噪音数量识别相差不大,保证了训练数据的平衡性。
分别使用远监督和交叉预测去噪得到的训练数据去训练得到模型,然后对AIMed语料进行预测,结果如表2所示。
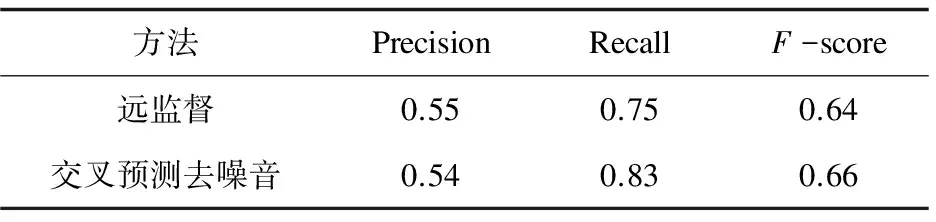
表2 测试结果对比
从表2可以看出,去噪后的模型在保持精确度的同时,召回率提高了8%,提升效果明显,并且整体F-score也提高了2%。说明交叉预测的方法对训练集中的噪音消除的效果较为明显,有效提高了模型的性能。
4 结束语
详细分析了基于远监督产生大规模文本进行蛋白质交互关系抽取的方法,针对训练数据存在噪音的问题,提出了一种交叉预测去噪的方法。通过对训练数据进行分组预测来清除其中的噪音,并通过人工标注语料进行测试。实验结果表明,同远监督相比,交叉预测有效清除了训练数据中的噪音,提高了模型的识别效果。
参考文献:
[1] PRASAD T S K,GOEL R,KANDASAMY K,et al.Human protein reference database-2009 update[J].Nucleic Acids Research,2009,37:767-772.
[2] BADER G D,DONALDSON I,WOLTING C,et al.BIND:the biomolecular interaction network database[J].Nucleic Acids Research,2001,29(1):242-245.
[3] SALWINSKI L,MILLER C S,SMITH A J,et al.The database of interacting proteins:2004 update[J].Nucleic Acids Research,2004,32:449-451.
[4] KERRIEN S,ALAMFARUQUE Y,ARANDA B,et al.Int Act-open source resource for molecular interaction data[J].Nucleic Acids Research,2007,35:561-565.
[5] CEOL A,ARYAMONTRI A C,LICATA L,et al.MINT,the molecular interaction database:2009 update[J].Nucleic Acids Research,2010,38:532-539.
[6] BUNESCU R, MOONEY R, RAMANI A,et al.Integrating co-occurrence statistics with information extraction for robust retrieval of protein interactions from Medline[C]//Proceedings of the workshop on linking natural language processing and biology:towards deeper biological literature analysis.[s.l.]:Association for Computational Linguistics,2006:49-56.
[7] KOIKE A, KOBAYASHI Y, TAKAGI T. Kinase pathway database:an integrated protein-kinase and NLP-based protein-interaction resource[J].Genome Research,2003,13(6a):1231-1243.
[8] 杨志豪,洪 莉,林鸿飞,等.基于支持向量机的生物医学文献蛋白质关系抽取[J].智能系统学报,2008,3(4):361-369.
[9] 唐 楠,杨志豪,林鸿飞,等.基于多核学习的医学文献蛋白质关系抽取[J].计算机工程,2011,37(10):184-186.
[10] GRIMES G R,WEN T Q,MEWISSEN M,et al.PDQ Wizard:automated prioritization and characterization of gene and protein lists using biomedical literature[J].Bioinformatics,2006,22(16):2055-2057.
[11] ANANIADOU S,KELL D B,TSUJII J.Text mining and its potential applications in systems biology[J].Trends in Biotechnology,2006,24(12):571-579.
[12] NIU Y,OTASEK D,JURISICA I.Evaluation of linguistic features useful in extraction of interactions from PubMed;application to annotating known, high-throughput and predicted interactions in I2D[J].Bioinformatics,2010,26(1):111-119.
[13] HAUSSLER D.Convolution kernels on discrete structures[R].California:University of California at Santa Cruz,1999.
[14] MINTZ M,BILLS S,SNOW R,et al.Distant supervision for relation extraction without labeled data[C]//Proceedings of the joint conference of the 47th annual meeting of the ACL and the 4th international joint conference on natural language processing of the AFNLP.[s.l.]:Association for Computational Linguistics,2009:1003-1011.
[15] 王宇伟,牛 耘.基于关系相似性的蛋白质交互作用识别[J].计算机技术与发展,2015,25(2):42-46.
[16] 吴红梅,牛 耘.基于特征加权的蛋白质交互识别[J].计算机技术与发展,2016,26(2):114-117.
