简州大耳羊早期胎儿到新出生阶段背最长肌中可变剪接的鉴定与分析
2018-04-08郭家中陶海溪李鹏飞张红平
郭家中,陶海溪,李鹏飞,李 利,张红平
(四川农业大学 动物科技学院,成都 611130)
可变剪接(alternative splicing,AS)是指真核生物体内多外显子基因通过选择不同的剪接位点,从而形成多个不同RNA转录本的生物学过程。可变剪接普遍存在于各种真核生物中。例如,基于人类大脑、肝、心脏等多个组织的转录组比较分析表明,90%以上的蛋白编码基因发生过可变剪接[1-2]。在低等动物中,Gibilisco等[3]在4种果蝇的多个组织中发现,大约有20%~37%的多外显子基因发生过可变剪接,而且不同性别的同一组织发生的可变剪接模式也有显著不同。在植物中,Shen等[4]发现在大豆的生长发育过程中大约有63%的基因发生了可变剪接,而且早期发育阶段发生的可变剪接多于晚期阶段。此外,已有研究表明,在动物体内比例最高的可变剪接类型是外显子跳跃[1,3,5],而在植物体内比例最高的可变剪接类型则是内含子保留[4,6],表明动植物体内可变剪接发生的分子机制具有差异。
已有研究认为,可变剪接形成的部分转录本并不具有功能,是生物体不精密调控产生的噪音[7-8];但越来越多的证据表明,可变剪接形成的大量功能不同的成熟转录本[2,9],在高等动物的组织发育和疾病的发生发展过程中扮演着重要作用。在肌肉发育方面,Singh等[10]通过RNA-Seq数据发现敲弱Rbfox2的表达后, Mef2d 和 Rock2基因的可变剪接体丧失活性,导致小鼠成肌细胞融合过程受阻,证明单个可变剪接因子所调控的可变剪接事件是小鼠成肌细胞融合过程的必要条件之一。最近,Du等[11]在强直性肌营养不良的小鼠骨骼肌中发现了由CUG重复引起的可变剪接紊乱。另外,可变剪接在动物心肌的发育过程中也扮演着重要作用。例如,ASF/SF2基因的敲除影响CaMKIIδ基因的转录本类型,从而导致小鼠生长过程中的细胞死亡[12]。
截至目前,尽管有关畜禽骨骼肌生长发育过程中的转录组变化已经被陆续报道[13-15],但是对骨骼肌发育过程中可变剪接的时序变化特征及其作用机制鲜有研究,而近几年迅猛发展的高通量测序数据为在全基因组水平上研究可变剪接提供了丰富的数据资源。本试验采用转录组测序数据分析简州大耳羊妊娠期第45天、60天和105天胎儿以及出生后第3天羔羊背最长肌中的可变剪接事件,从而为山羊骨骼肌生长发育的分子机制研究提供基础数据和理论参考。
1 材料与方法
1.1 组织样品的收集和高通量测序
本研究从四川简州大耳羊原种场选取一批成年简州大耳羊空怀母羊,进行同期发情处理并配种。根据配种记录,分别采集母羊妊娠后第45天(E45-1、E45-2和E45-3)、第60天(E60-1和E60-2)、第105天(E105-1、E105-2和E105-3)胎儿和出生后第3天(B3-1、B3-2和B3-3)羔羊的背最长肌样本(n=11)。分别提取总RNA,构建高通量测序文库,采用HiSeq 2500测序仪进行读段长度为125 bp的双末端测序(SRR3567144)。
1.2 序列比对和转录本组装
原始测序数据通过FastQC软件[16]进行质量评估,使用rMATS(v3.2.2 beta)软件[17]剪切尾部低质量序列后,获得长度为120 bp的高质量读段。采用STAR(v2.5)软件[18]将高质量读段比对到山羊参考基因组(https://www.ncbi.nlm.nih.gov/genome/10731?genome_assemb-ly_id=281266)[19]。利用Stringtie(v1.3.3)软件[20]组装转录本并评估基因表达量(默认参数)。
1.3 可变剪接的鉴定及基因富集分析
使用Astalavista(v4.0)软件[21]对每个样本中的可变剪接事件进行鉴定、分类和统计。根据说明,Astalavista 软件能够鉴定可变剪接外显子跳跃(skipping exon,SE)、5′端可变剪接位点(alternative 5 splice site,A5SS)、3′端可变剪接位点(alternative 3 splice site,A3SS)、内含子保留(intron retention,RI)及其他类型。利用自编的Python程序提取发生可变剪接的基因名称和数量,然后应用Gene Ontology Consortium(http://geneontology.org/)进行GO功能注释和富集分析(数据库版本日期2017-09-26)。
1.4 可变剪接因子基因表达量的分析
真核生物体内的可变剪接过程受到RNA结合蛋白(RBMs)、核内不均一核糖核蛋白 RNA聚合酶Ⅱ转录物(HNRNPs)和丝氨酸/精氨酸富集剪接因子(SRSFs)等多种类型转录因子的共同调控[22-24]。据此,本研究比较分析RBMs、HNRNPs和SRSFs3个家族的成员在简州大耳羊背最长肌中的表达变化模式。
2 结果与分析
2.1 读段的比对和转录本组装
对4个发育阶段的简州大耳羊背最长肌测序原始读段进行质量控制后,总共获得526 497 414条高质量读段。由表1可见,在各样本中,88.18%~96.62%的高质量读段成功比对到山羊参考基因组。其中,比对到基因组唯一位置的读段所占比例为77.36%~83.33%。上述高深度的测序结果为山羊背最长肌发育中可变剪接的鉴定提供了可靠的数据基础。
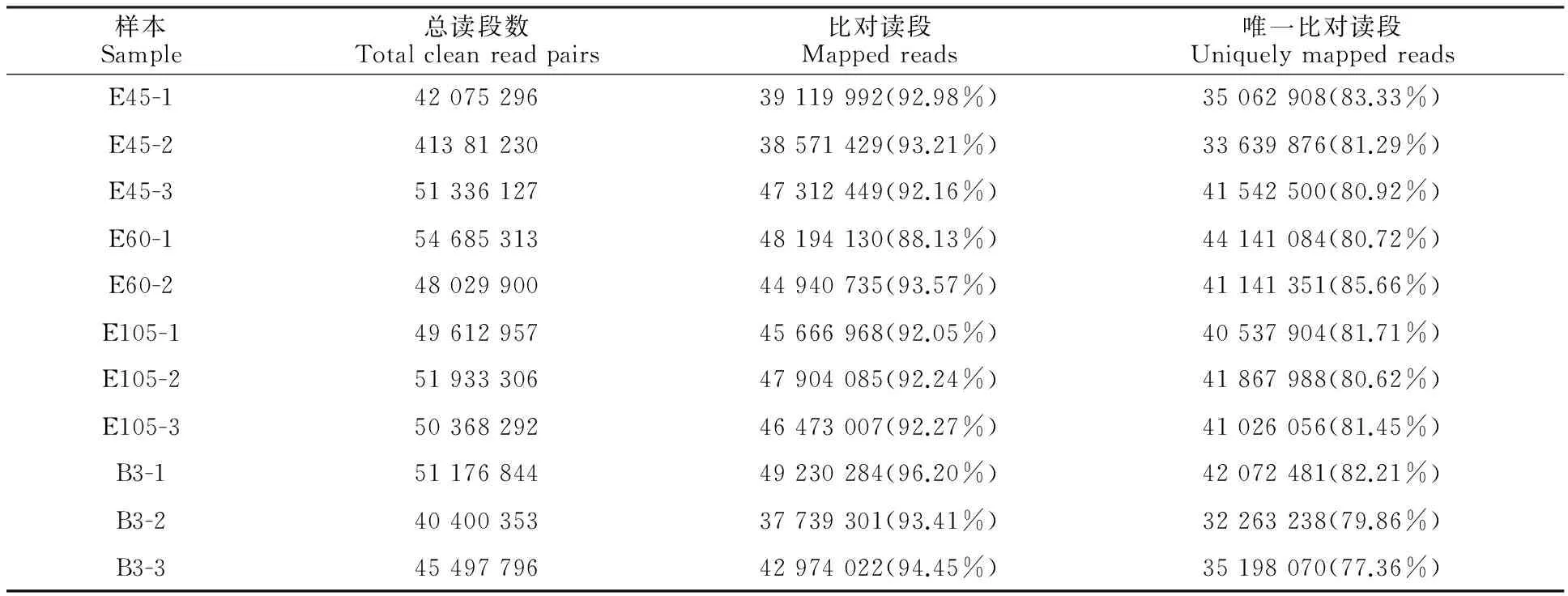
表1 11个文库读段比对结果Table 1 Mapping results of clean reads in eleven libraries
2.2 全基因组水平的可变剪接事件鉴定
采用Stringtie软件进行转录本组装,共获得2 050 999条转录本。将上述转录本与山羊参考基因组进行比较,共鉴定出137 308个可变剪接事件,577种不同类型的可变剪接组合模式。其中,以跳跃外显子、内含子保留、可变的3′端剪接位点、可变的5′端剪接位点4种基本类型的可变剪接数量最多。在各个阶段鉴定出的可变剪接类型中,SE的比例最高,占各样本总量的27.80%~34.44%;其次,A3SS、RI和A5SS分别为18.24%~20.78%,12.14%~22.10%和10.49%~11.21%(图1)。值得注意的是,随着生长发育的进行,RI类型的比例在持续降低,尤其是在出生后第3天的可变剪接数目较妊娠期显著地降低(P<0.05),检测出的可变剪接总数也显著减少(P<0.05)。另外,在妊娠期第45天和妊娠期第60天鉴定出可变剪接数量最多的基因是伴肌动蛋白(Nebulin,NEB),其数量别为71和79;妊娠期第105天和出生后第3天鉴定出可变剪接数量最多的基因是遮掩蛋白(Obscurin,OBSCN),其数量分别为131和218。

图1 4个发育阶段可变剪接事件的数量及比例Fig.1 Numbers and proportions of alternative splicing events at four developmental stages
基因组注释结果显示,鉴定出的可变剪接事件来自于4 562个已注释基因,占参考基因组中已注释基因总数(27 199)的16.77%(图2)。妊娠期第45天、妊娠期第60天、妊娠期第105天和出生后第3天的可变剪接分别来自于2 779、3 388、3 006和2 127个已注释基因。其中1 319个基因在4个发育阶段均发生了可变剪接,表明在出生前的背最长肌发育过程中大量基因发生了可变剪接。各个阶段发生可变剪接的基因比较结果显示,妊娠期第60天的特异性基因最多(615个),表明此阶段转录表达较活跃;妊娠期第45天、第105天和出生后第3天依次为361、283和137个。
2.3 可变剪接基因的功能富集分析
功能注释结果(图3)表明,4个阶段均发生可变剪接的1 319个基因分别显著富集(P<0.05)在 mRNA加工(GO:0006397)、腺苷亲核转酯反应的RNA剪接(GO:0000377)、RNA调控(GO:0043484)等291个生物学过程。在分子功能方面,上述基因显著富集在RNA结合(GO:0003723)、转录因子结合(GO:0008134)、钙黏着蛋白结合(GO:0045296)等64个GO类别中。在细胞组成方面,上述基因显著富集在核斑(GO:0016607)、收缩纤维(GO:0043292)、肌原纤维(GO:0030016)等95个GO类别。上述结果表明,各个发育阶段的可变剪接事件主要参与到大分子代谢过程和基因表达调控等过程。
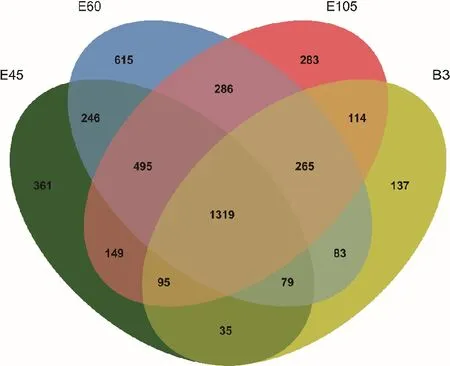
图2 4个发育阶段发生可变剪接的基因数量Fig.2 Numbers of AS genes at four developmental stages
对4个阶段发生可变剪接的特异性基因进行GO功能注释和富集分析,仅有妊娠期第45天和妊娠期第60天的基因富集结果显著。由图4可见,妊娠期第45天的特异性基因主要富集在5个GO类别中,包括生物学过程中的细胞组成结构调节(GO:0051128);细胞组成中的细胞质基质(GO:0005829)、胞内细胞器(GO:0044446)、膜内膜结合细胞器(GO:0043231);分子功能中的蛋白质结合(GO:0005515)。上述结果表明,妊娠期第45天的背最长肌中发生可变剪接的特异性基因主要功能为调节细胞内物质合成。
如图5所示,妊娠期第60天特异性基因显著富集在RNA聚合酶Ⅰ转录本延长(GO:0006362)、染色体组织(GO:0051276)、有丝分裂(GO:0000278)、细胞定位(GO:0006996)等24个GO生物学过程类别中。在细胞组成方面,上述基因显著富集在中心体(GO:0005814)、含磷基团转移酶复合物(GO:0061695)、细胞核(GO:0005730)、转移酶复合物(GO:1990234)、微管组织中心(GO:0005815)、微管骨架(GO:0015630)等35个GO类别。在分子功能方面,上述基因显著富集在嘌呤依赖性解旋酶活性(GO:0070035)、ATP-依赖性解旋酶活性(GO:0008026)、作用于RNA的催化活性(GO:0140098)、转移酶活性(GO:0016740)、杂环化合物结合(GO:1901363)、有机环状化合物结合(GO:0097159)等9个GO类别。
2.4 可变剪接因子的表达差异分析
转录组测序结果显示,RBMs、HNRNPs和SRSFs3个家族中58个基因的平均表达量大于5 FPKM。对上述基因的表达量进行单因素方差分析,结果表明,包括 HNRNPA0、 RBM15B、 SRSF9等19个基因在4个阶段中呈现显著性差异表达(P<0.05)。 如图6所示,15个可变剪接调控因子的表达量在发育过程中呈现降低趋势。而 RBM7和 RBM48的表达量表现为在出生前逐渐升高,出生后急剧下降。
3 讨 论
发生在转录后水平的可变剪接是形成高等动植物体内蛋白质多样性和复杂性的重要机制之一。Wang等[1]报道,在人类大脑、肝脏、肌肉等多个组织中,大约有95%基因发生了可变剪接。尽管如此,在不同物种中发生可变剪接基因的比例具有差异。例如,相关研究表明,猪、牛、鸭基因组分别有80.6%[25]、21%[26]、46.12%[27]基因发生了可变剪接。本研究利用转录组测序数据,在妊娠期第45天、妊娠期第60天、妊娠期第105天和出生后第3天共4个发育阶段的简州大耳羊背最长肌共鉴定出137 308个可变剪接事件,发生可变剪接的基因占山羊注释基因组总数的16.77%。该结果表明,可变剪接广泛发生在山羊骨骼肌发育过程中;值得注意的是,上述比例低于人、猪和鸭中的结果。一方面,可能是因为不同组织中发生可变剪接的基因比例不同。例如,在人类各组织中,大脑发生可变剪接的基因比例最高,而肌肉、脂肪组织相对较低[1]。另一方面,山羊参考基因组不够完善也是导致上述结果的一个重要原因。此外,本研究中外显子跳跃类型的可变剪接比例最高,这与在其他动物中发现的结果相一致。
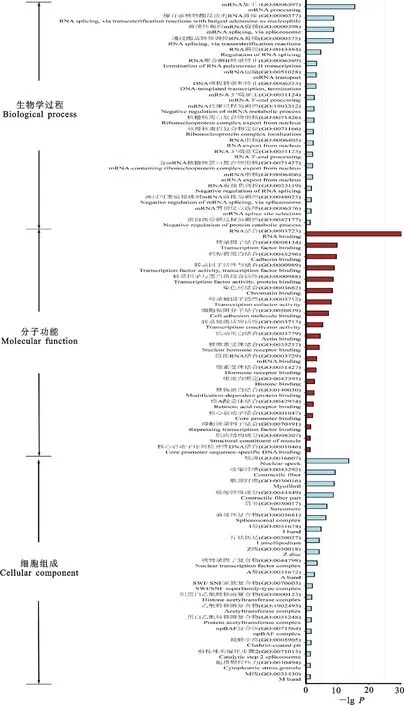
图3 4个发育阶段共有可变剪接基因的GO功能富集分析Fig.3 GO Functional enrichment analysis of the overlapped As genes across four stages
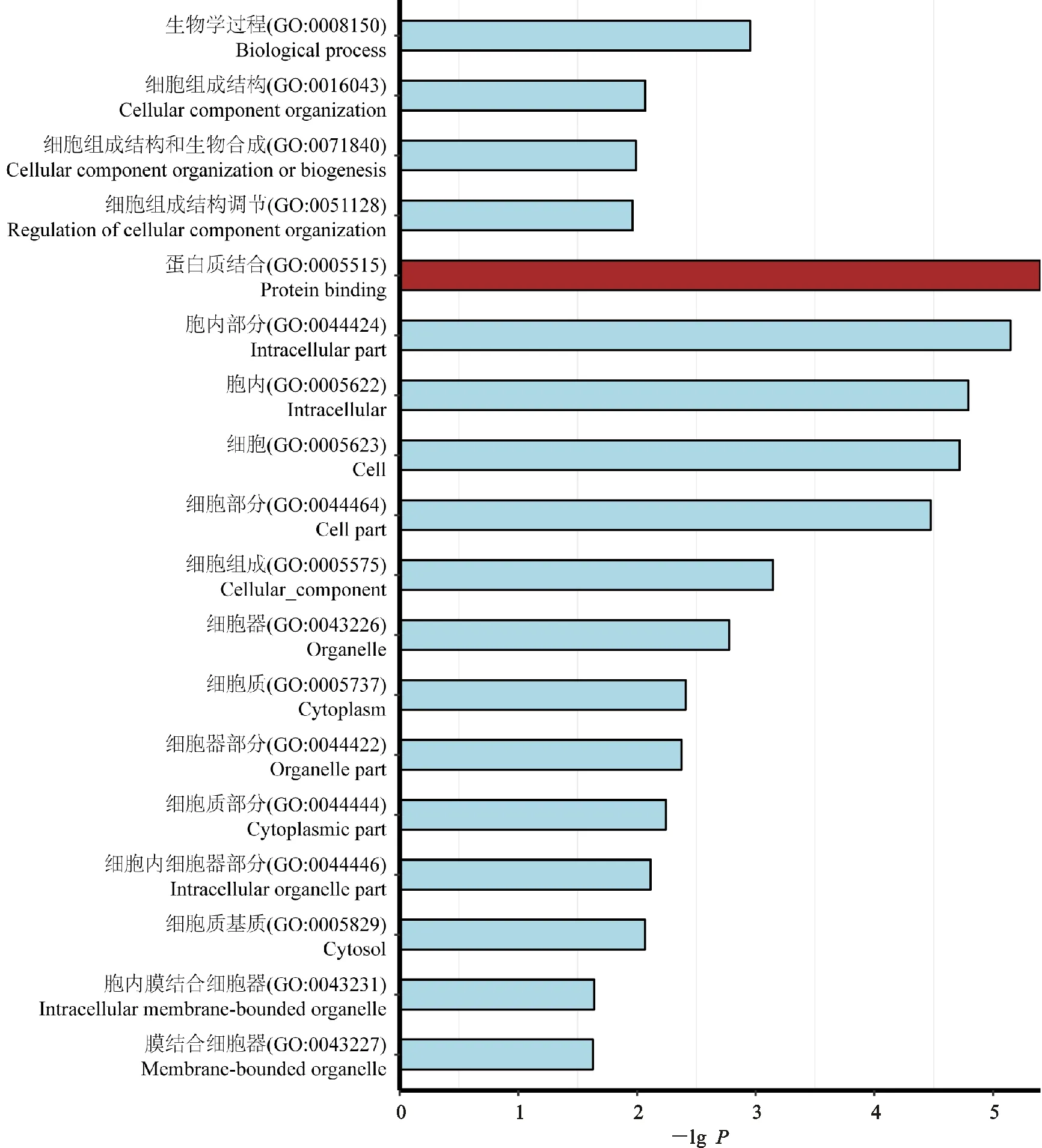
图4 妊娠期第45天特异性基因GO功能富集分析Fig.4 GO Functional enrichment analysis of unique AS genes at day 45 of gestation
已有研究表明,哺乳动物骨骼肌肌纤维数量在其出生前已经稳定,出生后主要是肌纤维的增长和变粗,故出生前是哺乳动物骨骼肌发育关键阶段[28]。例如,Greenwood等[29]和Nissen等[30]对猪早期胚胎和出生后胎儿肌肉的冷冻切片观察发现,初级纤维和次级纤维的增殖在出生前就已经停止,出生后纤维只有长度和体积的变化。在本研究中,4个发育阶段均发生可变剪接的基因进行GO功能富集分析后,细胞组成结果主要与肌肉发育相关,如肌原纤维、收缩纤维、肌节、A带和I带等,表明肌纤维在上述阶段持续发育,可变剪接基因参与到肌肉发育过程中。Wilson等[31]的研究表明,绵羊胫骨前肌在胚胎期第32天出现原发性肌管,胚胎期第38天出现少量的次级肌管,发育至第62天次级肌管形成。本研究中,山羊胎儿背最长肌在妊娠期60天的可变剪接数量和可变剪接调控因子的表达量均高于其他阶段,表明背最长肌发育进入活跃期,大量细胞在该阶段增殖和分化。同时GO功能注释和富集分析结果也显示,可变剪接基因主要富集在细胞增殖和大分子合成相关过程,表明可变剪接在背最长肌肌纤维发育的分化调控过程中具有重要作用。
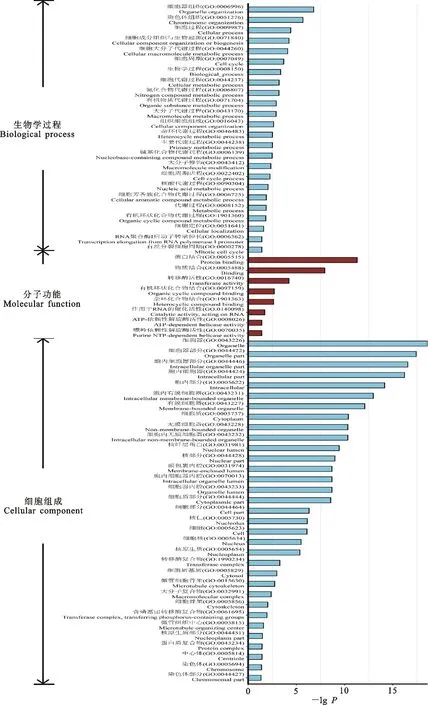
图5 妊娠期第60天特异性基因GO功能富集分析Fig.5 GO Functional enrichment analysis of unique AS genes at day 60 of gestation
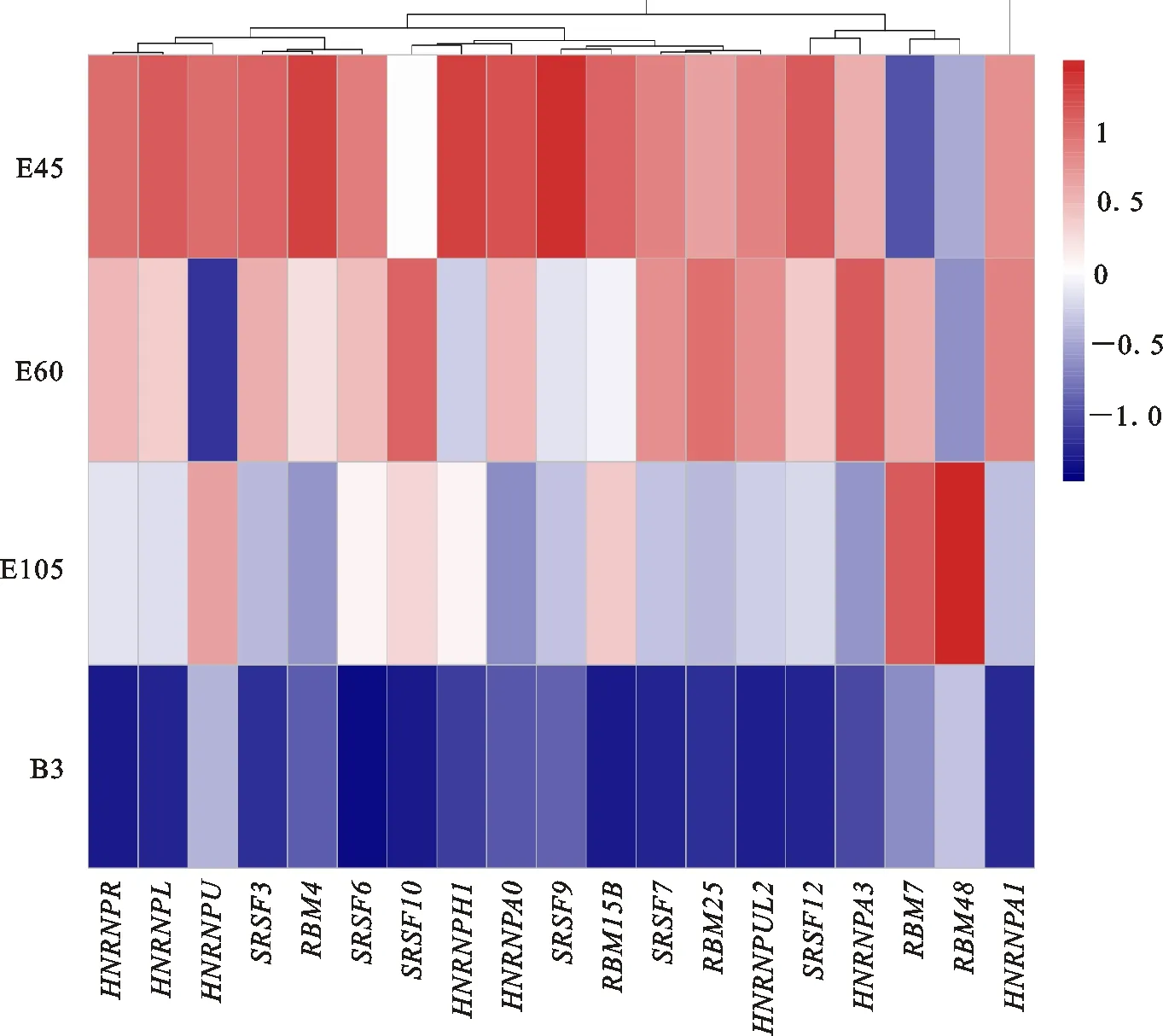
图6 19个可变剪接因子基因在4个发育阶段中的表达量热图Fig.6 Heatmap of expression profiles of 19 alternative splicing factors across four developmental stages
在高等动植物体内,可变剪接是一个受到RBMs、HNRNPs和SRSFs等多种因子协同调节的生物学过程[20-22]。例如, HNRNP A/B蛋白可以结合HIV-1病毒的3′端剪切位点和第3外显子活化区域,从而抑制其某一种转录本的表达[32-33]。Motta-Mena等[34]发现HNRNP 蛋白可以直接抑制 CD45基因的第4外显子,并且通过抑制邻近的剪接增强子影响第5外显子的表达。最近,Liu等[35]在 HNRNPA1基因敲除小鼠中证明, HNRNPA1对肌肉相关基因的表达和选择性剪接起着至关重要且不可替代的作用。在本研究中, HNRNPA1、HNRNPL和 HNRNPR等基因的表达量都呈现出随生长发育而逐渐降低的趋势。该结果表明,在生长发育早期,可变剪接因子的高表达保证了背最长肌增殖分化所需要的丰富转录本[36]。综上所述,可变剪接事件的时序变化特征与山羊背最长肌的发育过程密切相关[37]。
参考文献Reference:
[1]WANG E T,SANDBERG R,LUO S,etal.Alternative isoform regulation in human tissue transcriptomes[J].Nature,2008, 456(7221):470.
[2]PAN Q,SHAI O,LEE L J,etal.Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing[J].NatureGenetics,2008,40(12):1413-1415.
[3]GIBILISCO L,ZHOU Q,MAHAJAN S,etal.The evolution of alternative splicing inDrosophila[J].Biorxiv,2016,2(21):054700.
[4]SHEN Y,ZHOU Z,WANG Z,etal.Global dissection of alternative splicing in paleopolyploid soybean[J].ThePlantCell,2014,26(3):996-1008.
[5]LIN L,PARK JW,RAMACHANDRAN S,etal.Transcriptome sequencing reveals aberrant alternative splicing in Huntington’s disease[J].HumanMolecularGenetics,2016,25(16):3454-3466.
[6]LI W,LIN W D,RAY Petal.Genome-wide detection of condition-sensitive alternative splicing inArabidopsisroots[J].PlantPhysiology,2013,162(3):1750-1763.
[7]PICKRELL J K,PAI A A,GILAD Y,etal.Noisy splicing drives mRNA isoform diversity in human cells[J].PLoSGenetics,2010,6(12):e1001236.
[8]TRESS M L,ABASCAL F,VALENCIA A.Alternative splicing may not be the key to proteome complexity[J].TrendsinBiochemicalSciences,2017,42(2):98-110.
[9]NILSEN T W,GRAVELEY B R.Expansion of the eukaryotic proteome by alternative splicing[J].Nature,2010,463(7280):457-463.
[10]SINGH R K,XIA Z,BLAND C S,etal.Rbfox2-coordinated alternative splicing of Mef2d and Rock2 controls myoblast fusion during myogenesis[J].MolecularCell,2014,55(4):592-603.
[11]DU H,CLINE M S,OSBORNE R J,etal.Aberrant alternative splicing and extracellular matrix gene expression in mouse models of myotonic dystrophy[J].NatureStructural&MolecularBiology,2010,17(2):187-193.
[12]XU X,YANG D,DING J H,etal.ASF/SF2-regulated CaMKIIδ alternative splicing temporally reprograms excitation-contraction coupling in cardiac muscle[J].Cell,2005,120(1):59-72.
[13]ZHAO X,MO D,LI A,etal.Comparative analyses by sequencing of transcriptomes during skeletal muscle development between pig breeds differing in muscle growth rate and fatness[J].PloSOne,2011,6(5):e19774.
[14]ZHAN S,ZHAO W,SONG T,etal.Dynamic transcriptomic analysis in hircine longissimus dorsi muscle from fetal to neonatal development stages[J].Functional&IntegrativeGenomics,2017(S1):1-12.
[15]WANG Y,ZHANG C,FANG X,etal.Identification and profiling of microRNAs and their target genes from developing Caprine skeletal muscle[J].PloSOne,2014,9(5):e96857.
[16]VESELOVSKA L,SMALLWOOD S A,SAADEH H,etal.Deep sequencing and de novo assembly of the mouse oocyte transcriptome define the contribution of transcription to the DNA methylation landscape[J].GenomeBiology,2015,16(1):209.
[17]SHEN S,PARK J W,LU Z X,etal.rMATS:robust and flexible detection of differential alternative splicing from replicate RNA-Seq data[J].ProceedingsoftheNationalAcademyofSciences,2014,111(51):E5593-E5601.
[18]DOBIN A,DAVIS C A,SCHLESINGER F,etal.STAR:ultrafast universal RNA-seq aligner[J].Bioinformatics,2013,29(1):15-21.
[19]BICKHART D M,ROSEN B D,KOREN S,etal.Single-molecule sequencing and chromatin conformation capture enable de novo reference assembly of the domestic goat genome[J].NatureGenetics,2017,49(4):643-650.
[20]PERTEA M,PERTEA G M,ANTONESCU C M,etal.StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J].NatureBiotechnology,2015,33(3):290-295.
[21]FOISSAC S,SAMMETH M.ASTALAVISTA:dynamic and flexible analysis of alternative splicing events in custom gene datasets[J].NucleicAcidsResearch,2007,35(2):W297-W299.
[22]MANLEY J L,KRAINER A R.A rational nomenclature for serine/arginine-rich protein splicing factors(SR proteins)[J].Genes&Development,2010,24(11):1073-1074.
[23]ELLIOTT D J,OGHENE K,MAKAROV G,etal.Dynamic changes in the subnuclear organisation of pre-mRNA splicing proteins and RBM during human germ cell development[J].JournalofCellScience,1998,111(9):1255-1265.
[24]HWANG B,LIM J H,HAHM B,etal.hnRNP L is required for the translation mediated by HCV IRES[J].BiochemicalandBiophysicalResearchCommunications,2009,378(3):584-588.
[25]冉茂良,陈斌,李智,等.基于RNA-seq测序数据鉴定和分析猪基因组可变剪接事件[J].中国科学(生命科学),2016(3):274-284.
RAN M L,CHEN B,LI ZH,etal.Identification and analysis of alternative splicing events in sus scrofa using RNA-Seq data[J].ScientiaSinica(Vitae),2016(3):274-284.
[26]CHACKO E R S.Genome-wide analysis of alternative splicing in cow:implications in bovine as a model for human diseases[J].BMCGenomics,2009,3(3):S11.
[27]徐铁山,顾丽红,侯水生,等.应用RNA-seq数据开展鸭基因组可变剪接的鉴定与分析[J].中国家禽,2016(17):10-16.
XU T SH,GU L H,HOU SH SHetal.Identification and analysis of alternative splicing in duck genome using RNA-seq data[J].ChinaPoultry,2016(17):10-16.
[28]PICARD B,LEFAUCHEUR L,BERRI C,etal.Muscle fibre ontogenesis in farm animal species[J].ReproductionNutritionDevelopment,2002,42(5):415-431.
[29]GREENWOOD P,HUNT A,HERMANSON J,etal.Effects of birth weight and postnatal nutrition on neonatal sheep:Ⅱ.Skeletal muscle growth and development[J].JournalofAnimalScience,2000,78(1):50-61.
[30]NISSEN P,DANIELSEN V,JORGENSEN P,etal.Increased maternal nutrition of sows has no beneficial effects on muscle fiber number or postnatal growth and has no impact on the meat quality of the offspring[J].JournalofAnimalScience,2003,81(12):3018-3027.
[31]WILSON S,MCEWAN J,SHEARD P,etal.Early stages of myogenesis in a large mammal:formation of successive generations of myotubes in sheep tibialis cranialis muscle[J].JournalofMuscleResearchandCellMotility,1992,13(5):534-550.
[32]BILODEAU P S,DOMSIC J K,MAYEDA A,etal.RNA splicing at human immunodeficiency virus type 1 3′ splice site A2 is regulated by binding of hnRNP A/B proteins to an exonic splicing silencer element[J].JournalofVirology,2001,75(18):8487-8497.
[33]ZHU J,MAYEDA A,KRAINER A R.Exon identity established through differential antagonism between exonic splicing silencer-bound hnRNP A1 and enhancer-bound SR proteins[J].MolecularCell,2001,8(6):1351-1361.
[34]MOTTA MENA L B,HEYD F,LYNCH K W.Context-dependent regulatory mechanism of the splicing factor hnRNP L[J].MolecularCell,2010,37(2):223-234.
[35]LIU T Y,CHEN Y C,JONG Y J,etal.Muscle developmental defects in heterogeneous nuclear Ribonucleoprotein A1 knockout mice[J].OpenBiology,2017,7(1):160303.
[36]GABUT M,S AMAVARCHI-TEHRANI P,WANG X,etal.An alternative splicing switch regulates embryonic stem cell pluripotency and reprogramming[J].Cell,2011,147(1):132-146.
[37]KALSOTRA A,COOPER T A.Functional consequences of developmentally regulated alternative splicing[J].NatureReviewsGenetics,2011,12(10):715-729.
