基于CNN-BLSTM-CRF模型的生物医学命名实体识别
2018-04-04李丽双郭元凯
李丽双,郭元凯
(大连理工大学 计算机科学与技术学院,辽宁 大连 116023)
0 引言
命名实体识别是自然语言处理中的重要任务之一。近年来,神经网络在通用领域的命名实体识别表现出了很好的性能。相比于统计机器学习方法或基于规则的方法,基于神经网络的深度学习方法具有泛化性更强、更少依赖人工特征的优点。因此,许多基于神经网络的通用领域命名实体识别模型被提出。例如Collobert[1]等首次使用CNN与CRF结合的方式在通用命名实体识别领域的CONLL2003语料上取得了较好的效果。Huang[2]等构造了一个采用人工设计的拼写特征的BLSTM-CRF模型,在CONLL2003语料上达到了88.83%的F-值。Chiu 和Nichols[3]等建立了CNN-LSTM模型在CONLL2003语料上达到了91.62%的F-值。虽然神经网络在通用命名实体识别领域中展现出了较好的性能,但在生物医学命名实体识别领域中的应用仍存在问题。相比于一般领域的命名实体,生物医学命名实体识别有以下几个难点: (1)包含的实体数量和种类多; (2)待识别的实体可能会由许多单词修饰,导致实体的边界难以划分; (3)生物医学语言没有一套统一的命名方式,所以待识别的实体可能会有多种表述方式; (4)待识别的实体经常存在缩写、嵌套、大小写混合、含有特殊字符的情况。也正是因为如此,生物医学命名实体识别的许多方法依旧依赖人工特征和领域知识。
目前生物医学命名实体识别的方法主要分为浅层机器学习和深层神经网络的方法。浅层机器学习方法主要包括条件随机场模型(CRF)、隐马尔可夫模型(HMM)、最大熵模型(ME)、支持向量机(SVM)等。例如,Li[4]等通过使用丰富的人工特征基于CRF进行实体识别,在Biocreative Ⅱ GM语料上达到了87.28%的F-值。Manabu[5]等将CRF、HMM、ABNER、LingPipe等模型融合,在Biocreative Ⅱ GM语料上达到了最高F-值88.87%。此外,Wang[6]等验证了基于CRF的Gimli方法,在JNLPBA2004语料上F-值达到了72.23%。Zhou和Su[7]通过丰富的领域知识和人工特征采用CRF在JNLPBA2004语料上F-值提高到了72.55%。Liao[8]等构建了skip-chain CRF模型用于生物医学命名实体识别,该模型能够充分考虑到较远距离具有依赖关系的生物医学信息,在JNLPBA2004语料上达到了73.20%的F-值。但是传统的浅层机器学习方法在很大程度上依赖于人工特征的设计,人工特征和领域知识在提高模型性能的同时也导致整个模型的鲁棒性和泛化能力下降。
为了减少复杂的人工特征,有相关研究利用词向量结合浅层机器学习方法进行生物实体识别。如Tang[9]等采用CRF模型进行生物实体识别,在基本人工特征的基础上加入不同的词向量特征,在BioCreative Ⅱ GM和JNLPBA语料上的F-值分别为80.96%和71.39%。Chang[10]等利用少量人工特征和词向量结合的方式构建CRF模型并添加后处理,在JNLPBA语料上达到了71.39%的F-值。虽然词向量在一定程度上能够提高浅层机器学习方法的性能,但是与其他最好的系统相比仍然存在一定的差距,这主要是因为这些词向量本身包含的特征信息有限,并不能完全取代复杂的人工特征,而且难以处理长距离依赖关系。
在使用深度神经网络进行生物医学命名实体识别的研究中,Yao[11]等首先在无标注的生物文本上利用神经网络生成词向量,然后建立多层神经网络,在JNLPBA语料上F-值为71.01%。Li[12]等采用双向长短期记忆网络(BLSTM)方法在Biocreative Ⅱ GM的语料上达到了88.6%的F-值,同时在JNLPBA语料上达到了72.76%的F-值。上述研究虽然没有使用领域知识和人工特征,但是词向量对于字符级特征不能很好表示,因此识别性能有待提高。本文提出一种基于CNN-BLSTM-CRF神经网络模型,该模型首先利用CNN训练出单词的字符级特征,然后与从大规模背景语料训练得到的词向量进行组合,再将组合的词向量送入BLSTM-CRF深层神经网络进行训练,从而得到一个利用字符级特征和词向量的生物实体识别模型。在Biocreative Ⅱ GM和JNLPBA2004语料上的实验结果表明,在未使用任何人工特征的情况下,该模型在两个语料上都达到了目前的最好效果,F-值分别是89.09%和74.40%。
1 CNN-BLSTM-CRF模型
1.1 模型整体框架
图1为本文的CNN-BLSTM-CRF模型框架。CNN-BLSTM-CRF共由三部分组成: CNN模块,BLSTM模块和CRF模块。首先通过查询词向量表将输入的语句转换为相应的词向量序列,然后对于语句中的每一个单词,通过查询字符向量表获得每个字符的字符向量,由字符向量组成单词的字符向量矩阵。CNN对字符向量矩阵进行卷积和池化,获得每个单词的字符级特征。每个单词的字符向量和词向量进行拼接,拼接后的词向量输入BLSTM进行实体识别。最后CRF模块将BLSTM的输出解码出一个最优的标记序列。
1.2 CNN模块
卷积神经网络中的卷积层能够很好地描述数据的局部特征,通过池化层可以进一步提取出局部特征中最具有代表性的部分。Santos[13]等利用CNN对字符进行处理得到CharWNN 用于词性标注工作(POS),并取得了较好的效果。Chiu 和Nichols[3]等采用CNN抽取字符级特征在通用实体识别领域达到了很好的效果。因此,本文提出利用CNN抽取生物医学文本中单词的字符级特征,通过字符级特征与词向量相结合的方法来提高模型的性能。这里使用的CNN模块与Chiu[3]不同之处如下: (1)本文并没有采用Chiu[3]额外设计一些人工的字符特征与字符向量拼接的方法; (2)本文对于不同类型的字符设置并随机初始化了不同的字符向量,以区分字符的大小写、字符类型(字母、数字、标点、特殊字符)。例如,大写字母A与小写字母a分别对应两组不同的字符向量。
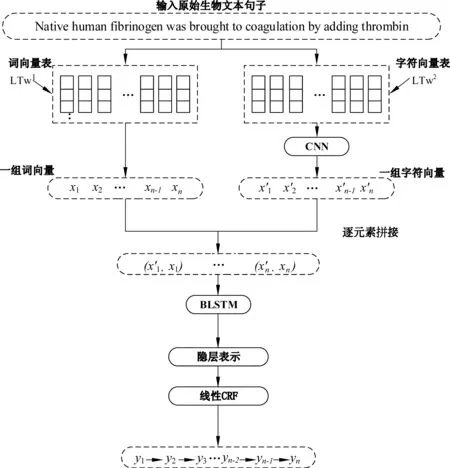
图1 生物医学命名实体识别的CNN-BLSTM-CRF模型
CNN的结构如图2所示,主要由字符向量表、卷积层、池化层组成。字符向量表将一个单词中的每个字符转化成为对应的字符向量。首先,由单词的每个字符的字符向量组成单词的字符向量矩阵。其次,为了解决由于单词长度不同导致字符向量矩阵大小不同的问题,以最长的单词为准,在单词的左右两端补充占位符(padding),使得所有字符向量矩阵大小一致。最后,字符向量表在模型的训练过程中通过反向传播算法不断更新。
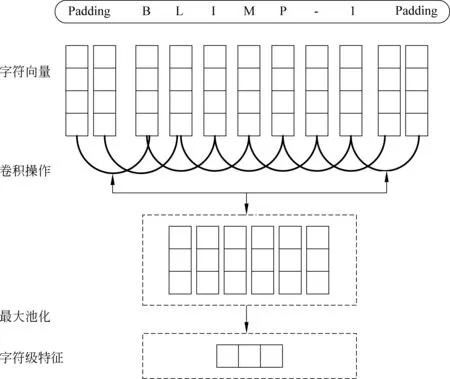
图2 字符级卷积神经网络模型
卷积层使用一个大小是T的卷积核在单词的字符向量矩阵上进行卷积来提取出局部特征,卷积核大小T决定了可以提取单词周围T个词的特征,最后通过池化获得单词的字符级特征向量。
1.3 BLSTM模块
长短时记忆网络(LSTM)[14]是一种特殊的循环网络(RNN)模型,克服了传统RNN模型由于序列过长而产生的梯度弥散问题。LSTM模型通过特殊设计的门结构使得模型可以有选择地保存上下文信息,因此LSTM具有适合生物医学命名实体识别的特点。LSTM网络的主要结构可以形式化地表示为:
(1)

为了能够有效利用上下文信息,我们采用双向LSTM(BLSTM)结构。双向LSTM对每个句子分别采用顺序(从第一个词开始,从左往右递归)和逆序(从最后一个词开始,从右向左递归)计算得到两套不同的隐层表示,然后通过向量拼接得到最终的隐层表示。
1.4 线性CRF模块
CRF能够通过考虑相邻标签的关系获得一个全局最优的标记序列。本文将CRF融合到BLSTM模块中,对BLSTM的输出进行处理,获得全局最优的标记序列。对于一个句子S={W1,W2,…,Wn}送入网络中训练,定义矩阵P是BLSTM层的输出结果,其中P的大小n×m,n是单词个数,m是标签的种类。定义pij代表句子中第i个单词的第j个标签的概率。对于一个预测序列y={y1,y2,…,yn},它的概率可以表示为:
式中,矩阵A是转移矩阵,例如Aij表示由标签i转移到j的概率,y0、yn则是预测句子起始和结束的标记,因此A是一个大小为m+2的方阵。所以在原语句S的条件下产生标记序列y的概率为:
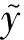
在训练过程中标记序列的似然函数:
其中,YX表示所有可能的标记集合,包括不符合BIOES标记规则的标记序列。通过式(4)得到有效合理的输出序列。预测时,由式(5)输出整体概率最大的一组序列:
1.5 训练参数
训练过程中,优化器采用RMSprop,相比于随机梯度下降SGD模型的训练速度更加快速;学习率选取0.001。同时,通过实验发现在双向LSTM的输入和输出部分增加Dropout可以减轻模型过拟合的问题,Dropout[15]值选取了0.5。整体的模型训练通过GTX1080进行加速。
2 实验结果
为了说明本文CNN-BLSTM-CRF模型的有效性和泛化性,分别选用了Biocreative Ⅱ GM和JNLPBA2004语料进行了实验。所有的实验都是基于预训练得到的200维词向量和相同的参数进行的。
2.1 语料介绍
Biocreative Ⅱ GM和JNLPBA2004语料详细信息见表1。此外,JNLPBA的语料不同于Biocreative Ⅱ GM语料,JNLPBA待识别的实体有五种,分别是DNA、RNA、Cell_line、Cell_type、Protein,所以相对于Biocreative Ⅱ GM语料,只需识别出基因实体,JNLPBA则还需对识别出的实体给出准确的类别。
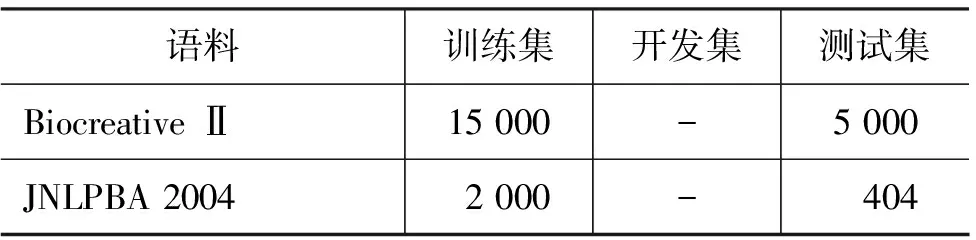
表1 语料介绍
在语料处理方面,为了能够清楚地表示语料中待识别的命名实体,我们采用了BIOES标记的方式代替BIO2去标记实体。因为根据Ratinov 和Roth[16]、Dai[17]、Lample[18]等的研究采用BIOES的标记效果好于BIO2方式,能更加清楚地划分实体的边界。
2.2 实验结果及分析
CNN-BLSTM-CRF模型在Biocreative Ⅱ GM语料上进行的生物命名实体识别结果如表2所示,下面通过对比实验的结果来分析各个模块在模型中起到的作用。
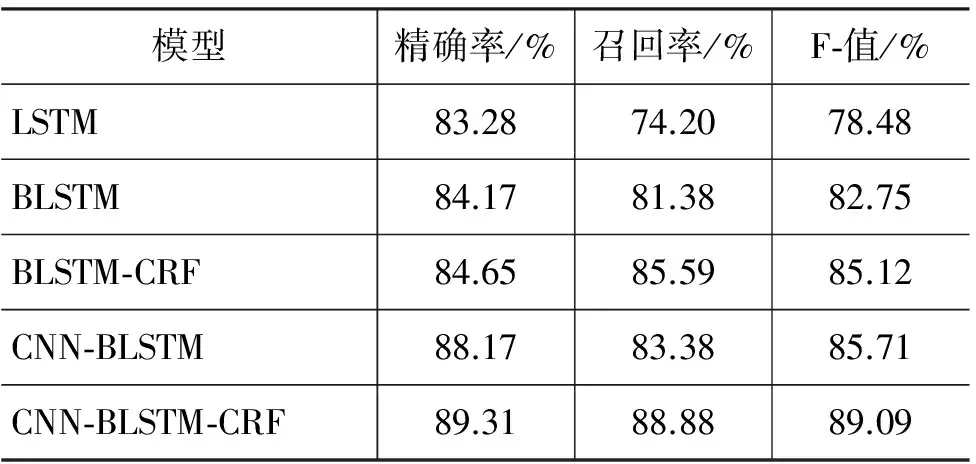
表2 Biocreative Ⅱ GM结果
(1) BLSTM模块
为了验证BLSTM结构的有效性,进行了BLSTM模型与LSTM模型的对比实验。根据实验结果,BLSTM模型在Biocreative Ⅱ GM语料上的F-值为82.75%,精确率为84.17%,召回率为81.38%,比LSTM模型的F-值高出了4.27%。无论是召回率还是精确率,双向LSTM递归神经网络明显优于单向的网络,主要由于BLSTM模型比LSTM模型更加充分地利用了上下文信息。
(2) CNN 模块
为了验证CNN模块抽取的字符级特征的有效性,进行了CNN-BLSTM模型与BLSTM模型的对比实验。实验结果表明,CNN-BLSTM相对于BLSTM精确率提升了4.00%,召回率提升了2.00%,F-值提升了2.96%。表明CNN抽取的字符级特征的有效性。由于我们通过CNN模块抽取的字符级向量能够一定程度上表示形态特征,所以对于具有大小写混合、包含特殊字符、边界模糊特点的这类生物实体能够充分获取相关特征,从而提高识别的F-值。例如“LTRran1+ kinase”“fructose-2,6-bisphosphatase”“G alpha i-2”等实体被CNN-BLSTM正确识别,而BLSTM则不能正确识别。由此可见CNN模块的加入使得模型对存在含有特殊字符的实体有效果提升。
(3) 线性CRF 模块
为了验证CRF模块的有效性,进行了BLSTM-CRF模型与BLSTM模型的对比实验。实验结果表明,BLSTM-CRF模型相比于BLSTM模型,召回率、精确率、F-值分别提高了4.21%、0.48%、2.73%。由于线性CRF能够充分利用相邻标签的关系,在全局优化输出的标签序列中,对长度较大及带有修饰词汇的生物命名实体的识别性能较高。例如“mammalian glycoprotein hormone receptors、PITIM-compelled multi-phosphoprotein complex、human urokinase-type plasminogen activator gene”这类生物医学实体被BLSTM-CRF正确识别,而BLSTM模型则不能正确识别。CRF模块的加入解决了一部分含有修饰词、长度较大实体的识别问题。
2.3 与现有其他工作的对比
(1) Biocreative Ⅱ GM语料
表3给出了Biocreative Ⅱ GM语料上本文模型与先进系统的对比结果。下面从是否采用人工特征进行分析。
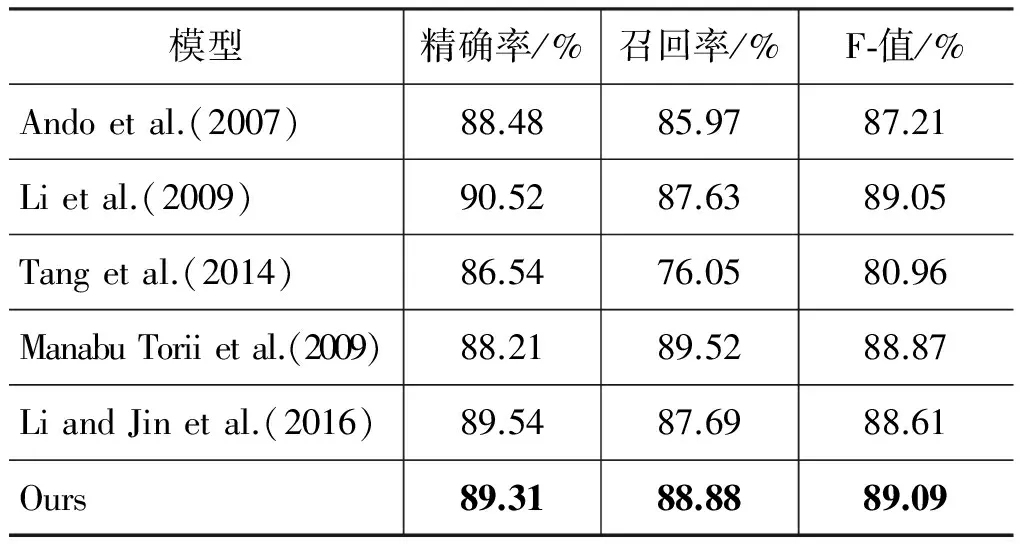
表3 在Biocreative Ⅱ GM语料上的模型对比
在使用人工特征的方法中,Ando[19]等从大规模未标注数据中学习新的特征表示,结合字典和大量设计的人工特征在Biocreative Ⅱ GM评测上取得了第一名,达到了87.21%的F-值。Li和Lin et al[4]等通过使用丰富的人工特征,例如: 词性、形态特征、词干等用CRF进行实体识别,达到了89.05%的F-值。Tang[9]等通过使用词表示特征的方式达到了80.96%的F-值。Manabu Torii[5]等通过将CRF、HMM、ABNER、LingPipe等模型融合,在Biocreative Ⅱ GM语料上达到了88.87%的F-值。而本文方法自动学习词向量和字符向量,未采用任何基于人工总结的特征,取得了比采用大量领域知识和人工特征方法更好的结果。
在不依赖人工特征的方法中,Li和Jin[12]通过构建带有双词向量和句子向量的BLSTM模型达到了88.61%的F-值。该方法充分利用了词向量所表示的语义但未能表达字符级形态特征。本文的CNN-BLSTM-CRF模型则通过CNN卷积获得了字符级的词的形态特征,并与词向量组合,达到了89.09%的F-值,比Li[9]的结果高了0.48%。
通过以上对比分析可以看出,我们的模型在未使用任何人工特征的情况下,在Biocreative Ⅱ GM取得了目前的最好结果。
(2) JNLPBA语料
为了说明我们模型的泛化能力,表4给出了JNLPBA语料上与其他先进模型的对比实验。同样从是否采用人工特征方面进行分析。
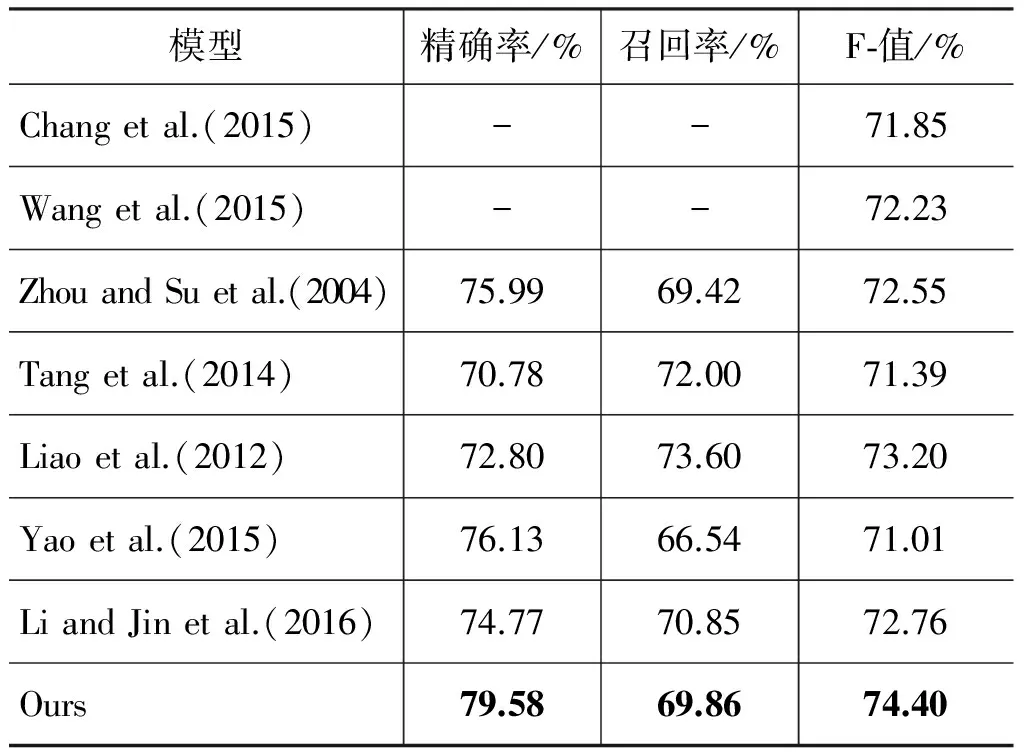
表4 在JNLPBA语料上的模型对比
在使用人工特征方面,良好设计的人工特征起到了很好的作用。例如,Chang[10]等通过一些人工设计的特征和词向量送入CRF模型进行训练达到了71.85%的F-值。此外Wang[20]等验证了基于CRF的Gimli方法达到了当时的最高F-值72.23%,Zhou和Su[7]通过丰富的领域知识和人工特征使F-值提高到了72.55%。Tang[9]等通过使用词表示特征的方式达到了71.39%的F-值。Liao[8]等构建了能充分考虑到长距离依赖关系的skip-chain CRF模型,在JNLPBA2004语料上达到了73.20%的F-值。 而本文提出的CNN-BLSTM-CRF模型,未采用任何人工特征,在JNLPBA语料上取得了更好的结果。
同样也有探索不依赖人工特征的深层神经网络方法,Yao[11]等通过使用多层神经网络学习特征表示,达到了71.01%的F-值。Li和Jin等人[12]通过构建带有双词向量和句子向量的BLSTM模型,达到了72.76%F-值。本文的方法F-值为74.40%,比Yao[11],Li和Jin[12]等分别高出了3.39%,1.64%。由表4可以,CNN-BLSTM-CRF模型比目前最好的系统Liao[8]F-值提高1.20%。
3 结论
本文针对生物医学命名实体识别任务,提出了通过CNN网络获得字符级特征来补充词向量,进而构建CNN-BLSTM-CRF神经网络模型的方法,在Biocreative Ⅱ GM和JNLPBA语料上取得了目前最好的性能。主要结论如下:
(1) 在生物医学命名实体识别任务中,人工特征和领域知识对于结果的影响很大。但是构建合适的人工特征需要大量的特征选择实验,导致了系统的成本提升、泛化能力下降。因而本文构建了CNN-BLSTM-CRF深层神经网络模型,在不使用任何人工特征的情况下,获得了比使用大量丰富特征和领域知识的浅层机器学习方法更好的结果。
(2) 本文提出了利用CNN网络卷积来获得表示单词形态特征的字符向量,用以补充词向量的不足。通过字符向量的加入,使得模型对于含有特殊字符、大小写混合这类实体能够更有效地识别,从而提高了模型的性能。
(3) 为了获得更加准确的识别结果,我们通过CRF对CNN-BLSTM网络的输出进行解码,获得最优的标记序列。CRF的融入提升了对于含有多修饰词、边界模糊的生物医学实体的识别性能。
综上,在生物医学命名实体识别任务上,本文提出的通过CNN网络获得字符级特征来补充词向量,以及BLSTM 与CRF模型的融合,都是有效提高识别性能的途径。
[1]Pinheiro P H O, Collobert R. Recurrent convolutional neural networks for scene parsing[J], 2014(1): 82-90.
[2]Huang Z, Xu W, Yu K. Bidirectional LSTM-CRF models for sequence tagging[J]. arXiv preprint arXiv:1508.01991,2015.
[3]Chiu J P C, Nichols E. Named entity recognition with bidirectional LSTM-CNNs[J]. arXiv preprint arXiv:1511.08308,2015.
[4]Li Y, Lin H, Yang Z. Incorporating rich background knowledge for gene named entity classification and recognition[J]. BMC Bioinformatics, 2009, 10(1): 223.
[5]Torii M, Hu Z, Wu C H, et al. BioTagger-GM: a gene/protein name recognition system[J]. Journal of the American Medical Informatics Association, 2009, 16(2): 247-255.
[6]Wang X, Yang C, Guan R. A comparative study for biomedical named entity recognition[J]. International Journal of Machine Learning & Cybernetics, 2015: 1-10.
[7]Zhou G D, Jian S. Exploring deep knowledge resources in biomedical name recognition[C]//Proceedings of International Joint Workshop on Natural Language Processing in Biomedicine and ITS Applications. Association for Computational Linguistics, 2004: 96-99.
[8]Liao Z, Wu H. Biomedical named entity recognition based on skip-chain CRFS[C]//Proceedings of the Industrial Control and Electronics Engineering (ICICEE),2012: 1495-1498.
[9]Tang B, Cao H, Wang X, et al. Evaluating word representation features in biomedical named entity recognition tasks[J]. BioMed Research International, 2014: 1-6.
[10]Chang F, Guo J, Xu W, et al. Application of word embeddings in biomedical named entity recognition tasks [J]. Digital Inf. Manage, 2015, 13(5): 321-327.
[11]Yao L, Liu H, Liu Y, et al. Biomedical named entity recognition based on deep neutral network[J]. International Journal of Hybrid Information Technology, 2015, 8(8): 279-288.
[12]Li L, Jin L, Jiang Y, et al. Recognizing biomedical named entities based on the sentence vector/twin word embeddings conditioned bidirectional LSTM[C]//Proceedings of China National Conference on Chinese Computational Linguistics. Springer International Publishing, 2016: 165-176.
[13]Santos C D, Zadrozny B. Learning character-level representations for part-of-speech tagging[C]//Proceedings of the 31st International Conference on Machine Learning (ICML-14), 2014: 1818-1826.
[14]Hochreiter S, Schmidhuber J. Long short-term memory[J]. Neural Computation, 2014, 9(8):1735-1780.
[15]Srivastava N, Hinton G E, Krizhevsky A, et al. Dropout: A simple way to prevent neural networks from overfitting[J]. Journal of Machine Learning Research, 2014, 15(1): 1929-1958.
[16]Ratinov L, Roth D. Design challenges and misconceptions in named entity recognition[C]//Proceedings of the 13th Conference on Computational Natural Language Learning. Association for Computational Linguistics, 2009: 147-155.
[17]Dai H J, Lai P T, Chang Y C, et al. Enhancing of chemical compound and drug name recognition using representative tag scheme and fine-grained tokenization[J]. Journal of Cheminformatics, 2015, 7(1):S14.
[18]Lample G, Ballesteros M, Subramanian S, et al. Neural architectures for named entity recognition[J], 2016, arXiv preprint arXiv:1603.01360.
[19]Ando R K. BioCreative Ⅱ gene mention tagging system at IBM watson[C]//Proceedings of the Second BioCreative Challenge Evaluation Workshop, 2007,(23): 101-103.
[20]Wang X, Yang C, Guan R. A comparative study for biomedical named entity recognition[J]. International Journal of Machine Learning and Cybernetics, 2015: 1-10.
