樟树NBS-LRR类抗病基因家族分析与CcRNL基因克隆
2018-03-14郑永杰伍艳芳李江章挺汪信东
郑永杰 伍艳芳 李江 章挺 汪信东
(江西省林业科学院 国家林业局樟树工程技术研究中心,南昌 330032)
抗病基因(R基因)在植物抵御病原菌的入侵过程中起着关键的作用。在植物与病原菌的相互作用中,植物会分泌由R基因编码的R蛋白,直接或间接识别病原菌分泌的效应因子(Avr),激活信号传导并引发一系列防御反应,从而抑制病原菌对植物的侵染过程。NBS-LRR类基因是目前植物中成员数量最多的一类R基因家族,NBS-LRR类基因主要由核苷酸结合位点(Nucleotide-binding site,NBS)和富亮氨酸重复结构(Leucine-rich repeat,LRR)编码组成。NBS结构域主要负责ATP和GTP的水解及信号的传导[1],而LRR结构域则负责病原菌蛋白的识别和相互作用[2]。根据N末端序列特征,NBS类抗病基因主要分为TNL(TIR-NBS-LRR)抗病基因和CNL(CC-NBS-LRR)抗病基因。有研究发现部分CNL类抗病基因在CC结构域内存在编码RPW8基因的区域,命名为RNL类抗病基因。拟南芥RPW8类基因对白粉病具有广谱抗性[3],异源表达能够增强烟草对白粉病菌[4]和水稻对稻瘟病菌的抗性。病原微生物侵染植物时,RPW8类基因通过激活水杨酸(Salicylic acid,SA)信号通路,诱发抗病反应[5-6]。
目前,不同植物中克隆超过140个抗病基因,其中80%属于NBS-LRR类基因。NBS-LRR抗病基因家族已成为植物抗病研究的热点,已在拟南芥[7]、水稻[8]、杨树[9]、葡萄[10]和木薯[11]等植物中展开。樟树(Cinnamomum camphoraL. Presl.)是自然界中较少受病原体侵害的优良抗性种质资源,但在生长过程中亦受部分病菌侵袭、尤其对苗期生长威胁较大。文献报道,由壳梭孢菌(Fusicoccumsp.)引起的壳梭孢溃疡病、拟茎点霉菌(Phomopsissp.)引起的拟茎点溃疡病和球二孢(Lasiodiplodiasp.)引起的球二孢溃疡病,严重时樟树发病率和死亡率将分别高达90%和80%以上[12-16];子囊菌亚门茶藨子葡萄座腔菌(Botryosphaeria ribis)与半知菌亚门桑茎点菌(Phoma moricola)引起的烂皮病和由胶孢炭疽病(Colletotrichum gloeosporioidesPenz.)引起的炭疽病严重时也可致樟树整株死亡[17-18];变色拟盘多毛孢(Pestalotiopsis versicolor(Speg.)Stey.)引起的拟盘多毛孢枝枯病及根腐病对樟树生长危害也较大[16]。樟树白粉病主要发生在苗期,在不通风的苗圃中可致大面积爆发,引起幼株死亡,成年樟树对白粉病具有较好的抗性。目前樟树白粉病菌还未分离鉴定。研究樟树抗病基因家族不但有助于自身病害防治而且可为其它易受病害侵袭的植物提供优良抗源。在前期研究工作中,江香梅等[19]利用Illumina测序技术完成了樟树5个化学类型的叶组织转录组测序。本研究在此基础上对樟树叶组织转录组数据中NBS-LRR家族基因进行了鉴定和生物信息学分析,并对当中2个RNL类基因进行了全长克隆和表达分析,以便进一步开展樟树中NBS-LRR基因的功能研究。
1 材料与方法
1.1 材料
樟树幼苗感白粉病叶片和正常叶片均于2016年5月取自江西省林业科学院苗圃中,取样材料经液氮中迅速冷冻,-80℃超低温冰箱保存备用。
1.2 方法
1.2.1 基因序列鉴定 基于前期研究获取的樟树叶转录组注释数据,筛选出注释结果为NBS-LRR的Contig序 列, 利 用GENSCAN(http://genes.mit.edu/GENSCAN.html)预测基因,并且翻译为蛋白序列;进一步将得到的蛋白序列分别通过Pfam(http://pf am.xfam.org/)和 SMART(http://smart.embl-heidelberg.de/)验证;去除非全长序列,得到候选的具有全长ORF的NBS-LRR序列。
1.2.2 基因序列的生物信息学分析 利用Muscle软件对樟树NNS-LRR序列进行多重比对分析;利用MEGA7.0.14软件输出系统发育树构建结果;利用在线分析工具ProParam(http://web.expasy.org/protparam/)对樟树NBS-LRR的基本理化性质进行分析;利用MEME寻找樟树NBS-LRR蛋白序列结构域的潜在保守元件。
1.2.3 樟树CcRNL1和CcRNL2基因序列扩增 樟树总RNA的提取使用RNAiso for Polysaccharide-rich Plant Tissue(TaKaRa公司)试剂盒,具体步骤参考说明书,然后用DNase I消化去除基因组DNA。取RNA 样品 2 μg,以 Oligo(dT)Primer为引物进行反转录合成cDNA(PrimeScriptTMII 1stStrand cDNA Synthesis Kit,TaKaRa)。利用 Primer 6.0 对目的基因序列进行引物设计(表1),采用LA Taq保真酶PCR扩增目的基因。PCR反应体系为:cDNA 50 ng,10×LA PCR Buffer II(Mg2+free)5 μL,MgCl2(25 mol/L)5 μL,dNTP Mixture(2.5 mol/L)8 μL,上下游引物(10 μmol/L)各1 μL,加超纯水至终体积50 μL。PCR 扩增程序:94℃ 预变性 3 min;94℃ 变性45 s,56℃ 退火 30 s,72℃ 延伸 2.5 min,35 个循环;72℃ 延伸10 min。1%琼脂糖凝胶电泳检测PCR产物,回收目标大小扩增条带(天根DNA回收试剂盒),回收产物链接到pMD18-T 载体上(TaKaRa),转化到大肠杆菌TOP10中,菌液PCR鉴定阳性克隆,委托上海生工进行目标基因测序。
1.2.4 樟树CcRNL1和CcRNL2基因的表达模式分析 以樟树正常叶和感白粉病叶组织的cDNA为模板,通过实时定量(qRT-PCR)和半定量RT-PCR(SqRT-PCR)检测CcRNL1和CcRNL2基因相对表达量。用Oligo6软件进行RT-PCR引物设计,樟树Actin基因为内参基因(表1)。qRT-PCR 反应体系 :2×SYBR premix Ex TaqTM 12.5 μL,50×ROX Reference DYE 0.5 μL,上下游引物(10 μmol/L)各0.5 μL,cDNA 25 ng,加超纯水至终体积 25 μL。扩增程序 :95℃ 2 min ;95℃ 15 s,60℃ 30 s,40 个循环;添加溶解曲线。在实时荧光定量PCR仪(Bio-RAD,CF×96TMReal-Time System)上进行操作,每个样品重复3次。实验数据统计参照2-△△CT计算相对表达量[20]。SqRT-PCR反应体系为:cDNA 20 ng,10×PCR Buffer II(Mg2+free)2 μL,MgCl2(25 mol/L)1.5 μL,dNTP Mixture(2.5 mol/L)1.3 μL,上下游引物(10 μmol/L)各1 μL,加超纯水至终体积 20 μL。扩增程序 :94℃ 预变性 3 min ;94℃ 变性45 s,56℃ 退火 30 s,72℃ 延伸 30 s,30 个循环。1%琼脂糖凝胶电泳检测PCR产物。

表1 引物序列信息
2 结果
2.1 樟树NBS-LRR基因的鉴定
基于樟树叶组织转录组序列的注释结果,筛选注释为NBS-LRR的序列,利用Pfam和SMART在线网站验证后获得具有NBS保守结构域的NBS-LRR蛋白序列。进一步去除非全长的NBS-LRR序列之后,在樟树转录组数据中共筛选到28个具有全长序列的NBS-LRR基因(表2)。樟树NBS-LRR家族成员的ORF长度为2 118-3 378,编码氨基酸个数为705-1 125,分子量在80.85-128.10 kD,理论等电点为5.35-9.03,不稳定系数为44.05-55.84、为不稳定蛋白;平均疏水性为负值,为亲水性蛋白。樟树NBSLRR蛋白不存在信号肽序列,为非分泌蛋白。蛋白质结构域预测结果显示,樟树NBS-LRR蛋白都具有特征结构域NBS结构域,C末端都具有LRR结构域,其中26条序列N末端存在螺旋卷曲(Coiled-coiled,CC)结构,属于CNL类基因;2条序列N末端存在RPW8结构域,属于RNL类基因;未发现TNL类基因。
2.2 樟树NBS-LRR基因系统进化树及保守结构域分析
进一步对樟树NBS-LRR基因进行聚类分析,利用Mega 7.0.14软件以邻接法构建了樟树NBS-LRR蛋白序列的系统进化树,并且利用在线分析工具MEME鉴定樟树NBS结构域的Motif特征。进化树结果(图1)显示,28个NBS-LRR蛋白被分为5个亚组。樟树的NBS保守结构域包含8个Motif(表3),其中6个Motif为已知元件,分别为P-loop、GLPL、RNBS-A、RNBS-B、RNBS-C和Kinase-2元件,2个为未知元件。
2.3 樟树CcRNL基因的克隆与序列分析
基于对樟树NBS类抗病基因家族的生物信息学分析,本研究挑取N末端存在RPW8结构域的RNL类基因(CL4083.Contig1_All 和 CL4083.Contig2_All)进行克隆。PCR扩增如图2显示,扩增的目的基因ORF条带大小约2 400 bp,分别将其命名为CcRNL1和CcRNL2。经过测序,CcRNL1序列长度为2 460 bp,CcRNL2序列长度为2 466 bp。序列比对结果显示,CcRNL1和CcRNL2序列在氨基酸水平上相似性为71%,除含有NBS基因家族保守结构域中的P-loop,Kinase-2,GLPL motif和C末端的LRR motif外,在N末端具有RPW8 motif,均属于NBS基因家族的RNL类基因(图3)。
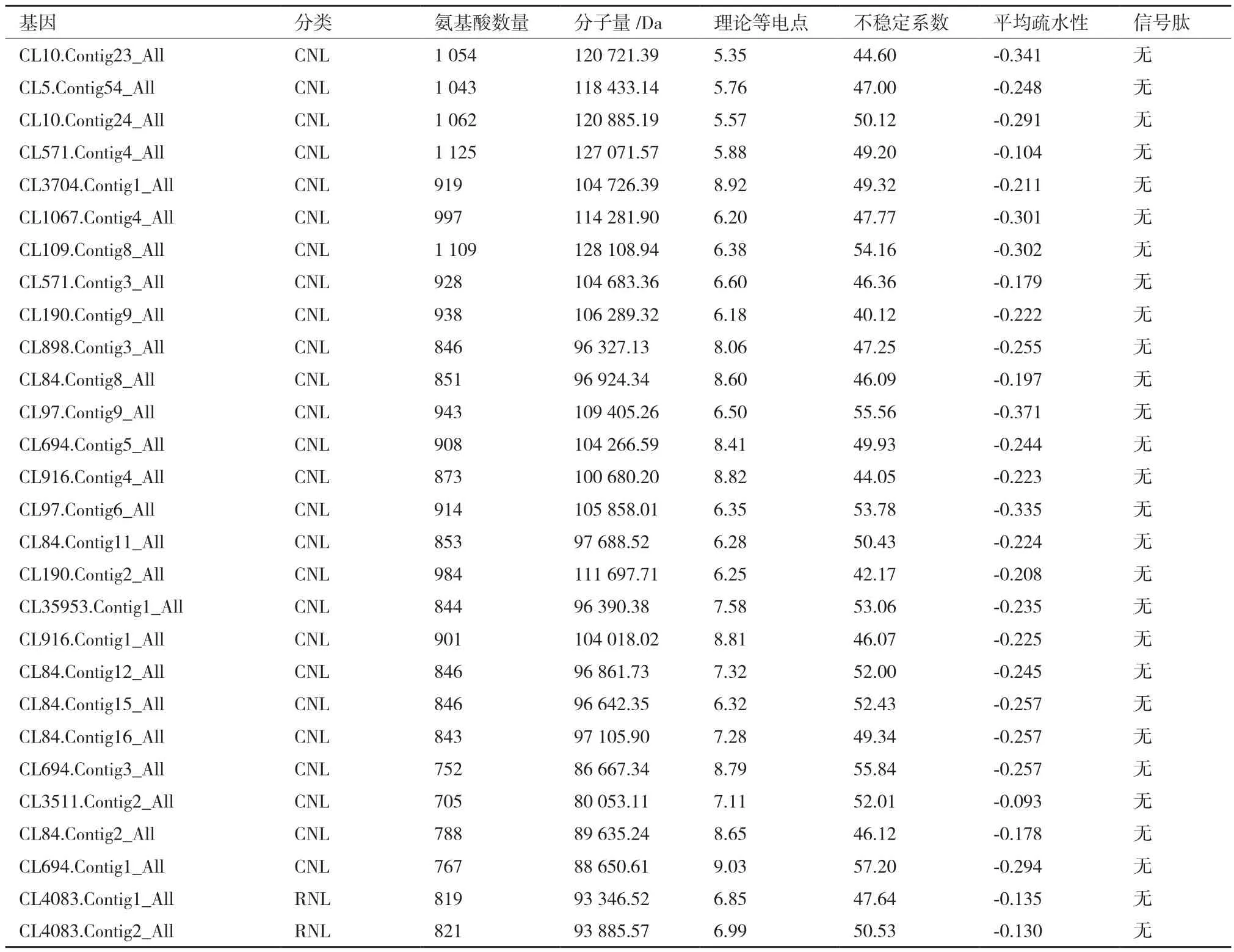
表2 樟树NBS-LRR基因家族生物信息学分析
2.4 樟树CcRNL蛋白的系统进化分析
为研究樟树CcRNL蛋白的系统进化关系,从NCBI数据库下载典型的植物RNL蛋白及近源蛋白氨基酸序列,与本研究获得的CcRNL基因的氨基酸序列进行多重比对,并采用邻接法(NJ)构建系统发育树。结果(图4)显示,RNL蛋白主要分为ADR1亚家族和NRG1亚家族两类,其中樟树CcRNL蛋白与拟南芥ADR1蛋白聚为一类,属于ADR1类亚家族,且与油棕(Elaeis guineensis)的亲缘关系最近。
2.5 CcRNL基因的表达模式分析
为进一步探究CcRNL1和CcRNL2的功能,利用实时定量PCR和半定量PCR对两者在樟树感白粉病叶片和正常叶片中的表达模式进行分析。结果(图5)显示:2个基因在樟树正常叶和感白粉病叶中均有表达,但相对表达水平有所差异,其中CcRNL1在樟树感白粉病叶中的表达明显高于正常叶,而CcRNL2在感白粉病和正常叶中的表达没有明显差异。由此可见CcRNL1可被樟树白粉病菌诱导表达,推测其可能与响应樟树白粉病胁迫反应有关,但还有待于进一步实验验证。
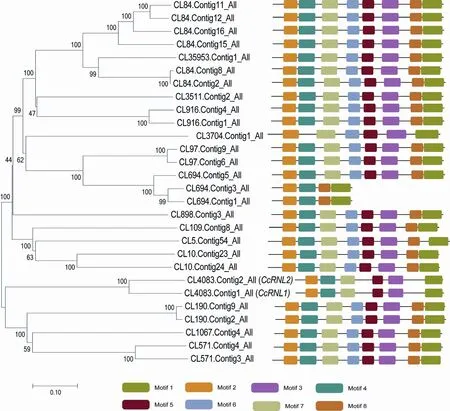
图1 樟树NBS-LRR抗病基因系统发育关系分析
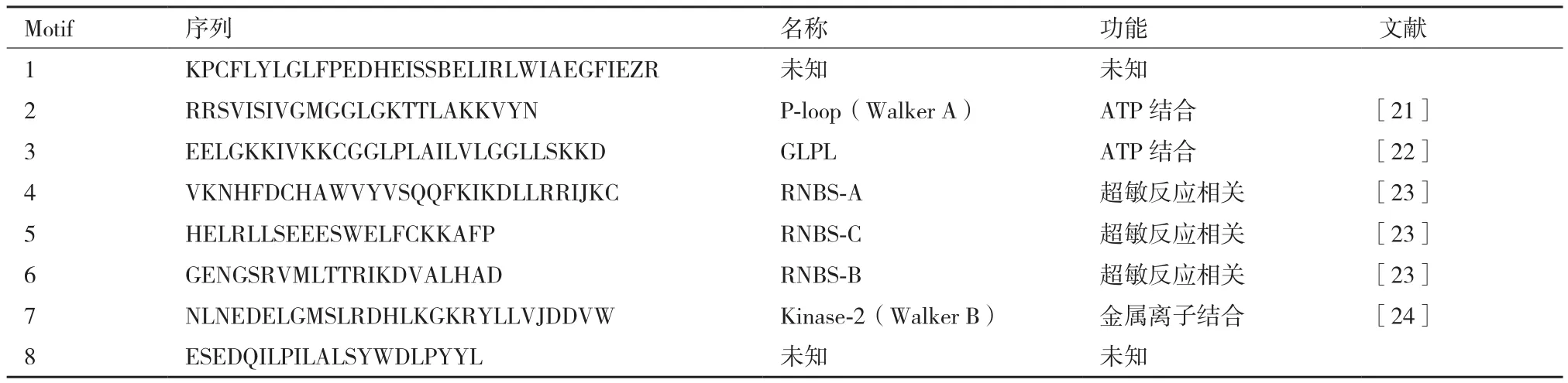
表3 樟树NBS结构域保守元件分析
3 讨论

图2 樟树CcRNL1和CcRNL2基因的扩增图
NBS-LRR基因家族是植物中发现的成员最多的抗病基因家族,也是目前植物抗病性研究的重点和热点。樟树是自然界中较少受病原体侵害的优良抗性种质资源,挖掘樟树R基因尤其具有广谱抗性的R基因对其它易感病植物新抗性种质创制具有重要意义。但目前樟树基因组没有公布,给樟树优良抗性基因的挖掘和功能研究带来了困难。本实验室通过对樟树5种化学类型的叶转录组深度测序,构建了含55 955条樟树unigene序列的数据库。基于此数据库,借助生物信息学的手段对樟树NBS-LRR抗病基因进行了研究。利用叶组织转录组数据一共获得了28个具有完整ORF的NBS-LRR序列,根据氨基酸保守结构域的不同划分为2类,包括26条CNL基因和2条RNL基因,基因数量偏少,可能与组织差异表达有关。另外,叶转录组中未发现TNL类抗病基因,这和水稻[8]、高粱[25]的情况类似,体现了樟树作为基底被子植物系统发育特征,即进化地位介乎于单子叶植物和真核双子叶植物之间。

图3 CcRNL1和CcRNL2蛋白序列比对
目前,已从不同植物中克隆得到NBS-LRR类基因。例如,冯艳芳等[26]克隆了花生AhDNrp基因,该基因属于CNL类抗病基因,在花生根部特异表达,且参与花生黄曲霉抗病反应过程。Peart等[27]克隆了NRG1基因,并且利用病毒诱导沉默技术(VIGS)验证了NRG1基因对烟草花叶病毒(TMV)表现出抗性。本研究克隆了两个樟树RNL类的基因,分别命名为CcRNL1和CcRNL2,序列长度为2 460 bp和2 466 bp,两者在氨基酸水平上相似性71%。结构域分析发现,该类基因除含有典型的NBS结构域和LRR结构域之外,在N端还存在RPW8结构域。RNL类基因划分为ADR1类亚家族和NRG1类亚家族两类,聚类分析结果显示CcRNL属于ADR1类亚家族。研究表明,拟南芥ADR1通过激活SA信号通路,参与白粉病和霜霉病的宿主防御反应[28]。在本研究中,樟树幼苗受白粉病菌侵袭后,CcRNL1基因的表达量相比正常生长个体显著上升,说明该基因可能参与了响应白粉病菌胁迫反应过程,是否通过SA信号通路有待进一步研究。

图4 NBS-LRR抗病基因系统发育关系分析
4 结论

图5 实时定量和半定量检测CcRNL1和CcRNL2白粉病诱导表达
本研究以樟树叶组织转录组数据为基础,利用生物信息学方法在转录组水平上对NBS-LRR基因家族进行了鉴定和分析。在樟树叶组织转录组中鉴定获得了28个NBS-LRR基因,包括26个CNL类基因和2个RNL类基因。在樟树叶组织中克隆了2个RNL类R基因,分别命名为CcRNL1和CcRNL2,聚类分析结果显示二者都属于ADR1类亚家族成员。半定量分析显示CcRNL1受樟树白粉病菌诱导表达,推测其可能参与了响应白粉病菌胁迫反应过程。
[1] Van OG, Mary G, Kasiem MM, et al. Structure-function analysis of the NB-ARC domain of plant disease resistance proteins[J]. J Exp Bot, 2008, 59(6):1383-1397.
[2] Leister RT, Katagiri F. A resistance gene product of the nucleotide binding site-leucine rich repeats class can form a complex with bacterial avirulence proteins in vivo[J]. The Plant Journal, 2000,22(4):345-354.
[3] Xiao S, Ellwood S, Calis O, et al. Broad-spectrum mildew resistance in Arabidopsis thaliana mediated by RPW8[J]. Science, 2001,291(5501):118-120.
[4] Wang W, Devoto A, Terner JG, et al. Expression of the membraneassociated resistance protein RPW8 enhances basal defense against biotrophic pathogens[J]. Mol Plant Microbe Interact, 2007, 20(8):966-976.
[5] Xiao S, Brown S, Patrick E, et al. Enhanced transcription of the Arabidopsis disease resistance genes rpw8. 1 and rpw8. 2 via a salicylic acid-dependent amplification circuit is required for hypersensitive cell death[J]. Plant Cell, 2003, 15(1):33-45.
[6] Moreau M, Westlake T, Zampogna G, et al. The Arabidopsis oligopeptidases TOP1 and TOP2 are salicylic acid targets that modulate SA-mediated signaling and the immune response[J].The Plant Journal, 2013, 76(4):603-614.
[7] Meyers BC, Kozik A, Griego A, et al. Genome-wide analysis of NBSLRR-encoding genes in Arabidopsis[J]. Plant Cell, 2003, 15(4):809-834.
[8] Yang S, Feng Z, Zhang X, et al. Genome-wide investigation on the genetic variations of rice disease resistance genes[J]. Plant Mol Biol, 2006, 62(1-2):181-193.
[9] Kohler A, Rinsldi C, Duplessis S, et al. Genome-wide identification of NBS resistance genes in Populus trichocarpa[J]. Plant Mol Biol, 2008, 66(6):619-636.
[10] Yang S, Zhang X, Yue JX, et al. Recent duplications dominate NBS-encoding gene expansion in two woody species[J]. Mol Genet Genomics, 2008, 280(3):187-198.
[11] Lozano R, Hamblin MT, Prochnik S, et al. Identification and distribution of the NBS-LRR gene family in the Cassava genome[J]. BMC Genomics, 2015, 16(1):360-374.
[12] 郭立中, 韦石泉. 樟树的一种新病害—樟树溃疡病病原菌鉴定[J]. 植物病理学报, 1995, 25(1):28-28.
[13] 邓先琼, 郭立中. 樟树溃疡病病原菌再鉴定[J]. 湖南村业科技, 2005, 32(1):39-40, 43.
[14] Harsh N, Chandra S. A new twig canker disease of Cinnamomum camphora[J]. Indian Journal of Forestry, 2008, 31(2):217-219.
[15] 邓先琼, 郭立中. 樟树溃疡病研究[J]. 湖南农业大学学报:自然科学版, 1998, 24(4):300-304.
[16] 王明生, 吴小芹, 王焱, 等. 上海市樟树病害种类调查及病害特征[J]. 中国森林病虫, 2011, 30(2):24-28.
[17] 高智辉, 宋晓斌, 王云果, 等. 樟树烂皮病调查及病原菌生物学特性研究[J]. 西北林学院学报, 2008, 23(6):122-125.
[18] 葛建明, 张伟, 管丽琴, 等. 香樟炭疽病菌生物学特性及其植物源农药的筛选[J]. 上海交通大学学报:农业科学版,2005, 23(4):401-405.
[19] 江香梅, 伍艳芳, 肖复明, 等. 樟树 5 种化学类型叶片转录组分析[J]. 遗传 , 2014, 36(1):58-68.
[20] Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod[J].Methods, 2001, 25(4):402-408.
[21] Tameling WI, Elzinga SD, Darmin PS, et al. The tomato R gene products I-2 and MI-1 are functional ATP binding proteins with ATPase activity[J]. Plant Cell, 2002, 11:2929-2939.
[22] Leister RT, Ausubel FM, Katagiri F. Molecular recognition of pathogen attack occurs inside of plant cells in plant disease resistance specified by the Arabidopsis genes RPS2 and RPM1[J]. Proc Natl Acad Sci, 1996, 93(26):15497-15502.
[23] Tameling WI, Vossen JH, Albrecht M, et al. Mutations in the NB-ARC domain of I-2 that impair ATP hydrolysis cause autoactivation[J]. Plant Physiology, 2006, 140(4):1233-1245.
[24] Traut TW. The functions and consensus motifs of nine types of peptide segments that form different types of nucleotide binding sites[J]. The FEBS Journal, 1994, 222(1):9-19.
[25] Mace E, Tai S, Innes D, et al. The plasticity of NBS resistance genes in sorghum is driven by multiple evolutionary processes[J].BMC Plant Biology, 2014, 14(1):253-267.
[26] 冯艳芳, 耿丽丽, 韩榕, 等. 花生 NBS-LRR 类基因的克隆及表达特性分析[J]. 生物技术通报, 2016, 32(8):90-95.
[27] Peart JR, Mestre P, Lu R, et al. NRG1, a CC-NB-LRR protein,together with N, a TIR-NB-LRR protein, mediates resistance against tobacco mosaic virus[J]. Curr Biol, 2005, 15(10):968-973.
[28] Grant JJ, Chini A, Basu D, et al. Targeted activation tagging of the Arabidopsis NBS-LRR gene, ADR1, conveys resistance to virulent pathogens[J]. Mol Plant Microbe Interact, 2003, 16(8):669-680.
