杂交三倍体泥鳅GISH技术反应体系的建立及应用
2018-03-07周贺庄子昕查荒源梁雨婷徐其征张蕊林忠乔李雅娟
周贺,庄子昕,查荒源,梁雨婷,徐其征,张蕊,林忠乔,李雅娟
(大连海洋大学辽宁省省级高校海洋生物资源可持续利用重点实验室,农业部北方海水增养殖重点实验室,辽宁大连116023)
泥鳅Misgurnus anguillicaudatus和大鳞副泥鳅Paramisgurnus dabryanus分别隶属于鳅科Cobitidae、花鳅亚科Cobitinae的泥鳅属Misgurnus和副泥鳅属Paramisgurnus[1],是广泛分布于中国各地的两种小型淡水养殖鱼类。泥鳅具有多倍体现象,据报道,除普通二倍体 (2n=50)外,还存在天然三倍体(3n=75)、天然四倍体 (4n=100)和天然六倍体(6n=150) 等[2-5]。 贾光风等[6]、 李雅娟等[7-9]通过对中国长江流域特有的天然四倍体泥鳅的起源及形成机制进行研究,证实了天然四倍体泥鳅是含有四套染色体组的遗传四倍体 (4n=100),且雌雄均能产生正常的2n配子。二倍体和四倍体杂交,理论上可获得100%的三倍体,而三倍体预期不育的鱼类具有生长快、抗病力强等特点,在生产上具有一定的应用价值[10]。杂交三倍体鱼类将杂交育种和三倍体育种相结合,是实现鱼类产业化生产的一条切实可行的途径。因此,2011年笔者利用天然四倍体泥鳅与大鳞副泥鳅杂交获得了杂交三倍体,并对杂交后代进行了基因组原位杂交 (Genomic in situ hybridization,GISH)分析,为杂交后代是三倍体的结论提供了分子细胞学证据[6],但关于天然四倍体泥鳅与大鳞副泥鳅杂交后代的遗传构成目前尚未见报道。本研究中,以大鳞副泥鳅与天然四倍体泥鳅杂交子代及亲本为研究对象,对GISH试验参数进行优化,建立了杂交三倍体泥鳅GISH技术反应体系,并进行GISH分析,旨在为杂交后代的鉴定提供最直观的证据,也为大规模生产三倍体泥鳅提供有效途径。
1 材料与方法
1.1 材料
试验用天然四倍体泥鳅采自湖北省武汉市,大鳞副泥鳅购自大连市农贸市场,均在实验室水箱中培育,培育温度为 (25±1)℃。
1.2 方法
1.2.1 人工催产及授精 选取性腺发育良好的经倍性鉴定后的天然四倍体泥鳅雌、雄各3尾,大鳞副泥鳅雌、雄各3尾,试验开始后,于当日20:00注射绒毛膜促性腺激素 (HCG),雌鱼注射HCG剂量为20~25 IU/g,雄鱼减半。次日8:00,按照杂交组合 (天然四倍体泥鳅♀×大鳞副泥鳅♂,大鳞副泥鳅♀×天然四倍体泥鳅♂)人工采集精液、卵子,干法授精,并将受精卵置于温度为 (25±1)℃的曝气水中培养,每隔1 h挑出死卵,并更换曝气水。
1.2.2 染色体标本制备 受精卵孵化约20 h发育至眼泡期后期,用0.025 g/L的秋水仙素浸泡1~1.5 h(30个胚胎/小瓶)后,用8 g/L的柠檬酸钠溶液 (约5 mL) 低渗20 min,温度为 (25±1)℃。用-20℃预冷的卡诺固定液 (甲醇与冰醋酸的体积比为3∶1,现配现用)处理3次。最后将样品置于卡诺固定液 (-20℃)中保存备用。采用冷滴片法制备单个胚胎染色体。参照Levan等[11]方法进行核型分析。
1.2.3 亲本DNA提取及浓度和质量检测 采用传统的酚仿抽提法提取亲本基因组DNA,并利用Eppendorf D30核酸蛋白测定仪测定其浓度和质量。
1.2.4 探针DNA和封阻DNA片段化 以大鳞副泥鳅DNA为探针DNA,天然四倍体泥鳅DNA为封阻DNA。具体方法如下:将大鳞副泥鳅和天然四倍体泥鳅DNA分装于6支离心管中,每管5 μL。将盛有大鳞副泥鳅DNA的3支离心管置于高压蒸汽灭菌锅中,设置梯度试验温度分别为100、105、110℃,持续时间依次设为1、2、3 min(片段化DNA持续的时间不包括前期升温及后期降温的时间)。将盛有天然四倍体泥鳅DNA的3支离心管置于高压蒸汽灭菌锅中,设置试验温度分别为110、115、120℃,持续时间依次设定为5、10、15 min(片段化DNA持续的时间不包括前期升温和后期降温的时间)。随后用琼脂糖凝胶进行电泳检测,分析不同温度及持续时间条件下DNA片段长度。
1.2.5 探针DNA与封阻DNA浓度比例 (probe/blocking DNA radio,P/B)的优化 P/B比设为1 ∶10、 1∶20、 1∶25、 1∶50。
1.2.6 GISH分析
1.2.6.1 探针DNA标记 对经检测合格的大鳞副泥鳅DNA采用优化后处理温度为105℃、处理时间为2 min进行探针DNA片段化。利用缺口平移试剂盒 (Roche 11745824910)在15℃低温水浴锅中进行标记,标记时间为120 min。标记后放入烘箱 (65℃)中,处理时间20 min,使酶失活。
1.2.6.2 封阻DNA的片段化 采用优化后处理温度为110℃、处理时间为10 min,对检测合格的天然四倍体泥鳅全基因组DNA进行封阻DNA片段化。
1.2.6.3 基因组原位杂交
(1)杂交液制备。将标记后的探针DNA与片段化后的封阻DNA按照筛选出的最佳混合比,即1∶25进行混合,加入体积分数为100%酒精60 μL和3 mol/L醋酸铵3 μL沉淀后于-30℃下静置30 min,以15 000 r/min离心30 min,去掉上清液;加入150 μL体积分数为70%的酒精再次以15 000 r/rim离心3 min,去掉上清液;室温下晾干后加入50 μL杂交缓冲液 (50%硫酸葡聚糖 10 μL+20×SSC 1.83 μL+100%去离子甲酰胺 27.5 μL+ddH2O 10.67 μL)。
(2)探针DNA变性。于83℃下处理7 min,迅速移到冰水中10 min以上 (变性过程中的温度与时间控制需要精确)。
(3)染色体标本变性。取出老化完成的染色体标本,放入提前65℃下预热的变性液 (pH为7.0的70%甲酰胺/2×SSC)中3 min,迅速移入-20℃预冷的体积分数为70%的酒精中7 min,再依次移入体积分数为90%的酒精中7 min、体积分数为100%的酒精中10 min。
(4)杂交。室温下晾干染色体标本,将50 μL杂交液全部滴加在事先标记好的染色体区域,覆盖20 mm×20 mm的封口膜,放入2×SSC湿盒,于37℃培养箱中孵育18 h以上。
1.2.6.4 漂洗、对比染色与观察 具体步骤如下:
(1)洗脱。揭掉封口膜,染色体标本放入50%甲酰胺/2×SSC溶液中,42℃下手动漂洗20 min,再放入1×SSC溶液中42℃下手动漂洗7 min,重复3次,于2×SSC溶液中室温下静置20 s。
(2)滴加100 μL染色封阻液 (0.05 g BSA+1 mL 20×SSC+10 μL Tween20), 盖上封口膜, 放在2×SSC湿盒中,于37℃下恒温避光孵育30 min。
(3)FITC荧光信号检出。去除封口膜,加入100 μL Avidin-FITC 液 (PBS+1%BSA 与 Alexa-Fluor488的体积比为200∶4),盖上封口膜,于37℃下避光温育80 min。
(4)漂洗。揭掉封口膜,放在4×SSC/0.1%Tween20溶液中42℃下避光漂洗5 min,重复3次,再于4×SSC溶液中避光漂洗10 min。
(5)信号放大。滴加 100 μL信号增幅液(PBS+1%BSA与Biotinylated Anti-avidin D的体积比为125∶0.5),盖上新的封口膜,放在2×SSC湿盒中,于37℃下避光温育45 min。
(6)漂洗。步骤同本节中 (4)。
(7)加入100 μL Avidin-FITC液,于37℃下避光培养1 h。
(8)漂洗。步骤同本节中 (4)。
(9)对比染色。依次将染色体标本放入盛有体积分数为70%、90%、100%酒精的染色缸中常温脱水各 5 min, 再加入 50 μL 2.5 μg/mL的 DAPI/antifade溶液,用指甲油封片。水平放置于冰箱(-4℃)中避光保存。次日在荧光显微镜 (Leica DM2000)下观察并拍照。
2 结果与分析
2.1 DNA电泳
提取的亲本DNA的OD260nm/OD280nm值为1.8~2.0,符合试验要求。如图1所示,扩增出的条带清晰、完整。

图1 基因组DNA琼脂糖电泳结果Fig.1 Agarose gel electrophoresis of genomic DNA
2.2 GISH体系的建立
2.2.1 探针DNA的片段化 本研究中以大鳞副泥鳅为探针,片段化结果如图2所示。105℃高压灭菌2 min时,探针DNA片段显示亮度较高的弥散状条带,长度为500~1000 bp,保证了其可以在染色体DNA中穿梭,使之定位在同源序列处并与之杂交 (图2-A);而110℃高压灭菌3 min时,探针DNA片段长度位于500 bp以下,片段长度太短易导致特异性弱且信号不强 (图2-B)。
2.2.2 封阻DNA的片段化 本研究中以天然四倍体泥鳅为封阻DNA,片段化结果如图3所示,120℃高压灭菌5 min处理的封阻DNA片段长度仅在100 bp左右 (图3-A);110℃高压灭菌10 min处理的封阻DNA片段为100~500 bp时显示亮度较高的弥散状条带,符合要求 (图3-B);120℃高压灭菌15 min处理的DNA片段长度在100 bp以下,片段长度均太短,不符合要求 (图3-C)。

图2 探针DNA片段化电泳检测结果Fig.2 Electrophoresis of probe DNA fragmentation
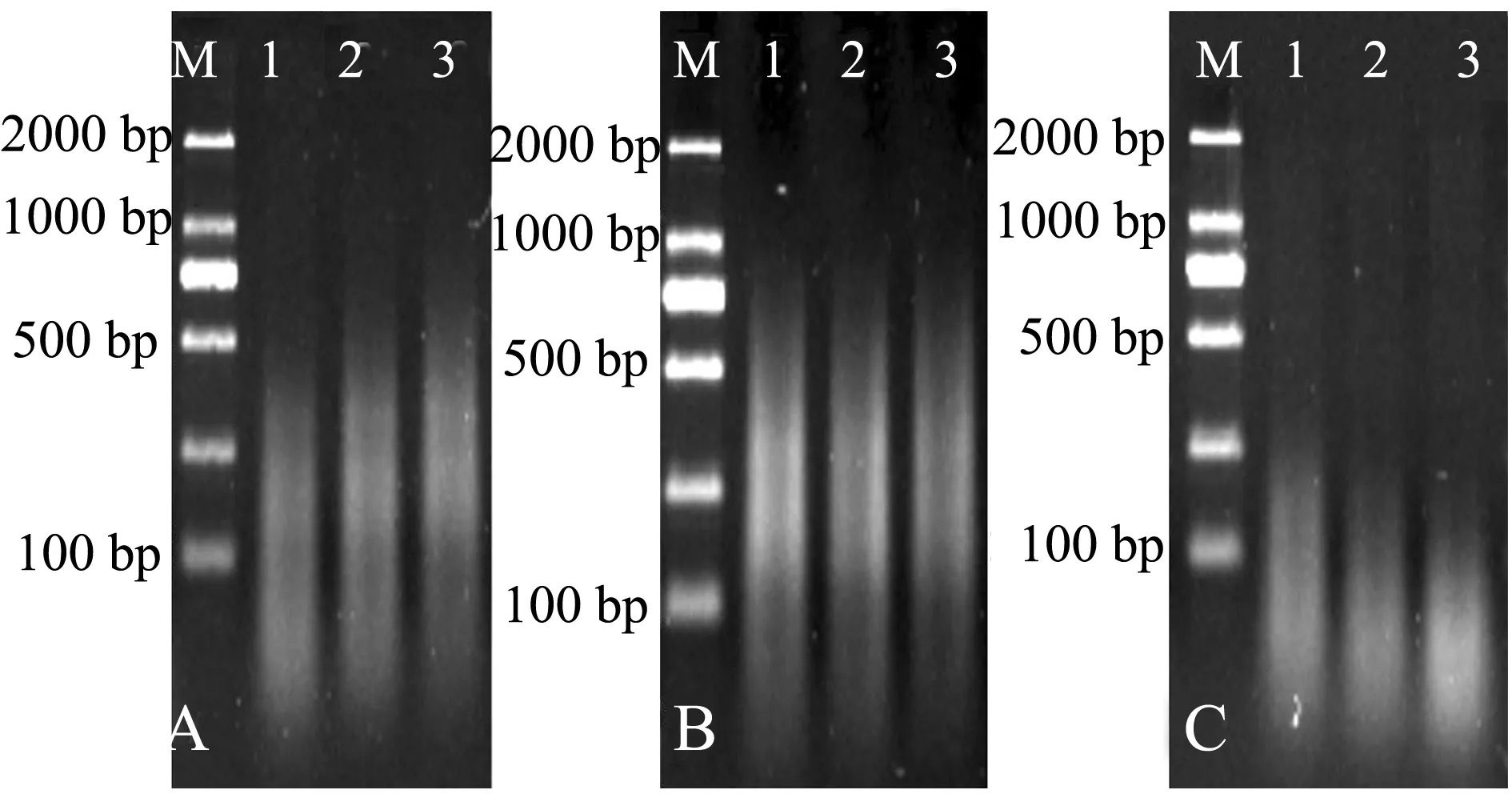
图3 封阻DNA片段化电泳检测结果Fig.3 Electrophoresis of blocking DNA fragmentation
2.2.3 探针DNA与封阻DNA浓度比例 (P/B)
从图4可见:当 P/B值为1∶10(图4-A)和1∶20(图4-B)时,杂交信号不清晰;当P/B值为1∶25(图4-C) 和1∶50(图4-D) 时, 均可以得到较好的杂交信号,但P/B值为1∶25时效果最好。
2.3 天然四倍体泥鳅与大鳞副泥鳅正、反杂交子代的GISH分析
利用已建立的GISH技术反应体系对天然四倍体泥鳅与大鳞副泥鳅的正、反杂交子代进行GISH分析,结果如图5所示。天然四倍体泥鳅♀×大鳞副泥鳅♂的杂交子代,存在有杂交信号的染色体24条,来源于亲本大鳞副泥鳅,且杂交信号均位于着丝点处,而无杂交信号染色体50条,来源于亲本天然四倍体泥鳅 (图5-A、B);大鳞副泥鳅♀×天然四倍体泥鳅♂杂交子代也存在有杂交信号的染色体24条,来源于亲本大鳞副泥鳅,且杂交信号均位于着丝点处,而无杂交信号染色体也为50条,来源于亲本天然四倍体泥鳅 (图5-C、D)。
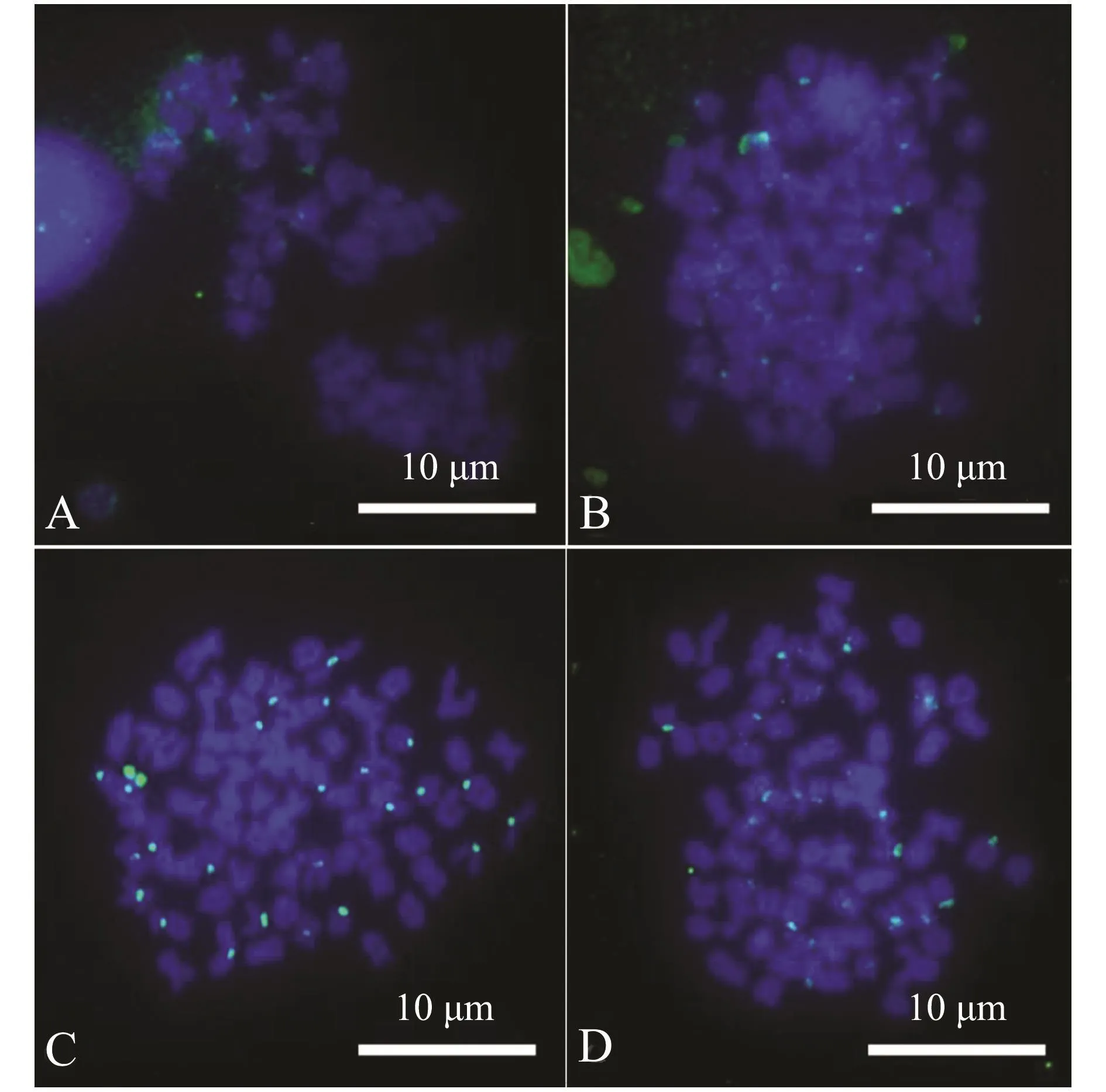
图4 大鳞副泥鳅♀×天然四倍体泥鳅♂后代P/B试验结果Fig.4 GISH of chromosomes in embryos from loach Paramisgurnus dabryanus♀×natural tetraploid loach♂
3 讨论
3.1 影响基因组原位杂交的因素
基因组原位杂交 (GISH)是以基因组DNA为探针与染色体标本进行的原位杂交,是用于检测目标DNA的一种技术,也是研究杂交物种染色体构成及进化的有效方法[12]。GISH试验有许多影响因素,但探针DNA和封阻DNA的制备及使用浓度是决定GISH成功与否的关键因素。GISH中无论是探针还是封阻均须通过染色体的立体结构完成杂交,探针DNA过长则会影响探针在杂交过程中无法正常移动到染色体需要杂交的位置上,导致杂交信号缺少或不清晰等问题;探针DNA长度过短,则会导致杂交信号无代表性,杂交信号过多且不清晰。因此,优化探针长度,是研究基因组原位杂交的一项必要技术,完善和发展该技术可显著提升GISH技术的成功率和分辨率。
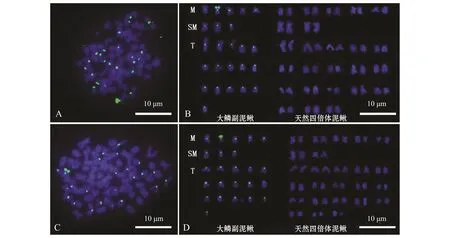
图5 杂交三倍体泥鳅GISH分析Fig.5 GISH of chromosomes in embryos from hybrid triploid loach
本试验中利用高温解旋原理,使基因组DNA片段化,结果显示,基因组DNA在105℃、持续时间2 min时,片段化后探针DNA片段长度为500~1000 bp,封阻DNA在温度为110℃、持续时间10 min时,片段长度为100~500 bp,说明用高压灭菌锅对基因组DNA进行片段化的方法可行。加入封阻DNA后主要有两方面的作用:首先,由于同源序列杂交,阻止探针DNA与非特异性位点杂交或非目标DNA杂交,封闭假阳性信号;其次,封阻DNA可阻止探针DNA与相似DNA杂交,区分目标DNA和非目标DNA[13]。探针DNA与封阻DNA浓度比例 (P/B)对杂交结果的清晰度及杂交程度均有决定性影响,是决定整个杂交效果的另一个关键因素。当封阻DNA过量时,封阻DNA与目标DNA杂交,只有少量的目标DNA与探针DNA杂交会产生杂交信号。Rampin等[14]曾经对Squalius alburnoides的染色体进行GISH分析,使用的P/B值为1∶20至1∶40,但其使用的探针DNA量只有100 ng,由于探针DNA的使用量过低,不得不减小杂交面积以得到较为清晰的杂交信号。本研究中,探针DNA与封阻DNA浓度比 (P/B)结果显示,当P/B=1∶25或1∶50时均可以获得明亮的杂交信号,P/B值过高或过低均使杂交信号不明显。另外,本试验中建立的GISH技术体系有两个局限性:其一,所有的特异性杂交信号均位于染色体着丝点处 (除一条染色体端部额外又有一簇杂交信号外),说明只有染色体着丝点处的序列与探针DNA产生了特异性反应,无法捕捉到染色体体臂间的交换及染色体变异等,其原因还不清楚;其二,建立的GISH技术体系现只用于泥鳅不同属间杂种,对于亲缘关系更近的种间杂种,该体系试验结果不能有效分开亲本染色体组,其原因主要是两种泥鳅亲缘关系太近。
3.2 基因组原位杂交在水产动物杂交育种上的应用
杂交育种技术是新品种开发和品种选育的有效途径和方法。在杂交育种过程中,杂交后代的真实性鉴定是必要的,因为只有知道杂交后代是否真正含有亲本血缘,才能确定杂交利用的效果和杂交方法可行性。GISH技术可以从分子水平上对染色体直接进行原位定位,明确了解杂种的核基因组组成,GISH技术已被广泛应用于杂种外缘染色体的检测[15-16],且在水产动物的研究中也有应用。毕克[17]运用GISH技术对华贵栉孔扇贝♀×栉孔扇贝♂杂交子代的染色体组成进行了分析;吕振明[18]对栉孔扇贝和虾夷扇贝杂交后代的染色体来源进行了GISH技术鉴定;Rita对栉水虱Asellus aquaticus进行了性染色体的GISH分析,并且捕捉到了来自Y染色体的变异[19]。综上所述,GISH技术在水产物种后代遗传学分析中同样具有独特的优势。本研究中,以大鳞副泥鳅DNA为探针,自然四倍体泥鳅DNA为封阻,对大鳞副泥鳅与天然四倍体泥鳅正、反杂交所得的杂交三倍体泥鳅进行了原位杂交,在染色体中期可清楚地检测到24条染色体具有荧光信号,来源于大鳞副泥鳅;另外50条染色体无杂交信号,来源于四倍体泥鳅。原位杂交结果清晰显示,后代分别继承了父、母本的染色体,为真正的杂交三倍体。因此,本试验表明,泥鳅属间杂种后代的鉴定可用GISH的方法加以有效检测。
本研究结果证明了大鳞副泥鳅与四倍体泥鳅属间杂交的真实性,该研究结果不仅为杂交三倍体泥鳅后代的鉴定提供了最直观的证据,也为中国天然四倍体泥鳅是含有四套染色体组的遗传四倍体并能产生2n的配子提供了分子细胞遗传学基础数据。因此,本试验结果对泥鳅三倍体育种研究具有一定的指导意义。
[1] 陈景星.中国沙鳅亚科鱼类系统分类的研究[J].动物学研究,1980,1(1):3-26.
[2] 李雅娟,印傑,王嘉博,等.中国におけるドジョウ倍数体の分布に関する研究[J].日本水産学会誌,2008,74(2):177-182.
[3] 周小云.湖北省多倍体泥鳅分布格局及泥鳅育种基础研究[D].武汉:华中农业大学,2009.
[4] 李雅娟,高敏,钱聪,等.德国镜鲤和不同倍性泥鳅银染核型的比较研究[J].东北农业大学学报,2012,43(12):127-133.
[5] Li Yajuan,Yu Zhuo,Zhang Mingzhao,et al.The origin of natural tetraploid loach Misgurnus anguillicaudatus(Teleostei:Cobitidae)inferred from meiotic chromosome configurations[J].Genetica,2011,139(6):805-811.
[6] 贾光风,李雅娟,钱聪,等.大鳞副泥鳅(2n♀)×泥鳅(4n♂)杂种后代染色体带型及FISH分析[J].东北农业大学学报,2011,42(9):72-75.
[7] 李雅娟,钱聪,印杰,等.不同倍性泥鳅杂交后代染色体数目组成的研究[J].大连海洋大学学报,2012,27(4):326-332.
[8] 李雅娟,田萍萍,李莹,等.中国洪湖不同倍性泥鳅的染色体组型及形态特征比较分析[J].大连水产学院学报,2009,24(3):236-241.
[9] 李雅娟,张明昭,于卓,等.黑龙江泥鳅、北方泥鳅和泥鳅的形态差异分析[J].大连海洋大学学报,2010,25(5):397-401.
[10] 青木宙,隆島史夫,平野哲也.魚類のDNA:分子遺伝学的アプローチ[M].東京:恒星社厚生閣,1997.
[11] Levan A,Fredga K,Sandberg A A.Nomenclature for centromeric position on chromosomes[J].Hereditas,1964,52(2):201-220.
[12] 王卫军,杨建敏,刘志鸿,等.异源精子诱导栉孔扇贝(Chlamys farreri)雌核发育二倍体早期胚胎的细胞学研究及GISH鉴定[J].海洋与湖沼,2009,40(3):325-329.
[13] Howell E C,Kearsey M J,Jones G H,et al.A and C genome distinction and chromosome identification in Brassica napus by sequential fluorescence in situ hybridization and genomic in situ hybridization[J].Genetics,2008,180(4):1849-1857.
[14] Rampin M,Bi K,Bogart J P,et al.Identifying parental chromosomes and genomic rearrangements in animal hybrid complexes of species with small genome size using genomic in situ hybridization(GISH)[J].Comparative Cytogenetics,2012,6(3):287-300.
[15] Schwarzacher T,Leitch A R,Bennett M D,et al.In situ localization of parental genomes in a wide hybrid[J].Annals of Botany,1989,64(3):315-324.
[16] Bailey J P,Bennett M D,Bennett M D,et al.Genomic in situ hybridization identifies parental chromosomes in the wild grass hybrid × Festulpia hubbardii[J].Heredity,1993,71(4):413-420.
[17] 毕克.杂交扇贝(华贵栉孔扇贝Chlamys nobilis♀×栉孔扇贝C.farreri♂)的分子细胞遗传学分析[D].青岛:中国海洋大学,2004.
[18] 吕振明.栉孔扇贝和虾夷扇贝杂交过程中的细胞与分子遗传学分析[D].青岛:中国海洋大学,2006.
[19] Barzotti R,Pelliccia F,Rocchi A.Sex chromosome differentiation revealed by genomic in-situ hybridization[J].Chromosome Research,2000,8(6):459-464.
