稗属植物种质资源及应用价值研究进展
2018-03-06张婉茹吴姝菊王晓萍哈尔滨师范大学生命科学与技术学院黑龙江哈尔滨150025
张婉茹,吴姝菊,王晓萍(哈尔滨师范大学生命科学与技术学院,黑龙江 哈尔滨 150025)
稗属(Echinochloa)植物也称稗子、稗草,隶属于禾本科(Gramineae),是一年生或多年生的草本植物[1]。全世界大约有35种,只有两个栽培种(E.frumentacea和E.utilis),其余均为野生种[2]。稗属植物繁殖力强、生态适应性广,大多数为田间杂草,因其含有丰富的营养物质、草质柔软、抗逆性强、适口性好,近年来多被作为牧草或饲料来研究利用。另外,稗属植物还蕴含丰富的基因资源,其巨大的应用价值正在被逐渐发掘和利用。为此,总结稗属植物种质资源研究和在饲草、优良基因等领域开发利用的进展,并对其日后的发展方向进行初步展望,旨在为稗属植物的进一步研究和利用提供一定的理论基础。
1 稗属植物种质资源及分布
目前,全世界稗属植物有50余种(包含亚种和变种),最早在欧洲和亚洲印度发现,现已广泛分布在全球热带和温带地区[3]。《中国植物志》最新英文修订版中记载我国稗属植物有8个种,6个变种(表1),在全国各地均有大量分布,其中北方地区最多[4]。稗属植物生态适应性强,在沼泽、沟渠旁、低洼荒地以及稻田中均有大面积分布。大部分稗属植物既喜潮湿又抗旱,不仅可以生活在水中与水稻(Oryzasativa)争夺营养,还能在旱地生长危害大豆(Glycinemax)、玉米(Zeamays)、棉花(Gossypium)和小麦(Triticumaestivum)等农作物[5]。
2 稗属植物种质资源研究进展
2.1 稗属植物系统进化研究进展
蔡联炳和郭延平[6]研究了禾本科植物叶片表皮结构细胞主要类型的演化和系统分类,将稗属划分到黍超族黍亚科。许瑾等[7]对稻属及其近缘种禾本科植物的叶绿体psbA-trnH基因进行扩增和测序,并分析它们的系统进化关系和序列差异,结果发现稗草和玉米、稻属的亲缘关系很近。一些重复DNA顺序具有基因组特异性,用它们作探针可以分析同属或同科物种的起源关系和亲缘关系。利用水稻基因组中克隆出的一些重复序列作探针,研究它们在禾本科植物中的分布情况和杂交多态性表现,在研究pRRD9重复顺序时发现稗草(E.crusgalli)与狗尾草(Setariaviridis)的亲缘关系较近[8]。
Yabuno[9-11]通过细胞遗传学研究认为六倍体野生稗E.crusgalli和E.colona分别是栽培稗E.utilis和E.frumentacea的祖先,四倍体稻稗(E.oryzicola)(2n=4x=36)作为稻田中最具代表性的杂草,被认为是六倍体野生稗和栽培稗(2n=6x=54)的基因组供体。Hilu[12]采用随机扩增多态性DNA标记技术对Yabuno提出的稗属植物系统发育进行验证,通过数据分析发现栽培稗E.utilis和E.frumentacea在遗传上有很大的区别,但它们分别和E.crusgalli、E.colona划分到了一组,四倍体稻稗则与E.utilis、E.crusgalli表现出了很近的遗传关系,该结果与细胞学结果基本一致。Aoki和Yamaguchi[13]以水稻基因sh4代表稗属植物同源基因组的复制,研究了亚洲稗属植物的遗传分化关系,结果证实了亚洲异源六倍体的稗与稻稗的两个基因组相同,而多年生四倍体E.stagnina有一个基因组与稻稗相同,另一个基因组来源不明。研究还发现,西半球的孔雀稗(E.cruspavonis)与亚洲的稗和稻稗具有较近的亲缘关系,而光头稗(E.colona)和稗属其他各种的亲缘关系都比较远[13]。Aoki和Yamaguchi[14]利用核DNA转录间隔区(ITS)和叶绿体基因序列来研究稗和稻稗的亲缘关系,ITS和叶绿体DNA结果都表明生长在西半球的稗与欧亚地区的稗在遗传上有明显差别,但与美国的孔雀稗和海岸稗(E.walteri)表现出密切关系,nrDNA ITS结果表明,欧亚的稗和稻稗之间没有差别,但二者的叶绿体DNA序列却有明显差别,该研究结果证明稻稗是稗的父本。
2.2 稗属植物遗传多样性研究进展
2.2.1形态学研究进展 形态学标记是指植株生长发育过程中,肉眼可见的外部形态特征,如植株高度、叶片形状、小穗长度等,是个体基因型的外在表现[15]。依据形态性状来研究植物的遗传变异是最直接最简便的方法,也是目前稗属植物分类鉴定的主要依据。花序、小穗、芒等形态特征是稗属植物鉴定中最重要的形态参数[16],但是,由于其生存范围广,同种类植物在不同的生存环境下,其形态特征如植株高度、小穗颜色、芒长等均有较大的差异。稗属植物的形态性状具有高度变异性,这使得研究者们对其划分界限产生了不同的观点,因此,稗属植物的分类鉴定依然没有统一的标准。
Ruiz-Santaella等[17]在观察稗草形态性状时发现,小穗芒长作为鉴别稗属植物重要特征之一,也会受环境条件影响而发生变化。Gupta等[18]从印度不同生态环境中收集了194份湖南稗子(E.frumentacea),并根据收集地域将这些种质分为5组,发现不同地理环境的稗属植物形态性状差异显著。对不同来源的34种稗属植物进行形态差异和遗传变异的比较发现,各品种之间的形态差异和遗传变异性都比较大,这些变异可能是由于农业耕作方式、作物生长特性和地理环境差异以及除草剂的施用引起的[19]。在我国重要农业区稻田稗草的多样性研究中,依据21项形态性状,从206份稗属植物中鉴别出8个类群,分析发现这些类群在叶长、叶宽、小穗芒长、小穗长度等9个性状方面有显著差异[20]。对一年生牧草稗的遗传多样性分析发现,其形态性状遗传变异显著,说明稗的遗传多样性丰富,有利于优质育种材料的选择[21]。
2.2.2细胞学标记 染色体是细胞内遗传物质的主要载体和遗传信息的携带者。染色体变异主要表现为染色体数目变异和结构变异,具体表现在核型(染色体数目、大小、有无随体、着丝点位置等)及带型(C带、N带、G带等)两个方面[22]。染色体数目和结构不仅是鉴定植物品种最重要、最准确的指标,也是稗属植物重要的细胞学特征。
Yabuno[9-11]在研究日本稗属植物染色体时发现一年生野生稗草E.oryzicola、E.hispidula的染色数为2n=36,E.crusgallivar.caudata、E.crusgallivar.praticola、E.crusgallivar.formosensis、E.colonum、E.oryzoides和栽培种E.frumentacea、E.utilis的染色体数则为2n=54(表2)。冯久焕和张廷璧[23]首次对国内稗属植物的细胞学进行了研究,发现9种(含变种)试验材料的染色体数目只有两种类型2n=36和2n=54,其中无芒稗、孔雀稗、旱稗的染色体数目是2n=36,稗、西来稗、长芒稗、硬稃稗和水田稗的是2n=54;经过核型分析发现无芒稗、旱稗、稗、硬稃稗和长芒稗的核型是2A型,西来稗、孔雀稗和水田稗的核型是2B型;此外,他们还发现各品种间的核型构成是不同的,尤其是随体染色体数目以及在核型中的顺序和位置,这对于稗属植物的分类鉴定具有重要意义。吴姝菊等[24-25]对黑龙江省4个品种的寒地稗草进行细胞学分析发现,它们的染色体数都是2n=54,核型类型都是2B型,并且这4种稗草的染色体数目与冯久焕和张廷壁[23]报道的稗相同,且染色体形态特征和结构也相似,但核型不同,推测这4种寒地稗草是稗的变种。目前,对稗属植物染色体数目及核型的研究相对较少,而对其带型的分析则未见报道。因为稗属植物的染色体很小,属于小染色体,而且相邻的染色体对之间的长度差别不明显,所以,很难把不同的染色体对区别开。核型分析有利于人们从细胞水平认识稗属植物的演化、种群间亲缘关系、遗传变异等,为稗属植物的分类提供理论基础。
2.2.3生化标记 生化标记主要包含同工酶标记和贮藏蛋白标记。蛋白质是基因表达的直接产物,电泳后产生的蛋白质条带可以在生化水平上反映出植物的遗传变异[26]。Asíns和Barreda[27]通过对伊比利亚半岛东部的35种田间稗草同工酶的研究发现,其群体内具有很高的遗传一致性,并结合形态学,认为E.hispidula和E.oryzoides是E.crusgalli的种下类群。Nakayama等[28]利用同工酶特征,对稗属植物进行分类鉴定,认为稗、紫穗稗和水田稗为同一个种,该结果与Yamaguchi等[29]测定的基因序列和RAPD扩增结果一致。蛋白质组成是由基因表达决定的,几乎不会受到外界环境因素的影响,其组成成分的不同能够反映出基因型的差异。因此,生化标记可以在一定程度上反映稗属植物的遗传多样性,而国内有关这方面的研究还属空白。
2.2.4分子标记 分子标记作为直接分析核苷酸序列差异的研究方法,和其他遗传标记相比较,具有数量庞大,不受植物体发育时期、检测部位、季节及环境的影响,多态性高,检测方法简单、快速等优势,目前常用的分子标记有十多种[30]。
Yasuda等[31]把PCR-RFLP技术应用在对日本稻稗和稗的分子鉴定中,利用该技术成功地把这两个种区别开,另外在这两种稗草的采集地区内没有发现种内变异。Altop和Mennan[19]通过形态标记和RAPD分子标记研究了土耳其不同地理环境中的稗属植物的遗传多样性,发现不同种在形态特征方面有很大的差异,如发芽速度、开花时间、叶面积、植株高度、小穗长度等;另外,用7个RAPD引物扩增出了55个条带,其中多态性条带所占百分比为74.54%,这说明了不同地理环境中的稗属植物在表型和基因型上都有着很大的差别。其他研究者同样使用RAPD分子标记证实了稗属植物种内变异丰富[12,32]。Kaya等[33]利用4个AFLP引物对对稗属植物的28个个体进行研究,平均每个引物对扩增出41.5个条带,平均多态信息含量(PIC)为0.26,在种内和种间都检测到了高水平的遗传变异。Danquah等[34]使用AFLP和SSR标记鉴定24种稗属植物,6个AFLP引物共扩增出909个条带,其中有775个条带呈现出多态性,占总扩增条带的85.3%。而微卫星的基因多样性为0.28~0.72,这与AFLP结果相似。并利用AFLP和SSR标记将光头稗从稗和孔雀稗中区分出来,该研究证明了AFLP和SSR不仅可以用于鉴别基因型,也可以帮助解决稗属的分类问题。刘德好等[35-36]通过正交试验法优化了稗属植物的ISSR-PCR反应体系,利用该反应体系筛选出9对条带清晰、重复性好、多态性丰富的ISSR引物,并利用这9对引物扩增出137条DNA谱带,其中有136个条带具有多态性,多态位点百分比高达99.3%,这表明稗属植物的种间多态性极其丰富。通过对ISSR标记结果进行聚类分析,最后把53份样本划分成九大类群。付菲[37]利用流式细胞术和分子标记技术对稗属植物进行研究,49份供试材料被划分成水田稗和稗两大类,而SNP标记又把稗分成4类,即稗原变种、细叶旱稗、光头稗和另一未知稗类。
3 应用价值研究
3.1 经济价值
栽培稗很早就被种植利用,考古学研究发现,早在4 000年以前,日本人就开始种植紫穗稗作为重要的粮食作物[2]。稗草对环境的适应能力很强,当周围环境恶化而不利于其生长时,稗草会抑制自身的营养生长,促进生殖生长,加快种子成熟[38]。稗草的繁殖能力也很强,一株成熟的稗草可产生3 000粒以上的种子,且种子在旱田中可存活2~3年,在水田中可存活5~7年,期间只要达到萌发条件便可出苗生长[39]。在高寒地区农牧交错带灌溉条件下种植稗草,其鲜草产量一般在50~100 t·hm-2,干草产量在20 t·hm-2左右,籽实产量可达5 t·hm-2[40]。稗草不仅草产量高,而且营养丰富,粗蛋白含量大部分高于稻谷(表3),纤维素含量也高,其鲜草可用于饲喂牛、羊、兔、鱼。稗草籽粒的营养也很丰富,其粗蛋白和几种主要的氨基酸含量与玉米、高粱(Sorghumbicolor)相似(表4),是很好的精饲料或酿酒的原料[41]。另外,稗草的茎叶纤维可作造纸原料,根和幼苗可入药,有止血功效,颖果还可以用来制糖和酿酒[5]。正是由于稗草的抗逆性强、适口性好、营养价值高,近年来逐渐引起许多国家学者的重视,认为稗草是发展绿色畜产品的新型优质饲草。在澳大利亚和新西兰,经常把紫穗稗作为一年生牧草种植,它可以弥补多年生牧草生产和品质的不足[2]。易凤银和彭科林[41]从中国农业科学院引入5个优良品种,并连续3年对其生育特性、经济性状和栽培技术进行研究,最终选育出3个适应性强、生长旺盛、草产量和产籽量高、营养丰富的品种,并认为这3个品种适宜在南方大面积推广种植。古忠玉[42]在黑龙江地区收集了40份稗子,并在合江和牡丹江进行试种,经鉴定选出4个优良品种,拉林小粒稗、绥化锁链稗、肇东粳稗和哈尔滨大散稗,其中绥化锁链稗和肇东粳稗属于粮草兼用类型。

表3 稗草和稻谷营养成分的对比[41,43]Table 3 Comparison of nutrient compositions of Echincohloa and paddy[41,43]
营养成分计算以干物质为基础。
Nutrient compositions are calculated based on dry matter.
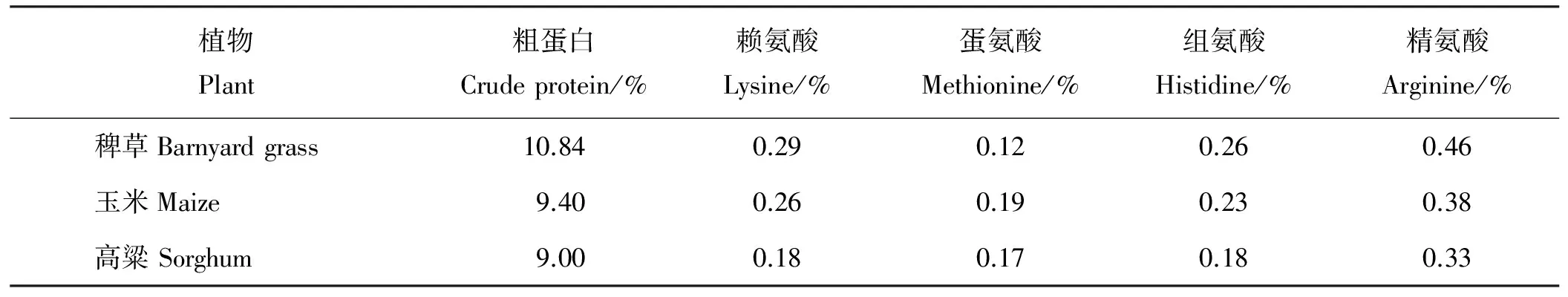
表4 稗草、玉米和高粱籽粒的营养成分的对比[41,43]Table 4 Comparison of nutrient components of barnyard grass, maize and sorghum grain[41,43]
3.2 优良基因的利用
稗属植物是C4植物,其光合效率明显高于C3植物,甚至比其他C4农作物还高,如高粱、玉米、甘蔗(Saccharumofficinarum)等[44]。为提高C3农作物的光合效率,科研人员正试图把稗属植物的C4光合酶基因导入水稻、小麦、烟草(Nicotianatabacum)等C3农作物中。张桂芳等[45]和王金明等[46]已经克隆出稗草的磷酸烯醇式丙酮酸羧化酶基因(ppc)和家稗的丙酮酸磷酸双激酶(PPDK)基因,研究了其结构和功能特点,为稗属资源的开发和利用奠定了理论基础。目前,张桂芳等[47]已经通过农杆菌介导法成功地把稗草根型ppc基因转入到水稻中,并且获得了高效表达的T6代转基因水稻。除光合效率比较高以外,稗草还具有一些比水稻更好的生物学特性,如耐旱、生长繁茂、竞争性强等。在研究稗草与水稻的竞争关系时发现,稗草利用光能和水分的能力均比水稻强,但蒸发和传导能力比水稻弱,所以稗草的耐旱性和耐水性都比水稻强[48]。研究者们把旱稻基因型旱65当作母本,把稗草长芒稗当作父本进行远缘杂交和回交,选育出“远F2-1”单株后代系,育性正常,杂种优势明显,具有穗大粒多、抗逆性强、光合效率高等优良性状[49-50]。远缘杂交是导入外源基因的有效途径,利用这种方法,把稗草的抗逆性基因和光合酶基因导入栽培旱稻中,培育出抗逆性强、光合效率高、结实率高的优良品种,必将促进稻类遗传育种的长远发展。
3.3 基因组DNA的利用
野生稗草具有很强的耐铁毒能力,利用穗茎注射DNA技术把稗草的总DNA转入到水稻中,可获得一些耐铁毒的变异株系,且这些株系的耐铁毒能力明显高过其受体[51],此方法可以作为培育耐铁毒水稻的一种新方法。而采用花粉管道法把稗草的总DNA转入到水稻受体R122中,可得到4个稳定的稗草DNA导入系,经研究发现,导入系在株高、株型、分蘖能力、粒形、稻瘟病抗性、稻米品质等生物学性状上产生了很大的变异[52]。通过花粉管通道法把稗草的总DNA转入到水稻恢复系渝恢1351中,可获得趋于稳定的D4代材料,个别材料的苗期耐寒力增强或中后期抗病能力增强[53],说明稗草在培育水稻新材料方面具有很大的潜质。
4 展望
过去多把稗属植物看作稻田中的恶性杂草,对稗属植物的研究主要集中在稗属植物对除草剂的抗药性和不同除草剂对稗属植物的防治效果等方面[54-55]。近年来,由于我国农田种草养畜产业的兴起和退牧还草工程的实施,稗草正逐渐成为我国主要栽培牧草之一[56]。稗草具有产量高、品质好、抗逆性强、可以多次刈割利用等特点,日后定会在牧草产业中占据重要位置[41]。我国稗属植物资源丰富,种类多样,但是缺乏对稗草资源的合理利用,尤其是稗属植物作为饲草方面的应用价值没有得到充分的开发,对此,未来我国对稗属植物的研究应主要集中在以下几个方面:1)我国稗属植物种类多样,但分布混乱,遗传基础复杂,为更好地推广和利用这些优质稗草,应加强对稗草种群分布、资源搜集和系统分类的研究。2)种质资源多样性是育种工作的基础[57],而有关稗属植物遗传多样性的研究报道很少。目前,分子标记技术在种质资源遗传多样性的研究中最为广泛[58],所以应加快稗属植物EST-SSR、SNP等最新分子标记的开发。3)随着测序技术的不断完善,基因组学研究得到空前发展,但是稗属植物的基因组学研究(如基因组大小、来源、演化等)仍十分有限。基因组学研究可以为种质资源收集和保护提供理论指导,也为阐明植物起源和演化、全面评估种质资源结构多样性提供核心理论和技术,同时大幅度提高基因发掘和种质创新效率[59]。禾本科一些模式生物如二穗短柄草(Brachypodiumdistachyon)、小麦等植物的全基因组序列测定都已经完成[60],因此,可以采用比较基因组学研究方法对稗草基因组开展一些研究工作。稻稗(E.oryzicola)(2n=4x=36)被认为是稗(E.crusgalli)(2n=6x=54)的祖先,可作为稗属的模式生物开展全基因组测序工作,这将成为推动稗属植物研究的重要事件。
References:
[1] 陈守良.中国植物志 第九卷.北京:科学出版社,1990:250-261.
Chen S L.Flora of China Volumes 9.Beijing:Science Press,1990:250-261.(in Chinese)
[2] 罗长富,闵瑾如.栽培稗草的起源及其生物学特性.草业科学,1990,7(5):21-23.
Luo C F,Min J R.Origin and the biology of cultivated baryard grass.Pratacultural Science,1990,7(5):21-23.(in Chinese)
[3] 周平,郭水良,印丽萍.稗属植物的分类与系统学研究概况.杂草科学,2013,31(1):1-4.
Zhou P,Guo S L,Yin L P.Status of research on taxonomy and systematics of the genusEchinochloa.Weed Science,2013,31(1):1-4.(in Chinese)
[4] Chen S L,Sylvia M P.Flora of China Volume 22.Beijing:Science Press and Missouri Botanical Garden Press,2013.
[5] 张桂芳,丁在松.野生植物资源:稗属植物.北京农业,2016(6):158-159.
Zhang G F,Ding Z S.Wild Plant Resources:Echinacea.Beijing Agriculture,2016(6):158-159.(in Chinese)
[6] 蔡联炳,郭延平.禾本科植物叶片表皮结构细胞主要类型的演化与系统分类和发育途径的探讨.西北植物学报,1995,15(4):323-335.
Cai L B,Guo Y P.Studies on constituent cells of leaf epidermis,systematics and phylogenetic path of the family Poaceae.Acta Botanica Boreali-Occidentalia Sinica,1995,15(4):323-335.(in Chinese)
[7] 许瑾,徐涛,肖正康,朱水芳,张万霞.禾本科psbA-trnH基因的序列分析及其在斑点野生稻鉴定中的应用.生物技术通报,2011(2):85-92.
Xu J,Xu T,Xiao Z K,Zhu S F,Zhang W X.Analysis ofpsbA-trnHgene sequences of Gramineae and its application in identification ofOryzapunctata.Biotechnology Bulletin,2011(2):85-92.(in Chinese)
[8] 韩爱东,林建东,缪汝槐,俞皓,刘良式.一些水稻的重复DNA顺序在稻属和其它禾本科植物中的多态性.植物学报(英文版),1997,39(6):577-581.
Han A D,Lin J D,Miao R H,Yu H,Liu L S.Distribution of repetitive DNA sequences derived from rice genome in genusOryzaand family Poaceae.Acta Botanica Sinica,1997,39(6):577-581.(in Chinese)
[9] Yabuno T.Morphological and cytological studies in the genusEchinochloa.Seiken Ziho,1953,6(6):66-71.
[10] Yabuno T.Cytotaxonomic studies on the two cultivated species and the wild relatives in the genusEchinochloa.Cytologia,1962,27(3):296-305.
[11] Yabuno T.Biosystematic study of the genusEchinochloa.Japanese Journal of Botany,1966,19(2):277-323.
[12] Hilu K W.Evidence from RAPD markers in the evolution ofEchinochloa,millets (Poaceae).Plant Systematics and Evolution,1994,189(3/4):247-257.
[13] Aoki D,Yamaguchi H.Oryzash4 gene homologue represents homoeologous genomic copies in polyploidyEchinochloa.Weed Biology and Management,2009,9(3):225-233.
[14] Aoki D,Yamaguchi H.Genetic relationship betweenEchinochloacrusgalliandEchinochloaoryzicolaaccessions inferred from internal transcribed spacer and chloroplast DNA sequences.Weed Biology and Management,2008,8(4):233-242.
[15] 齐晓芳,张新全,凌瑶,刘伟,彭燕.我国狗牙根种质资源研究进展.草业科学,2011,28(3):444-448.
Qi X F,Zhang X Q,Ling Y,Liu W,Peng Y.Research progress of theCynodondactylongermplasm in China.Pratacultural Science,2011,28(3):444-448.(in Chinese)
[16] Yamaguchi H,Umemoto S,Masanaga Y.Studies on barnyard grasses,especially on non-shattering form ofEchinochloaoryzicolaVasing. in Yunkui Plateau,China.Journal of Weed Science and Technology,1996,41(2):111-115.
[17] Ruiz-Santaella J P,Bastida F,Franco A R,De Prado R.Morphological and molecular characterization of differentEchinochloaspp. andOryzasativapopulations.Journal of Agricultural and Food Chemistry,2006,54(4):1166-1172.
[18] Gupta A,Mahajan V,Kumar M,Gupta H S.Biodiversity in the barnyard millet (Echinochloafrumentacea,Link,Poaceae) germplasm in India.Genetic Resources and Crop Evolution,2009,56(6):883-889.
[19] Altop E K,Mennan H.Genetic and morphologic diversity ofEchinochloacrusgalli,populations from different origins.Phytoparasitica,2011,39(1):93-102.
[20] 陆永良,刘德好,余柳青,刘都才,郭水良.中国主要农区稻田稗草分类与多样性研究.植物科学学报,2014,32(5):435-445.
Lu Y L,Liu D H,Yu L Q,Liu D C,Guo S L.Classification and diversity ofEchinochloain paddy fields of main agricultural regions in China.Plant Science Journal,2014,32(5):435-445.(in Chinese)
[21] 刘杰淋,唐凤兰,张月学,刘凤岐,朱瑞芬.一年生牧草稗遗传多样性的初步研究.黑龙江畜牧兽医,2014(13):103-104.
Liu J L,Tang F L,Zhang Y X,Liu F Q,Zhu R F.Preliminary study on the genetic diversity of annual barnyard grass as a forage grass.Heilongjiang Animal Science and Veterinary Medicine,2014(13):103-104.(in Chinese)
[22] 蒲艳艳,宫永超,李娜娜,田茜,丁汉凤.中国小麦作物遗传多样性研究进展.中国农学通报,2016,32(30):7-13.
Pu Y Y,Gong Y C,Li N N,Tian Q,Ding H F.A review of genetic diversity of wheat crops in China.Chinese Agricultural Science Bulletin,2016,32(30):7-13.(in Chinese)
[23] 冯久焕,张廷璧.国产稗属植物的细胞学研究.植物科学学报,1993,11(4):293-299.
Feng J H,Zhang T B.Cytological studies on Chinese species ofEchinochloa.Plant Science Journal,1993,11(4):293-299.(in Chinese)
[24] 吴姝菊.2种寒地稗草农艺学性状与细胞学研究.草地学报,2010,18(2):280-285.
Wu S J.Biological traits and cytology study of two kinds of barnyardgrass from cold areas.Acta Agrestia Sinica,2010,18(2):280-285.(in Chinese)
[25] 吴姝菊,李新玲,唐凤兰.大头稗(005)与鹅头稗(031)的生物学性状与细胞学研究.草业学报,2011,20(3):198-204.
Wu S J,Li X L,Tang F L.Studies of biological traits and karyotype of two barnyard grass variety.Acta Prataculturae Sinica,2011,20(3):198-204.(in Chinese)
[26] 王清郦,赵淑芬,孙启忠,温定英.胡枝子属植物遗传多样性及其应用价值研究进展.草业科学,2012,29(11):1771-1777.
Wang Q L,Zhao S F,Sun Q Z,Wen D Y.Progress on genetic diversity and application value ofLespedezaplants.Pratacultural Science,2012,29(11):1771-1777.(in Chinese)
[27] Asíns M J,Barreda D G D.Morphologic and isozyme variation in barnyardgrass (Echinochloa) weed species.Weed Technology,1999,13(2):209-215.
[28] Nakayama Y,Umemoto S,Yamaguchi H.Identification of polyploid groups in the genusEchinochloaby isozyme analysis.Journal of Weed Science and Technology,1999,44(3):205-217.
[29] Yamaguchi H,Utano A,Yasuda K,Yano A,Soejima A.A molecular phylogeny of wild and cultivatedEchinochloain East Asia inferred from non-coding region sequences oftrnT-L-F.Weed Biology and Management,2005,5(4):210-218.
[30] 吴则东,江伟,马龙彪.分子标记技术在农作物品种鉴定上的研究进展及未来展望.中国农学通报,2015,31(33):172-176.
Wu Z D,Jiang W,Ma L B.Research progress and future prospects of molecular marker technology applied in crop variety identification.Chinese Agricultural Science Bulletin,2015,31(33):172-176.(in Chinese)
[31] Yasuda K,Yano A,Nakayama Y,Yamaguchi H.Molecular identification ofEchinochloaoryzicolaVasing. andE.crus-galli(L.) Beauv. using a polymerase chain reaction-restriction fragment length polymorphism technique.Weed Biology and Management,2002,2(1):11-17.
[32] Roy S,Simon J P,Lapointe F J.Determination of the origin of the cold-adapted populations of barnyard grass (Echinochloacrusgalli) in eastern North America:A total-evidence approach using RAPD DNA and DNA sequences.Canadian Journal of Botany,2000,78(12):1505-1513.
[33] Kaya H B,Demirci M,Tanyolac B.Genetic structure and diversity analysis revealed by AFLP on differentEchinochloaspp. from northwest Turkey.Plant Systematics and Evolution,2014,300(6):1337-1347.
[34] Danquah E Y,Johnson D E,Riches C,Arnold G M,Karp A.Genetic diversity inEchinochloaspp. collected from different geographic origins and within rice fields in Cote d′Ivoire.Weed Research,2002,42(5):394-405.
[35] 刘德好,陆永良,娄玉霞,郭水良.正交试验法优化稗属植物ISSR-PCR反应体系.上海农业学报,2016,32(4):17-21.
Liu D H,Lu Y L,Lou Y X,Guo S L.Optimization of ISSR-PCR reaction system ofEchinochloaby orthogonal design.Acta Agriculturae Shanghai,2016,32(4):17-21.(in Chinese)
[36] 陆永良,刘德好,郭水良,余柳青.基于ISSR标记的中国稻田稗属植物分类.浙江农业学报,2014,26(5):1309-1314.
Lu Y L,Liu D H,Guo S L,Yu L Q.Classification ofEchinochloaspecies in Chinese paddy fields based on ISSR markers.Acta Agriculturae Zhejiangensis,2014,26(5):1309-1314.(in Chinese)
[37] 付菲.稗属植物的分类鉴定及其与水稻化感作用研究.杭州:浙江大学硕士学位论文,2016.
Fu F.Classification ofEchinochloaspecies and their allelopathy with rice.Master Thesis,Hangzhou:Zhejiang University,2016.(in Chinese)
[38] 冯文煦.杂草与作物间的竞争关系概述.杂草学报,1990,4(2):43-47.
Feng W X.An overview of the competitive relationship between weeds and crops.Journal of Weed Science,1990,4(2):43-47.(in Chinese)
[39] 钱希.苏北农垦稻区稗属植物的发生以及对生态环境的反应.杂草学报,1990,4(2):29-34.
Qian X.Occurrence ofEchinochloain the northern Jiangsu agricultural reclamation rice area and its response to ecological environment.Journal of Weed Science,1990,4(2):29-34.(in Chinese)
[40] 张月学,唐凤兰,严洪东.寒地牧草高产栽培关键技术.第2版.哈尔滨:黑龙江科技出版社,2004:53-107.
Zhang Y X,Tang F L,Yan H D.The Key Planting Techniques for High Yield Cultivation of Forage in Cold Area.The Second Edition.Harbin:Heilongjiang Science and Technology Press,2004:53-107.(in Chinese)
[41] 易凤银,彭科林.饲用稗草生育特性及栽培利用的研究.草业科学,1993,10(5):62-64.
Yi F Y,Peng K L.Study on the growth characteristics and culture utilization ofEchinochloacrusgalli.Pratacultural Science,1993,10(5):62-64.(in Chinese)
[42] 古忠玉.四个直接利用的稗子资源.中国种业,1983(1):27-28.
Gu Z Y.Four the direct use of barnyard grass resources.China Seed Industry,1983(1):27-28.(in Chinese)
[43] 熊本海,罗清尧,赵峰,庞之洪.中国饲料成分及营养价值表(2015年第26版)制订说明.中国饲料,2015(21):23-33.
Xiong B H,Luo Q R,Zhao F,Pang Z H.China feed composition and nutritional value table (26thedition, 2015)formulation of instructions.China Feed,2015(21):23-33.(in Chinese)
[44] 张丽.C3、C4不同作物光合生理特性比较的研究.北京:中国农业大学硕士学位论文,2004.
Zhang L.Comparison of photosynthetic characteristics for C3and C4plants.Master Thesis.Beijing:China Agricultural University,2004.(in Chinese)
[45] 张桂芳,赵明,丁在松,张丽,肖俊涛.稗草磷酸烯醇式丙酮酸羧化酶(PEPCase)基因的克隆与分析.作物学报,2005,31(10):1365-1369.
Zhang G F,Zhao M,Ding Z S,Zhang L,Xiao J T.Cloning and characterization of phosphoenolpyruvate carboxylase gene fromEchinochloacrusgalli.Acta Agronomica Sinica,2005,31(10):1365-1369.(in Chinese)
[46] 王金明,丁在松,张桂芳,赵明.家稗丙酮酸磷酸双激酶(PPDK)基因的克隆及序列分析.作物学报,2007,33(6):927-930.
Wang J M,Ding Z S,Zhang G F,Zhao M.Cloning and sequence analysis ofPPDKgene inEchinochloacrusgallivar.frumentacea(Roxb.) W. F. wight.Acta Agronomica Sinica,2007,33(6):927-930.(in Chinese)
[47] 张桂芳,丁在松,赵明.稗草(Echinochloacrusgalli)根型ppc基因对水稻的遗传转化及其对光合速率的调节效应.作物学报,2015(3):507-513.
Zhang G F,Ding Z S,Zhao M.Transformation of barnyardgrass (Echinochloacrusgalli) root type phosphoenolpyruvate carboxylase gene into rice (Oryzasativa) plants and their effects on photosynthesitic gas exchange.Acta Agronomica Sinica,2015(3):507-513.(in Chinese)
[48] 乔丽雅,王庆亚,张守栋,李扬汉.稗属(EchinochloaBeauv.)杂草的生物学特性研究进展.杂草科学,2002,20(3):10-14.
Qiao L Y,Wang Q Y,Zhang S D,Li Y H.Research advances in biological characteristics ofEchinochloaweed.Weed Science,2002,20(3):10-14.(in Chinese)
[49] 李慧敏,赵凤梧,李爱国,白丽荣,张建,赵明,王志敏,赵秀琴.旱稻(Oryzasativa)×长芒稗(Echinochloacaudata)远缘杂交后代结实率及杂种优势分析.核农学报,2003,17(1):11-15.
Li H M,Zhao F W,Li A G,Bai L R,Zhang J,Zhao M,Wang Z M,Zhao X Q.Study on seed set and heteroses of distant cross of upland rice(Oryzasativa)×long-awn-barnyard-grass(Echinochloacaudata).Acta Agriculturae Nudeatae Sinica,2003,17(1):11-15.(in Chinese)
[50] 赵凤梧,李慧敏,赵明,刘冬成,王志敏,赵秀琴,刘勇.旱稻×稗草远缘杂交研究.华北农学报,2003,18(9):76-80.
Zhao F W,Li H M,Zhao M,Liu D C,Wang Z M,Zhao X Q,Liu Y.Studies on distant hybridization of upland rice (O.sativa)×Barnyard Grass (E.caudata).Acta Agriculturae Boreali-Sinica,2003,18(9):76-80.(in Chinese)
[51] 周建林,李阳生,李达模.稗草DNA导入水稻产生的变异体的耐铁毒特性和RAPD分析.作物学报,2001,27(4):529-532.
Zhou J L,Li Y S,Li D M.Analysis on RAPD pattern and iron-toxicity tolerance of variants induced by introducingEchinochloacrusgalliDNA into rice.Acta Agronomica Sinica,2001,27(4):529-532.(in Chinese)
[52] 王丰,柳武革,李金华,刘振荣,李曙光,廖亦龙,彭惠普,毛兴学.水稻外源DNA导入系的创建及主要性状分析.分子植物育种,2004,2(4):489-494.
Wang F,Liu W G,Li J H,Liu Z R,Li S G,Liao Y L,Peng H P,Mao X X.Construction of exogenous DNA introgression lines and analysis of its main traits in rice.Moleeular Plant Breeding,2004,2(4):489-494.(in Chinese)
[53] 李经勇,唐永群.稗草DNA导入渝恢1351后代性状变异研究.中国稻米,2012,18(2):27-29.
Li J Y,Tang Y Q.Study on offsprings character variation of barnyardgrass DNA introduced into Yuhui 1351.China Rice,2012,18(2):27-29.(in Chinese)
[54] 李岗,吴声敢,吴长兴,赵学平,王强,陈丽萍,苍涛,俞瑞鲜,任海英.稗草对二氯喹啉酸抗性研究进展.杂草科学,2012,30(2):1-5.
Li G,Wu S G,Wu C X,Zhao X P,Wang Q,Chen L P,Cang T,Yu R X,Ren H Y.Research advances on resistance of barnyard grass (Echinochloacrusgalli) to quinclorac.Journal of Weed Science,2012,30(2):1-5.(in Chinese)
[55] 马国兰,柏连阳,刘都才,刘雪源,唐涛,彭亚军.抗二氯喹啉酸稗草对6种除草剂的多抗性分析及田间控制效果评价.草业学报,2014,23(6):259-265.
Ma G L,Bai L Y,Liu D C,Liu X Y,Tang T,Peng Y J.Control of herbicide resistantEchinochloacrusgalliindirect-seeded rice crop.Acta Prataculturae Sinica,2014,23(6):259-265.(in Chinese)
[56] 肖金玉,蒲小鹏,徐长林.禁牧对退化草地恢复的作用.草业科学,2015,32(1):138-145.
Xiao J Y,Pu X P,Xu C L.Effects of grazing prohibition on restoration of degraded grassland.Pratacultural Science,2015,32(1):138-145.(in Chinese)
[57] 杨发荣,黄杰,魏玉明,李敏权,何学功,郑健.藜麦生物学特性及应用.草业科学,2017,34(3):607-613.
Yang F R,Huang J,Wei Y M,Li M Q,He X G,Zheng J.A review of biological characteristics,applications,and culture ofChenopodiumquinoa.Pratacultural Science,2017,34(3):607-613.(in Chinese)
[58] 董雪妮,唐宇,丁梦琦,未丽,李金博,吴燕民,邵继荣,周美亮.中国荞麦种质资源及其饲用价值.草业科学,2017,34(2):378-388.
Dong X N,Tang Y,Ding M Q,Wei L,Li J B,Wu Y M,Shao J R,Zhou M L.Germplasm resources of buckwheat in China and their forage value.Pratacultural Science,2017,34(2):378-388.(in Chinese)
[59] 黎裕,李英慧,杨庆文,张锦鹏,张金梅,邱丽娟,王天宇.基于基因组学的作物种质资源研究:现状与展望.中国农业科学,2015,48(17):3333-3353.
Li Y,Li Y H,Yang Q W,Zhang J P,Zhang J M,Qiu L J,Wang T Y.Genomics-based crop germplasm research:Advances and perspectives.Scientia Agricultura Sinica,2015,48(17):3333-3353.(in Chinese)
[60] 凌宏清.小麦及其近缘种基因组测序研究进展与发展趋势.麦类作物学报,2016,36(4):397-403.
Ling H Q.Progress and perspectives of the genome sequencing in wheat and its relatives.Journal of Triticeae Crops,2016,36(4):397-403.(in Chinese)
