西藏冬青稞种质资源SNP标记的遗传多样性分析
2018-02-03孟霞曾兴权其美旺姆尼玛扎西
孟霞+曾兴权+其美旺姆+尼玛扎西


摘要 单核苷酸多态性(single nucleotide polymorphism,SNP)是等位基因间序列差异最为普遍的类型,可作为一种高通量的遗传标记。利用IlluminaiSelect 9k SNP芯片对96个西藏冬青稞种质资源进行了SNP检测。结果表明,在分布于7条染色体上的7 000个SNP位点中,5 026个检测到了多态性,多态性比率为71.8%;SNP聚类分析将96份西藏冬青稞种质资源聚成七大类群,种质资源间的遗传相似系数(GS),其变异范围为0.288~0.999,平均值为0.771。96份西藏冬青稞种质资源间亲缘关系较近,遗传基础比较单一。
关键词 西藏冬青稞;种质资源;单核苷酸多态性(SNP);遗传多样性
中图分类号 S512.3;S34 文献标识码 A 文章编号 1007-5739(2018)01-0040-02
Analysis of Genetic Diversity with SNP Markers on Winter Barley Germplasm Resources in Tibet
MENG Xia 1 ZENG Xing-quan 2 QIMEI WANGMO 2 NIMA TASHI 2 *
(1 College of Agriculture and Animal Husbandry,Tibet University,Linzhi Tibet 860000; 2 Academy of Agriculture and Animal Husbandry)
Abstract Single nucleotide polymorphism(SNP)is the most common type of sequence difference between athelets,which can act as a high-throughput genetic marker. SNP detections were applied on 96 winter barley germplasms samples from Tibet,with IlluminaiSelect 9k SNP chip. The results showed that among the 7 000 SNP loci distributed in seven chromosomes,polymorphism had been detected in 5 026 loci,the polymorphic ratio was 71.8%;96 winter barley germplasms from Tibet was clustered into 7 categories by SNP cluster analysis,genetic similarity(GS)coefficient between germplasms had a variation in the range of 0.288-0.999,with an average of 0.771. The genetic relationships between 96 Tibet winter barley germplasm were close,the genetic basis was relatively simple.
Key words winter barley from Tibet;germplasm resource;single nucleotide polymorphism(SNP);genetic diversity
大麥(H. vulgare L. ssp. vulgare)是最古老的作物之一。大麦是我国青藏高原地区主要的粮食作物,对该地区的农业生产有着举足轻重的作用。研究西藏大麦的遗传多样性及其遗传改良对大麦生产的稳步发展具有十分重要的意义。
近年来,随着DNA分子标记技术的出现,为在DNA水平上评估不同品种间的亲缘关系及计算品种间的遗传距离提供了新的方法和手段。Sun等[1]利用RAPD标记技术分析了西藏小麦、普通小麦和欧洲小麦遗传多样性,发现欧洲和西藏小麦遗传多样性远高于普通小麦;冯宗云等[2]利用微卫星标记研究西藏野生二棱大麦的遗传多样性,分析其地理分化。何智宏等为探明我国大麦亲本材料的遗传背景和群体结构特点,提高大麦种质材料的利用效率,利用SSR标记对大麦亲本材料进行遗传多样性和群体结构分析,发现需要引入新种质来拓展亲本的遗传基础。
单核苷酸多态性(single nucleotide polymorphisms,SNP)是普遍存在于生物基因组中的一种新型分子标记,是指在基因组水平上由单个核苷酸的变异而引起的DNA序列单个碱基多态性变化。SNP是二等位基因,具有在基因组中数量最多、分布密度高、无需电泳、可高通量自动化检测等特点[3-4]。大规模、高通量SNP芯片检测首先广泛应用于人类群体结构的遗传学研究和关联性分析[5],商业化的SNP芯片也已被应用于一些家畜全基因组关联分析、QTL定位和候选基因筛选[6-7]。在植物中,Clark等[8]利用高通量SNP芯片技术对模式植物拟南芥不同品系进行了多态性研究;近年来,随着大麦高通量测序研究工作的开展,SNP开发和利用进展迅速。大麦已经开发了基于Illumina技术平台的9k高通量SNP分析芯片,已开始应用于大麦遗传连锁图谱的构建、DNA指纹分析、群体结构和连锁不平衡分析以及基因定位等诸多领域。Ren[9]、Chao[10]和Wurschum等[11]利用发现SNP标记能够真实反映小麦品种间的亲缘关系,可以用于禾本科作物品种遗传多样性研究。
近年来,西藏大麦尚未有从全基因组水平上对其遗传构成和亲缘关系进行研究的报道。本研究采用Illumina 9k iSelect SNP标记技术对来自西藏的96个大麦品种的遗传构成进行分析,旨在了解这些品种在全基因组水平上的遗传多样性和亲缘关系,以期为大麦新品种选育的亲本选配提供理论依据。
1 材料与方法
1.1 供试材料
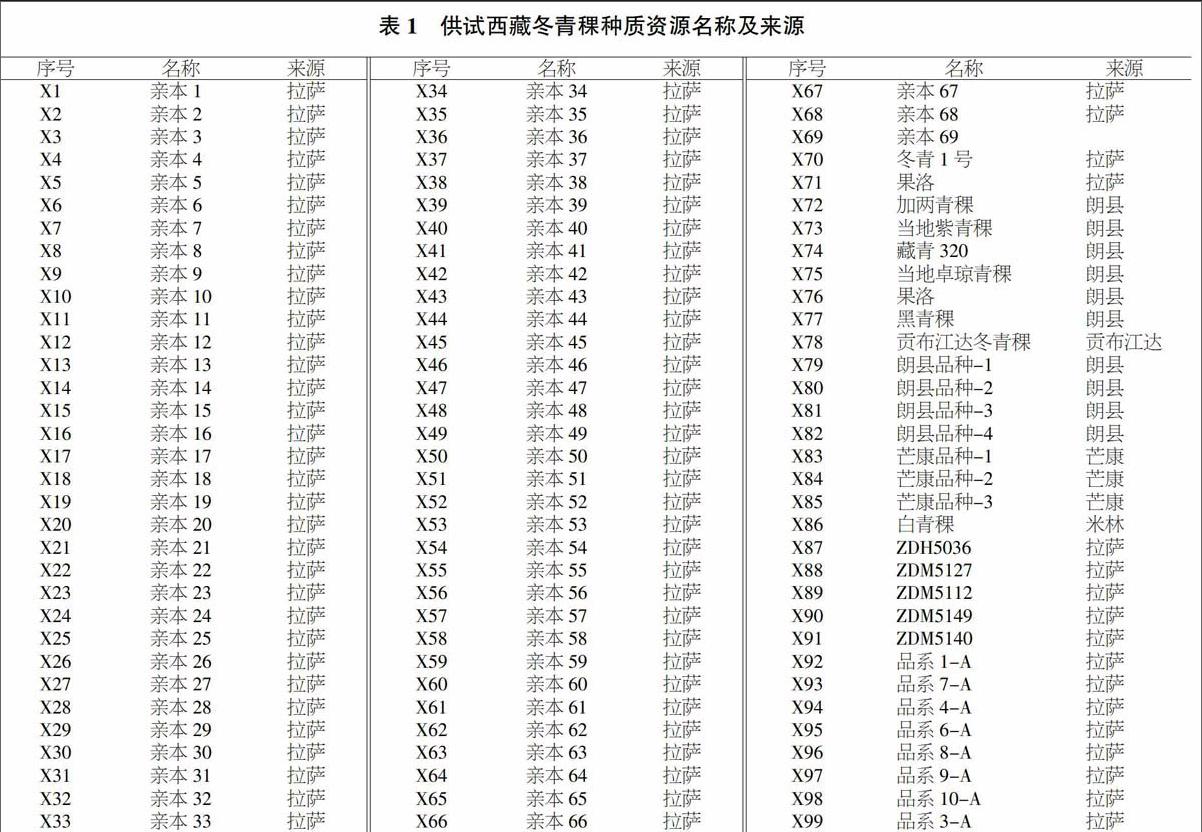
选用96份西藏冬青稞品种,对其进行编号,编号为X1~X99,其中X35、X61和X63缺号。供试材料为西藏农牧科学院提供的75份及从林芝、昌都收集的21份(表1)。
1.2 基因组DNA的提取
大麦基因组DNA的提取参照Saghai-Maroof等的CTAB抽提方法。
1.3 SNPs分析
利用Illumina 9k iSelect SNP标记对96个大麦品种进行了全基因组扫描,采用Genome Studio Polyploid Clustering Module v1.0 Software进行SNP分型。
2 结果与分析
2.1 多态性SNPs位点的分布
利用IlluminaiSelect 9k SNP芯片对96个大麦品种进行了SNP检测。在分布于7条染色体上的7 000个SNP位点中,5 026个检测到了多态性,多态性比率为71.8%。多态性SNP位点中有2 935个已经定位于各个染色体上,每条染色体上分布的多态性位点不等,从最少的4H染色体(244个)到最多的2H染色体(526个)(图1)。
2.2 西藏冬青稞种质资源间的遗传相似系数GS
利用NTSYS-pc 2.1遗传分析软件,根据SNP标记数据计算出冬青稞种质资源间的遗传相似系数(GS),其变异范围为0.288~0.999,平均值为0.771,其中X3与X44间的遗传相似性最低(0.288),表明这2个品种间亲缘关系最远,遗传差异较大;而X70与X72间的遗传相似性最高(0.999),表明这2个品种血缘关系最近,遗传差异较小。
根据所计算的4 560个遗传相似系数(GS),以0.18为组距进行次数分布分析(图2)。可以看出,96个品种间的遗传相似系数在0.78处分布密度最大。>0.78的遗传相似系数有1 946个,<0.7的有2 614个,分别占总数的42.68%和57.32%。遗传相似系数主要分布在0.65~0.89之间,数量为4 198个,占总数的92.06%;遗传相似系数为0.65的有240个,占总数的5.26%;遗传相似系数>0.89的有119个,占总数的2.61%。由此说明,西藏大麦品种间的遗传相似性高,品种间的差异较小,遗传基础相对狭窄。
2.3 聚类分析
根据小麦SNP遗传相似系数矩阵,利用NTSYS-pc2.1软件对96个小麦品种进行聚类分析(图3)。可以看出,96份西藏冬青稞种质资源聚成7大类群,其中Ⅰ、Ⅱ、Ⅵ、Ⅶ类只有1个品种,Ⅲ类有83个品种,Ⅳ类7个品种,Ⅴ类2个品种。从系谱和聚类结果可以看出,SNP标记划分的类别与品种来源具有较好的一致性。第Ⅰ类中,遗传相似系数在0.57处,X41和X44分别单独聚为一类,它们之间的遗传关系与其他材料间的遗传关系较远。
第Ⅲ类群包含83个品种,占试验品种总数的86.4%,品种间的遗传相似系数变幅在0.74~0.99之间,平均值为0.809,其中X70与X72之间的相似系数最大,在遗传基础上具有很高的亲缘性。
第Ⅳ类群包含7个品种,品种间的遗传相似系数变幅在0.74~0.99之间;第Ⅴ类群包含2个品种,品种间的遗传相似系数变幅为0.99。
第Ⅵ类群和第Ⅶ类分别包含1个品种,遗传相似系数在0.66处;X31单独聚为一类,遗传相似系数在0.69处;X23单独聚为一类。
3 结论与讨论
试验结果表明,西藏冬青稞种质资源间的遗传相似系数变幅为0.288~0.999,平均值为 0.771,且92.06%的供试材料间遗传相似系数在0.65~0.89之间,亲缘关系较近,遗传基础比较单一,大部分品种间的遗传差异较小,这将成为西藏大麦改良的限制因素。加强国内外育种单位的交流与合作,广泛收集不同类型的种质资源,不断创制新的适合本生态区的种质材料,加快新的育种技术应用,将是今后西藏大麦育种工作的重点。
4 参考文献
[1] SUN Q X,NI Z F,LIU Z Y,et al.Genetic relationships and diversity among Tibetan wheat,common wheat and European spelt wheat revealed by RAPD markers[J].Euphytica,1998,99:205-211.
[2] 冯宗云,张义正,张立立.应用微卫星标记研究西藏野生二棱大麦的遗传多样性及地理分化[J].高技术通讯,2003(10):16-53.
[3] NASU S,SUZUKI J,OHTA R,et al.Search for and analysis of single nucleotide polymorphisms(SNPs)in rice(Oryza sativa,Oryza rufipogon)and establishment of SNP markers[J].DNA Research,2002,9:163-171.
[4] 鄒喻苹,葛颂.新一代分子标记-SNPs及其应用[J].生物多样性,2003,11(5):370-382.
ZHOU Y P,GE S.A novel molecular marker-SNPs and its application[J].Biodiversity Science,2003,11(5):370-382.
[5] NISHIDA N,KOIKE A,TAJIMA A,et al.Evaluating the performance of Afrymetrix SNP array 6.0 platform with 400 Japanese individuals[J].BMC Genomics,2008,9:431.
[6] KARLSSON E K,BARANOWSKA I,WADE C M,et al.Efficient mapping of mendelian traits in dogs through genome-wide association[J].Nat Genet,2007,39:1321-1328.
[7] ANDERSON L.Genome-wide association analysis in domestic animals:a powerful approach for genetic dissection of trait loci[J].Genetica,2009, 136:341-349.
[8] CLARK R M,SCHWEIKERT G,TOOMAJIAN C,et al.Common sequence polymorphisms shaping genetic diversity in Arabidopsis thaliana[J].Science,2007,317(5836):338-342.
[9] REN J,CHEN L,SUN D,et al.SNP-revealed genetic diversity in wild emmer wheat correlates with ecological factors[J].BMC Evolutionary Biology,2013,13:169.
[10] CHAO S,DUBCOVSKY J,DVORAK J,et al.Population-and genome-specific patterns of linkage disequilibrium and SNP variation in spring and winter wheat(Triticum aestivum L.)[J].BMC Genomics,2010,11:727.
[11] WURSCHUM T,LANGER S M,LONGIN C F,et al.Population structure,genetic diversity and linkage disequilibrium in elite winter wheat assessed with SNP and SSR markers[J].Theor Appl Genet,2013,126:1477-1486.
