过表达H3K9me3去甲基化酶对猪克隆胚胎体外发育效率的影响
2018-01-25石俊松蔡更元刘德武吴珍芳李紫聪
吴 霄,李 果,敖 政,石俊松,蔡更元,刘德武,吴珍芳,李紫聪
(1.华南农业大学动物科学学院/国家生猪种业工程技术研究中心,广东 广州 510642;2.广东温氏种猪科技有限公司育种部,广东 新兴 527439)
猪克隆技术已成功应用于优良种畜扩繁、转基因动物生产等领域,但目前猪克隆胚胎发育至出生的效率只有0.3%~1%,导致猪克隆技术的效率仍然很低,这严重限制了猪克隆技术的发展及推广[1]。大量研究表明,克隆效率低下的主要原因是体细胞核的表观重编程错误或不完全[2]。重编程涉及大量复杂的表观遗传修饰,包括DNA甲基化、组蛋白修饰、X染色体失活、基因组印迹等。
组蛋白甲基化是一种重要的组蛋白修饰,其主要发生在赖氨酸和精氨酸侧链,例如H3K4me3、H3K9me3、H3K27me3这些修饰影响着基因表达的转录抑制或转录起始。目前,H3K9me3已经被发现是诱导多能干细胞(induced Pluripotent Stem Cells,iPSC)重编程的主要障碍。Soufi等[3]研究发现,许多重编程失败的区域往往是H3K9me3的富集区域,并且通过敲除相关的组蛋白甲基转移酶SUV39H1/2来阻断H3K9me3,可以“显著加速”重编程过程。在小鼠的体细胞克隆中,H3K9me3被确定是供体细胞基因组重编程的一个主要障碍,其在克隆胚胎发育早期沉默,导致克隆胚胎难以正常发育[4-5]。Matoba 等[6]通过异位表达H3K9me3去甲基化酶KDM4D去除了H3K9me3修饰,不仅可以重激活大多数的重编程抵抗区(reprogramming resistant regions,RRRs)的表达,还将小鼠克隆胚胎的囊胚率提高到84.9%。此外,Chung等[7]向小鼠克隆胚胎显微注射人源KDM4A的mRNA,仍然可以大幅度提高(90.3%)克隆小鼠的囊胚率。
由上可知,供体细胞来源的H3K9me3是导致克隆胚胎基因沉默的一种重要表观遗传标记[8]。已有研究表明,猪的体细胞存在高水平的H3K9me3修饰[9],这也可能是阻碍猪克隆胚胎发育的一个因素。目前,通过在猪克隆胚胎中去除H3K9me3修饰提高其发育效率的相关研究未见报道。鉴于猪H3K9me3去甲基化酶基因的全长序列仍未确定,本研究在猪克隆胚胎中通过异位表达鼠源KDM4B/KDM4D mRNA的方法去除H3K9me3修饰,以期提高猪克隆胚胎的发育效率。
1 材料与方法
1.1 试验材料
1.1.1 质粒、细胞株和猪克隆胚胎 小鼠KDM4B/KDM4D-pcDNA3.1(+)真核表达载体由苏州金唯智生物技术有限公司合成;猪胎儿成纤维细胞采自广东温氏食品集团华农温氏股份有限公司水台原种猪场;猪克隆胚胎由广东温氏食品集团股份有限公司研究院现代育种技术中心克隆实验室负责提供。
1.1.2 主要试剂 细胞与胚胎免疫荧光检测蛋白表达试剂盒购自Life Technologies公司;组蛋白H3K9me3免疫荧光所用抗体购自Abcam生物公司;体外转录试剂盒mMESSAGE mMACHINE®T7 Kit购自 Life Technologies公司;PrimeSTAR HS DNA Polymerase购自Clontech公司;普通质粒DNA小提试剂盒(E.Z.N.A®Mini Plasmid Kit)、DNA 纯化试剂盒(E.Z.N.A®DNA Cycle Pure Kit);胚胎注射级水购自Sigma公司;限制性内切酶HindIII、XhoI、BamHI购自广州昂科生物技术有限公司;溴化乙锭(Ethidium Bromide,EB)购自广州康龙生物科技有限公司;蛋白胨(Tryptone)、酵母浸出物(Yeast Extract)、琼脂糖(Agar)均购自北京鼎国生物技术有限公司。
1.2 试验方法
1.2.1 小鼠KDM4B/KDM4D基因克隆 从NCBI获悉小鼠KDM4B(NM_172132.2)、KDM4D(NM_173433.2)基因序列,序列大小分别是3279、1 551 bp。利用质粒分析软件,找到合适的酶切位点,将2条基因序列的编码区序列构建至pcDNA3.1(+)双核表达载体(图1)。交由苏州金唯智生物科技有限公司合成,共构建了2个基因表达载体。
1.2.2 酶切鉴定 将基因合成所得的穿刺菌接种于氨苄抗性平板中,37℃培养箱中培养12~16 h。挑取数个单克隆进行培养,进行菌液PCR鉴定,鉴定出的单克隆利用DNA小提试剂盒进行质粒抽提。抽提的质粒用限制性内切酶HindIII/XhoI进行酶切鉴定。
1.2.3 KDMB/D基因体外转录及加poly(A)尾 利用XhoI限制性内切酶线性化表达载体KDM4B/KDM4D,再利用DNA 纯化试剂盒纯化回收线性化载体。然后根据转录试剂盒进行体外转录,用E. coliPoly(A) Polymerase (NEB)对产物进行加poly A反应,获取稳定的mRNA用于后期猪克隆胚胎胞浆显微注射。
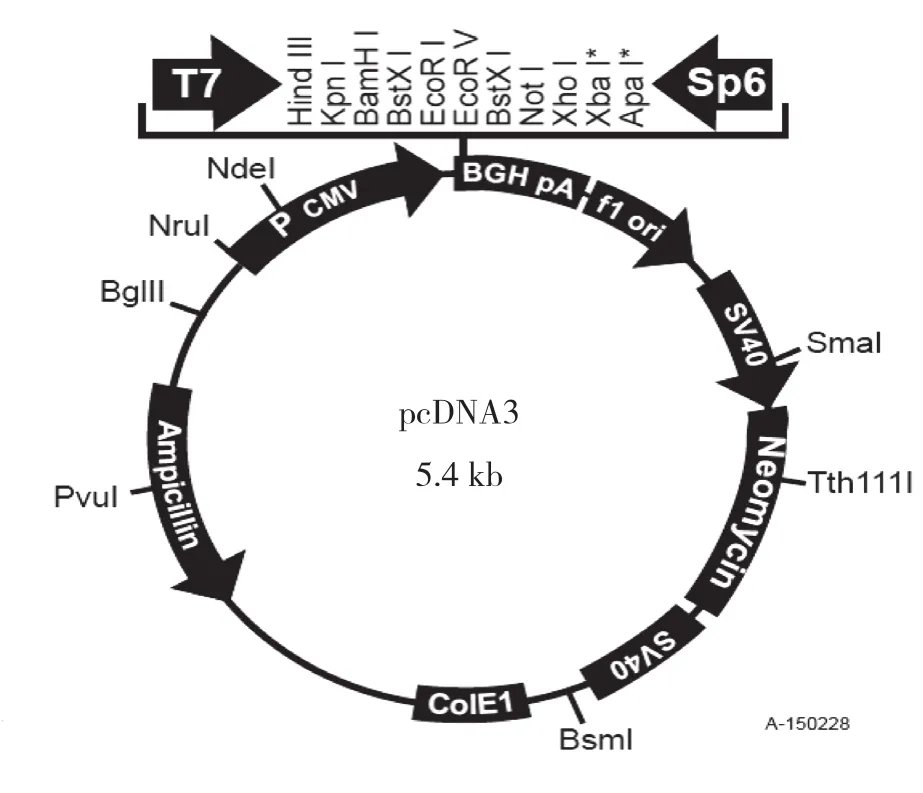
图1 pcDNA3.1(+)载体图谱
1.2.4 胞浆显微注射小鼠KDM4B/KDM4DmRNA 通过胞浆显微注射的方法将KDM4B/KDM4DmRNA注射入克隆胚胎中,mRNA 终浓度为2 000 ng/μL,每枚胚胎注射量为10 pL,对照组为不做处理的克隆胚胎。其中KDM4B/KDM4DmRNA注射组各注射了200个克隆胚胎。对注射组和对照组胚胎进行体外培养,统计24 h的胚胎卵裂率、72 h至8细胞期的胚胎数、168 h的囊胚数及发育至囊胚期的胚胎数。同时在胚胎发育至24 h时,在注射组和对照组中分别收集10个胚胎用于胚胎的组蛋白H3K9me3甲基化表达水平检测,KDM4B/KDM4D引物信息见表1。
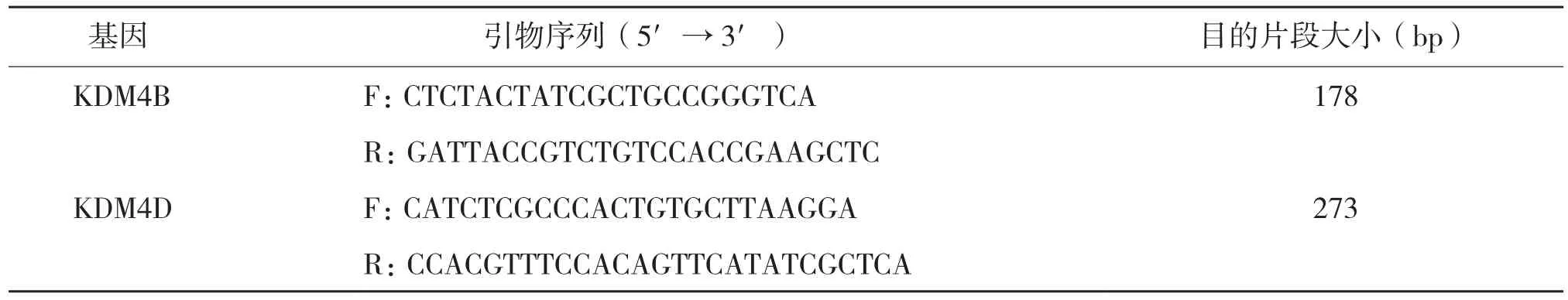
表1 KDM4B/KDM4D引物序列
1.2.5 免疫荧光法检测克隆胚胎中组蛋白H3K9me3表达水平 将收集的胚胎放入预热的0.5%链酶蛋白酶中作用1 min去透明带,然后用洗卵液洗涤3次;加入固定剂室温孵育15 min后用洗卵液洗涤3次;加入穿孔液室温孵育30 min后用洗卵液洗涤3次;加入封闭液4℃过夜;加入一抗稀释液孵育1 h后用洗卵液洗涤12次;在避光条件下加入二抗稀释液,37℃孵育1 h后用洗卵液洗涤12次;最后加入1μg/mL碘化丙啶(Propidium iodide,PI),室温孵育10 min后用洗卵液洗涤数次。将上述染色处理的胚胎转移到干净的载玻片上,以胚胎所在处为中心,在盖玻片大小范围内的四角做4个由凡士林/石蜡油组成的柱,盖上盖玻片,用指甲油封片,然后在荧光显微镜下观察,拍照记录。
1.2.6 数据处理及统计分析 基因相对表达数据分析采用2-△△CT方法计算相对表达量。体细胞克隆胚胎的体外发育数据(分裂率、囊胚率和囊胚细胞总数)用卡方检验进行分析,P<0.05表示差异显著。采用SPSS20软件作图。
2 结果与分析
2.1 KDM4B/KDM4D基因过表达载体构建
根据小鼠KDM4B(NM_172132.2)和KDM4D(NM_173433.2)基因序列,利用质粒分析软件查找合适的克隆位点,将KDM4B/KDM4D基因CDs序列克隆到商业化的克隆载体pcDNA3.1(+),共构建了pcDNA3.1(+)-KDM4B(8 695 bp,限制性内切酶位点为HindIII和XhoI)和 pcDNA3.1(+)-KDM4D(6 967 bp,限制性内切酶位点为HindIII和XhoI )两个克隆表达载体。
用限制性内切酶HindIII单酶切pcDNA3.1(+) -KDM4B 和 pcDNA3.1(+) -KDM4D 表达载体;HindIII和XhoI双酶切pcDNA3.1(+)-KDM4B质和pcDNA3.1(+) -KDM4D表达载体,酶切鉴定显示载体构建成功。将过表达载体进行测序,测序结果显示载体序列正确。
2.2 KDM4B/KDM4D基因过表达载体体外转录
利用XhoI酶将抽提好的KDM4B/KDM4D质粒进行线性化处理,纯化回收线性化载体,测定浓度,利用体外转录试剂盒获得KDM4B/KDM4DmRNA并进行加尾处理,对体外转录获得的mRNA进行凝胶电泳,结果(图2)显示KDM4B/KDM4DmRNA已成功转录。
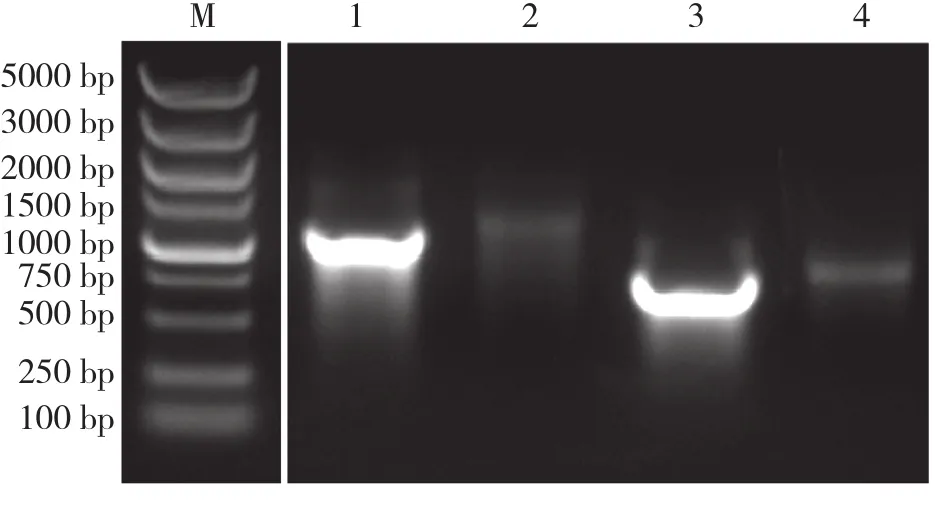
图2 KDM4B/KDM4D体外转录结果
2.3 胞浆显微注射KDM4B/KDM4D mRNA对猪克隆胚胎体外发育效率的影响
2.3.1 胞浆显微注射KDM4B/KDM4DmRNA 将24 h收集的克隆胚胎抽提RNA进行q-PCR检测KDM4B/KDM4D表达水平,结果(图3)显示注射组KDM4B/KDM4D基因相对表达量远高于对照组。
在1-cell期克隆胚胎中注射去甲基化酶KDM4BmRNA ,体外培养24 h后收集胚胎,检测组蛋白H3K9me3的表达水平,将KDM4BmRNA注射组与空白对照组克隆胚胎免疫荧光处理后,在紫外处理(Ultraviolet,UV)、生物染色处理(Biological stain,BS)下观察处理结果。免疫荧光实验结果(图4,彩插三)显示,实验组与对照组胚胎的H3K9me3的表达水平一致,并没有显著差异,说明通过克隆胚胎胞浆显微注射去人源和鼠源去甲基化酶基因mRNA的方式并不能有效去除组蛋白H3K9me3甲基化标记。
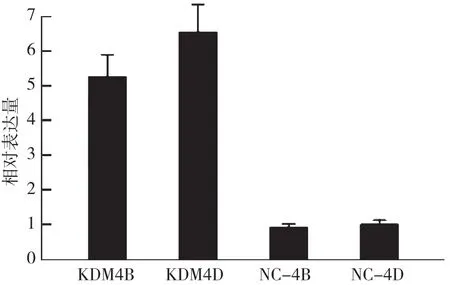
图3 注射后24 h KDM4B/KDM4D相对表达量
2.3.2 胞浆显微注射KDM4B/KDM4DmRNA猪克隆胚胎体外发育效率的变化 在1-cell期,对已激活的胚胎胞浆中显微注射KDM4B/KDM4DmRNA后,培养观察克隆胚胎的体外发育情况。统计克隆胚胎体外发育的卵裂率、囊胚率、囊胚细胞数等体外发育效率。结果(表2)表明,在1-cell期激活的胚胎胞浆中显微注射KDM4B/KDM4DmRNA的实验组与空白对照组比较,卵裂率、囊胚率没有显著变化;囊胚细胞数:KDM4B实验组与对照组相比显著提高,KDM4D实验组与对照组相比没有明显变化。对168 h的囊胚进行hocesst33342细胞染色,统计其囊胚细胞数,染色结果见图5(彩插三)。
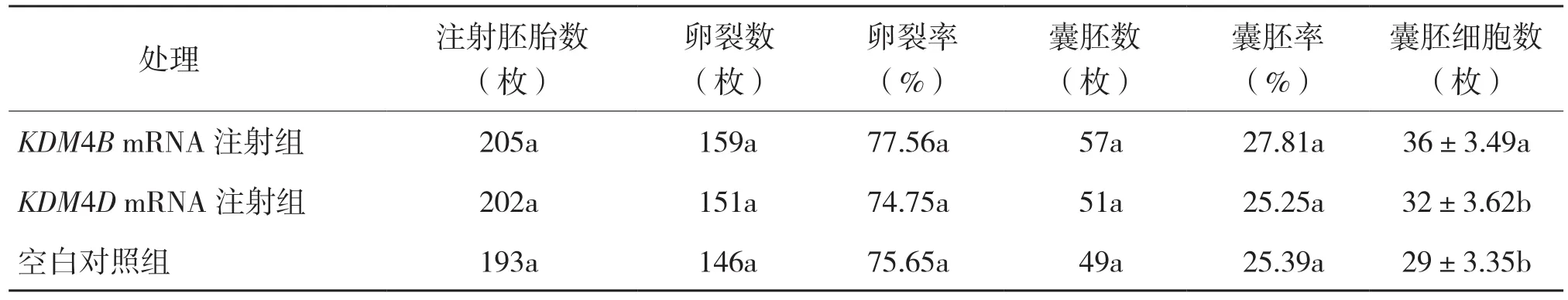
表2 猪克隆胚胎体外发育效率结果
3 讨论与结论
通过向体外转录的KDM4 mRNA显微注射入克隆胚胎来提高克隆效率的研究方法已经在小鼠上获得了成功[6-7]。Fukuda 等[10]也成功利用了KDM4B有效去除小鼠克隆胚胎的H3K9me3修饰,激活其所结合的RNF12基因和Xm-Xist的表达,使克隆胚胎发育效率得到提高。但该研究方法对猪克隆胚胎发育的影响鲜有报道,因此本研究向猪克隆胚胎显微注射KDM4B/KDM4DmRNA来进行去除猪克隆胚胎H3K9me3修饰的探索。在本研究中,虽然KDM4B/KDM4DmRNA的相对定量PCR结果显示KDM4B/KDM4DmRNA在克隆胚胎内成功表达,但是免疫荧光的结果显示H3K9me3修饰依然大量存在,而且注射组的体外发育效率与对照组相比没有显著差异,这说明向猪克隆胚胎显微注射鼠源的KDM4B/KDM4DmRNA没能有效去除H3K9me3修饰。这可能是由于注射的是鼠源KDM4B/KDM4D的mRNA,可能无法对猪克隆胚胎中的H3K9me3修饰起作用,虽然Chung等[7]将人源的KDM4AmRNA注射入小鼠克隆胚胎的研究表明了KDM4基因存在保守性,而在本研究中,异源mRNA对猪克隆胚胎发育效率的影响没有小鼠克隆胚胎那样有效。
获取猪源KDM4基因家族的完整序列,转录mRNA来进行显微注射,这对猪克隆效率的提高可能是一种更加具有针对性的研究策略。本研究中的KDM4BmRNA注射组的囊胚细胞数相比对照组有显著提高,这说明本研究仍具有进一步研究的潜力。同时,除了已报道的KDM4A/B/D,KDM4家族还包括了KDM4C、KDM4E及KDM4F等成员,这些基因在克隆效率上的运用尚未见报[11-12]。往后的研究可以对KDM4家族进行系统探索,寻找能在猪克隆胚胎发育中有效去除H3K9me3的一种或几种去甲基化酶。
人为去除H3K9me3修饰来提高胚胎克隆效率,除了增加组蛋白去甲基化酶的表达,还可以考虑抑制H3K9me3修饰的甲基化转移酶基因Suv39h1/h2和Setdb1的表达。Matoba等[6]通过向小鼠克隆胚胎转染siRNA来抑制其Suv39h1/h2的表达,将囊胚率从6.7%提高到49.9%。而Chen等[13]则在研究iPSC的过程发现Setdb1在H3K9me3修饰建立中的作用大于Suv39h1/h2。除了H3K9me3,还有许多其他的组蛋白修饰在影响着克隆胚胎发育。Cervera等[14]利用曲古抑菌素A提高H4K8的乙酰化状态,并提高了猪克隆胚胎的囊胚率(48.1%)。Huang等[15]发现H3K4me3在4-cell期和囊胚期的克隆胚胎中异常表达增加,这破坏了囊胚期H3K4me3-H3K27me3修饰的平衡,其认为这可能就是损害胚胎发育的原因之一。
克隆技术在猪的研究工作中具有重大的应用意义,如果能够克服克隆效率低下的难题,克隆技术才能更好地应用于猪的理论研究及生产实践中。利用高通量测序、免疫共沉淀、CRISPR/Ca9等前沿技术来揭示猪克隆胚胎中的表观遗传修饰图谱,寻找供体细胞重编程中的关键障碍,对异常表观遗传修饰进行修复或纠正,仍是今后研究的重点[16-17]。本研究通过向胚胎注射体外转录的mRNA,过表达去甲基化酶KDM4B/KDM4D,尝试去除猪克隆胚胎中的H3K9me3修饰,希望为今后的猪克隆技术研究应用提供理论依据。
[1] Liu Y,Li J,Løvendahl P,et al. In vitro manipulation techniques of porcine embryos:a meta-analysis related to transfers,pregnancies and piglets[J]. Reproduction Fertility &Development,2015,27(3):429-439.
[2] Niemann H. Epigenetic reprogramming in mammalian species after SCNT-based cloning[J]. Theriogenology,2016,86(1):80-90.
[3] Soufi A,Donahue G,Zaret K S. Facilitators and impediments of the pluripotency reprogramming factors' initial engagement with the genome[J].Cell,2012,151(5):994-1004.
[4] Popken J,Brero A,Koehler D,et al. Reprogramming of fibroblast nuclei in cloned bovine embryos involves major structural remodeling with both striking similarities and differences to nuclear phenotypes of in vitro fertilized embryos[J].Nucleus,2014,5(6):555-589.
[5] Hillringhaus L,Yue W W,Rose N R,et al.Structural and evolutionary basis for the dual substrate selectivity of human KDM4 histone demethylase family[J]. Journal of Biological Chemistry,2011,286(48):41616-41625.
[6] Matoba S,Liu Y,Lu F,et al. Embryonic development following somatic cell nuclear transfer impeded by persisting histone methylation[J]. Cell,2014,159(4):884-895.
[7] Chung,Young ,Matoba,et al. Histone Demethylase Expression Enhances Human Somatic Cell Nuclear Transfer Efficiency and Promotes Derivation of Pluripotent Stem Cells[J].Cell Stem Cell,2015,17(6):758-766.
[8] Ribeiro-Mason K,Boulesteix C,Brochard V,et al. Nuclear dynamics of histone H3 trimethylated on lysine 9 and/or phosphorylated on serine 10 in mouse cloned embryos as new markers of reprogramming[J]. Cellular Reprogramming,2012,14(4):283-294.
[9] Kim S Y,Kim T S,Park S H,et al. Siberian sturgeon oocyte extract induces epigenetic modifications of porcine somatic cells and improves developmental competence of SCNT embryos[J]. Asian-Australasian Journal of Animal Sciences,2014,27(2):266-277.
[10] Fukuda A,Tomikawa J,Miura T,et al. The role of maternal-specific H3K9me3 modification in establishing imprinted X-chromosome inactivation and embryogenesis in mice[J].Nature Communications,2014,5:5464.
[11] Yang L,Lin C,Liu W,et al. ncRNA-and Pc2 methylation-dependent gene relocation between nuclear structures mediates gene activation programs[J]. Cell,2011,147(4):773-788.
[12] Labbé R M,Holowatyj A,Yang Z Q. Histone lysine demethylase(KDM)subfamily 4:structures,functions and therapeutic potential[J]. American Journal of Translational Research,2014,6(1):1-15.
[13] Chen J,Liu H,Liu J,et al. H3K9 methylation is a barrier during somatic cell reprogramming into iPSCs[J]. Nature Genetics,2013,45(1):34-42.
[14] Cervera R P,Martígutiérrez N,Escorihuela E,et al. Trichostatin A affects histone acetylation and gene expression in porcine somatic cell nucleus transfer embryos[J]. Theriogenology,2009,72(8):1097-1110.
[15] Huang J,Zhang H,Wang X,et al. Impairment of preimplantation porcine embryo development by histone demethylase KDM5B knockdown through disturbance of bivalent H3K4me3-H3K27me3 modifications1[J]. Biology of Reproduction,2015,92(3):1-11,72.
[16] Inoue A,Lan J,Lu F,et al. Maternal H3K27me3 controls DNA methylation-independent imprinting[J]. Nature,2017,547(7664):419-424.
[17] Park K E,Park C H,Powell A,et al. Targeted gene knockin in porcine somatic cells using CRISPR/Cas Ribonucleoproteins[J].International Journal of Molecular Sciences,2016,17(6):810.
