津田芜菁BrSIZ1基因克隆、定位及表达
2018-01-18谷俊辰闫海芳
罗 云 马 璇 谷俊辰 闫海芳
津田芜菁基因克隆、定位及表达
罗 云 马 璇 谷俊辰 闫海芳*
东北林业大学生命科学学院, 黑龙江哈尔滨 150040
SIZ1是植物细胞蛋白质翻译后修饰SUMO化的E3连接酶, 参与植物蛋白相互作用、定位和抗逆反应。为研究BrSIZ1在津田芜菁中的表达特性, 本研究克隆了津田芜菁基因全长cDNA序列, 命名为(GenBank登录号为KY441465), 该基因全长2754 bp, 其ORF全长2571 bp, 编码856个氨基酸残基的多肽。构建了BrSIZ1-GFP表达载体进行亚细胞定位研究, 结果显示BrSIZ1-GFP定位于细胞核内, 可能在细胞核中发挥其功能。利用荧光定量PCR检测表明, 该基因表达量在叶片中最高, 在幼苗和红色根皮中次之, 表达具有组织特异性。而且在芜菁根皮中的表达受长波紫外线(UV-A)诱导, 在4°C、37°C胁迫的幼苗中, 表达量增加。
津田芜菁;; 基因克隆; 亚细胞定位; 表达分析
蛋白质小泛素类似修饰(small ubiquitin-related modifier, SUMO)是一种植物蛋白质翻译后修饰, 是蛋白质发挥生物学功能的重要调节机制。SUMO是一种小分子多肽, 修饰蛋白的过程称为SUMO化[1-2]。SUMO化途径与泛素化一样也有3种酶参与, SUMO活化酶(SUMO-activating enzyme, SAE/E1, 又名Ubc9), SUMO结合酶(SUMO-conjugating enzyme, SCE/E2)和SUMO E3连接酶(SUMO-E3, ligase)[3]。E3连接酶的主要作用是识别底物和提高靶蛋白的修饰效率, 在调节植物生长发育过程中发挥重要作用[4]。
目前在拟南芥中发现有两类SUMO E3连接酶, 分别为SIZ/PIAS (SAP and MIZ/protein inhibitor of activated STAT)和NES/MMS21 (non-SMC element/ methyl methanesulfonate sensitive)。SIZ/PIAS蛋白家族的特点是含有保守的结构域, 包括SAP、PINIT、SP-RING、SXS和核定位信号NLS, 还有植物所特有PHD[5-6]。SAP区域由一个螺旋-延伸-螺旋结构组成, 主要与DNA结合有关[7]。PHD是一个C4HC3的锌指结构, 只存在于植物SIZ/PIAS蛋白中, 与E3连接酶活性有关[6]。PINIT (Pro-Ile-Asn-Ile-Thr)区对于SIZ/PIAS蛋白存在于细胞核是必需的, 锌指结构域SP-RING赋予SUMO E3连接酶的活性[5]。SXS是SUMO结合区域[8]。由于SIZ1具有对底物识别的特异性功能, 使得它对植物的生长具有多种影响。目前研究表明: SIZ1参与调控植物根的生长、花期、花药发育及生物与非生物胁迫, 如抗病、抗寒、抗热、抗旱等[9-18]。津田芜菁(ssp.‘Tsuda’)膨大的肉质根不见光部分呈白色, 无花青素的合成; 而见光部分呈紫色, 有花青素的合成。单色光中只有在UV-A诱导下可合成花青素[19]。UV-A诱导后蛋白质组学分析发现SIZ1蛋白表达量增加。本研究根据已经报道的拟南芥序列, 通过RT-PCR结合RACE技术克隆了芜菁基因, 并研究了该基因的定位和表达, 以期为深入探讨SIZ1在UV-A诱导花青素合成和温度胁迫响应中的功能提供参考依据。
1 材料与方法
1.1 材料处理
津田芜菁(L. ssp.‘Tsuda’)种植于东北林业大学花卉生物工程研究所温室。长至60 d取其膨大白色根皮(不见光)、红色根皮(自然光)、去皮后根、叶片和长至5个月左右开放的花; 以UV-A (352 nm, 15 W m–2)光照处理一直处于土壤中生长的肉质根白色表皮3、6、12、24和48 h。取自然光下, 22°C萌发4天的幼苗, 将其移至4°C和37°C处理3、6、12 和24 h。迅速以液氮冷冻样品, 于–80°C保存备用。
1.2 花青素的测量
准备色素提取液(含1 % 盐酸的甲醇溶液), 取1 cm×1 cm的UV-A光照处理的根皮于提取液中, 4°C放置24 h[20]。在530 nm的光吸收下检测样品。以OD值表示花色素苷的含量, 生物学重复3 次。
1.3 RNA的提取和第1链cDNA的合成
将1.1准备的材料取出, 提取总RNA (上海宝生生物公司的TRIzol试剂), 取1 μg的总RNA用于cDNA第一链的合成。
1.4 BrSIZ1的cDNA克隆与序列测定
使用TaKaRa公司的RNA PCR Kit (AMV) Ver. 3.0试剂合成cDNA第1链。参考GenBank中亲缘关系相近物种拟南芥基因的核苷酸序列, 应用PrimerPremier 6.0设计引物BrSIZ1-F: 5′-AAA TCACAAGGGCAGACAAGGA-3′和BrSIZ1-R: 5′-A AAGGCTACCATCAGGTGCAT-3′, 克隆部分ORF序列。以cDNA第1链为模板。PCR条件为94°C 3 min预变性, 94°C 30 s, 55°C 30 s, 72°C 1 min, 30个扩增循环, 最后72°C延伸10 min。PCR扩增产物经0.8% (w/v)琼脂糖凝胶检测、回收目的条带, 与北京全式金公司的pEASY-T5 Zero Vector链接, 转化到大肠杆菌TOP10, 挑选克隆进行PCR鉴定, 由华大生物公司测序。测序引物分别为M13正反向引物。
在已获得部分ORF序列的基础上利用cDNA 末端快速扩增(RACE)法克隆3'-端序列[21], 以红色根皮1 μg总RNA为模板, 以oligo(dT)17接头引物5′-GACTCGAGTGCACATCG(T)17-3′为引物, 用反转录酶III (Invitrogen)合成第1链cDNA。参考已获得的ORF序列设计的5’引物BrSIZ1-5’F: 5′-AAATT GAAGTGAAACCTGAT-3′和接头引物5′-GACTCG AGTGCACATCG-3′进行PCR扩增。扩增条件为94°C预变性5 min, 随后30个循环, 每循环94°C变性30 s、55°C退火扩增30 s、72°C延伸90 s, 完成最后一个循环后, 72°C延伸 10 min。PCR扩增产物经琼脂糖凝胶检测、回收纯化, 测序同cDNA克隆。
根据获得的5'-cDNA序列设计基因特异性引物, 用5'/3'-RACE试剂盒(Roche)及基因特异性引物BrSIZ1-3’F: 5′-AAGCATACCATTGATCTGTAG-3′、oligo(dT)锚定引物5′-GACCACGCGTATCGATGTC GAC(T)16(A/C)-3′、PCR锚定引物5′-GACCACGCGT ATCGATGTCGAC-3′, 通过5'-RACE克隆5'-端序列。扩增条件、克隆方法同3'-RACE。
1.5 芜菁BrSIZ1基因生物信息学分析采用的方法
用NCBI的nucleotide BLAST程序(http://www. ncbi.nlm.nih.gov/blast/)进行基因序列同源性比对。用NCBI的protein BLAST程序(http://www. ncbi.nlm.nih.gov/blast/)分析BrSIZ1保守性功能域。用http://web.expasy.org/compute_pi/预测相对分子质量和等电点。用http://nls-mapper.iab.keio.ac.jp/cgi- bin/NLS_Mapper_form.cgi程序分析核定位信号(http://www.cbs.dtu.dk/services/SignalP/)。由MEGA5程序完成进化树的构建。
1.6 BrSIZ1在洋葱表皮细胞中的亚细胞定位
用加有酶切位点的引物对测序验证过的含有目的基因的质粒DNA进行扩增, 回收目的基因片段与pA7-GFP载体连接。通过真空渗透方法将重组质粒转入洋葱表皮细胞, 培养16 h后, 利用Zeiss 510 nete激光共聚焦扫描显微镜观察[22]。
1.7 荧光定量PCR检测芜菁BrSIZ1表达
用7500 Real-time PCR System (Applied Biosystems)检测基因的表达。引物为BrSIZ1-F: 5′-ACGGGCTCAACTCAAA-3′, BrSIZ1-R: 5′-TCTGGCGAGGAAATGAAA-3′, 内标引物为BrACTIN-F: 5′-GCTCAGTCCAAGAGAGG TATTC-3′, BrACTIN-R: 5′-GCTCGTTGTAGAAAGT GGATC-3′。试剂为SYBRPremix Ex(TaKaRa, 日本)。10 μL反应体系含5 μL SYBR Premix Ex, 0.2 μL ROX Reference Dye (50×)(TaKaRa, Japan), 4.5 μL ddH2O (TIANGEN, 中国), 0.17 μL cDNA, 0.33 μL引物混合物(正反向引物, 10 μmol L–1)。Real-time PCR程序为95°C 30 s预变性, 40个扩增循环(95°C 10 s, 60°C 31 s), 以及从60°C加热到90°C的溶解过程。表达水平计算公式为2–ΔCt, ΔCt = Ct(目的基因)–Ct()。每个样品设3个试验重复, 生物学重复3次。数据均用Meas±SE, 用SPSS19.0软件进行单因素方差分析,<0.05有意义。
2 结果与分析
2.1 芜菁BrSIZ1基因的cDNA克隆及序列分析
参考拟南芥基因序列设计引物, 以津田芜菁cDNA为模板, 克隆了基因序列, 将该序列用NCBI的BLAST程序检索数据库, 显示该序列与拟南芥具有极高的同源性, 高达90%以上, 命名为, GenBank登录号为KY441465。基因序列全长2754 bp, 开放阅读框ORF区长2571 bp, 该基因序列编码一种具有856个氨基酸残基的多肽。
氨基酸序列分析显示, BrSIZ1相对分子质量为94.3kD, 等电点为5.2。BLAST分析结果显示BrSIZ1含有5个保守的功能域, 分别是N-SAP(I)、PHD(II)、PINIT(III)、SP-RING(IV)功能域、SXS(V)功能域和一个NLS (图1和图2)。将津田芜菁BrSIZ1氨基酸序列与油菜(; XP_013697486.1)、亚麻芥(; XP_010443841.1)、拟南芥(; OAO90655.1)、遏蓝菜(; JAU05124.1)、甜杏(; ALI97585.1)、甘蓝(var.; XP_013628264.1)、醉蝶花(; XP_010535588.1)、枣(; XP_015874378.1)、葡萄(; XP_01065 1133.1); 胡桃(; XP_018819476.1)、甜瓜(; XP_008463667.1)、梅花(; XP_008225245.1)同源蛋白的氨基酸序列利用MEGA5.0软件, 根据邻位相接法(Neighbor- joining)构建系统进化树(图3)。结果表明, 津田芜菁BrSIZ1与油菜的同源蛋白亲缘关系最近, 与同属十字花科的亚麻芥、拟南芥、遏蓝菜、油菜和甘蓝聚为一支, 而其他的葡萄、甜瓜、枣、胡桃、甜杏和梅花聚在一支。

图1 BrSIZ1的结构功能域(AtSIZ1: SIZ1OAO90655.1)
功能域包括一个N-SAP(I), 一个PHD(II), 一个PINIT(III, 一个SP-RING(IV)功能域和一个SXS(V)功能域。
The domains include: an N-terminal SAP (I, scaffold attachment factor A/B/acinus/PIAS); the PHD (II, plant homeodomain); the putative PINIT core domain (III); the SP-RING (IV, SIZ/PIAS-RING) domain; and the SXS (V, Ser-X-Ser) domain.
2.2 BrSIZ1在洋葱表皮细胞中的亚细胞定位
对拟南芥、番茄等物种SIZ1蛋白定位研究发现, 该蛋白定位于细胞核, 所以本研究构建了-GFP瞬时表达载体(图4), 在洋葱表皮细胞中进行了亚细胞定位研究。将BrSIZ1-GFP转入洋葱表皮细胞中, 培养16 h后, 激光共聚焦显微镜观察。结果如图5所示, 在融合蛋白表达载体转化的洋葱表皮细胞中, 绿色荧光集中分布在细胞核中, 表明BrSIZ1蛋白定位于细胞核中。
2.3 芜菁BrSIZ1基因组织特异性表达
对基因在芜菁红色根皮、白色根皮、去皮根、花、叶和幼苗不同组织中的表达分析(图6)发现, 津田芜菁基因在检测组织中都表达, 白根皮中表达量最少, 叶子中的表达最高, 是在白根皮中的2.0倍。在有花青素合成的红色根皮、幼苗和花中次之, 分别是根皮的1.6、1.6和1.5倍。在白色根皮中表达量最少, 具有一定的组织表达特异性。

图2 津田芜菁BrSIZ1基因核苷酸序列及其推导的氨基酸序列(最后框内为核定位信号)

图3 不同植物SIZ1氨基酸序列系统进化树
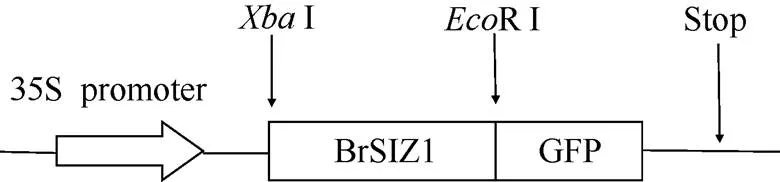
图4 pBrSIZ1-GFP表达载体示意图
2.4 UV-A光诱导BrSIZ1基因在芜菁根皮中的表达
取2个月龄的芜菁白色根皮进行UV-A光照诱导花青素合成, 分别取照射0、3、6、12、24和48 h检测根皮中花青素含量表明, 在UV-A光照6 h后花青素含量未见明显升高, 12 h后有微量花青素开始合成, 并且在24 h和48 h后花青含量持续增高(图7)。分别提取UV-A诱导不同时间后的津田芜菁根皮总RNA, 对基因进行定量PCR分析。结果显示UV-A光照6 h时的表达开始增加, 12 h时表达量最高, 表达量达到未处理时的2.8倍。之后24 h和48 h时表达量开始下降, 降低至未处理时的1.9倍和1.8倍(图8)。表明在根皮中的表达受UV-A光诱导,基因表达量与UV-A诱导花青素合成是一致的(图7和图8)。花青素合成和基因的表达趋势相比存在滞后, 这可能与启动花青素合成相关基因表达需要时间有关。
2.5 BrSIZ1基因在温度胁迫下的表达
取4 d龄津田芜菁幼苗在4°C低温和37°C高温处理, 检测基因的表达。结果表明在4°C低温处理3 h时,基因表达量开始增加, 24 h内基因表达量持续增加, 24 h时的表达量增加至未处理时的5.8倍(图9)。在37°C高温处理3 h时,基因表达量也开始增加直到处理12 h时达到最高, 达到未处理时的4.4倍, 处理24 h时表达量开始下降(图9)。说明基因在津田芜菁中参与温度胁迫应答。
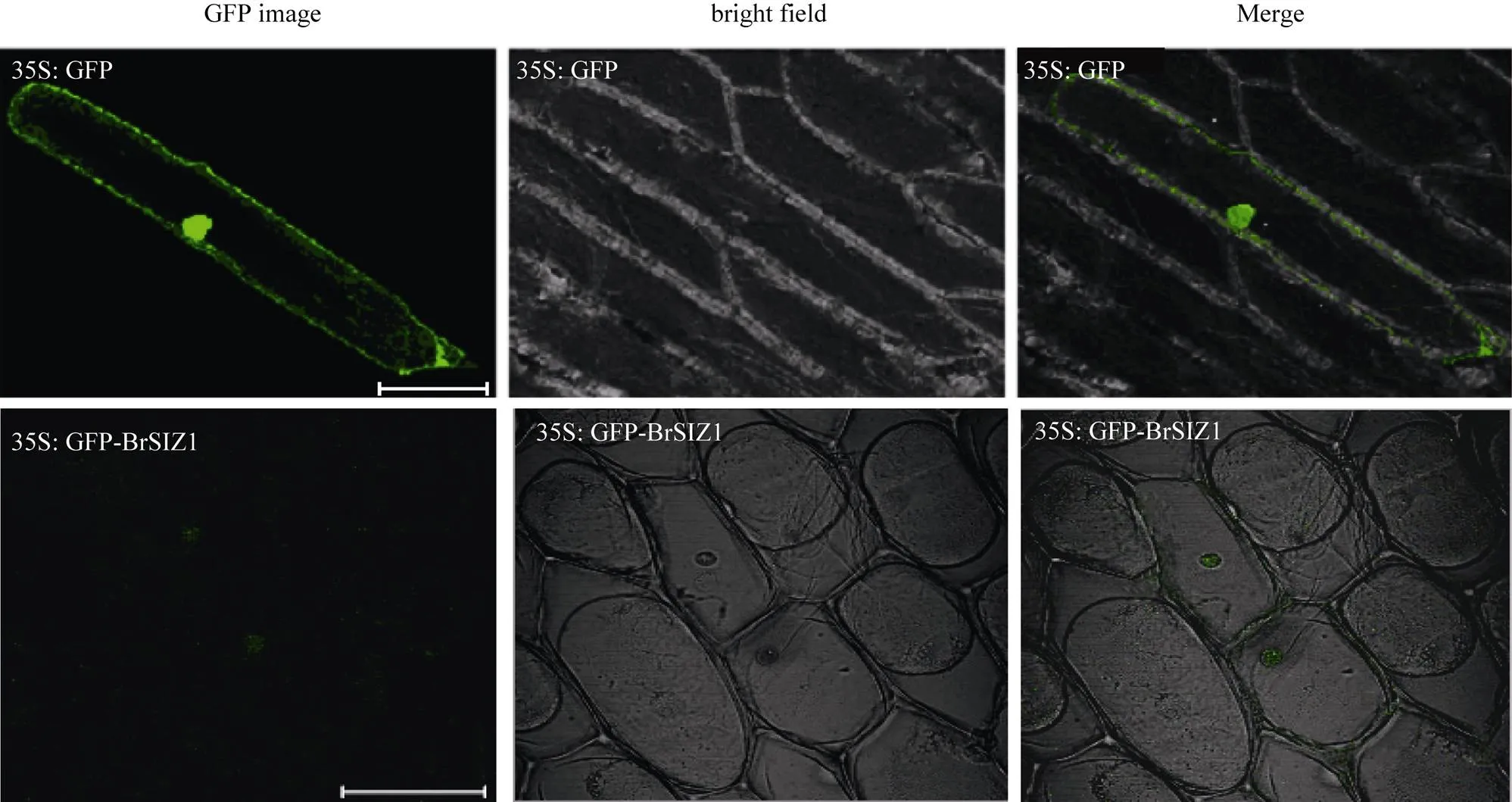
图5 GFP和BrSIZ1-GFP在洋葱表皮细胞中的亚细胞定位
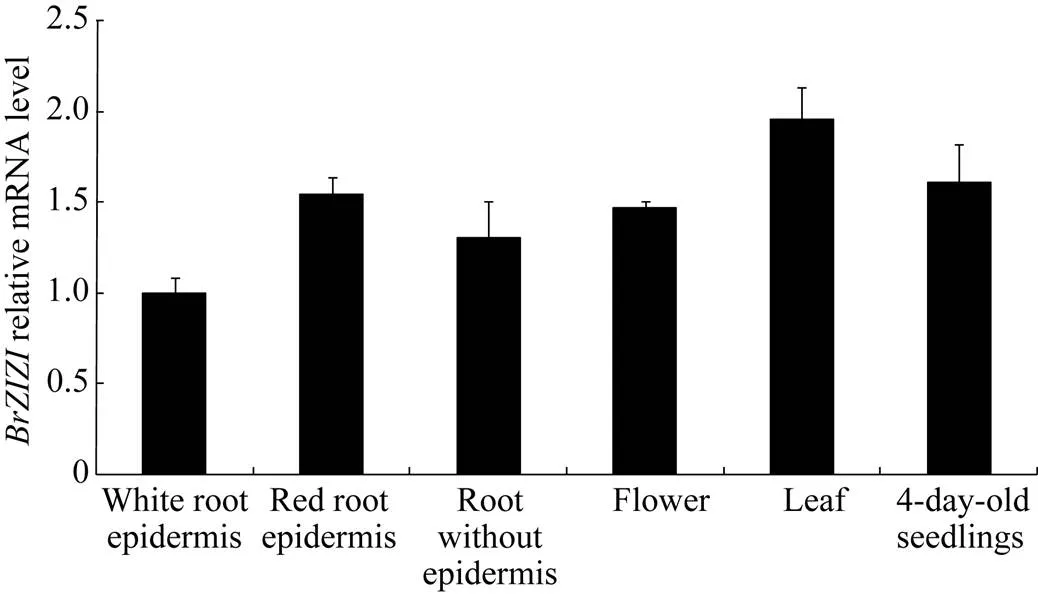
图6 定量PCR分析津田芜菁不同组织中BrSIZ1基因的表达
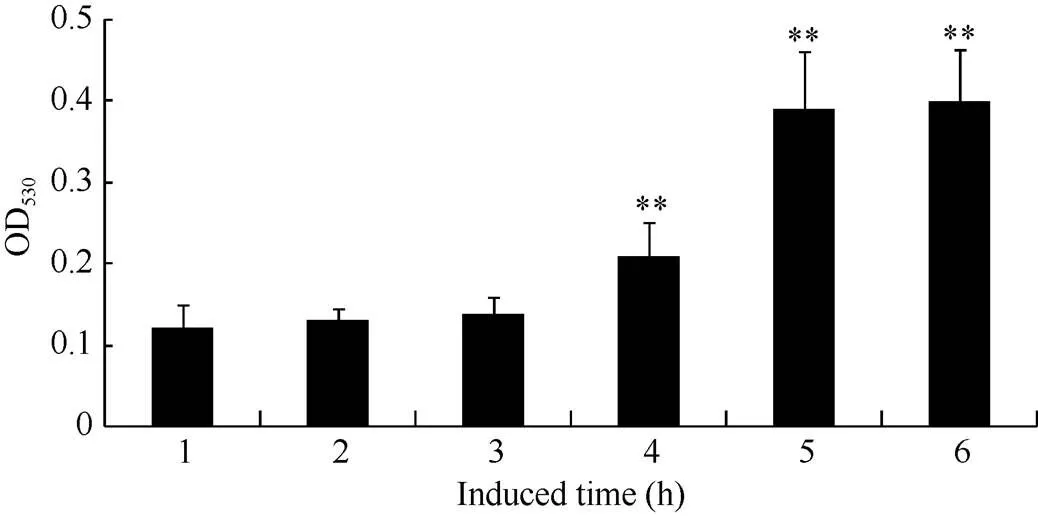
图7 UV-A光照诱导津田芜菁白色根皮花青素合成含量

图8 定量PCR分析UV-A光照诱导芜菁白根皮BrSIZ1基因表达

图9 定量PCR分析4°C、37°C处理芜菁4天幼苗BrSIZ1基因表达
3 讨论
本研究克隆了津田芜菁蛋白质翻译后修饰SUMO化的E3连接酶基因, 该基因cDNA序列含有一个2571 bp的开放读码框, 编码一个856个氨基酸的蛋白质。该蛋白含5个保守的功能域一个NLS。这些特点与已报道其他物种SIZ1蛋白结构相似[5-8]。BrSIZ1与油菜、亚麻芥、拟南芥进化上亲缘关系最近。BrSIZ1氨基酸序列在822~846氨基酸之间存在核定位信号, 该信号为双分型的NLS (bipartite NLS), 是由两簇碱性亮氨酸残基组成, 两簇碱性亮氨酸之间被10~12个非保守氨基酸残基间隔, 序列为KR(X)10-12RRKK[23]。BrSIZ1蛋白被定位于细胞核(图5), 与拟南芥、水稻、石斛兰等物种研究结果一致[6,11,24]。
SUMO化修饰参与植物生长发育调控,基因在芜菁的各个组织中都表达, 且在叶子、根皮、幼苗、花中表达量较高, 具有一定的组织表达特异性。与在拟南芥、石斛兰等物种中一样, 在根、茎、叶、花瓣的各组织中都表达, 参与调控植物多种组织的生长发育[16,24]。拟南芥SIZ1蛋白参与调节叶片细胞分裂、根部细胞增殖、调控植物开花时间等过程[9]。拟南芥SIZ1突变体发育迟缓, 叶序、叶片大小、形状以及许多生理反应上均表现出缺陷型[26]。芜菁基因在叶子、根表达量相对高, 可见SUMO E3连接酶在芜菁叶片和根等器官发育上发挥重要作用。最近研究表明SUMO调控分生组织骨架, 控制有丝分裂细胞周期到核内周期转变、着丝粒解凝、基因转录与沉默的关键分子开关, 这很可能就是SUMO作为真核生物细胞与个体生长发育重要调控机制的原因[25]。
津田芜菁根皮花青素的合成受UV-A光的特异诱导[19,22],基因在有花青素合成的根皮中表达, 且在UV-A光诱导的花青素合成过程中表达量增加, 说明BrSIZ1蛋白可能间接参与调控了花青素的合成。E3连接酶可以介导HY5 (ELONGATED HYPOCOTYL 5)、COP1 (constitutively photomorphogenic 1, COP1)在内的多种植物光信号通路相关蛋白的活性[19,22]。在黑暗中COP1蛋白与转录因子HY5等结合作用于苯基苯乙烯酮合酶(chalcone synthase, CHS)、叶绿素/结合蛋白CAB等基因启动子区, 抑制这些基因表达, 光下则脱离, 基因开始表达[26]。其中CHS是花青素合成途径中第一个关键酶, 津田芜菁BrSIZ1蛋白可能通过该途径调控花青素合成。
此外, SUMO化修饰还参与植物胁迫应答反应, 津田芜菁BrSIZ1蛋白在4°C低温和37°C高温中表达量增加, 参与温度胁迫应答。这与在拟南芥研究结果一致[14,18], 但目前还不清楚基因温度响应机制。
4 结论
克隆了津田芜菁基因, 该基因ORF全长2571 bp, 编码856个氨基酸多肽。BrSIZ1蛋白定位于细胞核。基因表达具有组织特异性, 且在UV-A光诱导的花青素合成和4°C、37°C胁迫的幼苗中, 表达量增加。
[1] Girdwood D W H, Tatham M H, Hay R T. SUMO and transcriptional regulation., 2004, 15: 201–210
[2] Johnson E S. Proteion modification by SUMO., 2004, 73: 355–382
[3] Novatchkova M, Budhiraja R, Coupland G. SUMO conjugation in plants., 2004, 220: 1–8
[4] Johnson E S, Gupta A A. An E3-like factor that promotes SUMO conjugation to the yeast septins., 2001, 106: 735–744
[5] Sharrocks A D. PIAS proteins and transcriptional regulation-more than just SUMO E3 ligases., 2006, 20: 754–758
[6] Bienz M. The PHD finger, a nuclear protein-interaction domain., 2006, 31: 35–40
[7] Aravind L, Koon E V. SAP—a putative DNA-binding motif involved in chromosomal organization., 2000, 25: 112–114
[8] Miura K, Jin J B, Hasegawa P M. Sumoylation, a post-translational regulatory process in plante., 2007, 10: 495–502
[9] Huang L X, Yang S G, Zhang S C. TheSUMO E3 ligase AtMMS21, a homologue of NSE2/MMS21, regulates cell proliferation in the root., 2009, 60: 666–678
[10] Jin J B, Jin Y H, Lee J. The SUMO E3 ligase AtS1Z1 regulates flowering by controlling a salicylic acid-mediated floral promotion pathway and through affects on FLC chromatin structure., 2008, 53: 530–540
[11] Thangasamy S, Guo C L, Chuang M H, Lai M H, Chen J, Jauh G Y. Rice SIZ1, a SUMO E3 ligase, controls spikelet fertility through regulation of anther dehiscence., 2011, 189: 869–882
[12] Kurepa J, Walker J M, Smalle J. The small ubiquitin-like modifier (SUMO) protein modification system inaccumulation of SUMO1 and -2 conjugates is increased by stress., 2003, 278: 6862–6872
[13] Lee J Y, Nam J, Park H C, Na G. Salicylic acid-mediated innate immunity inis regulated by SIZ1 SUMO E3 ligase., 2007, 49: 79–90
[14] Miura K, Jin J B, Lee J, Yoo C Y, Stirm T, Ashworth E N, Bressan R A, Yun D J, Hasegawa P M. SIZ1-mediated sumoylation of ICE1 controls CBF3/DREB1A expression and freezing tolerance in., 2007, 19: 1403–1414
[15] Yoo C Y, Miura K, Jin J B. SIZ1 (small ubiquitin-like modifier) E3 ligase facilitates basal thermotolerance inindependent of salicylic acid., 2006, 142: 1548–1558
[16] Catala R, Ouyang J, Abreu I A. The Arabidopsis E3 SUMO ligase SIZ1 regulates plant growth and drought responses., 2007, 19: 2952–2966
[17] Zhang S, Zhuang K, Wang S, Lv J, Ma N N, Meng Q W.A novel tomato SUMO E3 ligase, SlSIZ1, confers drought tolerance in transgenic tobacco., 2017, 59: 102–117
[18] Calderon-Villalobos L I, Nill C, Marrocco K. The evolutionarily conservedF-box protein AtFBP7 is required for efficient translation during temperature stress., 2007, 392: 106–116
[19] Zhou B, Li Y, Xu Z, Yan H, Homma S, Kawabata S. Ultraviolet A-specific induction of anthocyanin biosynthesis in the swollen hypocotyls of turnip ()., 2007, 58: 1771–1781
[20] Kawabata S, Kusahara Y, Li Y, Sakiyama R. The regulation of anthocyanin biosynthesis inunder low light conditions., 1999, 68: 519–526
[21] Frohman M A, Dush M K, Martin G R. Rapid production of full-length cDNAs from rare transcripts: amplification using a single gene-specific oligonucleotide primer., 1988, 85: 8998–9002
[22] Zhou B, Zhao X, Kawabata S, Li Y. Transient expression of a foreign gene by direct incorporation of DNA into intact plant tissue through vacuum infiltration., 2009, 31: 1811–1815
[23] Dingwall C, Robbins J, Dilworth S M, Roberts B, Richardson W D. The nucleoplasmin nuclear location sequence is larger and more complex than that of SV-40 large T antigen., 1988, 107: 841–849
[24] Liu F, Wang X, Su M Y, Yu M Y, Zhang S C, Lai J B, Yang C W, Wang Y Q. Functional characterization of DnSIZ1, a SIZ/ PIAS-type SUMO E3 ligase from Dendrobium., 2015, 15: 225–239
[25] Huang X, Ou-yang X, Deng X W. Beyond repression of photomorphogenesis: role switching of COP/DET/FUS in light signaling., 2014, 22: 96–103
[26] Lay H A, Sudip C, Ning W, Tokitaka O, Alfred B, Deng X W. Molecular interaction between COP1 and HY5 defines a regulatory switch for light control ofdevelopment., 1998, 1: 213–222
Cloning, Location, and Expression ofinL. subsp.‘Tsuda’
LUO Yun, MA Xuan, GU Jun-Chen, and YAN Hai-Fang*
College of Life Sciences, Northeast Forestry University, Harbin 150040, Heilongjiang, China
SIZ1, a SUMO E3 ligase involved in post-translation of proteins in plant cells, plays a role in protein interaction, location, and response to environmental stresses. In order to elucidate the expression profile of SIZ1 in Tsuda, cDNA ofgene was isolated from Tsuda. This gene was named(GenBank accession number KY441465).was 2754 bp in full length cDNA and 2571 bp in full length open reading frame (ORF), encoding a peptide with 856 amino acids. A BrSIZ1-GFP expression vector was constructed to analysis the subcellular localization. BrSIZ1-GFP was localized to nucleus, indicating that BrSIZ1 may play an important role in the nucleus. Quantitative-PCR analysis showed that thewas expressed meetly in leaf and secondly in young seedling and red root epidermis, showing tissue specificity. The expression of thewas induced by UV-A light in the root epidermis. The transcript level ofwas up-regulated when treated with temperature of 4°C or 37°C in young seedling.
;; gene cloning; subcellular location; expressing analysis
2017-03-07
2017-07-23;
2017-08-02.
10.3724/SP.J.1006.2018.000075
通信作者(Corresponding author): 闫海芳, E-mail: yanhaifang224@126.com
本研究由中央高校基本科研业务费专项(DL12CA10)和东北林业大学科研训练项目(KY2015053)资助。
This study was supported by the Fundamental Research Funds for the Central Universities (DL12CA10) and the Research Training Program of Northeast Forestry University (KY2015053).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20170802.2156.006.html
