高粱抗丝黑穗病分子遗传机制研究进展
2017-12-28白春明陆晓春
白春明,陆晓春
(辽宁省农业科学院创新中心,辽宁沈阳 110161)
高粱抗丝黑穗病分子遗传机制研究进展
白春明,陆晓春
(辽宁省农业科学院创新中心,辽宁沈阳 110161)
抗丝黑穗病是高粱品种审定考虑的最重要的指标之一。尽管接种能有效鉴定和培育抗丝黑穗材料或品种,但高粱抗丝黑穗病基因和分子机制还不清楚,限制了该抗病基因在育种和材料抗性鉴定中的应用。文中简要综述了高粱丝黑病遗传及基因定位,探讨了高粱丝黑穗病抗病分子机理,并介绍了高粱抗丝黑穗病全基因关联分析初步结果。高粱抗丝黑穗病基因克隆与分子标记鉴定有利于提高高粱丝黑穗病抗性材料鉴定的可靠性和精准性,提升抗丝黑穗育种效率,也将为高粱抗丝黑穗病分子机制研究提供候选基因。
高粱:丝黑穗病;基因克隆;GWAS
高粱[Sorghum bicolor(L.)Moench]又称蜀黍,属禾本科,是人类重要的粮食作物之一,世界总产在粮食作物中排在第五位,位于玉米、水稻、小麦、大麦之后(刘凤,2015;王成,2014)。具有C4植物的高光效、抗旱、耐涝和耐盐碱等特性,目前仍是非洲干旱等地区主要粮食作物。高粱不仅可以食用、饲用,还可以酿酒和生产生物乙醇等,用途广泛。酿酒是中国高粱的主导产业,近年来广东等省年进口近千万吨高粱用于饲用,高粱主导产业链有望随市场需求扩展。高粱在我国农业产业结构调整过程中发挥了重要作用,种植总面积稳中有升,为农民创收做出了重要贡献。
高粱丝黑穗病[Sporisorium reilianum(Kühn)Langdon&Fullerton]是我国高粱生产上的重要病害之一,尤其在高粱主产区东北三省和内蒙古是丝黑穗病的重发区。辽宁省高粱品种审定对于品种的丝黑穗病抗性采取一票否决制,每年都要通过接种,对参加区域试验的品种进行抗性鉴定。20世纪90年代我国高粱丝黑穗病菌的3号小种引发高粱丝黑穗病大爆发,平均发病率18%,高者达80%以上,产量损失严重(徐秀德,刘志恒.2012)。因此高粱丝黑穗病的防治对于高粱安全生产具有重要意义(Poland,etal.,2011;Wang,etal.,2012;Wang,et al.,2013;Liu,etal.,2014)。本文将对高粱丝黑穗病不同生理小种致病力、基因遗传和抗病分子机制等研究进行归纳,有助于人们更好的了解高粱丝黑穗病分子遗传机制进展,希望尽快能将抗丝黑穗病基因和分子标记用于高粱抗性品种培育和材料抗性鉴定中。
1 高粱丝黑穗病发生及生理小种变异
高粱的丝黑穗病是由孢堆黑粉菌[Sporisorium reilianum(Kühn)Langdon&Fullerton](属真菌界,担子菌门,孢堆黑粉菌属)引起的,是高粱主要土传真菌病害之一(Dodman,R.L.,et al.,1985)。丝黑穗病菌主要在高粱幼芽出土前侵染幼苗中胚轴,其次为胚芽鞘和胚根。高梁孢堆黑粉菌的菌丝从中胚轴或根部侵入到高梁体内之后,植株并不很快出现典型的可见病症,病原菌处于潜伏期,直到高梁幼穗开始分化时,潜伏在体内的菌丝才开始大量增生,最终在穗部产生黑粉病症(Leslie,J.F.,2013)(卢庆善,1999)。由于该病直接破坏高粱果穗,对高粱产量造成严重威胁(张春来等,2013),但也有人利用感病品种收集黑粉提取氨基酸等活性成分,深加工成保健品等(石太渊等,2012;王辉等,2006)。土壤中病菌含量、土壤温湿度、播种深度、出苗快慢、品种抗病性等因素都影响高梁丝黑穗病的发生(徐秀德,刘志恒.2012)。由于影响因素较多,按照抗性评价标准高粱丝黑穗病共分0~9级,材料和品种抗性鉴定通常采用多年和多点鉴定结果,尤其在鉴定抗性表型时,要保证抗性鉴定结果的可靠性,种植群体至少保证在100株以上。因此,抗丝黑穗病基因克隆将能极大提高抗性鉴定的可靠性、精准性,并减少抗性鉴定的田间工作量。
在不同国家发现的高粱丝黑穗病菌生理小种数量不同,美国有6个生理小种,其主要鉴别寄主为 Tx7078、SA281、Tx414和 TAM2571(Prom,etal.,2011),墨西哥有3个生理小种(Herrera,et al.,1986),在我国已有 4个生理小种(杨慧勇等,2014)。不同生理小种致病力有明显差异(Frederiksen,R.A.,1986)。1号小种对中国高粱“三尺三”和“护4号”致病力强,对甜高粱苏马克致病力弱,对白卡佛尔和 Tx3197A几乎不侵染;2号小种对Tx3197A、白卡佛尔和甜高粱苏马克致病力强,而对中国高粱和甜玉米致病力弱(吴新兰,1982)。3号生理小种能侵染1、2号小种不侵染的Tx622A和Tx622B,具有很强的致病力,中国生理小种与美国生理小种相比具有完全不同的毒力特征(徐秀德等,1994)。4号小种对A2V4、晋杂12号有较强的致病力,而对3号小种致病的7501B,Tx7078,B35不能侵染(张福耀等,2005;Zhang F.Y.,et al.,2011)。对4个生理小种都表现免疫或高抗的育种材料较少(杨慧勇等,2014)。从这些研究结果来看,高粱生理小种分化简单,但由于不同国家使用的鉴别寄主不同,我们尚不能确认各国不同生理小种是否有含有相同的致毒基因。从我国4个生理小种对不同品种的致病力来看,可推断高粱丝黑穗病不同生理小种抗病分子机制有可能符合基因一对一关系。尽管丝黑穗病3号生理小种是我国东北高粱产区主要流行的生理小种,但要获得广谱持久的抗丝黑穗品种,理论上应通过4个生理小种的抗性基因的聚合。因此,高粱不同丝黑穗病生理小种抗性的精细定位与聚合对于高粱抗丝黑穗病品种培育和安全生产具有重要意义。
2 高粱抗丝黑穗病遗传及基因定位
解析作物产量和抗性等性状的分子遗传机制,有助于人们更有效的利用种质资源培育新品种和利用新技术满足人类粮食安全的需要。作物重要农艺性状遗传的复杂程度决定着性状在育种中的选择效率和选择方法,也影响该性状基因定位效率。曹如槐(1988)等用人工接种1号小种,按照不完全双列杂交设计对17个抗性不同的品种丝黑穗病抗性进行了遗传研究,结果表明高粱对丝黑穗病的抗性遗传方式因品种而异,有的品种具数量性状遗传特点,有的则具有质量性状遗传特点,抗病性属数量性状遗传的品种,其抗性主要是受加性基因控制。徐秀德(2000)等在人工接种条件下,利用6个抗性不同的高粱种质不完全双列杂交设计,对高粱对丝黑穗病菌3号小种的抗性进行了遗传研究,结果表明多数试材的抗、感性遗传受质量性状控制,只要亲本之一为免疫,则F1表现免疫或高抗,可能是1~2对非等位主效基因的作用。也有研究表明高粱对丝黑穗病菌3号生理小种的抗性可能受2对彼此独立的非等位基因控制,并且基因之间存在着互作效应(Li,et al.,2012)。Oh等(1994)以SC325×RTx7078杂交的F2和F3材料,用 RFLP和 RAPD技术,对美国高粱丝轴黑粉菌5号小种的抗性位点Shs1进行分子标记,发现它与RFLP位点pSbTXS560和pS-bTXS1294,RAPD位点 OPG5连锁,定位于第8染色体94.1 cM处。邹剑秋(2010)等利用恢复系分离群体(2381R/矮四)和保持系分离群体(Tx622B/7050B),筛选得到了2个在抗病品系中稳定出现、可作为高粱抗丝黑穗病3号生理小种基因的SSR标记,Xtxp13和Xtxp145,其分别位于第2号染色体和第9号染色体上,距离抗病位点的遗传图距分别约为9.6 cM和10.4 cM。由于丝黑穗病不是美国等国外高粱生产的主要病害,总体上国内外丝黑穗病分子遗传相关研究还较少。从现有研究来看,尽管高粱丝黑穗病抗性遗传多数报道为质量性状,但由于丝黑穗病基因定位使用标记数量少,利用的是临时定位群体而不是永久群体,以及群体抗病性鉴定表型结果不准确等原因,目前这些定位结果可靠性及精细程度还有待提高。因此利用永久群体和测序技术是较快定位和克隆高粱抗丝黑穗病基因的有效途径。
3 高粱丝黑穗病抗病分子机理
植物分子免疫系统有通过识别受体PRR(pattern recognition receptor)引起的免疫反应 PTI(PAMPs-triggered immunity)和通过识别病原菌的效应蛋白引起免疫反应ETI(Effector-triggered immunity)两种模式。PTI免疫系统包括细菌的鞭毛、延伸因子EF-Tu以及真菌的几丁质,寡聚半乳糖醛酸、角质单体、系统素等引起的免疫反应。ETI免疫系统是病原菌在感染植物后,可以分泌效应蛋白到植物细胞内,效应蛋白通过抑制植物的PTI等方式使病原菌逃避植物的免疫反应,增加病原菌的致病性。植物的R基因编码的是一类NBS-LRR类的胞内受体蛋白,能够识别来源不同的病原菌分泌的效应蛋白,可能引起ETI的抗病反应。
有关高粱抗丝黑穗病的分子机理还不清楚,也没有抗病基因克隆的报道,但从高粱丝黑穗病小种专化性来看,有可能目前中国的4种生理小种抗性应属于ETI抗性系统。玉米丝黑穗病抗性遗传属于数量性状,从克隆的玉米丝黑穗病抗性基因来看,该基因ZmWAK是一类跨膜的激酶蛋白,在植物细胞内起到受体激酶传导外界信号的作用(Chen,et al.,2008;Zhao,et al.,2012;Zuo,et al.,2014),WAK可能作为受体类激酶“监视”果胶的完整性,识别病原菌攻击细胞壁过程中植物细胞产生的损伤相关分子模式DAMPs(damage-associated molecular patterns)从而诱导抗病反应的发生(Hematy,et al.,2009),进而继续诱导ETI的抗病反应。尽管高粱有玉米抗丝黑穗病基因ZmWAK的同源基因(sb05g027230)(氨基酸比对51.76%,见图1),但高粱丝黑穗病菌和玉米丝黑穗病菌有不同的致病力,玉米丝黑穗菌系不能侵染高粱,高粱丝黑穗病菌虽能侵染玉米,但侵染力极低,是同一个种的两个不同生理分化型(吴新兰,1982;姜钰等,2014),因此该基因也不能间接说明高粱抗丝黑穗病分子机制。高粱NBS-LRR抗病基因有200多个(Paterson,et al.,2009;Cheng,et al.,2010),但在这些 NBS基因中是否与抗丝黑穗病相关尚未报道。因此,精细定位和克隆高粱抗丝黑穗病基因不仅能利用分子标记提高抗性育种效率,也有利于丝黑穗病免疫分子机制研究。
4 高粱抗丝黑穗病全基因关联分析
传统植物抗病基因定位的主要方法是利用分离群体结合SSR等分子标记遗传图谱对抗病基因进行QTL定位。由于分离群体仅涉及两个特定的亲本材料,因此连锁分析只涉及到同一座位的两个等位基因(杨小红等,2007),同时在构建分离群体时由于杂交和自交次数的限制,发生的重组次数有限,QTL作图精度一般在10~30 cM之间(Flint-Garcia,et al.,2005)。随着近年来高通量测序技术的迅猛发展和多种作物全基因组测序的完成,基于测序技术进行基因定位和克隆植物数量性状基因已成为目前国际植物基因组学研究的热点,如水稻的14个农艺性状(Huang,et al.,2010)、玉米小斑病(Kump,et al.,2011)、玉米过敏性防御反应的全基因组关联分析(GWAS)(Olukolu,et al.,2014)、大豆 8个农艺性状利用GBS-GWAS定位(Sonah et al.,2015)、拟南芥对寄生疫霉抗性(Meng et al.,2015)等。Zhao,X.等 2015年利用GWAS和遗传图谱定位了大豆抗核菌病。Han,Y P.等人(2015)利用SLAF简化测序,进行大豆胞囊线虫抗性的GWAS分析,获得8个位点与已经报道的Rhg4及Rhg1是重合的,又发现3个新位点。Zhao,X.等人(2017)用200种不同的大豆品种对大豆胞囊线虫(SCN)HG类型2.5.7的抗性进行了筛选,通过GWAS揭示了13个SNP与SCN HG的2.5.7有关,总共30个基因被鉴定为潜在的SCN抵抗的候选基因。Wang等(2012)通过高密度的基因分型技术结合全基因组的关联分析,定位了玉米抗丝黑穗病相关的基因,对其中bin2.09位点进行了候选区段的丝黑穗病抗性基因的关联分析,并探讨可能存在的抗病机制。徐明良课题组前期研究中在bin2.09的位置上检测到一个主效 QTL,命名为 qHSR1(Chen et al.,2008;Zhao et al.,2012),Zuo,W.L.等(2014)人再利用重组个体后代检测法,建立亲本的BAC文库,测序后发现定位区间内黄早四(高感)比Mo17(抗病)缺失147Kb的DNA片段,进而进行抗病相关基因表达分析等确定ZmWAK为抗病候选基因。为了挖掘高粱抗丝黑穗病基因,辽宁省农业科学院分子遗传改良课题组基于简化基因组测序技术(SLAF-seq)获得了634份不同血缘的高粱材料的26万SNP,经4年重复接种高粱丝黑穗病菌,获得了236份材料抗感丝黑穗病的可靠表型数据,利用这些数据进行全基因组关联分析,在第8条染色体上鉴定出与抗丝黑穗病相关性的SNPs(图2),该区域对应5个候选基因,包括抗病相关基因,目前正在克隆和验证候选基因。
高粱与水稻和拟南芥等分子遗传研究模式植物相比,已经挖掘和鉴定的高粱重要农艺性状基因和分子标记数量少很多,但高粱基因组测序已经完成(Paterson et al.,2009),只有玉米基因组四分之一大小,而且高粱又是C4作物,因此高粱有潜力成为C4作物分子遗传研究的模式植物。

图1 玉米抗丝黑穗病基因ZmWAK与高粱同源基因氨基酸比对Figure 1 Comparison of head smut resistant gene ZmWAK in maize and homologous gene in sorghum
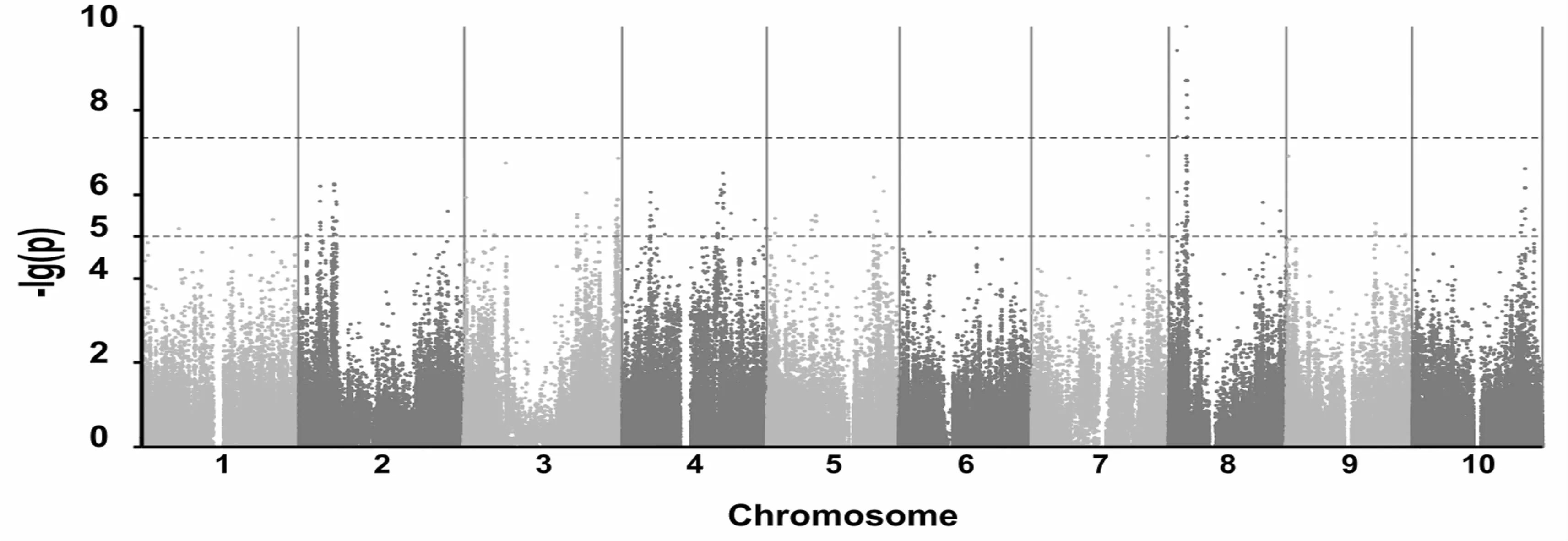
图2 高粱抗丝黑穗病混合线性模型全基因组关联分析Figure 2 Genome-w ide association study of head smut of sorghum using them ixed linear model
高粱丝黑穗病生理小种分化少,遗传简单,具有较大的遗传效应,只要抗性表型鉴定准确,容易利用基于测序的基因定位方法克隆该基因,从而为高粱抗丝黑穗病分子标记辅助育种提供实用分子标记,并为高粱抗丝黑穗病分子机制研究提供候选基因。
[1] 曹如槐,王晓玲,任建华,等.高粱对丝黑穗病的抗性及遗传研究[J].遗传学报,1988,15(3):170~173.
[2] 姜钰,徐秀德,胡兰,等.不同症状的玉米丝黑穗病病原菌rDNA-ITS分析[J].玉米科学,2014,22(2):150~154.
[3] 刘凤.高粱:从非洲来的“毒品”还是希望?[J].甘肃农业,2015(19):58~59.
[4] 卢庆善.高粱学(下)[M].北京:中国农业出版社,1999:393~397.
[5] 石太渊,于淼.高粱乌米的营养功能与加工利用研究[J].农业科技与装备,2012(8):67~70.
[6] 王成.高粱抗蚜性分子数量遗传研究[D].天津农学院,2014.
[7] 王辉,斯琴格日乐,马瑞辉,包海鹰.玉米黑粉菌孢子粉的无机元素和氨基酸含量测定[J].菌物研究,2006,4(1):20~23.
[8] 吴新兰.高粱丝黑穗病菌(Sphacelotheca reiliana(Kühn)Clint.)的生理分化[J].植物病理学报,1982,12(1):13~18.
[9] 徐秀德,董怀玉,杨晓光,等.高粱抗丝黑穗病菌3号小种遗传效应研究[J].杂粮作物,2000,(1):9~12.
[10] 徐秀德,刘志恒主编.高梁病虫害原色图鉴[M].北京:中国农业科学技术出版社,2012:51~62.
[11] 徐秀德,卢庆善,潘景芳.中国高粱丝黑穗病菌小种对美国小种鉴定寄主致病力测定[J].辽宁农业科学,1994(1):8~10.
[12] 杨慧勇,王华云,赵文博,等.高粱丝黑穗病抗源筛选及遗传多样性分析[J].山西农业科学,2014,42(12):1252~1255,1264.
[13] 杨小红,严建兵,郑艳萍,等.植物数量性状关联分析研究进展[J].作物学报,2007,33(4):523~530.
[14] 张春来,杨慧勇,柳青山,王花云,赵威军,张福耀,董良利.高梁抗丝黑穗病遗传与分子育种[J].山西农业科学,2013,41(3):201~206.
[15] 张福耀,平俊爱,杜志宏,等.山西高平高粱丝黑穗病致病力研究[J].植物病理学报,2005,35(5):475~477.
[16] 邹剑秋,李钥莹,朱凯,等.高梁丝黑穗病菌3号生理小种抗性遗传研究及抗病基因分子标记[J].中国农业科学,2010,43(4):713~720.
[17] Chen Y SQing Chao,Tan G Q,et al.Identification and fine mapping of a major QTL conferring resistance against head smut in maize[J].Theor Appl Genet,2008(117):1241~1252.
[18] Cheng X Jiang H Y Zhao Y,et al.A genomic analysis of disease-resistance genes encoding nucleotide binding sites in Sorghum bicolor[J].Genetics&Molecular Biology.2010(33):292~297.
[19] Dodman R L,Obst N R,Henzell R G.Races of sorghum head smut(Sporisorium reilianum)in South-East Queensland[J].Australasian Plant Pathology,1985,14(2):45.
[20] Flint-Garcia SA,Thuillet A C,Yu JM,etal..Maize association population:a high-resolution plat form for quantitative trait locus dissection[J].The Plant Journal,2005,44(6):1054~1064.
[21] Frederiksen R A,Odvody G N.Compendium of Sorghum Diseases[J].St Paul:American Phytopathology Society,1986:17~18.
[22] Han Y P,Zhao X,Cao G L,et al.Genetic characteristics of soybean resistance to HG type 0 and HG type 1.2.3.5.7 of the cyst nematode analyzed by genome-wide association mapping[J].BMCGenomics,2015,16(1):598.1~11.
[23] Hematy K,Cherk C,Somerville,S.Host-pathogen warfare at the plant cell wall[J].Current Opinion in Plant Biology,2009,12(4):406~413.
[24] Herrera JA,Vallejo A B.Distribution of races of head smut(Sporisorium reilianum)in the northeast and southwest areas of Mexico[J].Sorghum Newsletter,1986(29):86.
[25] Huang X H,Wei X H,Sang T,et al.Genome-wide association studies of14 agronomic traits in rice landraces[J].Nature Genetics,2010,42(11):961~967.
[26] Kump K L,Bradbury P J,Wisser R J,et al.Genome-wide association study of quantitative resistance to southern leaf blight in themaize nested associationmapping population[J].Nature genetics,2011,43(2):163~168.
[27] Leslie JF.Sorghum and Millets Diseases[M].Wiley-Blackwell,2013.
[28] Li Y Y,Zou JQ.,Ma CY,etal.Developmentof Head Smut Resistance-linked Sequence Characterized Amplified Regions Markers in Sorghum[J].Int.J.Agric.Biol,2012,14(4):613~616.
[29] Liu C L,Weng JW,Zhang DG,etal.Genome-wide association study of resistance to rough dwarf disease in maize[J].Eur JPlant Pathol,2014,139:205~216.
[30] Meng Y L,Huang Y H,Wang Q H,etal.Phenotypic and genetic characterization of resistance in Arabidopsis thaliana to the oomycete pathogen Phytophthora parasitica[J].Frontiers in Plant Science,2015(6):378.
[31] Oh B J,Frederiksen R A,Magill CW.Identification ofmolecularmarkers linked to head smut resistance gene(Shs)in sorghum by RFLP and RAPD analyses[J].Phytopathology,1994,84(8):830~833.
[32] Olukolu B A,Wang G F,Vontimitta V,et al.A Genome-Wide Association Study of the Maize Hypersensitive Defense Response Identifies Genes That Cluster in Related Pathways[J].Plos Genetics,2014,10(8):761~768.
[33] Paterson A H,Bowers JE.The Sorghum bicolor genome and the diversification of grasses[J].Nature,2009,457:551~556.
[34] Poland JA,Bradbury P J,Buckler E S,et al.Genome-wide nested association mapping of quantitative resistance to northern leaf blight in maize[J].proceedings of the national academy of sciences of the united states of America,2011,108(17):6893~6898.
[35] Prom L K,Perumal R,Erattaimuthu S,et al.Virulence and molecular genotyping studies of Sporisorium reilianum isolates in sorghum[J].Plant Disease,2011(95):523~529.
[36] Sonah H,O'Donoughue L,et al.Identification of loci governing eight agronomic traits using a GBS-GWAS approach and validation by QTLmapping in soya bean[J].Plant Biotechnology Journal,2015,13(2):211~221.
[37] Wang M,Yan JB,Zhao JR,et al.Genome-wide association study(GWAS)of resistance to head smut in maize[J].Plant Science,2012(196):125~131.
[38] Wang Y H,Acharya A,Burrell A M,et al.Mapping and candidate genes associated with saccharification yield in sorghum[J].NRC Research Press,2013,56(11):659~665.
[39] Zhao X,Han Y P.Loci and candidate gene identification for resistance to Sclerotinia sclerotiorum in sobean(Glycinemax L.Merr.)via association and linkage maps[J].The Plant Journal,2015,82(2):245~255.
[40] Zhao X,Teng W.Loci and candidate genes conferring resistance to soybean cyst nematode HG type 2.5.7[J].BMC Genomics,2017(18):462.1~10.
[41] Zhao X R,Tan G Q,Xing Y X,et al.Marker-assisted introgression of qHSRl to improve maize resistance to head smut[J].Molecular Breeding,2012(30):1077~1088.
[42] Zhang F Y,Ping JA,Du Z H,et al.Identification of a new race of Sporisorium reilianum and characterization of the reaction of sorghum lines to four races of the head smut pathogen[J].Phytopathology,2011,159(5):342~346.
[43] Zuo W L,Chao Q,Zhang N,et al.A maize wall associated kinase confers quantitative resistance to head smut[J].Nature Genetics,2014(18),1~7.
Research Progress on Molecular Genetic Mechanism of Head Smut Resistance of Sorghum
BAIChun-ming,LU Xiao-chun(Innovation Center,Liaoning Academy of Agricultural Sciences,Shenyang,Liaoning 110161)
Sorghum resistance head smut is one of the most concerned important indexes of sorghum varieties certification.Although head smut resistance varieties can be effectively identified and cultivated by the use of soil inoculation,the head smut resistance gene and molecularmechanism of sorghum is still unclear to date,and also limited the resistance genes application in the breeding and material resistance identification.In this review,we summarized the advance of the genetic location and resistance molecularmechanism of the head smut of sorghum,and briefly presented thewhole genome association analysis of sorghum head smut in our laboratory.The of gene cloning of sorghum resistance to head smutwill improve reliability and precision of identification of head smut resistance in sorghum lines,and also provide candidate gene to study its molecular mechanism.
Sorghum;Head Smut;Gene cloing;GWAS
S435.14
B
1002-1728(2017)06-0039-05
10.3969/j.issn.1002-1728.2017.06.008
2017-12-01
国家高粱产业技术体系(CARS-06-01-06);国家自然青年科学基金(31601627);辽宁省科学技术计划项目(2014027018)
白春明(1982-),女,副研究员,主要从事高粱抗病分子育种研究。E-mail:baichunming82@163.com通讯作者:陆晓春(1970-),男,研究员,主要从事作物分子育种研究。E-mail:luxiaochun2000@126.com
