2016年-2017年广东省部分地区猪繁殖与呼吸综合征病毒ORF5基因的遗传变异分析
2017-12-26于林洋董建国张乐宜刘燕玲梁鹏帅宋长绪
于林洋,董建国,2,张乐宜,刘燕玲,梁鹏帅,王 磊,宋长绪*
(1.华南农业大学动物科学学院,广东广州 510642;2.信阳农林学院牧医工程学院,河南信阳 464000)
2016年-2017年广东省部分地区猪繁殖与呼吸综合征病毒ORF5基因的遗传变异分析
于林洋1,董建国1,2,张乐宜1,刘燕玲1,梁鹏帅1,王 磊1,宋长绪1*
(1.华南农业大学动物科学学院,广东广州 510642;2.信阳农林学院牧医工程学院,河南信阳 464000)
为研究广东地区猪繁殖与呼吸综合征病毒(PRRSV)流行株ORF5基因的遗传演化情况,采用RT-PCR方法对2016年-2017年采自广东省地区猪场疑似PRRS猪肺脏组织样品中的PRRSV ORF5基因进行了扩增,并对这些毒株的核苷酸和氨基酸序列进行了生物信息学分析。结果表明,成功扩增出12株PRRSV流行毒株的ORF5基因片段。遗传演化分析表明,扩增出的毒株有10株与国内高致病性毒株亲缘关系较近,属于同一分支;2株与广东地区新报道的毒株QYYZ和GM2亲缘关系较近,属于同一分支。氨基酸序列比对结果表明,新分离的毒株在GP5抗原表位上存在广泛的变异。结果表明,该地区PRRSV毒株已经发生广泛变异。研究结果不仅揭示了广东地区流行的PRRSV ORF5基因的遗传演化特性,也为该地区PRRS的防控提供了科学依据。
猪繁殖与呼吸综合征病毒;ORF5 基因;变异分析
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是以引起母猪流产、弱胎和木乃伊胎为主的繁殖障碍性疾病和以各种年龄猪呼吸系统疾病为特征的一种重大传染病[1]。该病20世纪80年代首次报道,随后短短几十年间,该病在世界范围内广泛传播,给世界养殖业带来了巨大的经济损失[2]。
PRRS的病原是猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV),1991年在新西兰首次证实引起PRRS的病原是PRRSV,随后1992年美国也证实[3-5]。PRRSV是有囊膜的单股正链RNA病毒,属于套式病毒目、动脉炎病毒科、动脉炎病毒属成员[6]。PRRSV全长大约15 kb,包含至少10个开放阅读框(ORF):ORF1a,ORF1b,ORF2a,ORF2b,ORFs3-7以及最近发现的ORF5a[7]。ORF1a和ORF1b位于5′ UTR的下游,编码复制酶多聚蛋白pp1a和pp1b,pp1a和pp1b进一步水解成非结构蛋白(NSP)NSP1α,NSP 1β,NSP 2~NSP 6,NSP 7α和NSP 7β,NSP 8 ~ NSP 12[8]。ORF2a,ORF2b,ORFs3-7位于基因组的3′端,分别编码结构蛋白GP2,E,GP3,GP4,GP5,GP5a,M,N[9]。
根据基因组的差异性,PRRSV被分为两个基因型:以Lelystad virus为代表毒株的欧洲型PRRSV(1型)和以VR-2332为代表的北美型PRRSV(2型)。令人惊讶的是,PRRSV在两个大陆上似乎经历了不同的进化。这两种类型的毒株核苷酸同源性仅有60%左右,但是能够引起相似的症状[10]。PRRSV RNA聚合酶易于出错的特性和PRRSV不断地重组以及外界选择压力的共同作用,导致该病毒快速发生变异[11-12]。在所有基因中,NSP2区域和GP5基因是变异最大的区域,研究者经常选择NSP2和GP5基因进行PRRSV遗传演化分析[13]。
1995年中国首次证实有PRRS的存在,随后疾病迅速蔓延至中国大部分地区,给养猪业造成了巨大的经济损失。2006年,中国暴发了以持续高热、皮肤发红、高发病率和高病死率为特征的非典型性PRRS。该病迅速传遍全国,对中国养猪业造成了史无前例的打击。进一步研究证实该病是由NSP2区域存在30个不连续氨基酸的缺失为特征的高致病性PRRSV(HP-PRRSV)引起[14-15]。2014年,中国多地区报道NADC30-like毒株流行,致使现在市面上的商品化疫苗效果较差,给我国PRRS防控带来了巨大困难[16-19]。
本研究对2016年-2017年来自广东地区的可疑PRRSV猪肺脏病变组织进行病毒RNA/DNA的提取,并进行PRRSV ORF5的RT-PCR扩增。选取阳性PCR产物进行基因序列分析。同时选取国内外具有代表性的PRRSV毒株与所测定的基因序列进行遗传进化分析。根据分析结果得出广东省PRRSV的遗传进化趋势,为广东省内PRRSV防控提供有力的参考依据。
1 材料与方法
1.1 材料
1.1.1 病料 样品来自广东省疑似暴发PRRS的自然发病猪场,采集发病猪肺脏组织于-20℃冻存备用。
1.1.2 细胞、菌体及载体 本试验所使用Marc-145细胞,克隆宿主菌E.coliDH5α感受态细胞以及阳性对照病毒株GDzj株均由华南农业大学动物科学学院猪病防控研究室保存;pMD-19T克隆载体购自宝生物工程(大连)有限公司。
1.1.3 主要试剂 病毒DNA/RNA提取试剂盒为Magen公司产品;反转录试剂盒为Promega公司产品;RT-PCR相关试剂盒为Toyobo生物科技有限公司和Vazyme公司产品;DNA Marker DL 2 000等为TaKaRa公司产品;胶回收试剂盒为Promega公司产品;EB替代染料为广州华奇盛生物科技有限公司产品;pEASY-Blunt Simple Cloning Vector和Trans-5α感受态细胞均为北京全式金生物技术有限公司产品。
1.2 方法
1.2.1 引物设计与合成 根据GenBank中发布的VR-2332等PRRSV毒株全基因组序列进行生物信息学分析,选取保守区域设计1对特异性引物用于扩增ORF5基因序列。GP5-F:5′-GTGTCAGGCATTGTGGCTGTG-3;GP5-R:5′- CATTATTGGCGTGTAGGTGATAGAAAA-3′,扩增大小为821 bp。引物由苏州金维智生物公司合成。
1.2.2 ORF5基因的扩增及克隆 取0.5 g左右的肺脏组织剪碎置于无菌匀浆器中,加入1 mL灭菌PBS,于冰浴中匀浆处理后转移至1.5 m LEP管中,反复冻融3次,12 000 r/min离心5 min,取上清置于一新的1.5 mL EP管中。按TRIzol试剂盒操作说明书提取病料组织总RNA。以提取的总RNA为模板,采用AMV反转录酶进行RNA反转录制备cDNA。然后以cDNA为模板,GP5-F/GP5-R为引物,使用TOYOBO生物科技有限公司高保真酶对目的片段进行扩增。扩增体系为50 μL,KOD-Plus-Neo 1 μL,10×PCR buffer for KOD-Plus-Neo 5 μL,2 mmol/L dNTP 5 μL,25 mmol/L MgSO43 μL,cDNA模板4 μL,上游引物2 μL,下游引物2 μL,RNase Free H2O 28 μL。PCR扩增循环参数为:94℃ 2 min;98℃ 10 s,62℃ 30 s,68℃ 30 s,35个循环;72℃延伸10 min。取5 μL的PCR产物进行10 g/L琼脂糖凝胶电泳的观察。PCR阳性样品用凝胶回收后克隆于pEASY-Simple T1 Cloning Vector载体上,阳性重组质粒由苏州金维智生物公司测序。
1.2.3 序列比对与分析 利用Lasergene及Mega 5.0软件将对不同病毒株的ORF5基因序列进行序列比对并构建进化树,同时将GP5氨基酸序列与其他国内外代表毒株的GP5氨基酸序列进行比对分析。PRRSV代表毒株的基因序列均下载自GenBank。
2 结果
2.1 ORF5 基因的 RT- PCR 扩增结果
用特异性扩增PRRSV ORF5基因序列的引物对采集的20份疑似 PRRS 病料样品进行RT-PCR 扩增,图1结果显示,12份病料样品为阳性,编号分别为GDzj,GDkp,GDqy,GDjy,GDmz,GDsg,GDyf,GDgz,GDmm,GDhy,GDhf,GDhz。琼脂糖凝胶电泳检测结果与预期相符。
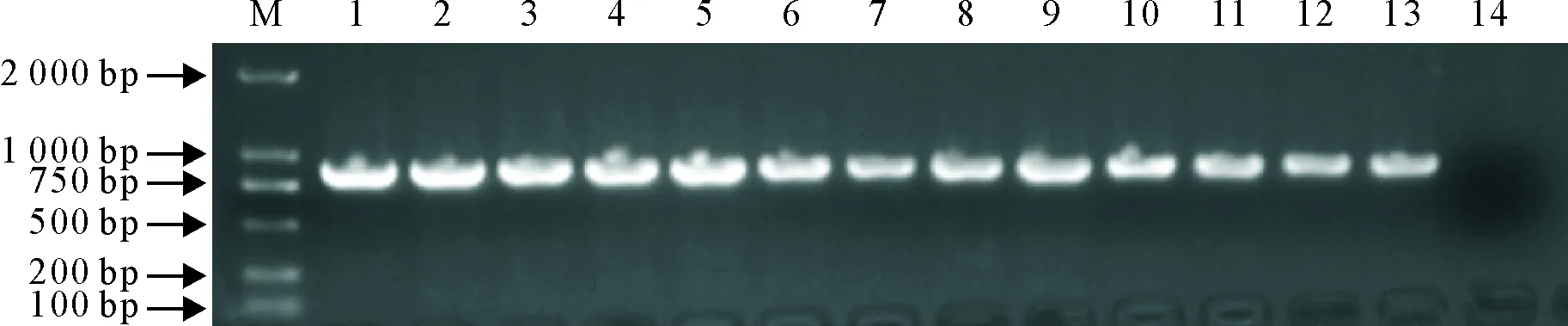
M.DNA标准DL 2 000;1~12.GDzj,GDkp,GDqy,GDjy,GDmz,GDsg,GDyf,GDgz,GDmm,GDhy,GDhf,GDhz;13.阳性对照;14.阴性对照
M.DNA Marker DL 2 000;1- 12.GDzj,GDkp,GDqy,GDjy,GDmz,GDsg,GDyf,GDgz,GDmm,GDhy,GDhf,GDhz;13.Positive control;14.Negative control
图1疑似PRRS病料样品ORF5基因RT-PCR扩增结果
Fig.1 RT-PCR amplication of PRRS ORF5 gene in samples
2.2 ORF5基因核酸遗传进化分析
对扩增得到的12份阳性样品的PCR产物进行测序,测序结果显示均获得了预期大小的目的片段。为了进一步分析这些毒株间的遗传演化情况,运用分子生物学软件对分离毒株和其他国内外具有代表性的毒株进行进化树的构建(图2),结果显示,整个遗传进化树可分为两大支:以Lelystad virus为代表的欧洲型分支和以VR2332为代表的北美型分支,以VR-2332为代表的北美型分支又可进一步分为以GM2和QYYZ为代表的subgroup 1,以美国经典毒株VR-2332为代表的subgroup 2,以NADC30为代表的subgroup 3,以中国经典毒株CH-1a为代表的subgrouup 4,以中国高致病性毒株HuN4、JXA1和JXwn06为代表的subgroup 5。其中GDhf和GDhz属于subgroup 1,GDhy属于subgrouup 4,其余毒株属于subgrouup 5。
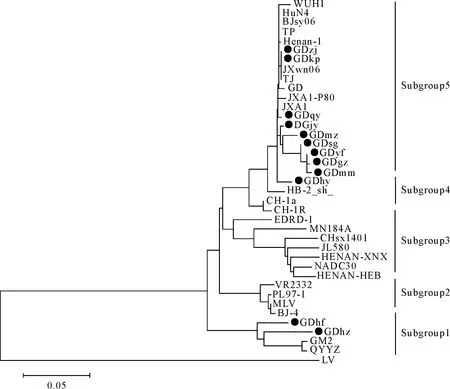
图2 PRRSV ORF5基因遗传进化分析
2.3 ORF5基因同源性分析
用DNAStar软件进行分离毒株PRRSV ORF5基因同源性分析(图3),结果显示,分离毒株之间的同源性在90.6%~95%,分离毒株与美洲型代表株VR-2332同源性在83.1%~89.4%,与Ch-1a同源性在83.9%~95.9%,与HUN4和JXA1同源性在83,6%~99.8%,与NADC30同源性在84.4%~86.4%,与QYYZ同源性在82.6%~93.2%。其中GDhf和GDhz与QYYZ同源性最高,在91.0%~93.2%,其余毒株与HUN4和JXA1同源性最高,为96.2%~99.8%。这些毒株与欧洲型Lelystad virus同源性仅为62.9%~64.0%。
2.4 PRRSV GP5氨基酸序列比对
目前已经确定的北美型毒株GP5蛋白上有3个表位,1个为中和表位(aa37-45),2个非中和表位(aa27-30和aa180-197)。除此之外,GP5上还有2个重要的抗原相关区域,1个是胞外区域(aa27-41),1个是C末端区域(aa180-197)。如图4所示,与北美型毒株VR-2332相比,所获得的12个广东毒株,在中和表位(aa37-45),仅有1个毒株GDmz存在G38A的突变。在非中和表位区域aa27-30,有4个毒株GDsg,GDyg,GDgz,GDmm在29位由G突变为A,GDhf和GDhz由G突变为T。在非中和表位aa180-197,除GDhf和GDhz外,其他10株有T183C和G197C突变;不同于其他10株,GDhf和GDhz存在G195A和A196T突变,与QYYZ突变一样;除此之外,GDhf还存在A189G的突变。
3 讨论
自从中国分离第1株PRRSV CH-1a以来,PRRS已经变成了养猪业最重要的疾病之一,给养殖业造成巨大经济损失[14-15]。由于PRRSV造成的免疫抑制和宿主的免疫系统选择压力的影响,病毒很难彻底被宿主清除,病毒基因组不断发生突变导致新的突变毒株越来越多[11-12]。

图3 PRRSV ORF5基因同源性分析
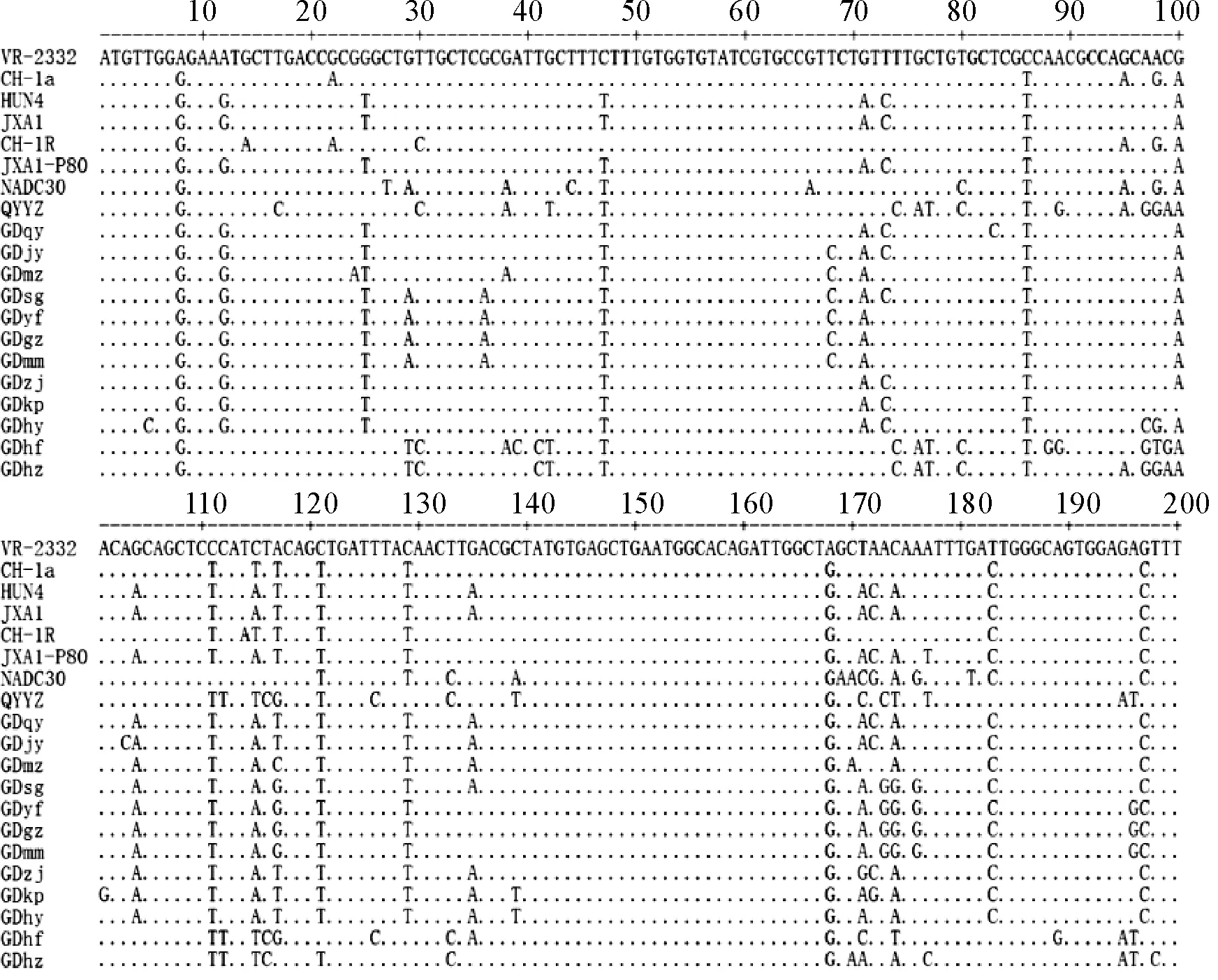
图4 PRRSV GP5蛋白氨基酸比对分析
从广东省各个地区发病猪场采集病料鉴定测序,得到12个病毒株的ORF5基因序列。从ORF5进化分析可以看出,这12个毒株属于两个不同的亚群,GDhf和GDhz与近些年广东地区分离的变异毒株GM2和QYYZ同源性最近,属于同一分支,其他毒株与中国2006年出现的HP-PRRSV毒株同源性最高,属于同一分支。有报道显示近些年NADC30-like毒株在中国广泛流行,给中国养猪业造成巨大的经济损失[16-19]。检测结果没有发现NADC30-like毒株。这些结果表明广东地区广泛流行HP-PRRSV毒株和新的与NADC30不同的变异毒株。
PRRSV GP5蛋白为囊膜糖蛋白,是病毒主要的结构蛋白,具有较高的免疫原性和中和活性[20]。有研究证实PRRSV上有3个抗原表位,其中在氨基酸37-45位(aa37-45)为中和表位;在氨基酸27-30位(aa27-30)和氨基酸180-197(aa180-197)位是2个非中和表位[21]。将测得的ORF5毒株序列与国内外代表毒株相比,在获得的12个毒株中,仅有GDmz在中和表位(aa37-45)区域存在G38A突变。突变较大的是非中和表位,有4个毒株在aa27-30区域G29A突变,2个毒株存在G29T突变;有10个毒株在aa180-197区域存在T183C和G197C的突变,另外两个毒株GDhf和GDhz同源性与最近广东地区新分离的毒株QYYZ同源性最高,不同于其他毒株,这2个毒株在非中和表位aa180-197区域G195A和A196T。这些位点的突变很可能影响GP5蛋白的免疫原性,导致疫苗免疫效价降低,影响疫苗的免疫特性,致使猪场的免疫失败。
目前中国广大地区均有NADC30-like毒株不断出现的报道,有研究者证实我国的NADC30-like毒株是由中国高致病性毒株和NADC30样毒株间重组产生的,具有高致病性[22]。也有研究者证实分离的NADC30-like毒株致病性较低[23]。这些结果表明PRRSV在我国发生了广泛的变异。我们检测的12株毒株中有2株与最近广东分离的QYYZ毒株同源性最高,该毒株具有高致病性特点,但与高致病性毒株亲缘关系较远,是否分离的这两株也具有高致病性有待于进一步研究证实[24]。另外,10株毒株与国内高致病性毒株亲缘关系较近,属于同一分支。我们的研究结果表明PRRSV仍在不断变异,且不同地区分离的毒株存在较大的差异,这些结论有助于更全面地了解广东地区PRRSV的流行特征,便于疾病的防控。
[1] Hopper S A,White M E C,Twiddy W.An outbreak of blue-eared pig disease (porcine reproductive and respiratory syndrome) in four pig herds in Great Britain [J].Vet Rec,1992,131(7):140-144.
[2] Keffaber K K.Reproductive failure of unknown etiology [J].Am Assoc Swine Pract Newsl,1989(1):1-9.
[3] Albina,E.Epidemiology of porcine reproductive and respiratory syndrome (PRRS):an overview [J].Vet,Microbiol,1997,55:309-316.
[4] Keffaber K K.Reproductive failure of unknown etiology [J].Am Assoc Swine Vet,1989,1( 2):1-9.
[5] Wensvoort G,Terpstra C,Pol J M,et al.Mystery swine disease in The Netherlands:the isolation of Lelystad virus [J].Vet Q,1991,13:121-130.
[6] Meulenberg J J,Hulst M M,de Meijer E J,et al.Lelystad virus,the causative agent of porcine epidemic abortion and respiratory syndrome (PEARS),is related to LDV and EAV [J].Virology,1993,192:62-72.
[7] Johnson C R,Griggs T F,Gnanandarajah J,et al.Novel structural protein in porcine reproductive and respiratory syndrome virus encoded by an alternative ORF5 present in all arteriviruses [J].J Gen Virol,2011,92:1107-1116.
[8] Beerens N,Selisko B,Ricagno S,et al.De novo initiation of RNA synthesis by the arterivirus RNA-dependent RNA polymerase [J].J Virol,2007,81:8384-8395.
[9] Johnson C R,Griggs T F,Gnanandarajah J,et al.Novel structural protein in porcine reproductive and respiratory syndrome virus encoded by an alternative ORF5 present in all arteriviruses [J].J Gen Virol,2011,92:1107-1116.
[10] Forsberg R.Divergence time of porcine reproductive and respiratory syndrome virus subtypes [J].Mol Biol Evol,2005,22:2131-2134.
[11] Li Y,Wang X,Bo K,et al.Emergence of a highly pathogenic porcine reproductive and respiratory syndrome virus in the Mid-Eastern region of China [J].Vet J,2007,174:577-584.
[12] Yuan S,Nelsen C J,Murtaugh M P,et al.Recombination between North American strains of porcine reproductive and respiratory syndrome virus [J].Virus Res,1999,61:87-98.
[13] Cha S H,Chang C C,Yoon K J.Instability of the restriction fragment length polymorphism pattern of open reading frame 5 of porcine reproductive and respiratory syndrome virus during sequential pig-to-pig passages [J].J Clin Microbiol,2004,42:4462-4467.
[14] Li Y,Zhou L,Zhang J,et al.Nsp9 and Nsp10 contribute to the fatal virulence of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China [J].PLoS Pathog,2014,10:e1004216.
[15] Nelsen C J,Murtaugh M P,Faaberg K S.Porcine reproductive and respiratory syndrome virus comparison:divergent evolution on two continents [J].J Virol,1999,73:270-280.
[16] Li X,Bao H,Wang Y,et al.Widespread of NADC30-like PRRSV in China:Another Pandora's box for Chinese pig industry as the outbreak of highly pathogenic PRRSV in 2006 [J].Infect Genet Evol,2017,49:12-13.
[17] Zhang Q,Jiang P,Song Z,et al.Pathogenicity and antigenicity of a novel NADC30-like strain of porcine reproductive and respiratory syndrome virus emerged in China [J].Vet Microbiol,2016,197:93-101.
[18] Li X,Wu J,Tan F,et al.Genome characterization of two NADC30-like porcine reproductive and respiratory syndrome viruses in China [J].Springerplus,2016,5(1):1677.
[19] Sun Z,Wang J,Bai X,et al.Pathogenicity comparison between highly pathogenic and NADC30-like porcine reproductive and respiratory syndrome virus [J].Arch Virol,2016,161(8):2257-2261.
[20] Zhang Q Y,Xu X J,You S M,et al.Emerging of two new subgenotypes of porcine reproductive and respiratory syndrome viruses in Southeast China [J].Microb Pathog,2016; 97:27-33.
[21] Kijona F K,Gholamreza H,Denis K G,et al.Genetic variation and phylogentic analyses of the ORF5 gene of acute porcine respiratory and reproductive syndrome viruse isolate [J].Vet Microbiol,2001,83(3):249-263.
[22] 郭天准,常洪涛,崔丹丹,等.2014-2015 年河南地区PRRSV 的分子检测及 NSP2、ORF5 基因变异分析 [J].病毒学报,2016,32(3):298-307.
[23] Plagemann P G.GP5 ectodomain epitope of porcin reproductiveand respiratory syndromevirus,strain Lelystad virus [J].Virus Res,2004,102(2):225-230.
[24] Lu W H,Tun H M,Sun B L,et al.Re-emerging of porcine respiratory and reproductive syndrome virus (lineage 3) and increased pathogenicity after genomic recombination with vaccine variant [J].Vet Microbiol,2015,175:332-340.
GeneticVariationsofORF5GenesofPorcineReproductiveandRespiratorySyndromeIsolatedinGuangdongRegionsfrom2016to2017
YU Lin-yang1,DONG Jian-guo1,2,ZHANG Le-yi1,LIU Yan-ling1,LIANG Peng-shuai1, WANG Lei1,SONG Chang-xu1
(1.CollegeofAnimalScience,SouthChinaAgricultureUniversity,Guangzhou,Guangdong,510642,China; 2.CollegeofAnimalHusbandryandVeterinary,XinyangCollegeofAgricultureandForestry,Xinyang,Henan,464000,China)
To investigate the genetic variations of ORF5 genes of porcine reproductive and respiratory syndrome virus (PRRSV) isolated in Guangdong regions,the lung samples of pigs suspected to be PRRSV positive from pig farms in Guangdong during 2016 to 2017 were collected and the ORF5 of the PRRSV in these samples were amplified by RT-PCR.The ORF5 gene sequences and deduced amino acid sequences were furtherly analyzed.Twelve complete ORF5 gene sequences were amplified,and phylogenetic analysis results showed that 10 isolates had close genetic relationship with the highly pathogenicitic PRRSV (HP-PRRSV) strains and they belonged to the same cluster.Other 2 isolates had close genetic relationship with the newly isolated strain QYYZ and they belonged to the same cluster.The amino acids alignment results indicated that the epitope of GP5 had extensive amino acid mutations.These results indicated that the region's PRRSV strains have experienced extensive evolation.These results not only revealed that the genetic evolution characteristics of PRRSV ORF5 gene in Guangdong,but also provided a scientific basis for furtherly controlling the PRRS.
Porcine reproductive and respiratory syndrome; ORF5 gene; variation analysis
2017-04-27
国家支撑计划项目(2015BAD12B02-5); 广州市科技计划项目(201508020062);现代农业产业共性技术创新团队项目(2016LM2150)
于林洋(1992-),男,河南驻马店人,硕士,主要从事动物分子病毒学和免疫学研究。*
S82.659.6
A
1007-5038(2017)12-0023-06
