红唇薄鳅2个野生群体的遗传多样性研究
2017-12-18刘红艳段辛斌田辉伍刘绍平陈大庆
刘红艳,熊 飞,段辛斌,田辉伍,刘绍平,陈大庆
( 1.江汉大学 生命科学学院,湖北 武汉 430056; 2. 中国水产科学研究院 长江水产研究所,湖北 武汉 430223 )
红唇薄鳅2个野生群体的遗传多样性研究
刘红艳1,熊 飞1,段辛斌2,田辉伍2,刘绍平2,陈大庆2
( 1.江汉大学 生命科学学院,湖北 武汉 430056; 2. 中国水产科学研究院 长江水产研究所,湖北 武汉 430223 )
采用9 个微卫星DNA标记对长江支流岷江下游厥溪和长江上游干流朱杨溪红唇薄鳅的2个野生群体遗传多样性及遗传分化情况进行了研究。共检测出73条等位基因,其中厥溪群体中有55条等位基因,朱杨溪群体中有69条等位基因,两群体共有等位基因数为51条。厥溪群体和朱杨溪群体的平均等位基因数分别为6.111和7.667,平均观测杂合度分别为0.665和0.668;平均期望杂合度分别0.684和0.712,平均多态性信息指数分别为0.621和0.656,从各参数结果来看,朱杨溪群体的遗传多样性水平高于厥溪。AMOVA结果显示,大多数遗传变异存在于红唇薄鳅群体内(98.68%),群体间的遗传变异为1.32%,固定系数不显著。基因流为9.42,表明两群体间基因交流频繁。UPGMA 聚类显示,厥溪和朱杨溪群体中的个体相互混杂。这些结果表明长江上游厥溪和朱杨溪群体未出现遗传分化。
红唇薄鳅;微卫星;遗传多样性
红唇薄鳅(Leptobotiarubrilabris),属鲤形目、鳅科、薄鳅属,为长江上游特有鱼类[1]。在鳅科中个体较大,仅次于长薄鳅(L.elongata),喜流水环境。20世纪80年代在长江上游干流及支流分布较广泛,但随着长江三峡工程的截流及金沙江梯级水电站的开发,破坏了其栖息地和产卵场的环境,加之环境污染、过度捕捞等原因,其天然资源量锐减[2-5]。目前关于红唇薄鳅的研究资料相当少,仅见红唇薄鳅泌尿系统的组织学观察[6]和种群生态与线粒体遗传学的研究[7]。研究遗传多样性有助于了解物种的进化历史和种群恢复潜力,因此,为了有效保护和恢复红唇薄鳅种群资源,有必要对其遗传多样性进行研究。
微卫星DNA是由2~6个核苷酸组成的核心单元串联重复形成,广泛分布于真核生物的整个基因组中,同一座位上,由于核心单元的重复数不同,在群体中表现出多态性[8-9]。目前,微卫星DNA标记已广泛应用于鱼类种群遗传学研究[10-14]。本研究利用微卫星DNA标记技术,对长江支流岷江下游厥溪和长江上游干流朱杨溪红唇薄鳅的2个野生群体的遗传多样性进行研究,查明其遗传多样性水平,遗传分化和基因流情况,以期为红唇薄鳅的保护和资源恢复提供科学依据。
1 材料与方法
1.1 样本采集和DNA提取
2011年1月—2013年1月进行长江上游红唇薄鳅资源分布调查,样品采集。样本采集地为长江支流岷江下游的厥溪和长江上游干流的朱杨溪,其中厥溪样本为19尾,朱杨溪样本为30尾。剪取部分鳍条于95%乙醇中保存带回实验室。取保存于乙醇中的鳍条组织,置于0.9%的生理盐水浸泡约12 h(期间更换生理盐水3~4次),鳍条组织去除乙醇后,剪碎置于离心管中,然后加入DNA 提取缓冲液[0.4 mol/L NaCl,10 mmol/L Tris-HCl (pH 8.0),2 mmol/L EDTA (pH 8.0) ]600 μL,终含量为2%的十二烷基硫酸钠和终质量浓度为0.2 mg/mL的蛋白酶 K,混匀后56 ℃消化5~6 h,期间取出混匀数次,DNA经酚—氯仿抽提,2倍体积无水乙醇沉淀,75%酒精洗涤,灭菌蒸馏水溶解。获得的DNA用核酸蛋白分析仪检测其纯度和质量浓度,最终DNA模板质量浓度定量在20 ng/μL,-20 ℃保存备用。
1.2 微卫星DNA标记分析
采用本实验室筛选的重复性稳定、多态性好的9个微卫星DNA标记进行群体遗传研究,其中7个为红唇薄鳅微卫星DNA标记(LR26、LR27、LR31、LR33、LR34、LR35、LR37)[15], 2个为同属鱼类长薄鳅的微卫星DNA标记(CB12和CB18)[16]。PCR反应总体积为10 μL,包括Taq DNA 聚合酶0.5 U(Fermentas),dNTP 0.2 mmol/L,MgCl22.0 mmol/L,1×Buffer,引物0.2 μmol/L,DNA 模板约20 ng,补充灭菌蒸馏水到10 μL。PCR 扩增程序为95 ℃预变性5 min,接着进行35个循环:94 ℃变性40 s、58 ℃退火30 s、72 ℃延伸40 s,最后72 ℃延伸10 min。反应在PTC100型PCR仪上进行。扩增产物用非变性的8%聚丙烯酰胺凝胶电泳进行分离,并通过银染法进行检测。
1.3 数据分析
根据DNA Marker标记和微卫星DNA的迁移率,读取各等位基因条带。利用CONVERT 1.3.1软件[17],将各群体等位基因条带矩阵转化相关分子遗传学软件所需的格式。用Popgene 1.2软件[18]计算各遗传多样性参数,包括各位点的等位基因数目、观测杂合度、期望杂合度。用PIC-CALC 0.6软件计算多态性信息指数。
分子变异方差分析用AMOVA软件[19]计算,检测群体内和群体间的遗传变异情况,计算中考虑微卫星等位基因的大小差异,所有多重比较中的P值均进行泊松校正。用Arlequin 3.1软件[20]计算群体间遗传距离。用 NTSYS[21]中的SHAN程序进行UPGMA 聚类,构建群体间的遗传关系。
2 结 果
2.1 微卫星DNA标记的PCR 扩增
9个微卫星DNA标记在2个红唇薄鳅群体中均表现为多态性。共检测出73条等位基因,其中在厥溪群体中有55条等位基因,在朱杨溪群体中有69条等位基因,两群体共有等位基因数为51条。每个位点检测到的等位基因数分别为2~12不等,平均值为8.111。引物LR35的在朱杨溪群体中的等位基因数最多,为12条,引物CB12在厥溪群体中的等位基因数最少,为2条(表1)。
2.2 遗传多样性
厥溪和朱杨溪的平均等位基因数分别为6.111和7.667,平均观测杂合度分别为0.665和0.668,平均期望杂合度分别0.684和0.712,平均多态性信息指数分别为0.621和0.656(表1)。可以看出,朱杨溪群体各遗传多样性参数均高于厥溪群体的,表明朱杨溪群体的遗传多样性水平高于厥溪。
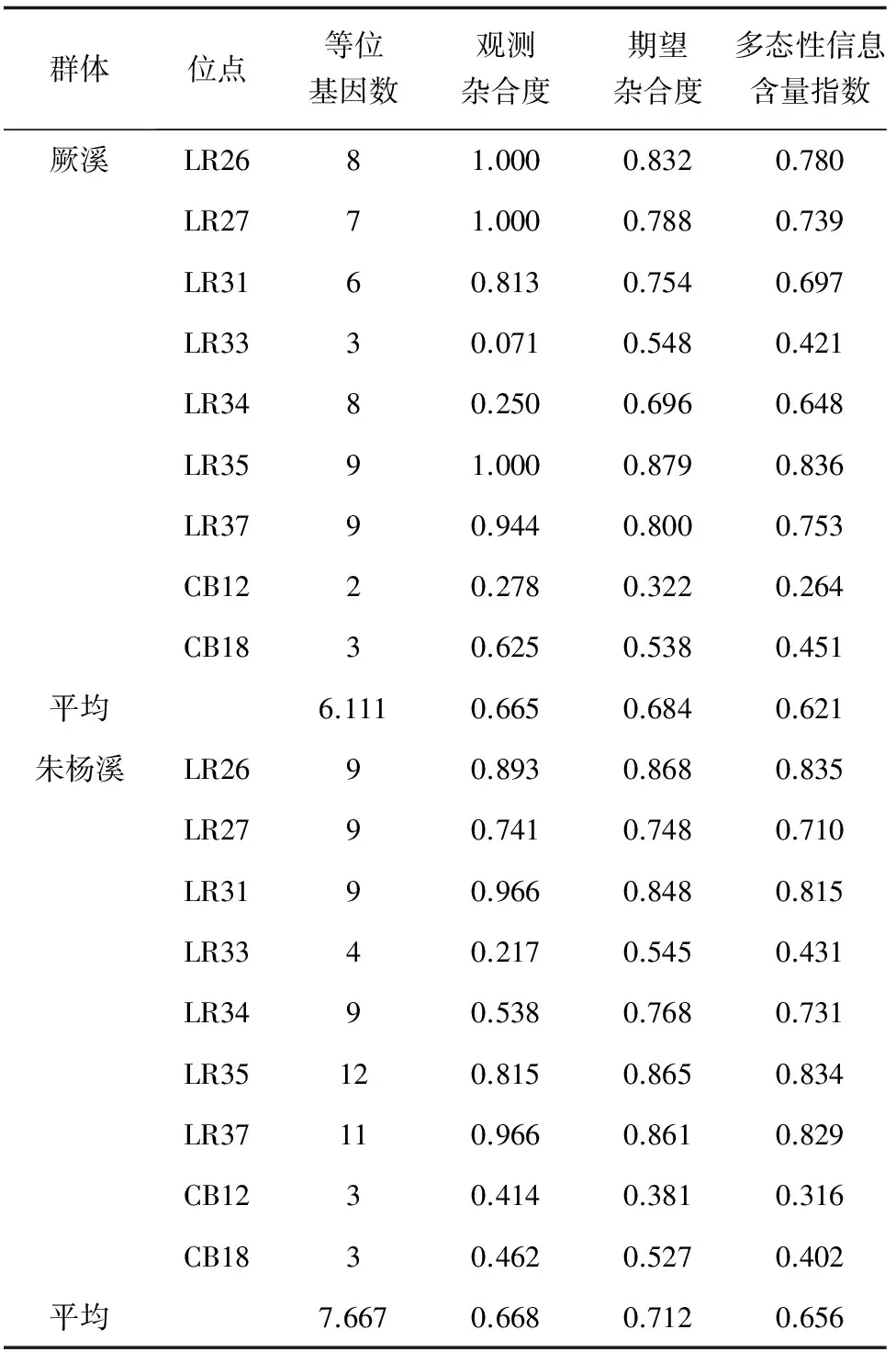
表1 红唇薄鳅群体微卫星多态性数据
2.3 遗传变异与聚类分析
厥溪和朱杨溪的Nei′s遗传距离为0.0633,遗传相似指数为0.9367,2个群体间的遗传距离较小,遗传相似度较高。分子方差分析(表2)显示,大多数遗传变异存在于红唇薄鳅群体内(98.68%),群体间的遗传变异为1.32%,固定系数遗传分化指数不显著,表明厥溪和朱杨溪群体未出现遗传分化。基因流为9.42,表明2个群体间基因交流频繁。
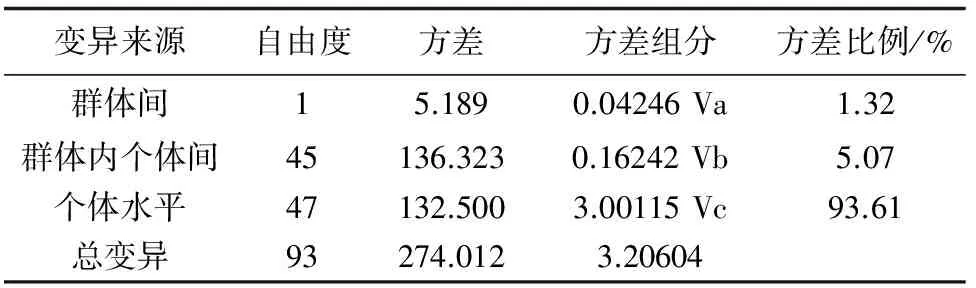
表2 红唇薄鳅群体分子变异方差分析
UPGMA 聚类图显示(图1),厥溪和朱杨溪群体中的个体相互混杂,并未按相应的地理位置进行聚类,表明这2个长江上游群体没有出现明显的遗传分化。
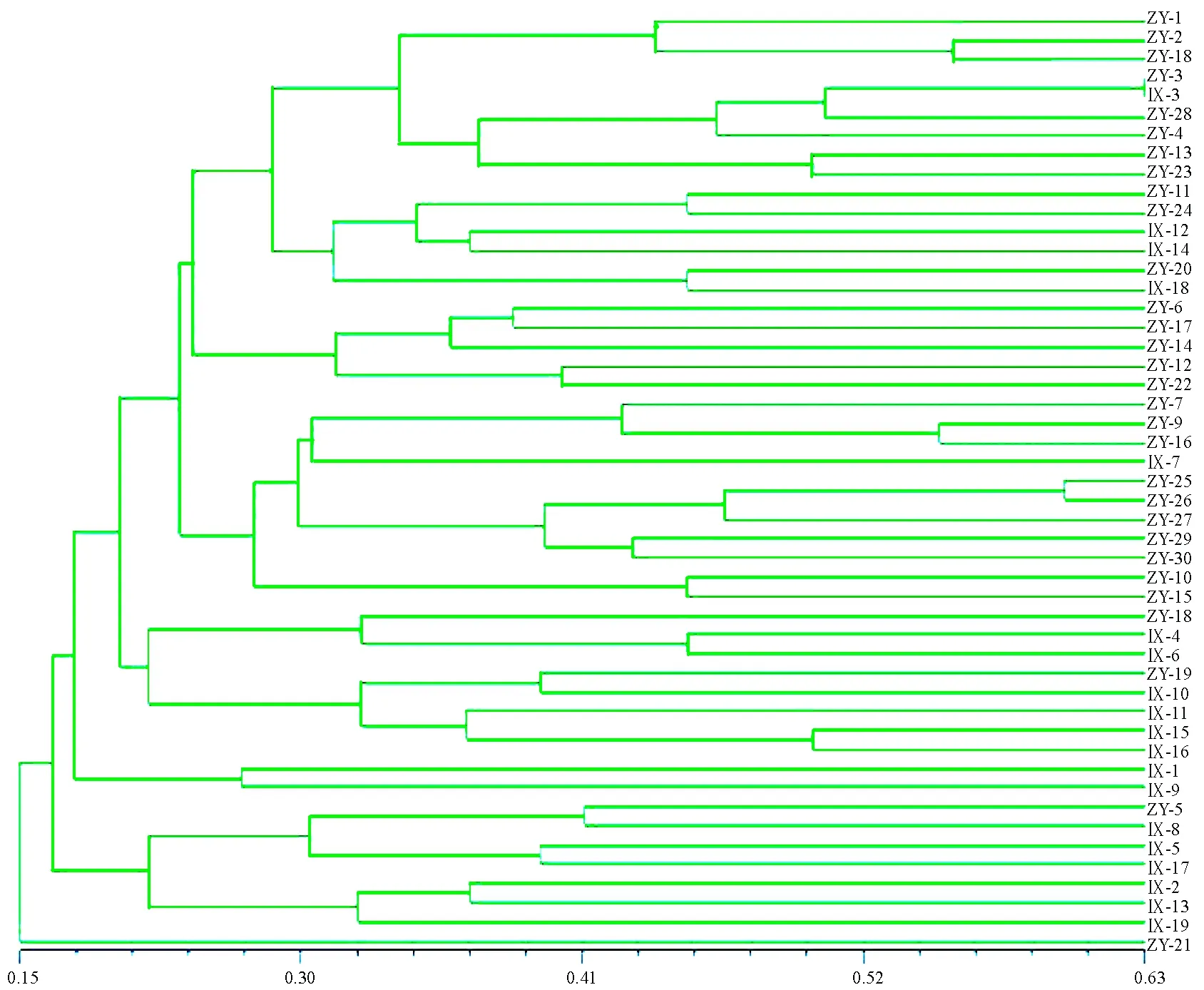
图1 基于Nei′s遗传距离的UPGMA聚类图
3 讨 论
3.1 红唇薄鳅群体的遗传多样性
厥溪和朱杨溪的平均等位基因数分别为6.111和7.667个,这与DeWoody等[22]得出的淡水鱼类微卫星等位基因数目约为7.5个的结论接近。同时,多态信息指数也是衡量基因片段多态性的理想指标,当多态信息指数>0.5 时,该座位为高度多态性;当0.25<多态信息指数<0.5 时, 该座位为中度多态性;当多态信息指数<0.25 时,该座位为低度多态性[23]。在厥溪群体和朱杨溪群体微卫星位点中有3个中度多态性位点(LR31、CB12和CB18),其余6个位点为高度多态性位点。从本研究选用的9 个微卫星DNA标记的多态性来看,红唇薄鳅2个野生群体的多态信息含量较高,属于中度及高度多态性,这说明红唇薄鳅的等位基因多样性较为丰富。
杂合度是指群体在多个基因座上遗传变异的表现,被认为是评价群体的遗传多样性的一个最适参数,按照Hardy-Weinberg定理,杂合度在随机交配群体中≤0.5[24]。本研究中,2个红唇薄鳅群体的观测杂合度和期望杂合度均大于0.5,表明红唇薄鳅发生近交的可能性不大。红唇薄鳅2个群体的平均观测杂合度和平均期望杂合度分别为0.667及0.698,低于同喜流水生活的长江宜宾圆口铜鱼(Coreiusguichenoti)(观测杂合度0.753, 期望杂合度0.728)[25]、鲢鱼(Hypophthalmichthysmolitrix)(观测杂合度0.805)[26]的杂合度,高于长江水系的草鱼(Ctenopharyngodonidellus)(观测杂合度0.4000~0.5741,期望杂合度0.4773~0.6489)[26]、鲤鱼(Cyprinuscarpio)(期望杂合度0.267~0.419)、铜鱼(C.heterokon)(观测杂合度0.4251~0.5158)等鱼类的杂合度[28],表明红唇薄鳅的群体遗传多样性比较丰富,有着较大的进化适应的潜力,这与田辉伍利用线粒体Cyt b和控制区DNA研究结果一致[7]。
3.2 红唇薄鳅基因流和遗传分化
红唇薄鳅2个群体间的遗传距离较小,这说明2个群体间的遗传相似程度较高。分子方差分析也显示,2个群体间遗传变异只占总遗传变异的较小的比例(1.32%),并且遗传分化指数值不显著,也说明红唇薄鳅群体间差异较小,遗传变异主要发生在群体内。UPGMA也支持厥溪和朱杨溪群体没有出现遗传分化,这2个群体均属于同一种群。这可能与红唇薄鳅的生活习性和产卵习性有关,红唇薄鳅属于喜流水环境的鱼类,在繁殖季节,亲鱼有上溯到相应的产卵场习性,产漂流性卵,卵随着河水向下漂流孵化,而后其幼鱼和成鱼则溯流回到上游,因而在这个过程中,各分布区群体之间有更多的遗传交流机会。本研究中基因流较大,也证明了群体间的基因交流频繁。这与线粒体Cyt b和控制区DNA的研究结果一致[7]。与红唇薄鳅有着相似生活习性和同为产漂流性卵的中华沙鳅(Botiasuperciliaris)在长江上游也未出现群体的遗传分化[29]。另外,产漂流性卵的铜鱼、圆口铜鱼和草鱼也未发生显著的遗传分化[27-28,30-31]。
3.3 种质保护
本研究中,虽然2个红唇薄鳅群体都保持了较高的遗传多样性, 但朱杨溪群体的遗传多样性水平要显著高于厥溪群体,说明岷江厥溪群体遗传资源已遭到了一定程度的破坏,需要加强保护。目前红唇薄鳅资源量正在不断地减少,长江上游梯级大坝的开发仍在持续的进行,大坝的建立将限制红唇薄鳅上下游群体之间的遗传交流,极大的改变水文条件,破坏其栖息地和产卵场,红唇薄鳅生存前景不容乐观,亟需进行长江特有物种的保护。物种的保护必须充分考虑到物种的遗传特征,本研究结果显示红唇薄鳅群体间没有显著遗传分化,可作为1个遗传单元来进行保护和管理。大坝截流后水库的形成,使原来的流水环境变成静水环境,鉴于红唇薄鳅独特的喜流水生活习性,有必要扩大采样范围和样本量进一步全面深入的研究红唇薄鳅遗传结构及多样性,以便为其种质资源的保护、管理以及种群恢复提供更加可靠的科学依据。
[1] 丁瑞华. 四川鱼类志[M]. 成都:四川科学技术出版社, 1994.
[2] 王文君, 谢山, 张晓敏, 等. 岷江下游产漂流性卵鱼类的繁殖活动与生态水文因子的关系[J]. 水生态学杂志, 2012, 33(6):29-34.
[3] 高天珩, 田辉伍, 叶超, 等. 长江上游珍稀特有鱼类国家级自然保护区干流段鱼类组成及其多样性[J]. 淡水渔业, 2013, 43(2):36-42.
[4] 熊飞, 刘红艳, 段辛斌, 等. 长江上游江津江段鱼类群落结构及资源利用[J]. 安徽大学学报, 2014, 38(4):95-102.
[5] 段辛斌, 田辉伍, 高天珩, 等.金沙江一期工程蓄水前长江上游产漂流性卵鱼类产卵场现状[J]. 长江流域资源与环境, 2015, 24(8):1358-1365.
[6] 史晋绒, 王永明, 谢碧文, 等. 红唇薄鳅泌尿系统的组织学观察[J]. 淡水渔业, 2014, 44(3):23-29.
[7] 田辉伍. 长江上游保护区长薄鳅和红唇薄鳅种群生态及遗传结构比较研究[D]. 重庆:西南大学, 2013.
[8] Li Y, Korol A B, Fahima T, et al.Microsatellites within genes: structure, function, and evolution [J]. Mol Biol Evol, 2004, 21(6):991-1007.
[9] Lawson M J, Zhang L. Distinct patterns of SSR distribution in theArabidopsisthalianaand rice genomes [J]. Genome Biol, 2006(7):14.
[10] 王长忠, 梁宏伟, 邹桂伟, 等. 长江中上游两个鲢群体遗传变异的微卫星分析[J]. 遗传, 2008, 30(10): 1341-1348.
[11] 吴勤超, 梁宏伟, 李忠, 等.黄颡鱼微卫星标记的筛选及三个野生群体的遗传结构分析[J]. 生物技术通报, 2010(3):154-163.
[12] Chen L, Michael W B, Xu S, et al.Microsatellite variation and significant population genetic structure of endangered finless porpoises (Neophocaenaphocaenoides) in Chinese coastal waters and the Yangtze River [J]. Mar Biol, 2010, 157(7):1453-1462.
[13] Ma H, Chen S, Yang J, et al. Genetic linkage maps of barfin flounder (Veraspermoseri) and spotted halibut (Veraspervariegatus) based on AFLP and microsatellite markers [J]. Mol Biol Rep, 2011, 38(7): 4749-4764.
[14] 刘云国,刘凌霄,王咏星. 茴鱼微卫星标记开发及保护遗传学研究进展[J]. 水产科学, 2016, 35(1):87-92.
[15] Duan X B, Liu H Y, Xiong F, et al.Development of microsatellite markers forLeptobotiarubrilaris, a threatened fish species in the Yangtze River [J].Conservation Genet Resour, 2013, 5(2):425-427.
[16] Liu H Y, Xiong F, Duan X B, et al.Isolation and characterization of polymorphic microsatellite loci from elongate loach (Leptobotiaelongata), a threatened fish species endemic to the Yangtze River[J]. Conservation Genet Resour, 2012, 4(1):129-131.
[17] Glaubitz J. CONVERT: A user-friendly program to reformat diploid genotypic data for commonly used population genetic software packages [J]. Molecular Ecology Notes, 2004, 4(2): 309-310.
[18] Raymond M,Rousset F. POPGENE (version 1.2), population genetics software for exact test and ecumenicism [J]. Journal of Heredity, 1995, 86(3):248-249.
[19] Excoffier L, Smouse P E, Quattro J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondria DNA restriction sites [J]. Genetics, 1992, 131(2):479-491.
[20] Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): an integrated software package for population genetics data analysis [J]. Evol Bioinform Online, 2005(1):47-50.
[21] Rohlf F J. NTSYS-pc numerical taxonomy and multivariate analysis system version 2.1[M]. New York:Department of Ecology and Evolution State University of New York, 2000.
[22] DeWoody J A, Avise J C. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals [J]. J Fish Biol, 2000, 56(3): 461-473.
[23] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. Am J Human Genet,1980, 32(3):314-331.
[24] 赵程, 梁旭方, 田昌绪, 等. 鳜属5 种鱼类微卫星遗传多样性分析[J]. 华中农业大学学报, 2015, 34(5): 76-80.
[25] 徐树英,张燕, 汪登强, 等. 长江宜宾江段圆口铜鱼遗传多样性的微卫星分析[J]. 淡水渔业, 2007, 37(3): 76-79.
[26] Wang C Z, Liang H W, Zou G W, et al.Genetic variation analysis of two silver carp populations in the middle and upper Yangtze River by microsatellite [J]. Heredity, 2008, 30(10):1341-1348.
[27] 廖小林.长江流域几种重要鱼类的分子标记筛选开发及群体遗传分析[D]. 武汉:中国科学院研究生院水生生物研究所, 2006.
[28] 廖小林, 俞小牧, 谭德清, 等. 长江水系草鱼遗传多样性的微卫星DNA 分析[J]. 水生生物学报, 2005, 29(2):113-119.
[29] 刘红艳, 陈大庆, 刘绍平, 等. 长江上游中华沙鳅遗传多样性研究[J]. 淡水渔业, 2009, 29(3):8-13.
[30] 袁希平, 严莉, 徐树英, 等.长江流域铜鱼和圆口铜鱼的遗传多样性[J]. 中国水产科学, 2008, 15(3): 377-384.
[31] Yan L, Wang D Q, Fang Y L, et al.Genetic diversity in the bronze gudgeon,Coreiusheterodonfrom the Yangtze River system based on mtDNA sequences of the control region [J]. Environ Biol Fish, 2008, 82(1):35-40.
GeneticDiversityinTwoWildPopulationsofRedlipLoachLeptobotiarubrilabris
LIU Hongyan1, XIONG Fei1, DUAN Xinbin2, TIAN Huiwu2, LIU Shaoping2, CHEN Daqing2
( 1. School of Life Sciences, Jianghan University, Wuhan 430056, China; 2.Yangtze River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Wuhan 430223,China )
Genetic diversity and genetic differentiation were investigated by 9 microsatellite loci in two wild populations (Juexi population and Zhuyangxi population) of redlip loachLeptobotiarubrilabrisin the upper reaches of the Yangtze River. A total of 73 alleles was detected, with 55 alleles in Juexi population and 69 alleles in Zhuyangxi population, in which 51 alleles were shared. The average number of alleles in Juexi and Zhuyang were 6.111 and 7.667, average observed heterozygosity 0.665 and 0.668, average expected heterozygosity 0.684 and 0.712, and the average polymorphism information index 0.621 and 0.656. These genetic parameters indicated that there was higher level of genetic diversity in Zhuyangxi than that in Juexi. AMOVA analysis revealed that most of the total variation occurred within populations (98.68%),a small amount of differentiation among populations (1.32%), without significance in Fst value (0.0132). The Nm value of 9.42 indicated that gene flow between the two sites was frequently. UPGMA clustering revealed that the individuals mixed together. The findings reflected that there was no significant differentiation in populations of redlip loach in the upper reaches of the Yangtze River.
Leptobotiarubrilabris; microsatellite; genetic diversity
10.16378/j.cnki.1003-1111.2017.02.013
2016-04-16;
2016-06-29.
国家自然科学基金资助项目(51109091);江汉大学出国留学基金资助项目.
刘红艳(1978-),女,副教授;研究方向:鱼类分子遗传. E-mail:lhy9603@126.com. 通讯作者: 段辛斌(1972-),男,副研究员;研究方向:鱼类资源学. E-mail: duan@yfi.ac.cn.
S965.199
A
1003-1111(2017)02-0192-05
