鼠尾草叶绿体基因组序列分析
2017-11-11何懿菡韩立敏刘玉萍王喆之
何懿菡 韩立敏 刘玉萍 田 娜 苏 旭 王喆之*
(1.陕西师范大学生命科学学院,药用资源与天然药物化学教育部重点实验室,西北濒危药材资源开发国家工程实验室,西安 710119; 2.青海师范大学生命与地理科学学院,西宁 810008; 3.陕西学前师范学院生命科学与食品工程学院,西安 710061)
鼠尾草叶绿体基因组序列分析
何懿菡1,2韩立敏3刘玉萍2田 娜1苏 旭2王喆之1*
(1.陕西师范大学生命科学学院,药用资源与天然药物化学教育部重点实验室,西北濒危药材资源开发国家工程实验室,西安 710119;2.青海师范大学生命与地理科学学院,西宁 810008;3.陕西学前师范学院生命科学与食品工程学院,西安 710061)
鼠尾草(Salviajaponica)是唇形科(Labiatae)鼠尾草属(Salvia)的一种多年生草本植物,具有十分重要的药用和经济价值。本文采用第二代测序技术Illumina Hiseq平台对鼠尾草的叶绿体基因组进行测序,同时以鼠尾草近缘物种丹参叶绿体基因组作为参考,组装得到完整叶绿体基因组序列。结果表明,鼠尾草叶绿体基因组序列全长153 995 bp,呈典型的四段式结构,其中LSC区长84 573 bp,SSC区长19 874 bp,两个IR区分别长24 774 bp;鼠尾草叶绿体基因组成功注释13组叶绿体基因,基因的种类、数目及GC含量等与唇形科中其它物种较为类似。这些研究结果丰富了鼠尾草属的叶绿体基因组数据,为今后鼠尾草属植物系统发育关系重建积累了基础性数据。
鼠尾草属;鼠尾草;叶绿体基因组;基因结构;系统进化
鼠尾草(Salviajaponica)是唇形科(Labiatae)、鼠尾草属(Salvia)中的一种多年生草本植物,在我国主要分布于横断山区,占鼠尾草属物种总数的52.8%,尤其是特有种数目较多,高达23种[1]。鼠尾草药材基原为全草,具有清热利湿、活血调经解毒消肿之功效[2],同时含有三萜、类固醇、酚类化合物等多种代谢产物[3],其根和叶含有咖啡酸、迷迭香酸、丹酚酸等8种水溶性成分,此外根中还含有丹参酮Ⅰ和丹参酮ⅡA等脂溶性成分[4]。
鼠尾草属植物因雄蕊形成具活动关节的杠杆状结构,与昆虫或蜂鸟的传粉行为高度适应,因而它们在唇形科有较为重要的系统地位[5],并且Bentham[6]还曾利用这一特征将其划分为四个亚属,至今这种分类系统仍被广泛采用。随着植物分子生物学测序技术和分析手段的迅速发展,叶绿体基因的psbA-trnH、matK和rbcL等多个片段被广泛用于鼠尾草属植物的DNA分子鉴定[7]。然而,先前鼠尾草属植物系统发育的分析均是基于叶绿体基因和核基因序列信息,DNA片段长度通常介于300~800 bp,信息位点较少,片段信息常常存在测定序列的分子进化速率与物种水平上的进化速率存在差异,以检测序列位点的多样性来代替物种的多样性,未能真实反映物种间的系统发育关系[8];加之,鼠尾草属种类繁多,种间变异复杂多样,从而导致对该属绝大部分物种的界定存在较多不足乃至张冠李戴。当前,随着高通量测序技术的发展,叶绿体基因组序列呈现出具有更强分辨率且适用于不同分类阶元的系统发育分析[9~10],可为药用植物物种鉴定和混伪品鉴别提供较为准确的方法。鼠尾草属细胞器基因组数据匮乏,进而限制了叶绿体基因组在物种鉴定和进化关系研究中的应用。药用植物丹参(Salviamiltiorrhiza)是鼠尾草属第一个获得完整叶绿体基因组的物种,通过与唇形目(Lamiales)已知叶绿体基因组数据的综合分析,发现它与芝麻(Sesamumindicum)亲缘关系最近[11]。鼠尾草和丹参均隶属于鼠尾草属,是该属的一个广布种,并且具有复杂的遗传多样性[12],加之它的物种起源及系统进化等诸多科学问题至今未见报道,尤其是目前鼠尾草叶绿体基因组研究尚属空白,据此本文以鼠尾草为研究对象,以丹参叶绿体基因组作为参考,通过对其叶绿体基因组的组装和分析,探讨鼠尾草叶绿体基因组的结构信息,旨在为鼠尾草种质资源的开发和利用提供科学依据,同时也可为今后鼠尾草属植物系统进化地位的确立及种质资源的鉴定提供基础性数据。
1 材料与方法
1.1 测序材料
鼠尾草种子撒播于室温大棚,覆膜待出芽培养3个月后,取幼嫩叶片作为提取DNA的材料。DNA的提取参照Biospin植物基因组DNA提取试剂盒(Bioer,杭州)说明书进行。
1.2 测序
将检测合格后的DNA,随机打断成1~2 kB的片段,富集大小一致的片段,连接至建库载体中构建DNA文库(DNA文库制备试剂盒,利用Illumina公司产品),然后利用Illumina HiSeq 2500基因组测序平台测序。
1.3 叶绿体基因组的组装与注释
测序获得的raw reads数据利用CLC Genomics Workbench 9.0(CLC Bio,Aarhus,Denmark)软件中Trim过滤去除低质量序列,过滤条件为Ambiguous limit=2、Quality limit=0.05;然后,以先前提交至NCBI的丹参叶绿体基因组(HF586694)作为参照序列,将Trim过滤后的8,877,028 reads用MITObim 1.7重建叶绿体基因组;最终得到由无gap的717 586条叶绿体序列,平均覆盖702个碱基,长度为157 839 bp的叶绿体基因组,应用Geneious 8.0.3首先进行丹参与鼠尾草序列的CLUSTAL比对,然后进行碱基修正,修改蛋白质和tRNA等注释,完成鼠尾草叶绿体基因组的分区,利用OGDraw 1.2(http://ogdraw.mpimp-golm.mpg.de/index.shtml)软件对鼠尾草叶绿体基因组序列进行绘图。
2 结果与分析
2.1 鼠尾草叶绿体基因组基本特征
使用Illumina Hiseq平台对鼠尾草叶绿体DNA提取样本进行测序,测序数据达2.4 G,目前基因组序列已提交至GenBank数据库(登录号KY646163)。鼠尾草叶绿体基因组序列全长153,995 bp,呈典型的四段式结构(图1),其中LSC区(large single copy region,大的单拷贝区)长84 573 bp,SSC区(small single copy region,小的单拷贝区)长19 874 bp,两个IR区(inverted repeat,反向重复区)分别长24 774 bp(表1)。总体看来A+T含量为62%,有明显的AT偏向性。IR区的GC含量(43.4%)明显高于LSC区(36.2%)和SSC区(32.0%),这主要由IR区包含的四个高GC含量的rRNA基因。
2.2 鼠尾草叶绿体基因组各分区基因组成
整条叶绿体基因组序列共编码135个基因,去除重复后为118个,包括89(去除重复83)个蛋白编码基因、37(去除重复30)个tRNA、8(去除重复4)个rRNA(表2)。6个蛋白编码基因、7个tRNA基因及4个rRNA基因在两个IR区各有一个拷贝。LSC区包含63个蛋白编码基因、22个tRNA基因,而SSC区只含有12个蛋白编码基因和1个tRNA基因(图1)。
表1鼠尾草叶绿体基因组碱基组成
Table1BasecompositionofchloroplastgenomeinS.japonica

T(U)(%)C(%)A(%)G(%)长Length(bp)GC(%)LSC32.618.531.217.78457336.2SSC33.916.734.115.31987432.0Ira28.222.628.320.82477443.4Irb28.320.828.322.62477443.4合计Total31.419.330.618.715399538.0CDS31.619.330.418.67828437.9
注:CDS.蛋白编码区 下同。
Note:CDS.Protein-coding regions The same as below.
表2鼠尾草叶绿体基因组各分区的基因组成
Table2GenecompositionofLSC,SSC,IRintheS.japonicachloroplastgenome
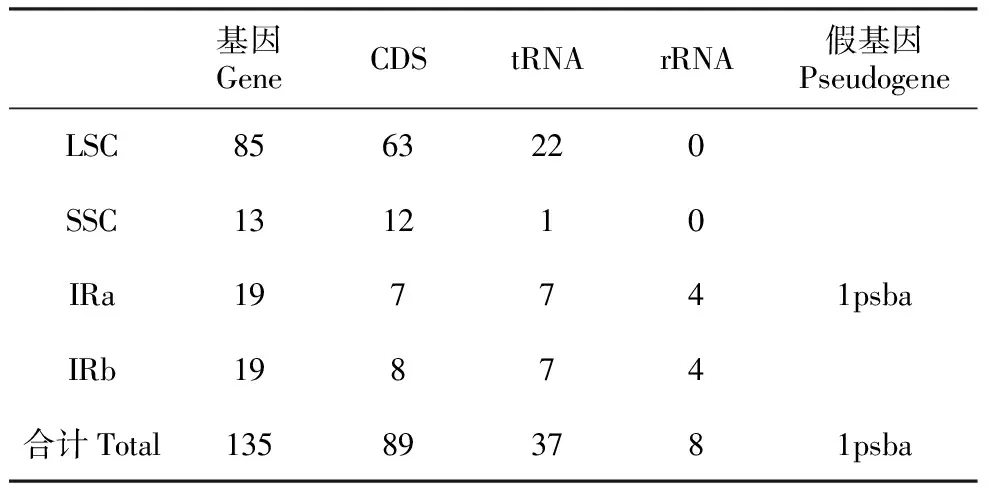
基因GeneCDStRNArRNA假基因PseudogeneLSC8563220SSC131210IRa197741psbaIRb19874合计Total135893781psba
2.3 鼠尾草叶绿体基因组中的基因归类
鼠尾草叶绿体基因组全部基因功能分类如表3所示,它共含有18个基因内含子,其中3个基因(clpP、rpsl2、ycf3)含有2个内含子。rpsl2是一个反式剪接基因,5′端位于LSC区,3′端在两个IR区各有一个拷贝。

图1 鼠尾草叶绿体基因组环形基因图 内侧基因顺时针;外侧基因逆时针转录;不同功能的基因以不同颜色表示。Fig.1 Gene map of chloroplast genome in S.japonica Genes drawn inside the circle are transcribed clockwise,and those outsides are counter clockwise.Genes belonging to different functional groups are color-coded.

基因分组Groupofgene基因名称Genename1光系统ⅠPhotosysteinⅠpsaA,psaB,psaC,pasalI,psalJ2光系统ⅡPhotosystemⅡpsbA,psbB,psbC,psbD,psbE,psbF,psbH,psbl,psbJ,psbK,psbL,psbM,psbN,psbT,psbZ3细胞色素b/f复合体Cytochromeb/fcomplexpetA,petB∗,petD∗,petG,petL,petN4ATP合酶ATPsynthaseatpA,atpB,atpE,atpF∗,atpH,atpI5NADH脱氢酶NADHdehydrogenasendhA∗,ndhB(x2),ndhC,ndhD,ndhE,ndhF,ndhG,ndhH,ndhI,ndhJ,ndhK6二磷酸核酮糖羧化酶大亚基RubisCOlargesubunitrbcL7核糖核酸聚合酶RNApolymeraserpoA,rpoB,rpoC1∗,rpoC28核糖体蛋白小亚基Ribosomalproteins(SSU)rps2,rps3,rps4,rps7(x2),rps8,rps11,rpsl2∗∗(x2),rpsl4,rps15,rpsl16∗,rpsl8,rpsl99核糖体蛋白小亚基Ribosomalproteins(LSU)rpl2∗(x2),rpll4,rpl16∗,rpl20,rpl22,rpl23(x2),rpl32,rpl33,rpl3610其他基因OthergenesclpP∗∗,matK,accD,ccsA,infA,cemA11未知蛋白Proteinsofunknownfunctionycfl,ycf2(x2),ycf3∗∗,ycf4,ycf15(x2)12转运RNATransferRNAs37tRNAs(6containanintron,7intheIRs)13核糖体RNARibosomalRNAsrrn4.5(x2),rrn5(x2),rrn16(x2),rrn23(x2)
注:*基因中有1或2个内含子
Note:*gene contains one or two introns.
表4鼠尾草叶绿体基因组内含子基因及外显子、内含子长度
Table4CharacterizationanalysisofgeneswithintronsinchloroplastgenomeofS.japonica
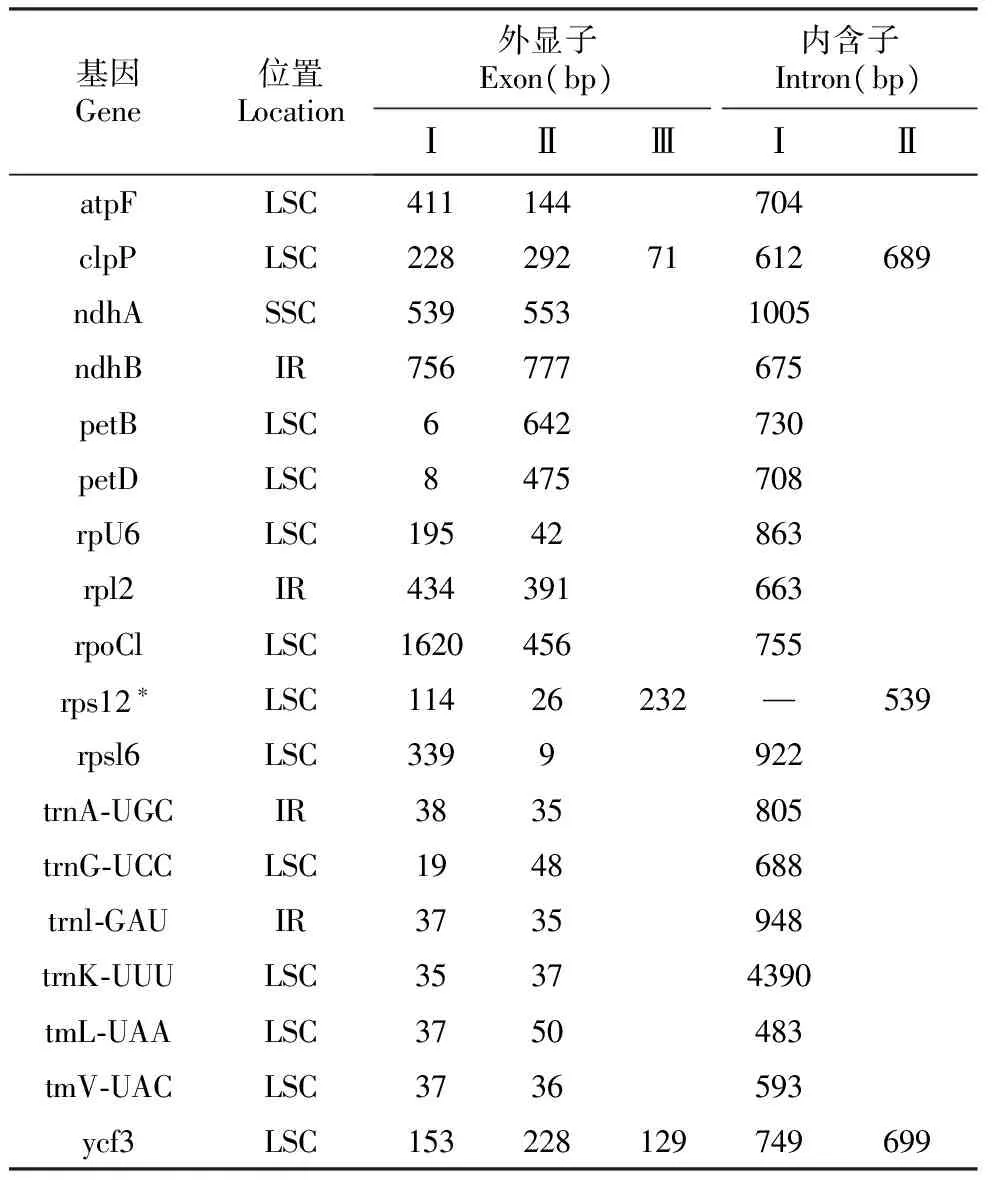
基因Gene位置Location外显子Exon(bp)内含子Intron(bp)ⅠⅡⅢⅠⅡatpFLSC411144704clpPLSC22829271612689ndhASSC5395531005ndhBIR756777675petBLSC6642730petDLSC8475708rpU6LSC19542863rpl2IR434391663rpoClLSC1620456755rps12∗LSC11426232—539rpsl6LSC3399922trnA⁃UGCIR3835805trnG⁃UCCLSC1948688trnl⁃GAUIR3735948trnK⁃UUULSC35374390tmL⁃UAALSC3750483tmV⁃UACLSC3736593ycf3LSC153228129749699
注:*rps12是一个反式剪接基因;5′端位于LSC区;3′端位于两个IR区
Note:*therps12 is a trans-spliced gene with the 5′ end located in the LSC region and the duplicated 3′ end in the IR regions.
2.4 鼠尾草叶绿体基因组内含子特征分析
52.3%、1.8%、6.0%的基因组序列分别编码蛋白、tRNA和rRNA基因,其余序列则归为非编码序列,包括内含子、基因间区及假基因。叶绿体基因组中内含子的基因特性如见表4所示,其中trnK-UUU内含子最长,多达4 390 bp,tmL-UAA内含子最短,为483 bp。
2.5 鼠尾草叶绿体基因组密码子使用情况
分析整个叶绿体基因组的密码子使用情况后发现共有2,797(10.6%)个密码子编码亮氨酸Leu,是编码率最髙的氨基酸;而只有292(1.1%)个密码子编码半胱氨酸Cys,是编码率最低的氨基酸(表5)。在所有密码子中,相对同义密码子使用频率(relative synonymous codon usage,RSCU)最高的为UGG,其次为UAU、GAU,最低的为ATA、CTG和GTG。
3 讨论
自地钱(Marchantiapolymorpha)完整叶绿体基因组发表以来[13],植物叶绿体基因组的研究得到国外内诸多学者的普遍关注,现成为叶绿体转化技术和植物亲缘关系鉴定的一个重要手段[14~15]。从先前的研究报道看,叶绿体基因组片段和核基因组序列已经普遍用于鼠尾草属植物的系统发育关系分析,并取得斐然成绩。

表5 鼠尾草叶绿体基因组密码子使用情况
注:相对同义密码子使用频率
Note:Rrelative synonymous codon usage,RSCU
譬如,Will等[16]通过ITS和ETS序列及叶绿体rpl32-trnL片段对非洲鼠尾草属41个类群系统发育的分析,认为非洲鼠尾草属物种并非单系起源(non-monophyletic);同样,Walker等[17]利用叶绿体rbcL和trnL-F片段对美洲鼠尾草属植物的研究,将其划分为3个独立的遗传谱系分支。在中国,李虔全[18]通过ITS序列构建系统发育树,认为中国鼠尾草属植物与日本鼠尾草属植物具有共同的祖先,新疆鼠尾草(S.deserta)与欧洲引进药鼠尾草(S.officinalis)具有较近的亲缘关系;然而,胡国雄[19]分子系统学的研究结果则表明鼠尾草属是一个包含了4个独立分支的多系类群,与Will等[20]的研究结果一致。尽管已有的分子系统学研究结果表明东亚鼠尾草属为单系类群,但不支持国产鼠尾草属的分类系统,国产鼠尾草属的分类系统不能通过合理地调整而与东亚鼠尾草的分子系统学研究结果相吻合,这可能与选取的研究样本和所选片段有关[19]。鼠尾草是鼠尾草属的一个广布种和具有重要经济及药用价值的物种,同时具有丰富的遗传多样性[12],因而加快对鼠尾草叶绿体基因组的分析,势必可为今后利用基因遗传标记、构建叶绿体超级条形码,揭示亚洲地区乃至世界范围内鼠尾草属的进化历史提供重要依据。目前,所有鼠尾草属植物的系统发育分析都是基于核基因和叶绿体基因序列片段信息,然而因片段信息常常存在测定序列的分子进化速率与物种水平上的进化速率存在差异,以检测序列位点的多样性来代替物种的多样性,这在一定程度上造成结果的不可靠性,基于基因组水平多态位点的检测结果更能真实反映物种系统发育关系[8]。我们以丹参叶绿体基因组序列作为参考,对鼠尾草叶绿体基因组的研究表明,它的全长为153 995 bp,同多数已公布的被子植物叶绿体基因组大小范围一样[21],由一对反向重复区(Inverted repeat,IR)分隔一个大的单拷贝区(Large single copy region,LSC)和一个小的单拷贝区(Small single copy region,SSC),呈典型的四段式结构[6],鼠尾草叶绿体的GC含量为38.0%,这与先前报道的丹参的研究结果一致[11]。同时,鼠尾草叶绿体基因组大小同丹参叶绿体基因组相似,隶属于是唇形目中较小的一族,叶绿体基因组大小的差异主要是因为LSC区的长度变异较大而导致的[11]。叶绿体基因组在基因组成及排列顺序上具有高度保守性,根据功能可将叶绿体基因分为三大类[22],第一类与转录、转译有关;第二类与光合作用系统有关;第三类是与氨基酸、脂肪酸、色素等其它生物合成相关,鼠尾草基因组成与之类似。通常,叶绿体基因组中往往具有假基因现象,本研究所测的鼠尾草叶绿体基因组也不例外,同样存在一些未知功能的基因,如ycf1基因,先前研究认为IRb/SSC边界往往可以扩张到ycf1基因中,从而导致ycf1假基因的发生[23~25]。此外,先前研究还发现,香豌豆属(Lathyrus)[26]和棉属(Gossypium)[27]植物的IR区都存在结构的变异,在进化过程中IR区部分的基因存在着丢失或萎缩现象,这些均有可能导致基因序列的变异,并极有可能致使物种进化事件的发生,因而我们推测它们很可能是鼠尾草叶绿体基因组中假基因产生的另一个重要原因之一。总之,本研究利用二代测序技术对鼠尾草的叶绿体基因组进行测序,并组装得到完整的叶绿体基因组序列,是继丹参后第二个鼠尾草属植物叶绿体基因组的建成,不仅可为今后鼠尾草种质资源的综合评价奠定了基础,而且也丰富了鼠尾草属的叶绿体基因组数据库,为种质资源的合理开发利用提供基础性数据。
1.魏宇昆,王琦,黄艳波.唇形科鼠尾草属的物种多样性与分布[J].生物多样性,2015,23(1):3-10.
Wei Y Q,Wang Q,Huang Y B.Species diversity and distribution ofSalvia(Lamiaceae)[J].Biodiversity Science,2015,23(1):3-10.
2.国家中医药管理局《中华本草》编委会.中华本草[M].上海:上海科学技术出版社,1999.
State Administration of Traditional Chinese Medicine.Chinese herbal medicine[M].Shanghai:Shanghai Science and Technology Publisher,1999.
3.Isom T,Noda Y.Studies on the chemical constituents ofSalviajaponicaandS.glabrescens[J].Nippon Kagaku Zassi,1991(3):244-246.
4.冯珂,赵鑫,郑丽玲,等.3种鼠尾草属植物有效成分分析[J].西北林学院学报,2010,25(6):140-143.
Feng K,Zhao X,Zheng L L,et al.Analysis of active ingredients in threeSalviaplants[J].Journal of Northwest Forestry University,2010,25(6):140-143.
5.Wester P,Claβen-bockhoff R.Pollination syndromes of new worldSalviaspecies with special reference to bird pollination[J].Annals of the Missouri Botanical Garden,2011,98(1):101-155.
6.Bentham G.Labiatarum,genera et species[M].London:Ridgway,1832.
7.陈士林,宋经元,姚辉,等.药用植物DNA条形码鉴定策略及关键技术分析[J].中国天然药物,2009(5):322-327.
Chen S L,Song J Y,YAO H,et al.Strategy and key technique of identification of Chinese herbal medicine using DNA barcoding[J].Chinese Journal of Natural Medicines,2009(5):322-327.
8.邓科君,张勇,鄢丹,等.鼠尾草属部分物种AFLP指纹图谱构建及遗传多样性分析[J].植物遗传资源学报,2010,11(4):483-486.
Deng K J,Zhang Y,Yan D,et al.Construction of AFLP fingerprint and genetic diversity analysis of the genusSalvia[J].Journal of Plant Genetic Resources,2010,11(4):483-486.
9.Clegg M T,Gaut B S,Learn G H J,et al.Rates and patterns of chloroplast DNA evolution[J].Proceedings of the National Academy of Sciences of the United States of America,1994,91(15):6795-6801.
10.林小涵,刘志华,李卿,等.药用植物叶绿体基因组研究[J].世界科学技术—中医药现代化,2010,12(3):442-446.
Lin X H,Liu Z H,Li Q,et al.Chloroplast genome of medicinal plants[J].World Science & Technology,2010,12(3):442-446.
11.Qian J,Song J Y,Gao H H,et al.The complete chloroplast genome sequence of the medicinal plantSalviamiltiorrhiza[J].PLoS One,2013,8(2):e57607.
12.Sudarmono S,Okada H.Genetic variation ofSalviajaponicathunb.(Lamiaceae)and its related species[J].Floribunda Journal,2011,4(2):33-39.
13.Umesono K,Inokuchi H,Ohyama K,et al.Nucleotide sequence ofMarchantiapolymorpha chloroplast DNA:a region possibly encoding three tRNAs and three proteins including a homologue ofE.coliribosomal protein S14[J].Nucleic Acids Research,1984,12(24):9551-9565.
14.Li X W,Gao H H,Wang Y T,et al.Complete chloroplast genome sequence ofMagnoliagrandifloraand comparative analysis with related species[J].Science China Life Sciences,2013,56(2):189-198.
15.邢少辰,Liu C J.叶绿体基因组研究进展[J].生物化学与生物物理进展,2008,35(1):21-28.
Xin S C,Liu C J.Progress in chloroplast genome analysis[J].Progress in Biochemistry and Biophysics,2008,35(1):21-28.
16.Will M,Claβen-bockhoff R.Why Africa matters:evolution of Old WorldSalvia(Lamiaceae)in Africa[J].Annals of Botany,2014,114(1):61-83.
17.Walker J,Sytsma K J,Wink M.Salvia(Lamiaceae) is not monophyletic:implications for the systematics,radiation,and ecological specializations ofSalviaand tribe mentheae[J].American Journal of Botany,2004,91(7):1115-1125.
18.李虔全.中国鼠尾草属植物的分子系统发育及其亚属间关系的研究[D].包头:内蒙古科技大学包头医学院,2013.
Li Q Q.Phylogenetic relationships ofSalvia(Lamiaceae)in China:evidence from DNA sequence datasets[D].Baotou:Baotou Medical College,2013.
19.胡国雄.唇形科鼠尾草属东亚分支的分子系统学研究[D].北京:中国科学院大学,2015.
HU G X.Phylogeny ofSalvia(Lamiaceae) in East Asia[D].Beijing:Chinese Academy of Sciences University,2015.
20.Will M,Claβen-bockhoff R.Time to splitSalvias.l.(Lamiaceae)-New insights from Old WorldSalviaphylogeny[J].Molecular Phylogenetics and Evolution,2017,109:33-58.
21.Yang M,Zhang X W,Liu G M,et al.The complete chloroplast genome sequence of date palm(PhoenixdactyliferaL.)[J].PLoS One,2010,5(9):e12762.
22.Wakasugi T,Tsudzuki T,Sugiura M.The genomics of land plant chloroplasts:Gene content and alteration of genomic information by RNA editing[J].Photosynthesis Research,2001,70(1):107-118.
23.Raubeson L A,Peery R,Chumley T W,et al.Comparative chloroplast genomics:analyses including new sequences from the angiospermsNupharadvenaandRanunculusmacranthus[J].BMC Genomics,2007,8:174.
24.Goulding S E,Wolfe K H,Olmstead R G,et al.Ebb and flow of the chloroplast inverted repeat[J].Molecular and General Genetics,1996,252(1-2):195-206.
25.Wang R J,Cheng C L,Chang C C,et al.Dynamics and evolution of the inverted repeat-large single copy junctions in the chloroplast genomes of monocots[J].BMC Evolutionary Biology,2008,8:36.
26.Magee A M,Aspinall S,Rice D W,et al.Localized hypermutation and associated gene losses in legume chloroplast genomes[J].Genome Research,2010,20(12):1700-1710.
27.Talat F.三个D组棉花叶绿体全基因组测序及其进化研究[D].北京:中国农业科学院,2013.
Talat F.Complete chloroplast genome sequences of Three D genome cotton species and their evolutionary implications[D].Beijing:Chinese Academy of Agricultural Sciences,2013.
Under the auspices of the National Natural Science Foundation of China(32170338);The Key Project of Natural Science Foundation of Shaanxi Preschool Normal College(2015ZDKJ005)
introduction:HE Yi-Han(1987—),female,Ph.D Candidates,engaged in research of medicinal plant secondary metabolic and molecular regulation.
date:2017-03-06
CompleteSequenceAnalysisofChloroplastGenomeofSalviajaponica
HE Yi-Han1,2HAN Li-Min3LIU Yu-Ping3TIAN Na3SU Xu2WANG Zhe-Zhi1*
(1.Key Laboratory of the Ministry of Education for Medicinal Resources and Natural Pharmaceutical Chemistry,National Engineering Laboratory for Resource Development of Endangered Crude Drugs in Northwest of China,College of Life Sciences,Shaanxi Normal University,Xi’an 710119;2.College of Geography and Life Science,Qinghai Normal University,Xining 810008;3.College of Life Sciences and Food Engineering,Shaanxi Xueqian Normal University,Xi’an 710061)
Salviajaponicais a perennial herb of the genusSalvia(Lamiaceae), which is of great medicinal and economic value. The chloroplast genome ofS.japonicawas sequenced using Illumina Hiseq platform. The complete chloroplast genome sequence was obtained bySalviamiltiorrhizachloroplast genome as a reference. The full-length chloroplast genome ofS.japonicawas 153 995 bp. The LSC was 84 573 bp and 19 874 bp in SSC, and the length of IR region was 24 774 bp. The chloroplast genome ofS.japonicasuccessfully annotated 13 groups of genes. The gene types, number and GC content were similar with other species of Lamiaceae. These results enrich the chloroplast data and also provide some valuable molecular data for reconstructing evolutionary history ofSalvia.
Salvia;Salviajaponica;Chloroplast genome;gene structure;phylogenetics and evolution
国家自然科学基金项目(32170338)资助;陕西学前师范学院自然科学基金重点项目(2015ZDKJ005)资助
何懿菡(1987—),女,博士研究生,主要研究方向为药用植物次生代谢及分子调控。
* 通信作者:E-mail:zhezhiwang@outlook.com
2017-03-06
* Corresponding author:E-mail:zhezhiwang@outlook.com
Q949.777.6
A
10.7525/j.issn.1673-5102.2017.04.013
