中间偃麦草TiAP1基因的克隆及生物信息学分析
2017-11-01田秋菊宋静杨艳琳许田王洪刚封德顺
田秋菊,宋静,杨艳琳,许田,王洪刚,封德顺
(山东农业大学农学院/作物生物学国家重点实验室,山东 泰安 271018)
中间偃麦草TiAP1基因的克隆及生物信息学分析
田秋菊,宋静,杨艳琳,许田,王洪刚,封德顺
(山东农业大学农学院/作物生物学国家重点实验室,山东 泰安 271018)
本研究从烟农15和中间偃麦草杂交所得的小偃麦异附加系SN6306中克隆得到TiAP1基因的全长序列,该序列全长1 272 bp,预测编码423个氨基酸。通过BlastP分析,发现其属于天冬氨酸蛋白酶家族。生物信息学分析发现,TiAP1蛋白含有信号肽且位于第16和17个氨基酸之间,同时含有两个木聚糖酶抑制剂结构域,分别位于该蛋白的N端和C端。此外,通过TMHMM预测,发现该蛋白无跨膜螺旋结构,因此是胞外蛋白。为了检测基因的亲缘关系,构建系统进化树,发现TiAP1基因与乌拉尔图小麦以及粗山羊草相应基因有较近的亲缘关系。上述结果为研究TiAP1基因的功能奠定了基础。
小偃麦异附加系;TiAP1;基因克隆;生物信息学分析;白粉病
小麦是目前全球种植最广的粮食作物之一。民以食为天,粮食问题一直关乎着国计民生,而小麦的产量与安全也关乎着国家的粮食安全。与水稻等农作物相比,小麦中含有更加丰富的营养物质。小麦与其他农作物相比有较长的生长期,抗灾能力强,经济效益相对较高,对解决吃粮问题有很重要的现实意义[1]。但是,在生产过程中,有很多生物和非生物胁迫因素影响小麦的产量和品质,比如:叶枯病、根腐病、黄叶病等。
小麦白粉病是常见小麦病害之一[2],其病原菌是一种特殊的病原真菌,属于禾本科布氏白粉菌(Blumeriagraminisf. sp.tritici)。它是活体营养专性寄生真菌,属于子囊菌亚门真菌的一种[3]。近几年来,白粉病蔓延迅速,至今为止,在全国所有麦区几乎都有爆发,减产可达 1/10至1/5,发病最严重时减产可达2/5至3/5[4],严重影响小麦丰产。
天冬氨酸蛋白酶(aspartic proticinase,APs)是四大类蛋白酶之一,该酶活性较强,即使在酸性环境下依然可以保持活性[5],其在真核生物以及微生物中分布广泛,主要参与机体的新陈代谢及生物调控[6]。多种生物体内都存在APs,例如:病毒、真菌、哺乳动物及植物[7]。它在不同的植物器官中表达,比如:种子、茎、叶片、花、根。作为一类蛋白水解酶,天冬氨酸蛋白酶的生物学功能具有多样性,其在不同的植物发育过程和部位行使的功能各有不同[8]。Kadek等[9]研究指出肉食性的猪笼草可以分泌一种液体以此来降解捕获的昆虫,为自身提供氮源,通过研究发现该液体中含有天冬氨酸蛋白酶,这种天冬氨酸蛋白酶最低可以在pH 2.5左右维持活性,且对于高pH值也显示了不同寻常的活性。该天冬氨酸蛋白酶序列与其他植物、动物APs有很大不同,它含有更多的半胱氨酸残基,这也可能是猪笼草能抗高温和耐酸的原因。Chen等[10]在水稻中发现了96个OsAP基因,其中66个在叶中表达,种子中的AP基因受激素和黑暗/光照的诱导有不同程度的上调表达。
近几年来,植物体内天冬氨酸蛋白酶基因的研究越来越引起人们的关注。本实验室在前期的研究中利用RNA-seq技术发现了一个新的天冬氨酸蛋白酶基因TiAP1,同时通过VIGS技术研究发现该基因可能与白粉病有关。本研究则在此基础上从小偃麦异附加系中克隆TiAP1基因全长,并对其进行生物信息学分析,以期为今后TiAP1基因的功能研究及在育种上的应用奠定基础。
1 材料与方法
1.1试验材料
供试材料为本实验室利用小麦近缘植物中间偃麦草(Thinopyrumintermedium,2n=42)与普通小麦烟农15(TriticumaestivumL.,2n=42)杂交得到的小偃麦异附加系SN6306、小麦烟农15以及中间偃麦草。
1.2材料处理
SN6306在蛭石和基质比列1∶1的条件下进行培养至三叶期,用镊子涂抹接种白粉病菌生理小种E09,在处理0、12、24、48、72 h和96 h时取叶片,将取得的叶片放入液氮中速冻后,迅速放于-80℃冰箱中冷冻低温保存,用于RNA的提取。
1.3 RNA的提取与反转录
所用材料为经白粉病处理12、24、48、72 h和96 h的SN6306叶片各个时间点的混合样,RNA提取步骤参照全式金的EasyPureTMPlant RNA Kit提取试剂盒说明书进行。RNA提取之后先用核酸测定仪 NanoPhotometer P360(Implent,德国)进行浓度测定,再用2%琼脂糖凝胶电泳进行RNA浓度和质量测定。
以提取的叶片总RNA为模板,利用全式金的TransScript®First-Strand cDNA Synthesis SuperMix(TransGen,China)反转录试剂盒进行反转录。具体过程如下:在RT管中加入总RNA约3 μg,Oligo(dT)2 μL,加水至终体积为9 μL,冰上操作,轻轻混匀;然后将管置于80℃ 3 min,冰上冷却2 min;接着向管中加入2×TS Reaction Mix 10 μL,TransScript®RT/RI Enzyme Mix 1 μL,使最终的反应体系为20 μL;接着反应体系按如下程序进行:42℃ 90 min,94℃加热2 min,冰上放置2 min,轻轻混匀,然后向体系中加入80 μL DNase-free水对第一链cDNA 进行稀释,并放于-20℃冰箱待用。
1.4TiAP1基因克隆
根据先前的RNA-seq结果中得到的TiAP1基因序列,设计特异引物对APF: 5′-AGGCAGACCTCACCCAAAC-3′、APR: 5′-ACAGAGAGAATACTACGAGACACGA-3′,以得到的cDNA为模板进行PCR反应。PCR反应体系(10 μL)为:cDNA 4 μL(约50 ng),10 μmol/L APF/R引物各0.5 μL,2×PCR Mix 5 μL。反应程序如下:首先94℃预变性5 min,然后进入Touch Down-PCR循环:94℃变性30 s,退火(65~55℃)1 min,后72℃延伸2 min,退火温度每个循环降低1℃,10个循环后降到55℃,再在55℃下进行25个循环,最后72℃延伸10 min,于10℃保存。PCR反应产物用1%琼脂糖凝胶进行电泳检测,利用EtBr进行染色,凝胶成像仪成像。用TaKaRa胶回收试剂盒切取目的条带进行胶回收,回收基因与克隆载体pMD18-T连接,将连接产物交由上海博尚生物技术有限公司进行测序。
1.5生物信息学分析
对TiAP1基因序列和蛋白序列进行生物信息学分析,具体为:TiAP1基因序列的开放阅读框(http://www.ncbi.nlm.nih.gov/gorf/gorf.html, ORFfinder)、TiAP1蛋白序列的信号肽(http://www.cbs.dtu.dk/services/SignalP/, SignalP)、蛋白亲疏水性(ProtScale, ExPASy)、蛋白结构(http://blast.ncbi.nlm.nih.gov/Blast.cgi, BLAST)等;蛋白质的多序列联配使用软件DNAMAN;系统进化树分析使用软件MEGA5。为了得到更多的TiAP1蛋白特征,进行跨膜螺旋预测(http://www.cbs.dtu.dk/services/TMHMM/)[13]、蛋白质三级结构模型预测(http://www.sbg. bio.ic.ac.uk/phyre2/html/page.cgi?id=index, Phyre2)[14,15]、蛋白质的Z值计算和蛋白质残基能量的计算(https://prosa.services.came.sbg.ac.at/prosa.php)[16]以及等电点、分子量等预测。
1.6TiAP1基因组序列的扩增比对
YN15、SN6306、中间偃麦草基因组DNA按照CTAB法提取,然后利用设计的APF/R引物以基因组DNA为模板扩增TiAP1的基因组序列。扩增体系:2×PCR Mix 5 μL,10 μmol/L APF/R各0.5 μL,模板DNA 1 μL,ddH2O补至10 μL。反应程序:首先94℃预变性5 min,然后进入Touch Down-PCR循环:94℃变性30 s,退火(72~60℃)1 min,后72℃延伸2 min,退火温度每个循环降低1℃,12个循环后降到60℃,再在60℃下进行25个循环,最后72℃延伸10 min,于10℃保存。PCR反应产物用1%琼脂糖凝胶进行电泳检测,利用EtBr进行染色,凝胶成像仪成像。用TaKaRa胶回收试剂盒切取目的条带进行胶回收,回收基因与克隆载体pMD18-T连接,将连接产物交由上海博尚生物技术有限公司进行测序。
2 结果与分析
2.1TiAP1基因的克隆
利用设计的APF/R引物对TiAP1基因进行扩增,得到的基因全长为1 272 bp(图1),编码423个氨基酸,通过BlastP分析,发现该基因属于天冬氨酸蛋白酶家族(图2、图3)。

M:DL2000 Marker;1:TiAP1扩增条带
图1TiAP1基因扩增结果
2.2TiAP1基因的多序列比对和聚类分析
利用TiAP1基因编码的氨基酸序列在NCBI中查找,在11个其它物种中找到了APs蛋白,对其进行多序列比对分析,结果表明TiAP1的氨基酸序列与这11个物种中的APs具有很高的相似性(图4)。对这共计12个APs蛋白进行进化树分析,发现TiAP1编码的氨基酸序列与乌拉尔图小麦以及粗山羊草中的APs序列相似,表现为亲缘关系(图5)。
2.3TiAP1基因组序列的扩增比对
利用设计的APF/R引物,以提取的YN15、SN6306、中间偃麦草DNA为模板进行PCR扩增。结果(图6)显示,在SN6306和中间偃麦草中扩出了大小一致的目的条带,而YN15中却未扩增出任何条带。测序分析发现,从SN6306和中间偃麦草中扩增到的DNA序列相似性将近99%,而该基因序列与小麦已测序数据库(https://wheat-urgi.versailles.inra.fr/Projects)中的AP基因相似性较低(图7、图8)。以上结果证明了TiAP1基因是来自于中间偃麦草的一个新基因。

图2TiAP1基因的核苷酸序列和推导的氨基酸序列

图3 TiAP1蛋白的BlastP分析

TiAP1(Elytrigiaintermedia(Host) Nevski中间偃麦草)、TuAP(EMS56884.1,Triticumurartu乌拉尔图小麦)、AtAP(EMT20017.1,Aegilopstauschii粗山羊草)、SiAP(XP_004953106.1,Setariaitalica狗尾草)、BdAP(XP_003578403.1,Brachypodiumdistachyon短柄草)、ZmAP(XP_008674331.1,Zeamays玉米)、NaAP(BAF98915.1,Nepenthesalata猪笼草)、VvAP(XP_002264056.1,Vitisvinifera葡萄)、SlAP(XP_004239638.1,Solanumlycopersicum番茄)、MaAP(XP_009410092.1,Musaacuminata野芭蕉)、PdAP(XP_008777368.1,Phoenixdactylifera海枣)、EgAP(XP_010907568.1,Elaeisguineensis油棕)。下图同。
图4 TiAP1与其他物种APs蛋白序列比对分析
2.4 TiAP1蛋白的生物信息学分析
分析发现TiAP1蛋白含有信号肽且位于第16和17个氨基酸残基之间(图9),但是没有跨膜螺旋,所以不是分泌蛋白(图10)。该蛋白含有两个木聚糖酶抑制剂结构域,分别位于该蛋白的N和C末端(图11)。
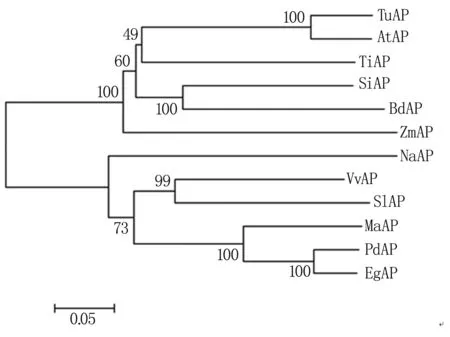
图5 TiAP1与其他物种APs蛋白序列的聚类分析结果
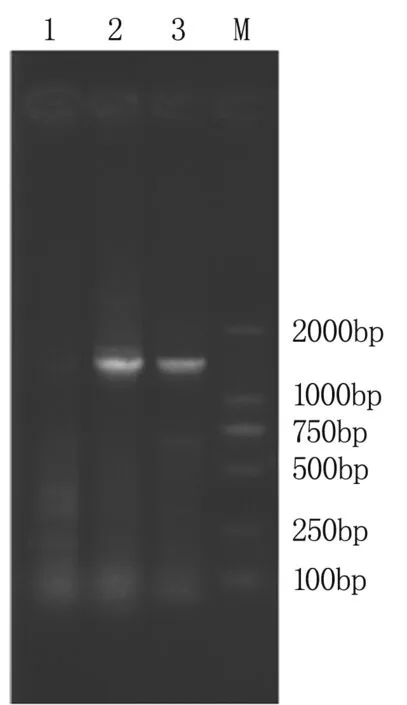
M:DL2000 Marker;1:YN15;2:SN6306;3:中间偃麦草
图6 APF/R引物对YN15、SN6306、中间偃麦草DNA的扩增结果
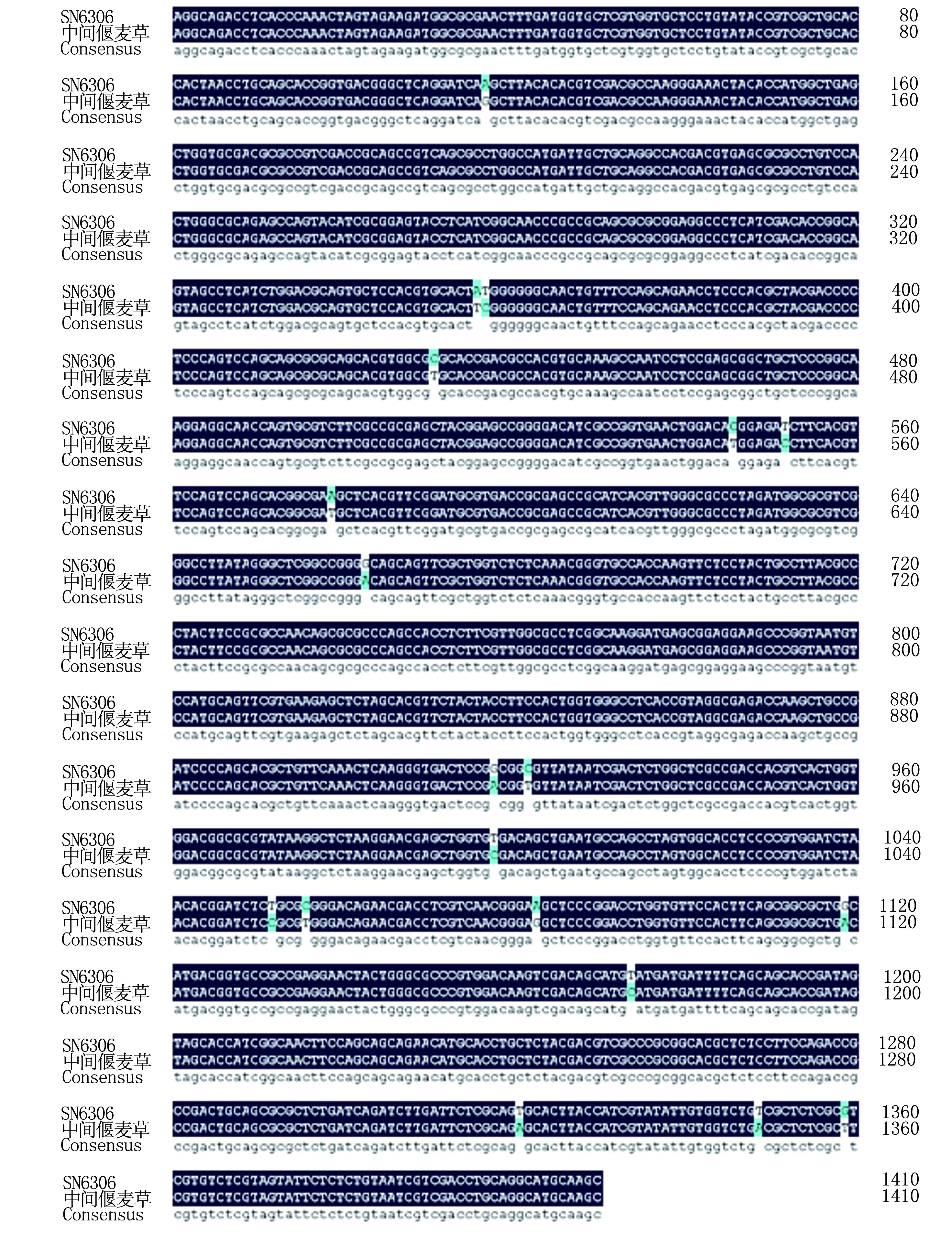
图7 SN6306、中间偃麦草TiAP1基因扩增序列比对
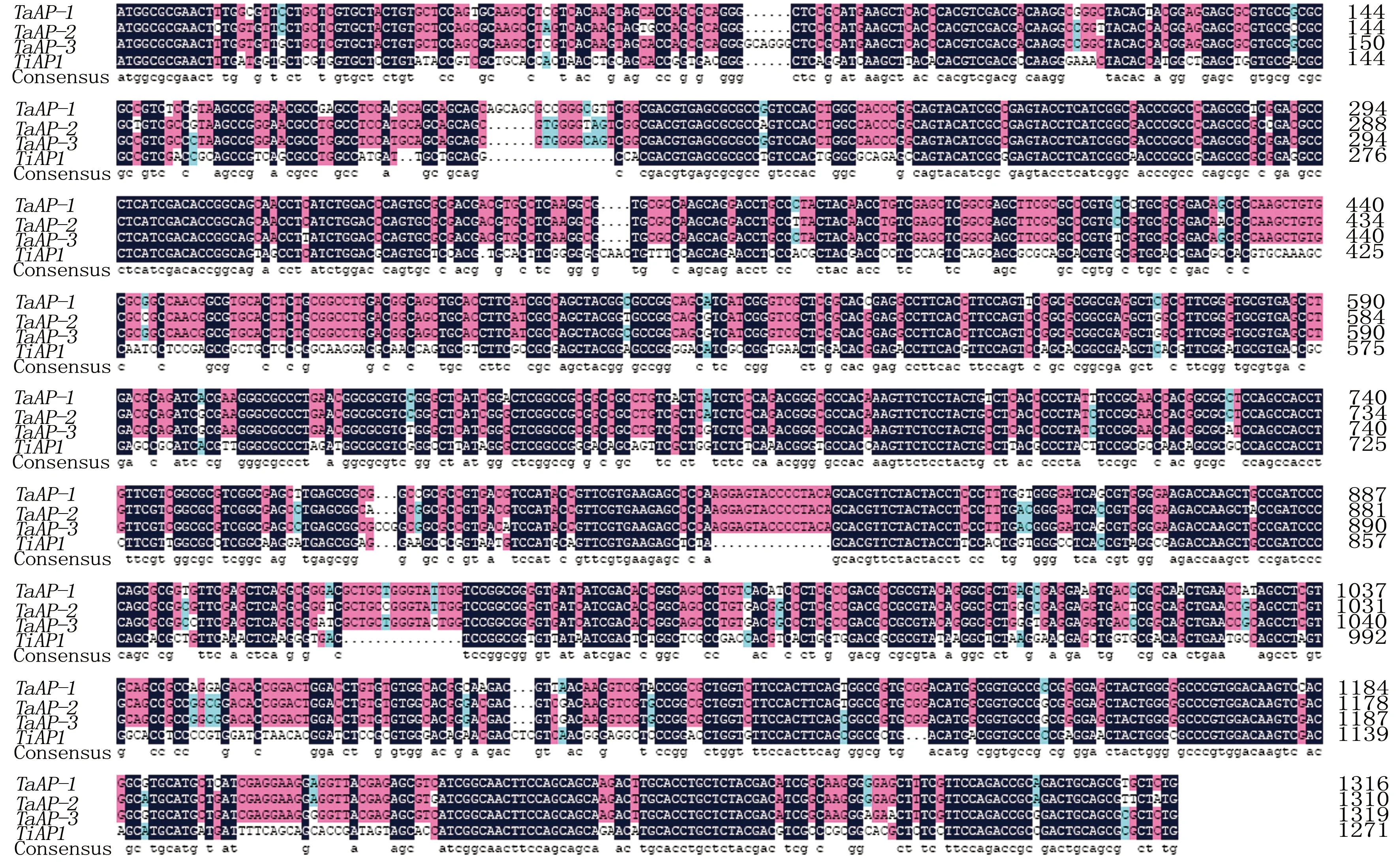
TaAP-1~ TaAP-3为小麦中的3个AP基因。
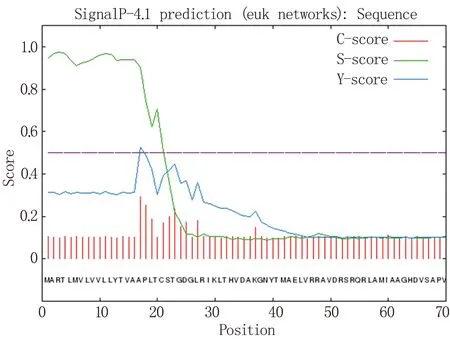
图9 TiAP1蛋白信号肽结构的预测
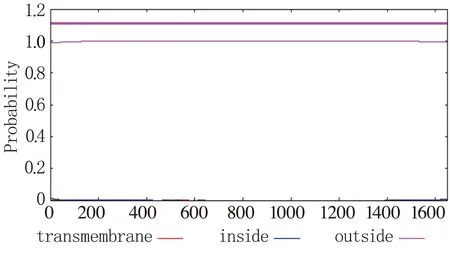
图10 TiAP1蛋白跨膜螺旋的预测
通过ProtParam对蛋白的理化特性进行了分析,比如:分子量等电点(pI)消光系数以及亲水系数等(表1)。通过SOPMA预测发现,在TiAP蛋白二级结构中延伸链和无规则卷曲所占比例较高(表1)。
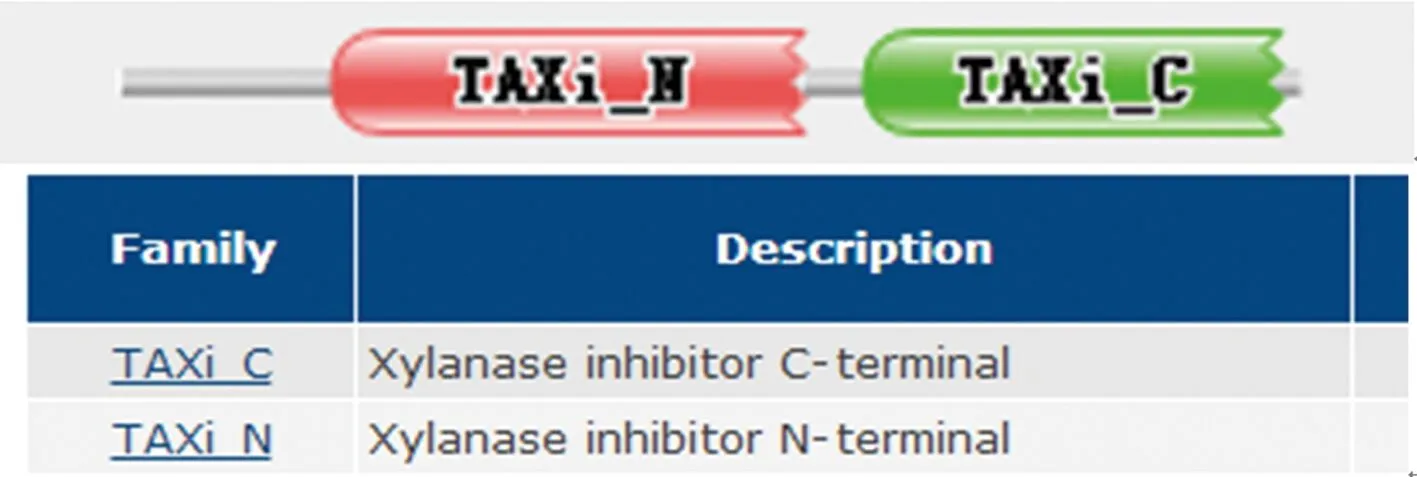
图11 TiAP1的蛋白结构域分析
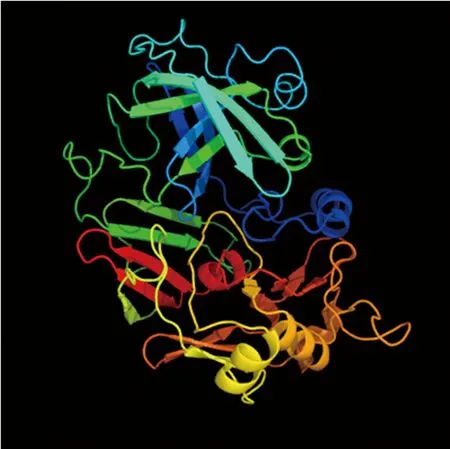
图12 TiAP1蛋白质三级结构预测
利用Phyre2对TiAP蛋白进行三级结构预测以及准确性分析(图12)。为了识别三级结构中的错误,用ProSA对三级结构进行检测。Z值是蛋白链通过X射线晶体照相或者核磁共振(NMR)计算出来的。Z值代表了整个模型的质量,Z>0,表明三级结构错误较多,而且不稳定,Z值为0或负数表明三级结构模型比较稳定。通过计算该蛋白三级结构模型的Z值为-8.52(图13),说明该三级结构模型比较理想。从图14可以看出氨基酸残基大部分都小于零,说明目的蛋白是可以构建三级结构模型的。
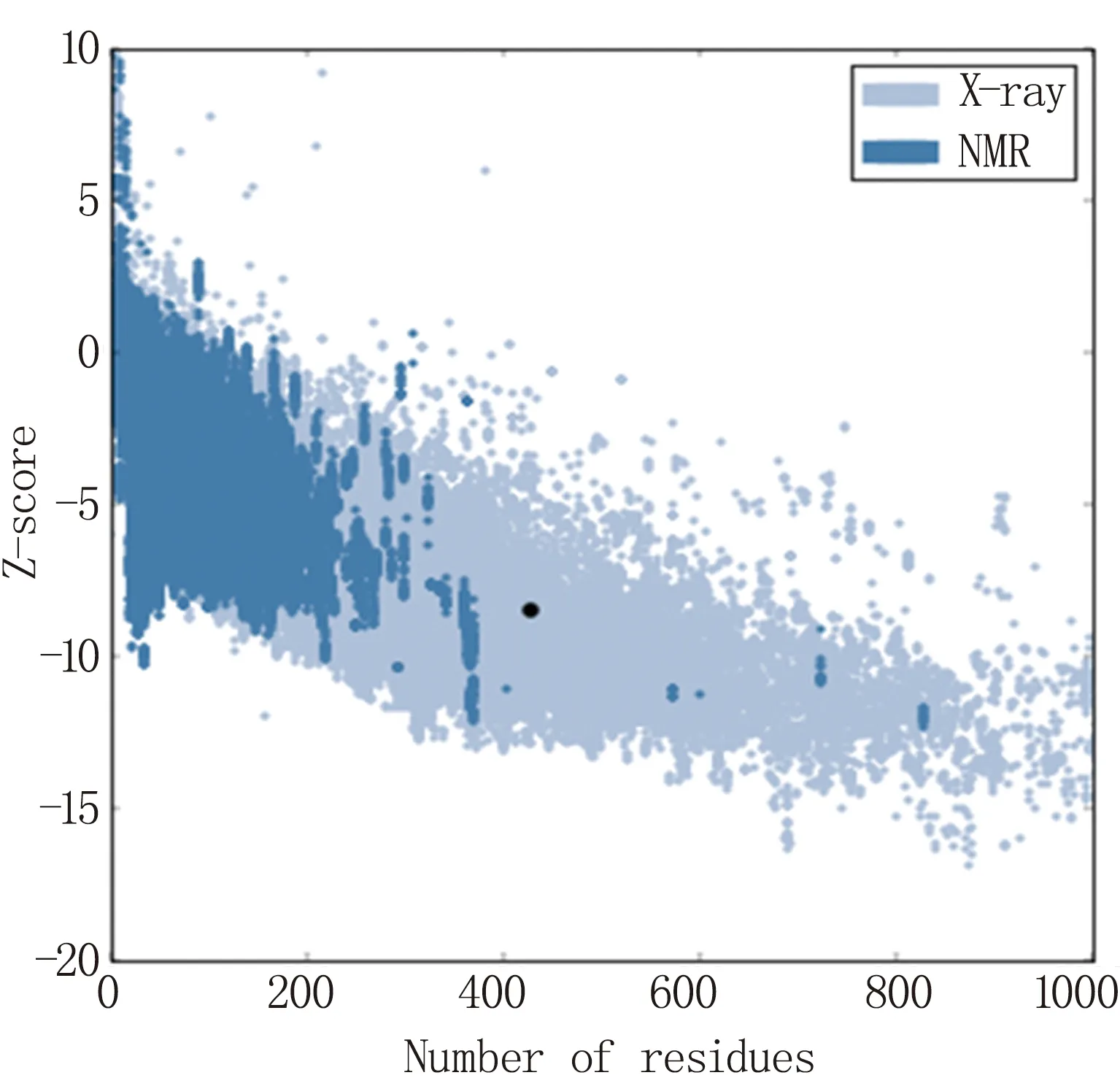
图13 蛋白质Z值的计算
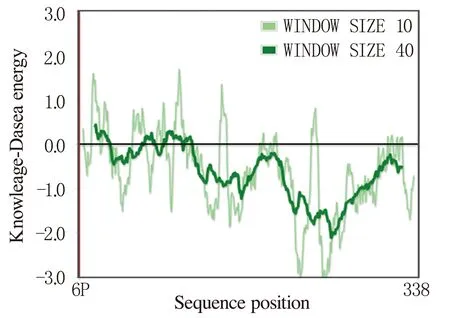
图14 TiAP1蛋白质中氨基酸残基能量图
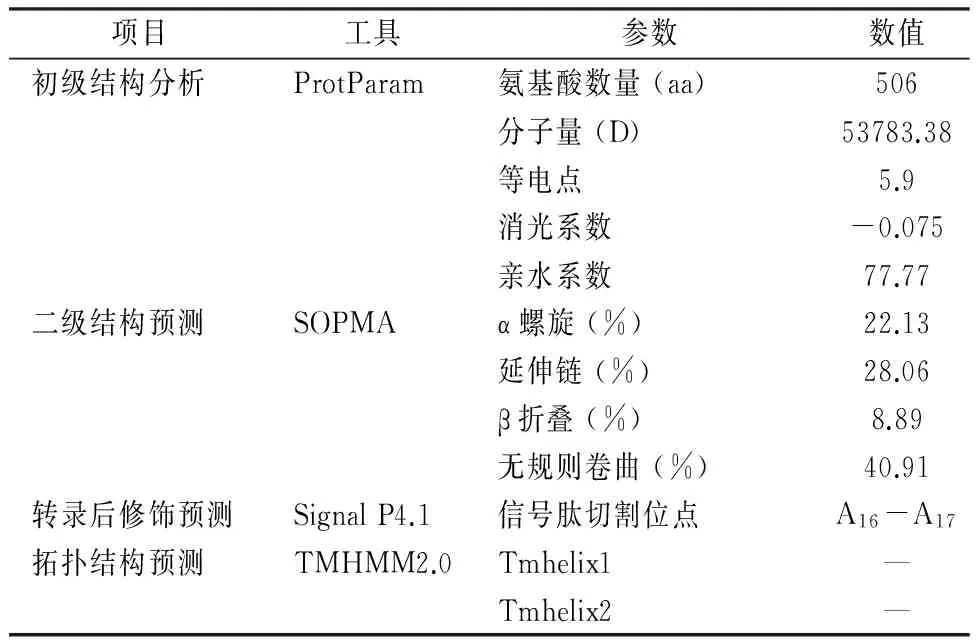
表1 蛋白质的初级结构、二级结构、翻译后修饰、跨膜结构预测
3 讨论与结论
本研究利用RNA-seq技术从烟农15和中间偃麦草杂交所得的小偃麦异附加系SN6306中克隆得到一个新的天冬氨酸蛋白酶基因TiAP1。该基因属于天冬氨酸蛋白酶家族,全长1 272 bp,编码423个氨基酸,含有信号肽,与乌拉尔图小麦以及粗山羊草具有较近的亲缘关系。利用HMHMM对跨膜螺旋进行预测,发现没有跨膜螺旋结构,因此属于胞外蛋白。对TiAP1蛋白的理化特性进行预测,目的蛋白等电点约为5.9,信号肽的切割位点位于第16和17个氨基酸之间。此外,通过对该蛋白的二级结构和三级结构进行预测,发现二级结构中延伸链和无规则卷曲所占的比例较高,三级结构构建的模型比较稳定。蛋白的空间结构很大程度上决定了蛋白质的生物学功能,因此了解蛋白质的结构对研究蛋白质的功能有重要意义。
有研究发现天冬氨酸蛋白酶基因在生物体内有重要作用。Contour-Ansel等[11]从普通大豆中克隆出一个天冬氨酸蛋白酶基因PvAP1,属于典型天冬氨酸蛋白酶;同时发现PvAP1受水分胁迫的影响,影响其转录水平和翻译后水平,由此推测天冬氨酸蛋白酶可能参与豆科植物的抗旱过程。Prasad等[12]在水稻中发现了一种编码典型天冬氨酸蛋白酶的基因OsCDR1,通过原核表达系统得到OsCDR1融合蛋白,利用融合蛋白处理拟南芥叶片,发现PR2转录本在处理和未处理的叶片中均有表达,而CDR1编码的没有活性的融合蛋白不能诱导PR2转录本在局部和系统中的表达,这表明OsCDR1有引起植物体系统防御反应的功能。
本研究较系统地研究了TiAP1基因的序列特征、系统进化等方面的内容,这在一定程度上为进一步研究TiAP1基因的作用机制奠定了基础。
[1] 郭绵英, 刘红茹.小麦白粉病的发生与综合防治措施[J].农技服务, 2012, 29(3):305.
[2] 于艳娟, 李国利, 王学臣. 小麦白粉病的发生原因与防治措施[J]. 天津农林科技, 2012 (4): 24-25.
[3] 牛祖彪. 白粉病菌诱导小偃麦异附加系SN6306差异表达基因的克隆与功能分析[D]. 泰安:山东农业大学, 2014.
[4] 陈芳. 武隆县小麦白粉病的发生原因及防治对策[J]. 南方农业, 2012, 6(10):43-44.
[5] Szecsi P B. The aspartic proteases[J]. Scandinavian Journal of Clinical Laboratory Investigation, 1992, 52(S210):5-22.
[6] Guevara M G, Oliva C R, Huarte M, et al. An aspartic protease with antimicrobial activity is induced after infection and wounding in intercellular fluids of potato tubers [J]. European Journal of Plant Pathology, 2002, 108(2):131-137
[7] Gomez-Gomez L, Felix G, Boller T. A single locus determines sensitivity to bacterial flagellin inArabidopsisthaliana[J]. Plant Journal, 1999, 18:277-284.
[8] 陈荣, 汲广东. 天冬氨酸蛋白酶家族的分类、功能和演化[J]. 鲁东大学学报, 2011, 27(4):328-334.
[9] Kadek A, Tretyachenko V, Mrazek H, et al. Expression and characterization of plant aspartic protease nepenthesin-1 fromNepenthesgracilis[J]. Protein Expression and Purification, 2014, 95:121-128.
[10] Chen J, Ouyang Y, Wang L. Aspartic proteases gene family in rice: gene structure and expression predicted protein features and phylo-genetic relation[J]. Gene, 2009, 442 (1/2):108-118.
[11] Contour-Ansel D, Torres-Franklin M L, Zuily-Fodil Y, et al. An aspartic acid protease from common bean is expressed ‘on call’ during water stress and early recovery [J]. Journal of Plant Physiology, 2010, 167(18): 1606-1612.
[12] Prasad B D, Creissen G, Lamb C, et al. Heterologous expression and characterization of recombinant OsCDR1, a rice aspartic proteinase involved in disease resistance[J]. Protein Expression and Purification, 2010, 72(2):169-174.
CloningandBioinformaticsofTiAP1GenefromThinopyrumintermedium
Tian Qiuju, Song Jing, Yang Yanlin, Xu Tian, Wang Honggang, Feng Deshun
(CollegeofAgronomy,ShandongAgriculturalUniversity/StateKeyLaboratoryofCropBiology,Taian271018,China)
The wheat-Thinopyrumalien addition line SN6306 was originated from the intergeneric hybridization between YN15 andThinopyrumintermedium(Host) Nevski. Through PCR amplification, the full length cDNA ofTiAP1 gene was obtained. Its open reading frame (ORF) was 1 272 bp and encoded a peptide of 423 amino acids. The BlastP analysis results showed that it belonged to the aspartic protease family. The bioinformatics analysis results showed that TiAP1 protein contained a signal peptide, whose cleavage site was located between the 16th and 17th amino acid, and two xylanase inhibitor domain structures, which located in the N and C end respectively. The TMHMM predicting results showed that the protein had no transmembrane helix structure. So the protein was not secreted protein. The phylogenetic tree showed thatTiAP1 gene was similar to the corresponding gene inTriticumurartuThum.ex Gandil andAegilopstauschii. These results would provide the reference for the research ofTiAP1 gene functions.
Wheat-Thinopyrumalien addition line;TiAP1; Gene clone; Bioinformatics analysis; Powdery mildew
S512.901
A
1001-4942(2017)10-0001-09
10.14083/j.issn.1001-4942.2017.10.001
2017-05-17
国家重点研发计划项目(2016YFD0102000);国家自然科学基金面上项目(31171552)
田秋菊(1990—),女,在读硕士研究生,研究方向为生物技术在小麦遗传改良中的应用。E-mail: qiujutian@yeah.net
封德顺(1970—),男,教授,主要从事小麦分子生物技术育种研究。E-mail: dsfeng@sdau.edu.cn
