使用CRISPR/Cas9技术构建新型重组伪狂犬病毒疫苗的初步研究
2017-11-01于之清李国新武吉强黄勤峰童光志
于之清,童 武,郑 浩,李国新,高 飞,王 涛,梁 超,叶 超,武吉强,黄勤峰,童光志
(中国农业科学院上海兽医研究所,上海200241)
使用CRISPR/Cas9技术构建新型重组伪狂犬病毒疫苗的初步研究
于之清,童 武,郑 浩,李国新,高 飞,王 涛,梁 超,叶 超,武吉强,黄勤峰,童光志
(中国农业科学院上海兽医研究所,上海200241)
本研究使用CRISPR/Cas9基因编辑技术结合同源重组,以伪狂犬病毒经典疫苗株Bartha-K61为骨架,将其gB基因替换为伪狂犬病毒变异株JS-2012的gB基因,经噬斑纯化、PCR筛选鉴定,获得重组病毒rPRV-BJB。体外生物学特性分析结果显示,重组病毒株rPRV-BJB与疫苗株Bartha-K61具有相似的生长特性和噬斑形态,可作为新型重组伪狂犬病病毒疫苗候选株。
变异伪狂犬病毒;CRISPR/Cas9;重组病毒;gB基因
Abstract:In this study, we utilized the speci fi c CRISPR/Cas9 gene editing system combined with homologous recombination to replace the gB of Bartha-K61 strain with that of JS-2012 strain. After several rounds plaque purification and PCR-screening, the acquired recombinant virus was identified by sequencing and designated as rPRV-BJB. rPRV-BJB and Bartha-K61 showed similar biological characteristics in vitro, indicating that rPRV-BJB is a promising PRV vaccine candidate that can be performed in following protective ef fi cacy evaluation.
Key words:PRV variant; CRISPR/Cas9;recombinant virus;gB gene
伪狂犬病毒(Pseudorabies virus,PRV)属于α疱疹病毒亚科(Alphaherpesvirinae),可感染不同年龄段的猪,以种猪繁殖障碍、育肥猪生长缓慢、哺乳仔猪高死亡率为特征。自2011年以来,我国许多Bartha-K61免疫猪场爆发伪狂犬病,造成巨大经济损失。国内多份研究表明,本次伪狂犬病爆发是由伪狂犬病毒变异株引起的,且变异毒株主要保护性抗原基因的核苷酸序列出现了多处变异[1,2]。研制新型伪狂犬病病毒疫苗株,控制变异伪狂犬病的流行成为当务之急。
PRV有11种囊膜糖蛋白,其中糖蛋白B(gB)[3-6]、糖蛋白C(gC)[7,8]和糖蛋白D(gD)[9]能够有效激发体内免疫保护反应,刺激机体产生中和抗体[10,11]。研究表明,gB能够有效激发体内体液免疫和细胞免疫,且能够针对PRV致死性攻击为易感动物提供有效免疫保护[5,12,13]。序列分析发现当前流行的伪狂犬病毒变异株JS-2012的gB基因与经典疫苗株Bartha-K61差异较大。那么,若以Bartha-K61毒株为骨架,将其gB基因替换为PRV变异株的gB基因,所获重组病毒是否能针对变异PRV为易感动物提供有效的免疫保护?
然而,PRV是DNA病毒,基因组庞大且结构复杂,GC含量高达73%,如何对其基因组进行高效精准的操作一直是待解决的难题。近年来,随着人们对CRISPR(clustered regularly interspaced short palindromic repeats,CRISPR)的认识逐渐加深,多种CRISPR/Cas9突变体以及商品化CRISPR/Cas9已成为病毒基因组编辑以及病毒致病机理研究的有力工具[14]。
本研究以经典疫苗株 Bartha-K61为基础,使用CRISPR/Cas9技术结合同源重组,将其gB 基因替换为PRV变异株 JS-2012的gB基因,获得重组病毒rPRV-BJB。rPRV-BJB株可作为新型重组伪狂犬病病毒疫苗候选株,为防控变异伪狂犬病的流行奠定了坚实基础。
1 材料和方法
1.1 细胞、病毒和质粒PK-15细胞由本实验室保存;PRV JS-2012(GenBank登录号:KP257591.1)变异株于2012年由中国农业科学院上海兽医研究所分离自江苏猪场,且该毒株由本实验室保存;PRV Bartha-K61(GenBank登录号:JF797217.1)来自于中国兽医药品监察所的中国兽医微生物菌种保藏中心。pCR®-Blunt II- TOPO®购自Invitrogen;lentiCRISPRv1来自张峰实验室[15,16];大肠杆菌TOPO10感受态细胞购自北京天根生物技术公司。
1.2 主要试剂和仪器FBS、DMEM以及EDTA-胰酶均购自Gibco公司;T4 DNA 连接酶、限制性内切酶购自 NEB 公司;PCR 所用高保真DNA聚合酶PrimeSTAR® HS DNA Polymerase以及LA Taq®with GC Buffer购自宝生物工程(大连)有限公司;AxyPrep DNA 片段回收试剂盒购自AxyGEN公司;质粒提取试剂盒Miniprep购自Qiagen公司;AXYPREP DNA GEL EXTRACTION KIT;酚氯仿、蛋白酶 K、RNase与低熔点琼脂糖购自Sigma公司;转染试剂Lipofactamine 3000购自Invitrogen 公司。
1.3 重组病毒构建设计原理根据Bartha-K61 gB基因序列,设计合成特异靶向Bartha-K61 gB基因的指导 RNA 序列,构建识别并剪切Bartha-K61 gB的CRISPR/Cas9质粒pCas9-Bar-gB1和 pCas9-BargB2。扩增Bartha-K61 gB基因两侧区域DNA片段作为左右重组臂,中间插入PRV变异株JS-2012 gB基因,构建重组转移载体pT-Bar-JSgB。将Bartha-K61基因组、pCas9-Bar-gB1、pCas9-BargB2和pT-Bar-JSgB共转染PK-15细胞,拯救的重组病毒命名为rPRV-BJB。
1.4 CRISPR/Cas9质粒构建根据 PRV Bartha-K61株(GenBank登录号:JF797217.1)的基因组序列,使用 CRISPR/sgRNA工具 Blue Heron Biotechnology. https∶//wwws.blueheronbio.com/external/tools/gRNASrc.jsp进行sgRNA设计,共设计了2条sgRNA,合成其互补引物对(表1),分别靶向Bartha-K61 gB基因的5'端和3'端区域。参照lentiCRISPRv1质粒的构建说明书,利用磷酸化酶(T4 PNK)对互补的gB-gRNA引物gB1-sgRNA-F/gB1-sgRNA-R和gB2-sgRNA-F/gB2-sgRNA-R进行退火磷酸化反应后,高温(95℃ 5 min)对 T4 PNK灭活和引物变性,然后逐步降温复性使引物互补形成双链,获得2条双链gB-gRNA DNA。以BsmBI酶切pCRISPR/Cas9 载体,胶回收大的载体片段。利用T4 DNA连接酶将2条双链gB-gRNA DNA分别连入上述BsmBI 酶切回收的载体中,转化TOP10感受态,提取质粒,以NotI和BamHI 双酶切鉴定,并将酶切阳性克隆进行测序分析。获得克隆质粒pCas9-BargB1和pCas9-Bar-gB2。

表1 靶向Bartha-K61基因组的CRISPR/Cas9 sgRNA序列Table 1 Sequences of sgRNAs target Bartha-K61 genome
1.5 同源重组供体质粒构建根据 PRV Bartha-K61株(GenBank登录号:JF797217.1)基因组序列以及PRV变异株JS-2012(GenBank登录号:KP257591.1)的基因组序列,用 Olio6.0 软件设计了3对引物dpLF/dpLR、dpRF/dpRR和dpgBF/dpgBR(见表2)。利用dpLF/dpLR与dpRF/dpRR 2对引物,以 PRV Bartha-K61 病毒基因组DNA为模板进行PCR扩增,获得Bartha-K61 基因上游 UL46 部分区域为同源左臂(Bar-L-arm)和gB基因下游UL28 部分区域为同源右臂(Bar-R-arm)的片段。利用引物 dpgBF/ dpgBR,以PRV JS-2012病毒基因组DNA为模板进行 PCR扩增JS-2012 gB基因片段JS-gB-DNA。以dpLF/dpgBR为引物、以回收得到的Bar-L-arm与gB-DNA 片段为模板进行Overlap PCR扩增,获得片段Bar-L-arm-JS-gB。以dpgBF/dpRR为引物、以回收得到的Bar-R-arm 与gB-DNA为模板进行OverLap PCR扩增,获得片段JS-gB-Bar-R-arm。将胶回收的Bar-L-arm-JS-gB和JS-gB-Bar-R-arm分别连入pCR®-Blunt II- TOPO®载体。转化感受态大肠杆菌TOP10,获得阳性质粒pBar-L-JSgB和pJSgB-Bar-R。将上述阳性质粒分别以NheI和BamHI进行双酶切反应。胶回收产物使用T4连接酶连接。4℃连接过夜后,转化感受态大肠杆菌TOP10,获得阳性供体质粒,命名为pT-Bar-JSgB。
1.6 重组病毒的拯救以1 MOI的剂量分别接种PRV Bartha-K61株于 PK-15细胞上,待细胞出现约70%病变时,弃去培养基,刮取细胞,使用酚氯仿法提取总基因组DNA。使用Lipofectamine® 3000 Transfection Reagent(Thermo Fisher)转染试剂,将上述1 μg 供体质粒pT-Bar-JSgB、1 μg pCas9-gB1、1 μg pCas9- gB2和2 μg 病毒基因组共转染PK-15细胞,转染后每天观察病变情况。当细胞出现大约80 %病变时,收取上清,-80℃保存备用。
1.7 重组病毒的筛选纯化将1.6收取的上清分别接种6 孔细胞培养板中的PK-15细胞,1 h后,弃去上清,覆盖含有2% FBS和1%低熔点琼脂糖的MEM,室温凝固后,置37℃、5% CO2培养箱中培养。60 h后,挑取单个噬斑,吹打入500 μL DMEM,放入-80℃储存备用。取上述50 μL病毒空斑溶液接种PK-15细胞,待细胞全部病变后,收取上清,提取病毒基因组。使用引物 screening-F(5'-CTCGA GGGACGCCTCCGCGGAGACGT-3')/screening-R(5'-CATCGACGGGCTGCTCGTGGGCGGCT GC-3')进行PCR鉴定,PCR阳性病毒液进行下一轮噬斑纯化。经过3轮噬斑纯化结合PCR筛选的阳性毒株进行测序分析。

表2 重组病毒供体质粒构建所需引物Table 2 The sequence of primers that utilized to construct donor plasmids
1.8 病毒生长曲线的绘制待T25细胞培养瓶中的PK-15细胞长至单层,将筛选到的重组病毒rPRVBJB株和疫苗 Bartha-K61株分别以1 MOI 感染剂量感染PK-15细胞。病毒感染细胞1 h后,弃去病毒液,并用PBS缓冲液清洗2遍后,每个T25细胞培养瓶中加入5 mL 含有2%FBS的DMEM培养基。在感染后每隔4 h收取300 μL细胞上清液进行病毒滴度测定,同时补加300 μL 2% FBS 的DMEM 培养基,直至感染后36 h,细胞呈现完全病变且大范围脱落。根据不同时间点病毒滴度(TCID50/mL)绘制病毒一步生长曲线。
1.9 空斑形成试验待Vero细胞于6孔板中长至单层,将重组病毒株rPRV-BJB和疫苗株Bartha-K61株 10倍梯度稀释后接种6孔板中的单层Vero细胞,每孔接500 μL,在37℃、5% CO2培养箱孵育1 h加入覆盖层,覆盖层为2 % FBS、1%(w/v)低熔点琼脂糖的MEM。37℃培养 60 h 后,小心去除6孔板中的半固体覆盖层,用5% (W/V)结晶紫染色。
2 结果
2.1 CRISPR/Cas9的构建将合成的sgRNA片段连接入pLentiCRISPRv1中,构建插入sgRNA的CRISPR/Cas9质粒pCas9-Bar-gB1和pCas9-Bar-gB2(图1)。结果显示,pCas9-Bar-gB1中含有sgRNA序列CGTGCGTGTCATCGTGGTCG,pCas9-Bar-gB2中含有sgRNA序列CAGCTCGCGGATATATCGCC。
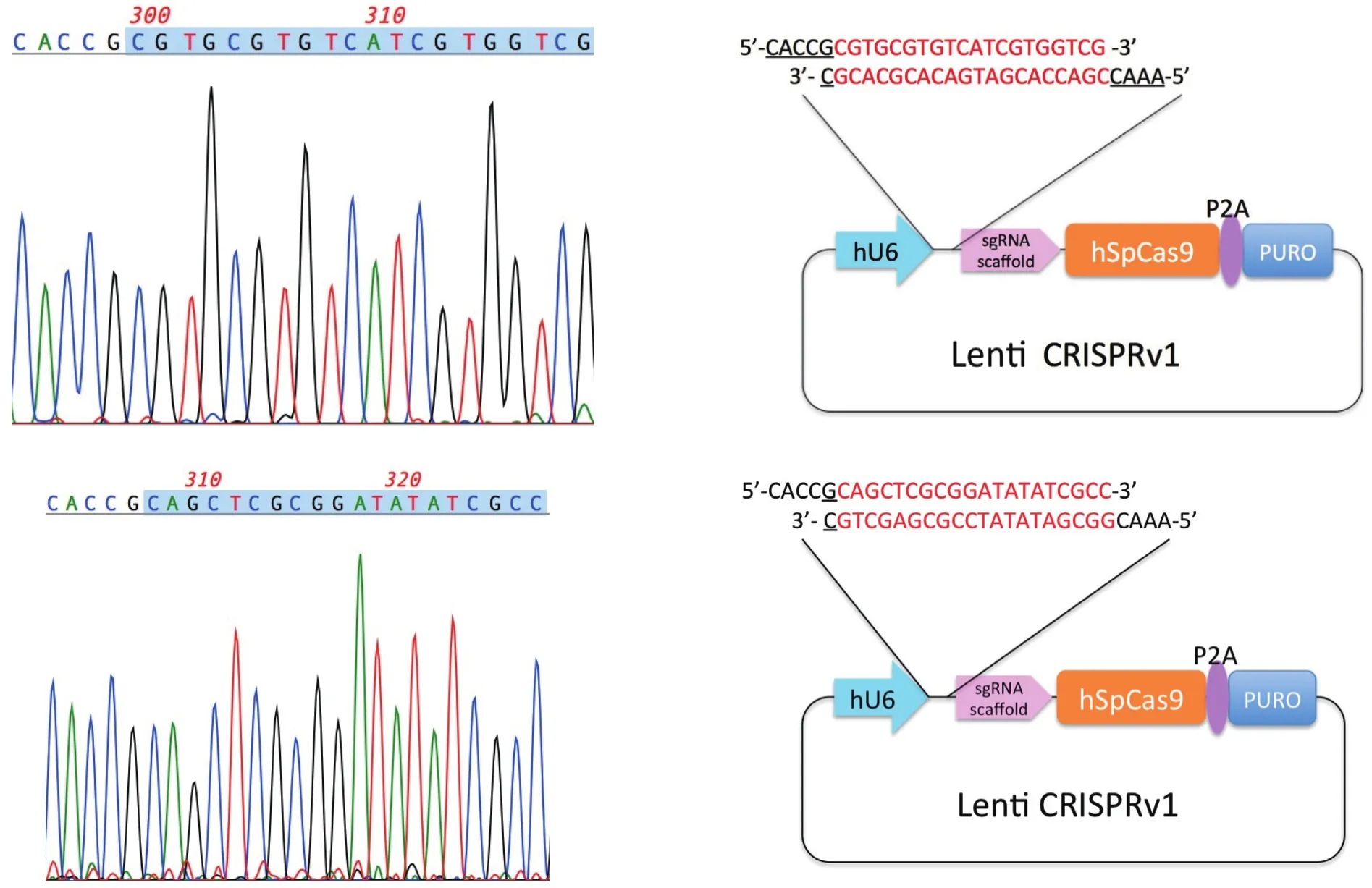
图1 pCas9-Bar-gB1和pCas9-Bar-gB2测序鉴定Fig. 1 Identi fi cation of pCas9-Bar-gB1 and pCas9-Bar-gB2 by sequencing
2.2 供体质粒的构建PCR扩增获得同源重组左臂(Bar-L-arm)、同源重组右臂(Bar-R-arm)及gB基因片段(JS-gB-DNA)(图 2A),经OverLap PCR获得拼接DNA片段Bar-L-arm-JS-gB和JS-gBBar-R-arm(图2B)。将PCR产物连接 pCR®-Blunt II- TOPO® 载体经测序鉴定,获得质粒pBar-L-JSgB和pJSgB-Bar-R。两质粒进行片段拼接后,经NheI和EcoRI双酶切鉴定,酶切产物经琼脂糖凝胶电泳鉴定后如图2C所示,阳性供体质粒称之为pT-Bar-JSgB(pTJSB)。
2.3 重组病毒的筛选纯化将疫苗株Bartha-K61基因组、供体质粒pT-Bar-JSgB、sgRNA质粒pCas9-Bar-gB1和pCas9-Bar-gB2共转染PK-15细胞,收获病变细胞上清进行噬斑纯化,单噬斑病毒再以PCR方法筛选阳性病毒克隆。如图3所示,重组阳性病毒经PCR扩出一条1200 bp的目的条带,而疫苗株Bartha-K61基因组不能扩增出条带(图3A)。将获得的阳性克隆病毒再进行噬斑纯化结合PCR鉴定,经过3轮噬斑纯化,PCR鉴定结果显示,所有鉴定的克隆均扩增出目的条带,均为阳性克隆(图3B)。对PCR阳性毒株的基因组进行序列分析,结果显示,gB核苷酸差异位点为特异性单峰且与变异株JS-2012的序列一致(图4),表明成功筛选到重组病毒rPRV-BJB。
2.4 重组病毒的生物学特性通过绘制一步生长曲线和噬斑形态来比较分析重组病毒rPRV-BJB和疫苗株Bartha-K61的体外生物学特性。一步生长曲线中,Bartha-K61 与 rPRV-BJB 相比,两者的病毒复制速率以及最终效价无显著性统计学意义,见图5。rPRV-BJB和Bartha-K61均在感染后复制速率较快,在感染后20 h 进入复制平台期,最终效价均可达到108TCID50/mL。另外,从病毒噬斑形态和大小上来分析,重组病毒株rPRV-BJB与疫苗株Bartha-K61的空斑形态较为相似,病毒噬斑均呈散落圆点状,边缘清晰,且两者以相同剂量接种 Vero 细胞后在相同面积下形成的单个噬斑分布均匀,噬斑数量也较为相近,如图6所示。这表明重组病毒株rPRV-BJB与疫苗株Bartha-K61具有较为相似的体外生物学特性,gB替换没有显著影响病毒的生物学特性。

图2 供体质粒的构建Fig. 2 Construction of donor plasmid
3 讨论
自20世纪70年代Bartha-K61疫苗株引入我国以来,广泛应用该疫苗使 PR 得到了有效控制[17,18],但2011年末,猪伪狂犬病在我国大规模爆发。研究表明,当前流行的PRV变异毒株发生了抗原变异,且经典疫苗株Bartha-K61不能针对变异伪狂犬病毒为猪群提供完全免疫保护[1,19-21]。本研究提供了疫苗株一种新型重组伪狂犬病病毒疫苗的构建策略,以Bartha-K61为骨架,使用CRISPR/Cas9基因编辑技术结合同源重组,将其gB基因全部替换为JS-2012的gB 基因,获得重组PRV疫苗候选株rPRV-BJB,可用于疫苗免疫保护效力评价,利于我国PRV变异毒株的流行防控。
高效的基因编辑技术以及精准简便的基因操作策略是构建 PRV 重组病毒的必要手段。CRISPR/Cas9 基因编辑技术在病毒基因编辑中应用日渐广泛,如对病毒基因组进行缺失构建缺失疫苗等。已有研究使用 CRISPR/Cas9 技术对 PRV 基因组进行基因缺失[22],但仍未有研究报道使用该技术直接对病毒基因进行大片段替换编辑,特别是对病毒的必需基因直接进行基因编辑。在此,我们使用 CRISPR/Cas9 结合同源重组技术直接对 PRV gB 基因进行大片段替换,并提供了一种简单高效的重组病毒筛选方法,扩宽了该技术在大病毒基因组的应用可能性,从而推动病毒的基础研究。

图3 重组病毒株rPRV-BJB的 PCR 初步筛选Fig. 3 The PCR-based method for screening rPRV-BJB

图4 纯化的重组病毒株rPRV-BJB gB基因区域序列分析(部分区域)Fig. 4 Sequence analysis of the puri fi ed recombinant viruses in gB gene region
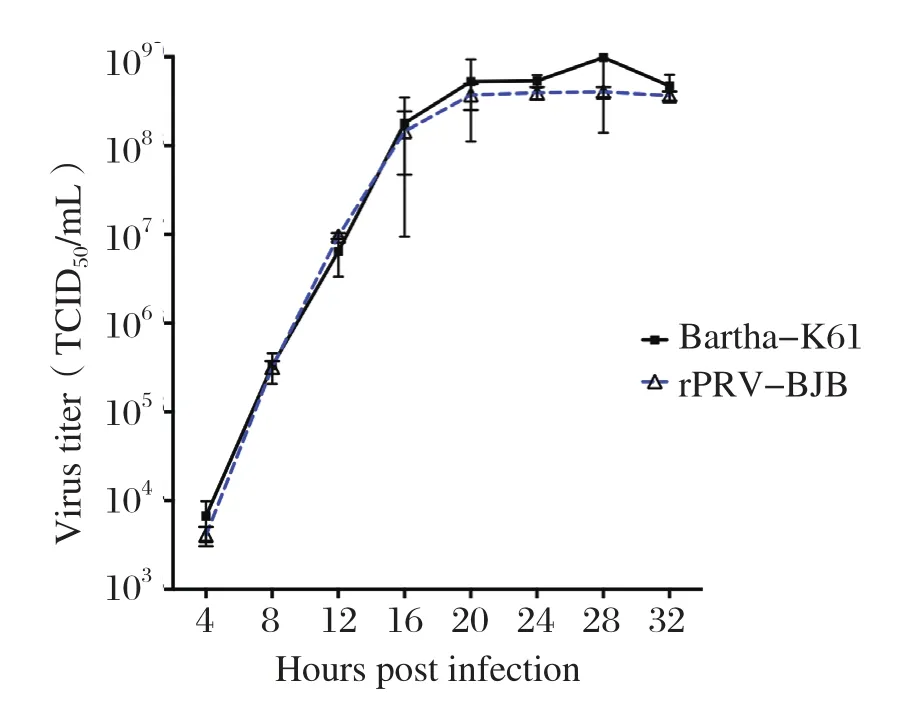
图5 重组病毒rPRV-BJB与疫苗株Bartha-K61的一步生长曲线Fig. 5 One-step growth curves of rPRV-BJB and Bartha-K61

图6 重组病毒rPRV-BJB与疫苗株Bartha-K61的噬斑形态比较Fig. 6 The morphology of plaque-form between the recombinant virus and parental virus
[1] An T Q, Peng J M, Tian Z J, et al. Pseudorabies virus variant in Bartha-K61-vaccinated pigs, China, 2012[J].Emerg Infect Dis, 2013, 19(11)∶ 1749-1755.
[2] Ye C, Zhang Q Z, Tian Z J, et al. Genomic characterization of emergent pseudorabies virus in China reveals marked sequence divergence∶ Evidence for the existence of two major genotypes[J]. Virology, 2015, 483∶ 32-43.
[3] Nakamura T, Ihara T, Nunoya T, et al. Role of pseudorabies virus glycoprotein II in protection from lethal infection[J]. Vet Microbiol, 1993(1-2), 36∶ 83-90.
[4] Robbins A K, Dorney D J, Wathen M W,et al. The pseudorabies virusgIIgene is closely related to the gB glycoprotein gene of herpes simplex virus[J]. J Virol,1987, 61(9)∶ 2691-2701.
[5] van Rooij E M, Haagmans B L, de Visser Y E, et al.Effect of vaccination route and composition of DNA vaccine on the induction of protective immunity against pseudorabies infection in pigs[J]. Vet Immunol Immunopathol, 1998, 66(2)∶ 113-126.
[6] Xuan X, Nakamura T, Ihara T,et al.Characterization of pseudorabies virus glycoprotein gII expressed by recombinant baculovirus[J]. Virus Res, 1995, 36(2-3)∶151-161.
[7] Ober B T, Summerfield A, Mattlinger C,et al. Vaccineinduced, pseudorabies virus-specific, extrathymic CD4+CD8+ memory T-helper cells in swine[J]. J Virol,1998, 72(6)∶ 4866-4873.
[8] Ober B T, Teufel B, Wiesmuller K H, et al. The porcine humoral immune response against pseudorabies virus specifically targets attachment sites on glycoprotein gC[J]. J Virol, 2000, 74(4)∶ 1752-1760.
[9] Mukamoto M, Watanabe I, Kobayashi Y,et al.Immunogenicity in Aujeszky's disease virus structural glycoprotein gVI (gp50) in swine[J]. Vet Microbiol, 1991,29(2)∶ 109-121.
[10] Gerdts V, Jöns A, and Mettenleiter T C Potency of an experimental DNA vaccine against Aujeszky's disease in pigs[J]. Vet Microbiol, 1999, 66(1)∶ 1-13.
[11] Hong W, Xiao S, Zhou R, et al. Protection induced by intramuscular immunization with DNA vaccines of pseudorabies in mice, rabbits and piglets[J]. Vaccine,2002, 20(7-8)∶ 1205-1214.
[12] van Rooij E M, Moonen-Leusen H W, de Visser Y E,etal. A DNA vaccine coding for gB and gD of pseudorabies virus (suid herpes type 1) primes the immune system in the presence of maternal immunity more efficiently than conventional vaccines[J]. Vaccine, 2006, 24(9)∶ 1264-1273.
[13] Yoon H A, Aleyas A G, George J A, et al. Differential segregation of protective immunity by encoded antigen in DNA vaccine against pseudorabies virus[J]. Immunol Cell Biol, 2006, 84(6)∶ 502-511.
[14] Dampier W, Nonnemacher M R, Sullivan N T,et al. HIV Excision Utilizing CRISPR/Cas9 Technology∶ Attacking the Proviral Quasispecies in Reservoirs to Achieve a Cure[J]. MOJ immunology, 2014, 1(4). pii∶ 00022.
[15] Sanjana N E, Shalem O and Zhang F. Improved vectors and genome-wide libraries for CRISPR screening[J]. Nat Methods, 2014, 11(8)∶ 783-784.
[16] Shalem O, Sanjana N E, Hartenian E,et al. Genome-scale CRISPR-Cas9 knockout screening in human cells[J].Science, 2014, 343(6166)∶ 84-87.
[17] Lomniczi B, Blankenship M L and Benporat T. Deletions in the genomes of pseudorabies virus vaccine strains and existence of four isomers of the genomes[J]. J Virol,1984, 49(3)∶ 970-979.
[18] Petrovskis E A, Timmins J G, Gierman T M, et al.Deletions in vaccine strains of pseudorabies virus and their effect on synthesis of glycoprotein gp63[J]. J Virol,1986, 60(3)∶1166-1169.
[19] Gu Z, Dong J, Wang J,et al. A novel inactivated gE/gI deleted pseudorabies virus (PRV) vaccine completely protects pigs from an emerged variant PRV challenge[J].Virus Res, 2015, 195∶ 57-63.
[20] Hu R M, Zhou Q, Song W B,et al.Novel pseudorabies virus variant with defects in TK, gE and gI protects growing pigs against lethal challenge[J]. Vaccine, 2015,33(43)∶ 5733-5740.
[21] Luo Y, Li N, Cong X,et al. Pathogenicity and genomic characterization of a pseudorabies virus variant isolated from Bartha-K61-vaccinated swine population in China[J]. Vet Microbiol, 2014, 174(1-2)∶ 107-115.
[22] Liang X, Sun L, Yu T, et al. A CRISPR/Cas9 and Cre/Lox system-based express vaccine development strategy against re-emerging Pseudorabies virus[J]. Scientific reports, 2016, 6∶ 19176.
PRELIMINARY STUDY ON UTILIZING CRISPR/CAS9 TO CONSTRUCT NEW RECOMBINANT PSEUDORABIES VARIANT VACCINE
YU Zhi-qing, TONG Wu, ZHENG Hao, LI Guo-xin, GAO Fei, WANG Tao, LIANG Chao, YE Chao, WU Ji-qiang, HUANG Qin-feng, TONG Guang-zhi
(Shanghai Veterinary Research Institute, CAAS, Shanghai 200241, China)
S852.659.1
A
1674-6422(2017)04-0006-07
2017-04-14
国家重点研发计划项目(2016YFD0500100)
于之清,女,硕士研究生,预防兽医学专业
童光志,E-mail:gztong@shvri.ac.cn
