猴头菇DNA条形码的构建及验证
2017-10-16皇甫永冠闫宝松张跃新宗宪春
皇甫永冠,闫宝松,张跃新,宗宪春
(1.东北林业大学盐碱地研究中心,哈尔滨 150040;2.黑龙江省林副特产研究所,黑龙江省非木质林产品研发重点实验室,黑龙江 牡丹江 157011;3 牡丹江师范学院,黑龙江 牡丹江 157000)
猴头菇DNA条形码的构建及验证
皇甫永冠1,闫宝松2,张跃新2,宗宪春3
(1.东北林业大学盐碱地研究中心,哈尔滨 150040;2.黑龙江省林副特产研究所,黑龙江省非木质林产品研发重点实验室,黑龙江 牡丹江 157011;3 牡丹江师范学院,黑龙江 牡丹江 157000)
为构建猴头菇品种专属的DNA条形码。应用分子生物学方法、选定β-tubulin基因作为条形码序列的目标基因,依据NCBI中猴头菇β-tubulin基因序列的全长,设计了一对用于扩增目标基因的引物:Betu-F1:ATGCGTGAAATCGTCCACC;Betu-R2:GAAGACGGGGGAAAGGAAC。应用上述引物对现有的19个猴头菇的品种进行PCR鉴定。测序后的序列结合BLAST、Clustal、MEGA、BioEdit等软件分析、比对得到多态性位点,分析多态性位点设计出构建条形码序列的特异性引物。经验证表明,猴头菇RT9品种的3个特有的条形码成功获得。试验结果表明,实验成功获得的DNA条形码序列可以将猴头菇RT9品种与猴头菇的其它品种区分鉴定。
猴头菇;DNA条形码;内含子多态性
Abstract:The aim was to construct the DNA barcode of Hericium erinaceus. Using molecular biology method,Selected β-tubulin gene as the target gene of bar code sequence,A pair of primers for amplification of the target gene was designed according to the full-length of the β-tubulin gene sequence of NCBI:Betu-F1:ATGCGTGAAATCGTCCACC;Betu-R2:GAAGACGGGGGAAAGGAAC.The above primers were used to identify the 19 species of Hericium erinaceus of PCR.Sequencing of the sequence with BLAST, Clustal, MEGA, BioEdit and other software analysis, comparison of polymorphic loci,Analysis of polymorphic loci was designed to construct a specific primer sequence. The results showed that the three unique bar codes of Hericium erinaceus RT9 were successfully obtained.The experiment result shows:the DNA barcode sequence obtained from the experiment can distinguish the RT9 and other varieties of Hericium erinaceus
Keywords:Hericiumerinaceus; DNA barcoding; Intron polymorphis
0 绪论
猴头菇因营养丰富、味道鲜美及具抗癌等功效,近些年来成为相关行业的研究热点,但对它的研究多集中在深加工等方面。中国现有的猴头菇有几十种到数百种,它们的营养成分及功效各不相同,而且名称混乱,给引种、栽培和开发利用工作带来较大的麻烦[1]。猴头菇菌种的区分鉴定对于今后的育种工作及种质资源的保护意义重大,但由于现在多数只是从形态学的角度来鉴定菌种,其准确性不是十分的可靠,研究表明,将分子标记应用在菌种鉴定上将会大幅度的增加鉴定效率。根据前人对物种的鉴定研究来看,DNA条形码技术在区分物种时具简单、快速、准确性高等优点,因此本实验结合内含子多态性,力争构建出猴头菇的专属DNA条形码,为今后食用菌行业的种质资源鉴定提供行之有效的手段。自Hebert等[2-3]提议使用线粒体基因细胞色素C氧化酶(COI)基因片段作为动物的DNA条形码后,DNA条形码技术得以迅速发展,在动物、植物、微生物的鉴定及分类上开始广泛的应用[4]。DNA barcoding技术在动物中的研究进展:COI基因通常作为动物的条形码[5-6]目前主要运用于鸟类[7-16]、鱼类[17-21]、昆虫[22-24]的分类鉴定。DNA barcoding在植物中研究进展:2009年国际生命条形码联盟植物工作组(Consortiumfor the Barcode of Life Plant Working Group)初步确定,并推荐使用叶绿体基因片段的rbcL和matK作为植物的DNA条形码基因[25]。DNA barcoding技术在微生物中的研究进展:目前主要在酿酒酵母(Saccharomyces cerevisiae)突变菌株的识别[26],毛霉属(Mucor)[27]、青霉属(Penicillium)[28]、口蘑属(Tricholoma)[29]等物种鉴别中有应用。相比于动物、植物,菌物DNA barcoding技术的研究起步相对较晚,研究的也较少,到目前为止菌物的DNA条形码的标准序列还没有选定。依据Zhao等[30]、Samson et al.[31],分别对丛赤壳类真菌、青霉亚属的代表性菌株进行的序列分析表明,β-tubulin基因在真菌鉴定中是一种理想的DNA barcoding标记基因,无论是在PCR鉴定还是测序上都表现出很高的成功率,而且不存在种间和种内的交叠,鉴定效果优于ITS。因此,本实验选定β-tubulin 基因作为构建猴头菇品种的条形码的目标基因,构建猴头菇品种的专属条线码。本研究针对19个猴头菇的品种构建猴头菇品种的DNA条形码,经验证成功构建的条形码序列在所有猴头菇品种中均适用,因此,DNA条形码技术在菌物鉴定中应用切实可行,为今后菌物菌种鉴定提供了技术手段。
1 材料与方法
1.1 实验材料
供试菌株见表1。

表1 供试菌种
续表1

序号名称采集地9野猴3黑龙江省牡丹江市三道关林区野生10RT9黑龙江省农业经济学院11RT10黑龙江省农业经济学院12RT11黑龙江省农业经济学院13RT12黑龙江省农业经济学院14RT13黑龙江省农业经济学院15RT14黑龙江省农业经济学院16RT16黑龙江省农业经济学院17RT17黑龙江省农业经济学院18RT18黑龙江省农业经济学院19RT19黑龙江省农业经济学院
1.2 实验方法
1.2.1 DNA条形码的构建
(1)选取材料:19个猴头菇品种(见表1)。
(2)获得及培养19个猴头菇品种的菌丝体。
(3)DNA的提取:本实验采用CTAB法对供试菌株的DNA进行提取。
(4)设计引物及扩增基因
设计引物
依据NCBI中猴头菇β-tubulin基因的全长设计了一对引物如下:Betu-F:ATGCGTGAAATCGTCCACC;Betu-R:GAAGACGGGGGAAAGGAAC。
扩增目的基因
PCR的体系:10×buffer 缓冲液 2 μL,d NTP(2.5mM each) 1.6 μL,上游引物(10μM) 1 μL,下游引物(10μM) 1 μL,r-Taq DNA 聚合酶(5U/ μL) 0.3 μL,基因组 DNA 1 μL,用灭过菌的去离子水补足至20μL。
PCR反应程序:94℃预变性5min;94℃变性30s,55℃退火30s , 72℃延伸1min20s,35个循环,72℃再延伸 20 min。
(5)T-A克隆
目的基因的回收及检测
利用琼脂糖凝胶DNA回收试剂盒对目标条带进行切胶回收,1%的凝胶浓度对回收的目的基因进行检测。
连接反应
体系10μL:2×Rapid ligation Buffer 5μL,pGEM-T Easy vector 1μL,PCR product,1μL,T4 DNA Ligase,1μL,ddH2O,2μL,16℃过夜连接不超过16h。
转化
热激转化:16℃连接过夜后,将10μL连接产物加入到感受态细胞中(DH5a)中,冰浴30min,42℃热激90s,在冰浴2min,向其中加入500μL B液体培养基,37℃180转震荡培养40~60min,取震荡培养后的菌液200μL涂布在加有AMP+IPTG+X-Gal的固体LB培养基的平板上(蓝白斑筛选),为防止因立即倒置培养,造成菌液流动现象,将涂布后的菌版在37℃正向培养1h,之后倒置过夜培养,筛选阳性单菌落。
筛选重组子
挑取蓝白斑筛选平板上白色的单菌落,至5mL含AMP 的LB液体培养基中,将震荡培养箱的温度调制37℃、转速调至180r/min,培养约6h左右至菌液浑浊,进行菌液PCR检测。
体系20μL:10×buffer 缓冲液 2 μL,d NTP(2.5mM each) 1.6 μL,上游引物(10μM) 1 μL,下游引物(10μM) 1 μL,r-Taq DNA 聚合酶(5U/ μL) 0.3 μL,菌液 1 μL,用灭过菌的去离子水定容至20μL。
PCR反应程序:94℃预变性5min;94℃变性 30s,60℃退火 30s,72℃延伸1min20s 35个循环,72℃再延伸 5 min。
PCR产物采用1%的琼脂糖凝胶电泳对其进行检测,阳性菌液保存备用。
重组子测序
取出保存的阳性菌液送至生物公司(promega)进行测序。
测序结果分析
测序成功的序列结合primmer5.0对其进行整理,用BLAST软件对单一序列进行比对,得到有效的序列,利用Clustal软件对有效的序列进行多重比对,找出每个品种的代表性序列,再将代表性序列应用MEGA 5.1的邻接法(Neighbor-Joining)构建系统发育树进、应用BioEdit软件做出更直观的多态性位点图,分析整体多态性位点的分布情况,再将系统发育进化树的每个大的分支的序列,应用BioEdit及Clustal软件进行多重比对,分析每个大分支的多态性位点,进而找出成功设计DNA条形码的多态性位点。
(6)确定特异引物并合成
分析多态性位点,结合primer软件设计每个特异品种的特有引物(如果后续的条形码验证成功,此引物则为条形码序列的特异引物)。将构建的特异引物送至生物公司(上海生工)合成。
1.2.2 DNA条形码的验证
(1)现有菌种中的验证:应用上述合成的特异性引物,分别对猴头菇19个品种的基因组DNA进行PCR验证,具体步骤如下:
①从样品组织中提取基因组DNA;
②用步骤1所提取的基因组DNA为模板,用上述合成的特异性引物,进行聚合酶链式反应;
③对步骤2扩增得到的PCR产物进行电泳检测,其特征是扩增片段长度,应与合成的特异性引物所扩增出目标条带大小一致。
(2)所有猴头菇品种中的适用性验证:把上述已构建成功的DNA条形码序列与GenBank数据库中已发表的含有猴头菇β-tubulin基因的序列(序列登录号:>gi|386872545|>gi|63333592|>gi|84626253|>gi|84626251| >gi|84626249| >gi|84626247|>gi|84626245|>gi|84626243| >gi|84626241|>gi|84626239|>gi|84626237| >gi|84626235| >gi|84626233| >gi|84626231|>gi|84626229| >gi|84626227|>gi|84626225| >gi|84626223| >gi|84626221|>gi|84626219|>gi|84626217|>gi|84626215|>gi|84626213|>gi|84626212| >gi|84626210| >gi|84626208|>gi|84626206|>gi|84626204| >gi|84626202|>gi|84626200| >gi|84626198| >gi|84626196| >gi|84626194| >gi|84626192|>gi|84626190|>gi|84626188| >gi|84626186| >gi|84626184|>gi|84626182| )进行比对如果构建成功的条形码序列与已发表的序列间存在差异,说明该条形码序列可以很好地将此品种与其他的猴头菇品种区分开。
2 结果与分析
2.1 DNA提取
提取的DNA采用1%的琼脂糖凝胶电泳进行检测,结果显示所有样本DNA均成功提取,见图1。

图1 DNA提取检测
2.2 目的基因切胶回收
依据NCBI中猴头菇β-tubulin基因的全长设计的一对引物Betu-F1:ATGCGTGAAATCGTCCACC;Betu-R2:GAAGACGGGGGAAAGGAAC,用其对目的基因进行扩增,PCR产物采用1%的琼脂糖凝胶电泳检测。所有样本均成功扩增,条带大小约1.3kb,切胶回收电泳结果见图2。
图2 目的基因切胶回收
2.3 阳性鉴定
在蓝白斑筛选的平板上挑取白色菌落,放入LB液体培养基内摇菌,做菌液PCR,PCR产物用1%琼脂糖凝胶电泳检测筛选出阳性菌。因为,所需要的阳性菌并非一次性检出,所以实验过程中将每次检测出的阳性菌液均进行保存,待实验结束后将所有检测出的阳性菌的菌液PCR产物集中电泳(见图3)。
图3 菌液阳性鉴定
2.4 系统发育进化树
应用MEGA软件对代表性序列进行分析得出系统发育进化树图(见图4),从图片上可直观看出猴头菇RT9品种是一条独立的分支,说明RT9品种与其它猴头菇品种间亲缘关系较远,构建条形码的成功率会增加。
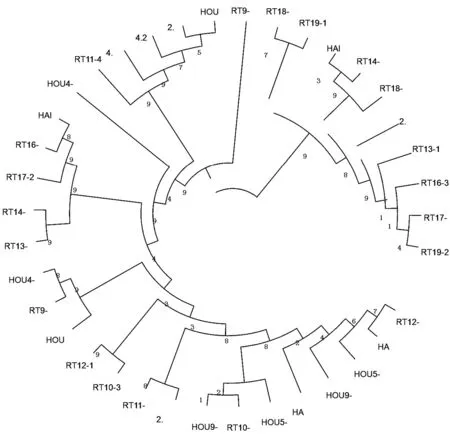
图4 系统发育进化树
注:图1中2.3、2.4、2.5为猴99的代表性序列;4.1、4.2为野猴3的代表性序列;RT16-2、RT16-3为RT16的代表性序列;HAI1、HAI3为海猴的代表性序列;RT17-1、RT17-2为RT17的代表性序列;RT18-2、RT18-3为RT18的代表性序列;RT19-1、RT19-2为RT19的代表性序列;RT10-3、RT10-4为RT10的代表性序列;RT13-1、RT13-2为RT13的代表性序列;RT12-1、RT12-2为RT12的代表性序列;RT9-1、RT9-3为RT9的代表性序列;RT11-4、RT11-5为RT11的代表性序列;RT14-3、RT14-4为RT14的代表性序列;HOU4-1、HOU4-2为猴4的代表性序列;HA2、HA5为HA的代表性序列;HOU2、HOU3为猴的代表性序列;HOU5-1、HOU5-2为猴5的代表性序列;HOU9-2、HOU9-3为猴9的代表性序列。
2.5 多态性位点分析
将测序后的所有序列应用BLAST、Clustal软件筛查分析,得到每个品种具有代表性的序列,结合MEGA软件构建系统发育进化树(见图4)。但是由于序列过多,导致分析多态性位点、设计特异性引物难度增加,为此,将系统发育进化树的每个大的分支的序列,应用Clustal软件分别进行多重比对,每个分支的多重序列比对结果见附录1,由于比对结果占篇幅庞大,附录1中仅列出构建成功的条形码序列所在的分支的多重比对结果,根据多重比对的结果分析多态性位点,在此基础上找出成功设计DNA条形码的多态性位点(附录1已标明),设计出每个品种特有的专属条形码引物。
2.6 条形码验证
分析多态性位点设计出了几十对用于构建条形码的引物,经逐一验证发现,猴头菇RT9的3个条形码成功构建(见图5-a;5-b),应用这三条条形码序列可以将RT9与猴头菇的其他18个品种区分开。对应的3个引物为CRT9-3F1:CGGTACGAGGATCGCGCAAAC;CRT9-3R1GCGACCGTCTT
CCCACA;(扩增序列长度为353bp)、CRT9-3F1:CGGTACGAGGATCGCGCAAAC;CRT9-3R2:GTGTCAAGTCGCAAAAAT(扩增序列长度为483bp)、RT9-2F:TCCTCACACACACTGGTAC;RT9-2R:GTACGGATGTTGCAGTAG(扩增序列长度为723bp),应用这3对引物扩增出的条形码序列。

图5-a DNA条形码验证
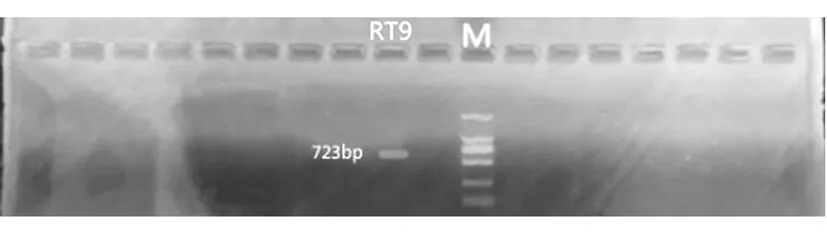
图5-b DNA条形码验证
2.7 3个条形码在整个猴头菇品种中的适用性检测
把上述已构建成功的3个猴头菇RT9的DNA条形码序列与GenBank数据库中已发表的含有猴头菇β-tubulin基因的序列(序列登陆号见1.2.2)进行比对,发现这3个条形码序列与已发表的序列间存在差异,因此应用这3个序列可以很好地将RT9与其它的猴头菇品种区分开,所以这3个序列可以作为鉴定猴头菇RT9的专属条形码。
3 讨论
3.1 构建条形码目标基因的选择
动物中通常作为条形码片段的基因为COI基因[5-6],也是目前研究最为完善的。最初作为植物条形码片段的基因为叶绿体基因片段的rbcL和matK[25],后来研究人员对高等植物、真菌、藻类的大量ITS2序列进行研究分析,结果显示,ITS2在种级分辨上表现出很高的鉴定成功率。所以,建议以ITS2可以作为新的DNA条形码,在鉴定绿色植物及部分真菌时使用[32-33]。
相比于动物、植物,菌物DNA条形码的研究及应用起步较晚,目前为止也没有明确作为菌物条形码片段的基因,前人研究发现,β-tubulin基因在丛赤壳类真菌的研究中表现出高的 PCR 和测序成功率,且没有种间及种内的交叠,在一些菌种鉴定过程中,β-tubulin基因表现出了ITS2还要高的鉴定成功率[30-31],因此,本实验选择了β-tubulin基因作为条形码片段的目标基因,并依据NCBI中猴头菇β-tubulin基因的全长设计了一对引物用于扩增目的基因。
3.2 假阳性分析
依据多态性位点合成的几十对用于扩增特异性条形码的引物,在条形码验证时仅有RT9的3个条形码成功获得,可能导致此结果的原因如下:①在测序时相关的碱基错读或缺失,②菌液PCR过程中可能发生碱基错配等原因。
3.3 实验体系优化
重组子鉴定过程中,开始阳性检出率非常低,存在非特异性扩增现象,经分析可能是如下原因:①可能实验体系存在问题,如,Taq酶、T载体等的使用是否合适,②可能由于PCR的退火温度过低,这样虽然可以增加扩增的产量,但引物和模板之间的错配现象也会相应增加,就会导致非特异性扩增。改进措施如下:①将实验原来用的生工的T载体更换为大连宝生物的pGEM-T载体。②将最初使用的Taq酶(上海生工)更换为r-Taq酶(大连宝生物)。③通过提高PCR的退火温度,由原来的55℃增至60℃。实验体系经上述优化后,阳性检出率低、非特异性扩增的现象均有所改进。
4 结论
本实验应用分子生物学的研究手段,成功构建出猴头菇RT9品种的3个条形码。经验证表明,这3个特异性条形码在所有猴头菇品种中均适用。因此,DNA条形码技术在菌物的鉴定中应用切实可行。
[1]Bunyard B A, Nicholson M S, Royse D J, et al. A systematic assessment of Morchella using RFLP analysis of the 28S ribosomal RNA gene[J]. Mycologia, 1994, 86(6):762-772.
[2]Hebert P D, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes.[J]. Proceedings of The Royal Society B: Biological Sciences, 2003, 270(1512): 313-321.
[3]Hebert P D, Ratnasingham S, De Waard J R, et al. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of The Royal Society B: Biological Sciences, 2003,270:96-99.
[4]裴男才,陈步峰.生物DNA条形码:十年发展历程、研究尺度和功能[J]. 生物多样性,2013,2l(5):616-627.
[5]Waugh J. DNA barcoding in animal species: progress, potential and pitfalls.[J]. BioEssays, 2007, 29(2):188-197.
[6]Kwong S, Srivathsan A, Meier R, et al. An update on DNA barcoding: low species coverage and numerous unidentified sequences[J]. Cladistics, 2012, 28(6):639-644.
[7]Hebert P D, Stoeckle M Y, Zemlak T S, et al. Identification of Birds through DNA Barcodes[J]. PLOS Biology, 2004, 2(10):1657-1663.
[8]蔡延森, 张修月, 岳碧松,等. 我国8种猛禽的DNA条形码技术研究[J]. 四川动物, 2009, 28(3):334-340.
[9]Lijtmaer D A, Kerr K C, Barreira A S, et al. DNA Barcode Libraries Provide Insight into Continental Patterns of Avian Diversification[J]. PLOS ONE, 2011, 6(7).doi:10.1371/journal.pone.0020744.
[10]马明义, 闫颖, 王译伟,等. 我国32种鸟类DNA条形码分析[J]. 四川动物, 2012, 31(5):729-733.
[11]Cai Y, Yue B, Jiang W, et al. DNA barcoding on subsets of three families in Aves[J]. Mitochondrial DNA, 2010,21(3-4):132-137.
[12]戴传银, 张瑞莹, 尹祚华,等. 部分山雀科鸟类的DNA条形码与物种识别[J]. 动物分类学报, 2010, 35(4):835-841.
[13]蔡延森, 张修月, 岳碧松,等. 我国8种猛禽的DNA条形码技术研究[J]. 四川动物, 2009, 28(3):334-340.
[14]屠云洁,高玉时,苏一军,等.我国部分地方鸡种COⅠ基因多态性及其分子系统进化 研究[J].安徽农业大学学报,2011,38(1):39-42.
[15]徐向明. 我国3个地方品种鸭线粒体DNA COⅠ基因的DNA条形码初步分析[J]. 畜牧与兽医, 2008, 40(11):51-53.
[16]虞德兵, 陆应林, 徐昊翔,等. 基于线粒体COI基因序列分析家鸭系统发育关系[J]. 南京农业大学学报, 2011, 34(6):109-114.
[17]Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia's fish species.[J]. Philosophical Transactions of the Royal Society B, 2005, 360(1462):1847-1857.
[18]Wong L L,Peatman E,L J G.DNA Barcoding of Cat?sh:Species Authentication and Phylogenetic Assessment[J].Plos One, 2011, 3(6):2578-2585.
[19]Bhattacharjee M J, Laskar B A, Dhar B, et al. Identification and re-evaluation of freshwater catfishes through DNA barcoding.[J]. PLOS ONE, 2012, 7(11).doi:10.1371/journal.pone.0049950.
[20]宁平, 刘静. 基于线粒体COI基因的DNA条形码探讨印度-西太平洋沿岸6种金线鱼属鱼类分类及亲缘关系[C]. 中国海洋湖沼学会鱼类学分会、中国动物学会鱼类学分会.2012年学术研讨会论文摘要汇编 2012:42-45.
[21]彭居俐, 王绪祯, 王丁,等. 基于线粒体CO1基因序列的DNA条形码在鲤科鲌属鱼类物种鉴定中的应用[J]. 水生生物学报, 2009, 33(2):271-276.
[22]Hajibabaei M, Janzen D H, Burns J M, et al. DNA barcodes distinguish species of tropical Lepidoptera[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(4):968-971.
[23]Hajibabaei M, Singer G A, Clare E L, et al. Design and applicability of DNA arrays and DNA barcodes in biodiversity monitoring[J]. BMC Biology, 2007, 5(1):24-24.
[24]潘程莹, 胡婧, 张霞,等. 斑腿蝗科Catantopidae七种蝗虫线粒体COI基因的DNA条形码研究[J]. 昆虫分类学报, 2006, 28(2):103-110.
[25]Hollingsworth P M, Forrest L L, Spouge J L, et al. A DNA barcode for land plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(31):12794-12797.
[26]Eason R G, Pourmand N, Tongprasit W, et al. Characterization of synthetic DNA bar codes in Saccharomyces cerevisiae gene-deletion strains.[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(30):11046-11051.
[27]Druzhinina I S, Kopchinskiy A G, Komon M, et al. An oligonucleotide barcode for species identification in Trichoderma and Hypocrea[J]. Fungal Genetics and Biology, 2005, 42(10):813-828.
[28]Seifert K A, Samson R A, Dewaard J R, et al. Prospects for fungus identification using CO1 DNA barcodes, with Penicillium as a test case[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(10):3901-3906.
[29]Mouhamadou B, Carriconde F, Gryta H, et al. Molecular evolution of mitochondrial ribosomal DNA in the fungal genus Tricholoma : Barcoding implications[J]. Fungal Genetics and Biology, 2008, 45(9):1219-1226.
[30]Zhao P, Luo J, Zhuang W, et al. Practice towards DNA barcoding of the nectriaceous fungi[J]. Fungal Diversity, 2011, 46(1):183-191.
[31]Samson R A, Seifert K A, Kuijpers A F A, et al. Phylogenetic analysis of Penicillium subgenus Penicillium using partial 尾-tubulin sequences. Stud Mycol[J]. Studies in Mycology, 2004, 2004(49):175-200.
[32]Gao T, Chen S. Authentication of the Medicinal Plants in Fabaceae by DNA Barcoding Technique[J]. Planta Medica, 2009, 75(4).doi:10.1055/s-2009-1216451.
[33]Chen S, Yao H, Han J, et al. Validation of the ITS2 Region as a Novel DNA Barcode for Identifying Medicinal Plant Species[J]. PLOS ONE, 2010, 5(1).doi:10.1371/journal.pone.0008613.
ConstructionandVerificationofDNABarcodeforHericiumerinaceus
Huangfu Yongguan1, Yan Baosong2, Zhang Yuexin2, Zong Xianchun3
(1.Northeast Forestry University/Saline and alkaline land resources and Environment Research Center,Harbin 150040;2.Heilongjiang Forest Products Research Institute,Heilongjiang Provincial Key Laboratory of Non-wood Forest Product Development,Mudanjiang, Heilongjiang 157011;3.Mudanjiang Normal University/College of life science and technology,Mudanjiang 157000)
2017-05-06
林业公益性行业科研专项经费(201404703-3)
皇甫永冠(1990-),女,博士,从事植物抗逆基因与逆境胁迫应答的分子机理研究,E-mail:595287087@qq.com.
S759.81
A
DOI.:10.13268/j.cnki.fbsic.2017.05.003
