侵染苣荬菜的中国胜红蓟黄脉病毒及伴随的卫星基因组结构特征
2017-09-29赵丽玲张晓云李婷婷
赵丽玲, 钟 静, 张晓云, 李婷婷, 丁 铭
(云南省农业科学院生物技术与种质资源研究所,云南省农业生物技术重点实验室, 昆明 650223)
侵染苣荬菜的中国胜红蓟黄脉病毒及伴随的卫星基因组结构特征
赵丽玲, 钟 静, 张晓云, 李婷婷, 丁 铭*
(云南省农业科学院生物技术与种质资源研究所,云南省农业生物技术重点实验室, 昆明 650223)
杂草是菜豆金色花叶病毒属病毒的重要中间寄主,常富集多种该属病毒及其卫星病毒。2014年,在云南红河常见杂草苣荬菜Sonchusarvensis上出现了疑似菜豆金色花叶病毒属病毒病症状。利用克隆、测序和生物信息学分析技术对其所含病毒进行分离鉴定,结果从1株病样中共获得了两条菜豆金色花叶病毒属病毒全序列、两条β卫星全序列和一条α卫星全序列。序列分析显示,两条菜豆金色花叶病毒属病毒全序列与中国胜红蓟黄脉病毒相似性最高,分别为99%和96%,确定为中国胜红蓟黄脉病毒的分离物。两条β卫星全序列与赛葵黄脉β卫星相似性最高,为97%,确定为赛葵黄脉β卫星的一个分离物。α卫星全序列与中国番茄黄化曲叶α卫星相似性最高,为86.3%,是中国番茄黄化曲叶α卫星的一个分离物。这是菜豆金色花叶病毒属病毒病害复合体在中国侵染苣荬菜的首次报道。
菜豆金色花叶病毒属病毒;α卫星;β卫星; 苣荬菜
菜豆金色花叶病毒属Begomovirus是双生病毒科Geminiviridae中包含种类最多,分布范围最广,造成经济损失最重的一个属[1-3]。按地理分布,菜豆金色花叶病毒属病毒可分为新世界和旧世界病毒。新世界菜豆金色花叶病毒属病毒为双组分病毒,即基因组包含DNA-A和DNA-B两种组分;旧世界菜豆金色花叶病毒属病毒既有双组分病毒也有单组分病毒。单组分的菜豆金色花叶病毒属病毒基因组仅包含DNA-A组分,有些单组分病毒与β卫星(betasatellite)或既与β又与α卫星(alphasatellite)相伴随形成病害复合体[4-5]。β卫星基因组大小约为辅助病毒的一半(约1.3 kb),需要依赖辅助病毒进行复制、包装、移动和介体传播,其互补链编码一个C1蛋白(即βC1),该蛋白能够引起典型的病毒侵染症状,并能抑制寄主植物的RNA沉默反应[6-7]。α卫星基因组大小也约为辅助病毒的一半(约1.3 kb),其病毒链编码一个Rep蛋白,该蛋白使其能自我复制,但需要辅助病毒进行包装、移动和介体传播[4,8]。
中国胜红蓟黄脉病毒Ageratum yellow vein China virus(AYVCNV)属于菜豆金色花叶病毒属成员,其基因组仅含DNA-A组分,与中国胜红蓟黄脉β卫星(Ageratum yellow vein China betasatellite,AYVCNB)相伴随[9-10]。DNA-A组分大小为2.7~2.8 kb,含有6个ORF,分别编码V1、V2、C1、C2、C3和C4基因。β卫星大小约1.3 kb,含有1个ORF,编码βCl蛋白,该卫星是引起胜红蓟黄脉症状所必需的,并能增加辅助病毒AYVCNV的积累量。AYVCNV于2006年首次报道,该病毒与AYVCNB卫星相伴随,引起海南省海口地区的胜红蓟黄脉病[9]。随后,在广西表现黄脉症状的胜红蓟中也发现存在大量的AYVCNV/AYVCNB[10]。近年的研究发现,该病毒不仅侵染胜红蓟,也侵染番茄(GenBank登录号:KC172826,KU975392,KU954377),威胁着番茄作物的生产。
苣荬菜Sonchusarvensis为菊科苦苣菜属多年生草本植物,几乎全世界均有分布,在我国常作为野菜被广泛食用,临床也常用于治疗咽喉肿痛、急性痢疾、炎症、产后出血、痔疮肿痛等症[11-13]。目前印度已有报道空心莲子草黄脉病毒Alternantherayellowveinvirus(AlYVV)病害复合体侵染苣荬菜,引起苣荬菜黄脉症状[14];中国广西有AYVCNV侵染该属另一植物苦苣菜的报道[15],但国内尚未有菜豆金色花叶病毒属病毒侵染苣卖菜的报道。本研究采集了表现叶脉黄化和增厚的苣荬菜病株,从中首次分离到菜豆金色花叶病毒属病毒,并对分离到的病毒核苷酸序列进行了比对和进化重组分析。
1 材料与方法
1.1 病害样品采集
2014年10月在云南省红河州河口县采集了1株苣荬菜病株(YN4368)。该病株具有明显叶脉黄化、增厚等疑似菜豆金色花叶病毒属病毒侵染的症状。
1.2 植物总DNA提取及PCR检测
采用CTAB法提取苣荬菜病株叶片总DNA[16]。利用菜豆金色花叶病毒属病毒简并引物PA/PB进行PCR检测,该引物扩增的目的条带为该属病毒基因共同区及外壳蛋白保守区约500 bp的基因片段[17]。与此同时,利用β卫星通用引物β01/β02和α卫星通用引物UNA101/UNA102对苣荬菜病株进行PCR检测,这两对引物的目的条带分别为其卫星全长[18-19]。PCR反应体系:10×PCR反应缓冲液(含Mg2+)2.5 μL、2.5 μmol/L dNTPs 2 μL、20 μmol/L上、下游引物各0.5 μL、TaqPlus DNA聚合酶(5 U/μL,上海申能博彩生物科技有限公司)0.5 μL、植物总DNA 125 ng,加双蒸水定容至25 μL。扩增条件:94℃预变性2 min;94℃变性45 s,50℃退火45 s,72℃延伸50 s(PA/PB引物)或90 s(β01/β02和UNA101/UNA102引物),30个循环;72℃延伸10 min。PCR产物经1.0%琼脂糖凝胶进行电泳检测。紫外灯下割取目的片段,利用Axygen DNA凝胶回收试剂盒分离纯化,回收产物连接到pGEM-T Easy(Promega, Madison, WI)载体上,通过热击法将连接产物转入大肠杆菌DH5α中,挑选阳性克隆,送上海立菲生物技术有限公司进行测序。
1.3 滚环扩增
为了获得高纯度的菜豆金色花叶病毒属病毒及其卫星基因组,对苣荬菜病样进行了滚环扩增(rolling circle amplification,RCA)。按照TempliphiTMKit(GE Healthcare)说明书上的步骤进行环状DNA的扩增。首先将1 μL植物总DNA样品(约50 ng)加入到5 μL样品缓冲液中,95℃变性3 min,立刻插入冰上放置10 min;然后加入5 μL反应缓冲液和0.2 μL酶(phi29 DNA聚合酶),在30℃反应18 h;最后在65℃下加热10 min,终止反应。
1.4 病毒DNA-A全长基因组克隆
根据测序获得的部分DNA-A序列,设计特异引物AYVCNV-F:5′-GACCCGCCGATATAGTCAT-3′和AYVCNV-R:5′-CGCTTCGACATAATTGCGA-3′扩增DNA-A全长。PCR反应体系:2×PCR反应缓冲液(含Mg2+)25 μL、2 μmol/L dNTPs 10 μL、20 μmol/L 上、下游引物各1 μL、KOD FX DNA聚合酶(1 U/μL,日本东洋纺)1 μL、250 ng 植物总DNA,加双蒸水定容至50 μL。扩增条件:98℃预变性2 min;98℃变性45 s,46℃退火45 s,68℃延伸3 min,30个循环;68℃延伸10 min。按1.2中的方法进行克隆测序。
1.5 病毒DNA-A及其卫星基因组结构和进化重组分析
通过DNASTAR Lasergene 7.1软件完成核酸序列拼接,运用DNAMAN 5.22 (Lynnon Biosoft, Quebec, Canada)和EditSeq(DNASTAR Lasergene 7.1)共同完成开放阅读框的寻找。序列相似性比对先通过BLAST(www.ncbi.nlm.nih.gov)进行初步比对,再选用MegAlign中的Clustal W方法(DNASTAR Lasergene 7.1)进行深入比对。进化树构建采用MEGA 5中的邻接法(neighbor-joining)完成,步差值设置为1 000[20]。重组分析通过RDP 4.46完成[21]。
2 结果与分析
2.1 病毒检测
以PA/PB为引物从YN4368样品中扩增到约500 bp的目的条带;以β01/β02和UNA101/UNA102为引物从YN4368样品中均扩增到约1 300 bp的目的条带。将获得的目的条带进行克隆测序,最终获得5条序列(YN4368-32、YN4368-34、YN4368-28β、YN4368-32β和YN4368-69α)。经BLAST比对后结果显示:YN4368-32与中国胜红蓟黄脉病毒YN1995(AYVCNV-YN1995,KU663132)的CP基因序列相似性高达98%,而YN4368-34与一点红黄脉病毒越南分离物Emiliayellowveinvirusisolate Vietnam(EmYVV-Vietnam,KC878472)相似性高达97%;YN4368-28β和YN4368-32β与赛葵黄脉β卫星Y249分离物(MaYVB-Y249,AJ971703)相似性高达97%;YN4368-69β与中国番茄黄化曲叶α卫星Y244分离物(TYLCCNA-Y244,AJ888449)相似性高达86.3%。以上结果表明,在苣荬菜病株YN4368中存在菜豆金色花叶病毒属病毒以及β和α卫星复合侵染。
2.2 病毒DNA-A全基因组结构及相似性比较
利用全长引物AYVCNV-F/R,以YN4368样品总DNA为模板未扩增到相应条带。改用RCA产物为模板,扩增到大小约为2 800 bp的条带。将目的条带进行克隆、测序后共获得2条DNA-A全长序列(YN4368-84和YN4368-88)。两条DNA-A都具有旧世界单组分菜豆花叶病毒属病毒典型的基因组结构特征,编码7个开放阅读框(ORF)。YN4368-84(GenBank登录号:KX759646)全基因组包含2 744个核苷酸(nucleotides, nt),其病毒链编码AV1(293~1 066 nt)和AV2(133~483 nt),其互补链编码AC1(1 515~2 603 nt)、AC2(1 208~1 615 nt)、AC3(1 063~1 467 nt)、AC4(2 153~2 575 nt)、AC5(271~543 nt),在AC1和AV2间含有一个273 nt的基因间隔区(intergenic region,IR)。YN4368-88(GenBank登录号:KX759647)全基因组包含2 752 nt,其病毒链编码AV1(294~1 067 nt)和AV2(134~484 nt),互补链编码AC1(1 516~2 604 nt)、AC2(1 209~1 616 nt)、AC3(1 064~1 468 nt)、AC4(2 190~2 447 nt)、AC5(272~544 nt),在AC1和AV2间含有一个281 nt的IR。
将序列YN4368-84与YN4368-88进行比对发现,两条序列相似性为90.8%。将这两条序列与GenBank中的其他病毒序列进行比对,发现序列YN4368-84与当地发现的中国胜红蓟黄脉病毒分离物Y4326(AYVCNV-Y4326,KU601622)和分离物YN4265(AYVCNV-YN4265,KU975392)相似性最高,为99%;序列YN4368-88与中国胜红蓟黄脉病毒分离物YN4356(AYVCNV-YN4356,KU975393)相似性最高,为96%。根据国际病毒分类委员会(ICTV)确定的菜豆金色花叶病毒属病毒种的分类标准,DNA-A的核苷酸相似性小于89%则认为是病毒新种,大于89%则确定为同种不同分离物[3]。另外,根据Fauquet等对同种不同成员间的分类标准,同种成员间的核苷酸相似性小于93%则表示这些成员属于不同的株系[22]。因此,YN4368-84DNA-A和YN4368-88DNA-A都属于中国胜红蓟黄脉病毒,但二者属于不同的株系,分别为中国胜红蓟黄脉病毒分离物YN4368-84(AYVCNV-YN4368-84)和中国胜红蓟黄脉病毒分离物YN4368-88(AYVCNV-YN4368-88)。以上结果表明,苣荬菜病株YN4368中存在中国胜红蓟黄脉病毒两种株系的共同侵染。
2.3 病毒DNA-A的进化和重组分析
将分离物AYVCNV-YN4368-84和AYVCNV-YN4368-88与其他菜豆金色花叶病毒属病毒构建进化树。结果显示,AYVCNV-YN4368-84与AYVCNV-Y4326聚于一个小分支,和其余AYVCNV等又共同聚于一个分支;AYVCNV-YN4368-88与AYVCNV-YN4356独自在一个小的分支,和其余AYVCNV等又共同聚于一个分支(图1)。该结果表明AYVCNV-YN4368-84和AYVCNV-Y4326亲缘关系最近;AYVCNV-YN4368-88与AYVCNV-YN4356亲缘关系最近;虽然AYVCNV-YN4368-84和AYVCNV-YN4368-88分离自同一寄主、属于同一种病毒,但两者在遗传进化中存在一定的距离。RDP重组分析显示,分离物AYVCNV-YN4368-84和AYVCNV-YN4368-88中并无明显的重组事件发生。

图1 AYVCNV-YN4368-84和AYVCNV-YN4368-88与报道的其他菜豆金色花叶病毒属病毒基于DNA-A的系统进化树Fig.1 Phylogenetic dendrogram based upon DNA-A nucleotide sequence alignment of AYVCNV-YN4368-84 and AYVCNV-YN4368-88 with other selected begomoviruses from GenBank
2.4 病毒伴随的β卫星分子结构及其相似性比较
序列YN4368-28β(KX759648)和YN4368-32β全长都为1 359 nt,序列同源性为99.9%,仅有2个核苷酸的差异,因此选择YN4368-28β进行基因注释和分析。YN4368-28β互补链上编码一个C1 基因(196~585 nt),在1 275~1 359 nt和1~14 nt区域有1个包含着茎环结构在内的卫星保守区域(satellite conserved region,SCR),在766~970 nt 区域内有一个富含A的区域(A含量为60.49%)。将YN4368-28β序列与GenBank中的其他β序列进行比对发现,该序列与MaYVB-Y249(AJ971703)相似性最高,为97%,与MaYVB其他分离物相似性为95%~96%,与其他β卫星序列相似性≤83%。根据ICTVβ卫星分类标准,全序列核苷酸同源性小于78%则认为是新的β卫星,大于则确定该分离物属于同一种β卫星[3],因此YN4368-28β卫星属于MaYVB的一个分离物,即赛葵黄脉β卫星分离物YN4368-28(MaYVB-YN4368-28)。将MaYVB-YN4368-28与分布在云南的其他β卫星构建进化树,结果显示MaYVB-YN4368-28与MaYVB-Y47和MaYVB-Y249聚于一个小分支,又与其他MaYVB共同聚于一个分支(图2)。该进化分析结果表明,MaYVB-YN4368-28与MaYVB-Y47和MaYVB-Y249亲缘关系较近。RDP重组分析结果显示,MaYVB-YN4368-28卫星序列中无明显的重组现象。
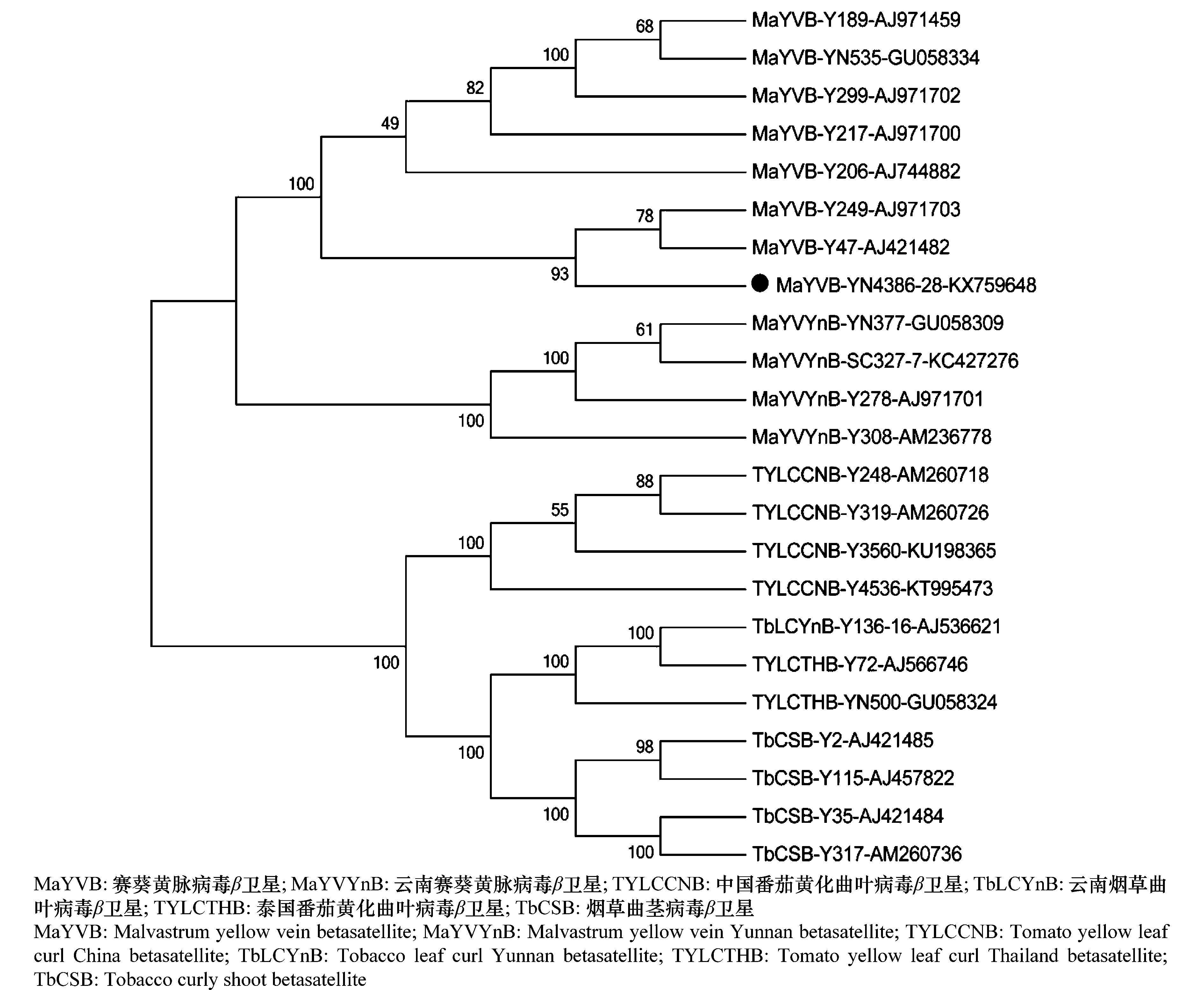
图2 基于MaYVB-YN4368-28与云南报道的其他β卫星分子构建的系统进化树Fig.2 Phylogenetic dendrogram based upon alignment of MaYVB-YN4368-28 with other betasatellites identified in Yunnan Province
2.5 病毒伴随的α卫星分子结构及其相似性比较
序列YN4368-69α(KX759649)全长为1 359 nt,病毒链上编码一个Rep基因(79~1 026 nt),具有一个包含TAGTATTAC序列的茎环结构,在1 025~1 214 nt 区域内有一个富含A的区域(A含量为48.95%)。将YN4368-69α与GenBank中的其他α序列进行比对发现,该序列与TYLCCNA-Y244相似性最高,为86.3%,与其他α卫星序列相似性≤85%。根据ICTVα卫星分类标准,全序列核苷酸同源性小于83%则认为是新的α卫星,大于则确定该分离物属于同一种α卫星[3],因此YN4368-69α卫星属于TYLCCNA(TYLCCNA-YN4368-69)。将TYLCCNA-YN4368-69与GenBank中其他α卫星构建进化树,结果显示TYLCCNA-YN4368-69独自成一支,与其他TYLCCNA共同聚于一个分支(图3)。该结果表明,TYLCCNA-YN4368-69与TYLCCNA的其他分离物亲缘关系相对较近,但又存在一定的距离。RDP重组分析结果显示,TYLCCNA-YN4368-69是由TYLCCNA-Y261和TbCSA-Y216重组而来,TYLCCNA-Y261是主要的母本,而TbCSA-Y216则是次要的母本(RDP,P=3.539×10-0.4;SiScan,1.107×10-0.6)。重组主要发生在TYLCCNA-YN4368-69的306~958 nt区段(Rep),该区段来自TbCSA-Y216。

图3 基于TYLCCNA-YN4368-69与云南报道的其他α卫星分子构建的系统进化树Fig.3 Phylogenetic dendrogram based upon alignment of TYLCCNA-YN4368-69 with other alphasatellites identified in Yunnan Province
3 结论与讨论
本研究从表现黄脉症状的苣荬菜中分离到中国胜红蓟黄脉病毒,利用分子克隆手段和生物信息学技术对该病毒进行分析,结果表明,该病毒与赛葵黄脉β卫星和中国番茄黄化曲叶α卫星相伴随,这是菜豆金色花叶病毒属病毒复合体侵染中国苣荬菜的首次报道。
杂草是菜豆金色花叶病毒属病毒及其传播介体烟粉虱的重要中间寄主,能同时被多种该属病毒及其卫星复合侵染。本研究从1株苣荬菜中分离到了两种株系的中国胜红蓟黄脉病毒、一种β卫星以及一种α卫星,表明苣荬菜是菜豆金色花叶病毒属病毒的寄主之一,能被该属病害复合体侵染。已有研究表明,杂草如同收纳菜豆金色花叶病毒属病毒的容器,通常一株杂草中能同时被多种病毒及其卫星复合侵染[14, 23-24]。菜豆金色花叶病毒属病毒复合侵染增加了重组和组分互换发生的频率,导致产生新分离物和新病毒[25-27]。因此需进一步对红河州河口县杂草开展系统调查和检测,加强对AYVCNV/MaYVB/TYLCCNA病害复合体发生率及病害严重度的调查研究,以防该类型的复合体造成病害的暴发流行。
AYVCNV在海南省海口和广西胜红蓟中最先发现[9-10],2012年在越南胜红蓟中也发现该病毒(AYVCNV-Vietnam 16,KC878475)。云南河口处于元江-红河流域向越南的出境处,是云南海拔最低的区域。本研究中发现来自云南红河的分离物AYVCNV-YN4368-84、AYVCNV-Y4326、AYVCNV-YN4265与AYVCNV-Vietnam 16亲缘关系最近。该结果表明,云南河口地区的AYVCNV-YN4368-84这一株系是AYVCNV-Vietnam 16的一个新的变种,且该株系很有可能是AYVCNV-Vietnam 16株系跨境传播传入该地。
假重组是导致菜豆花叶病毒属病毒遗传变异的一个重要原因之一,DNA-A和卫星之间的交换也称为假重组。已有研究表明,AYVCNV与AYVCNB形成AYVCNV/AYVCNB病害复合体或与AYVCNA形成AYVCNV/AYVCNA复合体[9-10,28]。本研究从黄脉症状的苣荬菜中分离到了AYVCNV/MaYVB/TYLCCNA病害复合体,这表明AYVCNV在自然条件下也能与其他异源β卫星和α卫星相伴随,并且该复合体的形成很有可能是通过假重组实现的。MaYVB最早于2008年从具有黄脉症状的赛葵上分离到,与MaYVV相伴随,参与植株典型症状反应,并能增加MaYVV在本氏烟中的积累[29]。本研究中,MaYVB是否是症状所必需,以及能否加强AYVCNV在苣荬菜中的积累还需进一步的研究。
[1] Harrison B D, Barker H, Book K R, et al. Plant viruses with circular single-stranded DNA [J]. Nature, 1977, 270(5639): 760-762.
[2] Varma A, Malathi V G. Emerging geminivirus problems: a serious threat to crop production [J]. Annals of Applied Biology, 2003, 142(2): 145-164.
[3] Brown J K, Fauquet C M, Briddon R W, et al. Virus taxonomy: Ninth report of the international committee on taxonomy of viruses[M]. San Diego: Elsevier Academic Press, 2012: 351-373.
[4] Nawaz-ul-Rehman M S, Fauquet C M. Evolution of geminiviruses and their satellite[J]. FEBS Letters, 2009, 583(12): 1825-1832.
[5] Xie Yan, Wu Peijun, Liu Pei, et al. Characterization of alpha satellites associated with monopartite begomovirus / beta satellite complexes in Yunnan, China [J]. Virology Journal, 2010, 7: 178.
[6] Briddon R W, Stanley J. Subviral agents associated with plant single stranded DNA viruses [J]. Virology 2006, 344(1): 198-210.
[7] Zhou Xueping. Advances in understanding begomovirus satellites[J]. Annual Review of Phytopathology, 2013, 51: 357-381.
[8] Saunders K, Bedford I D, Stanley J. Adaptation from whitefly to leafhopper transmission of an autonomously replicating nanovirus-like DNA component associated with ageratum yellow vein disease [J]. Journal of General Virology, 2002, 83(4): 907-913.
[9] Xiong Qing, Fan Sanwei, Wu Jianxiang, et al. Ageratum yellow vein China virus is a distinct begomovirus species associated with a DNAβmolecule [J]. Phytopathology, 2007, 97(4): 405-411.
[10] Jiao Xiaoyang, Gong Huanran, Liu Xuejian, et al. Etiology of Ageratum yellow vein diseases in South China [J]. Plant Disease, 2013, 97(11): 1497-1503.
[11] 石铸. 中国植物志[M]. 北京: 科学出版社, 1997: 60-69.
[12] 邵成雷. 苣荬菜的营养及药理作用[J]. 食品与药品, 2005, 7(6): 63-65.
[13] 李宁, 李铣, 杨世林. 过山蕨中有机酸类化学成分[J]. 沈阳药科大学学报, 2006, 23(7): 427.
[14] Mubin M, Shahid M S, Tahir M N, et al. Characterization of begomovirus components from a weed suggests that begomoviruses may associate with multiple distinct DNA satellites [J]. Virus Genes, 2010, 40(3): 452-457.
[15] 徐幼平. 广西双生病毒的分子鉴定[D]. 杭州: 浙江大学, 2006.
[16] Doyle J J, Doyle J L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochemistry Bulletin, 1987, 19: 11-15.
[17] Xie Yan, Zhou Xueping, Zhang Zhongkai, et al.Tobaccocurlyshootvirusisolated in Yunnan is distinct species ofBegomovirus[J]. Chinese Science Bulletin, 2002, 47(3): 197-200.
[18] Briddon R W, Bull S E, Mansoor S, et al. Universal primers for the PCR-mediated amplification of DNA beta-a molecule associated with some monopartite begomoviruses [J]. Molecular Biotechnology, 2002, 20(3): 315-318.
[19] Bull S E, Briddon R W, Markham P G. Universal primers for the PCR-mediated amplification of DNA 1: a satellite-like molecule associated with begomovirus-DNAβcomplexes [J]. Molecular Biotechnology, 2003, 23: 83-86.
[20] Tamura K, Peterson D, Peterson N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[21] Martin D P, Lemey P, Lott M, et al. RDP3: a flexible and fast computer program for analyzing recombination [J]. Bioinformatics, 2010, 26(19): 2462-2463.
[22] Fauquet C M, Briddon R W, Brown J K, et al. Geminivirus strain demarcation and nomenclature [J]. Archives of Virology, 2008, 153(4):783-821.
[23] 刘培. 云南赛葵上双生病毒的分子鉴定[D]. 杭州: 浙江大学, 2008.
[24] 李静. 我国六种双生病毒的分子鉴定及两种病毒的致病性研究[D]. 杭州: 浙江大学, 2010.
[25] Garcia-Andres S, Tomas D M, Sanchez-Campos S, et al. Frequent occurrence of recombinants in mixed infections of tomato yellow leaf curl disease-associated begomoviruses [J]. Virology, 2007, 365(1): 210-219.
[26] Albuquerque L C, Varsani A, Fernandes F R, et al. Further characterization of tomato-infecting begomoviruses in Brazil [J]. Archives of Virology, 2012, 157(4): 747-752.
[27] Rocha C S, Castillo-Urquiza G P, Lima A T M, et al. Brazilian begomovirus populations are highly recombinant, rapidly evolving, and segregated based on geographical location [J]. Journal of Virology, 2013, 87(10): 5784-5799.
[28] She Xiaoman, He Zifu, Yin Guohua, et al. A New alpha satellite molecule associated with Ageratum yellow vein China virus in the Philippines [J]. Journal of Phytopathology, 2015, 163(1): 54-57.
[29] Guo Wei, Jiang Tong, Zhang Xian, et al. Molecular variation of satellite DNAβmolecules associated withMalvastrumyellowveinvirusand their role in pathogenicity [J]. Applied and Environmental Microbiology, 2008, 74(6): 1909-1913.
(责任编辑: 田 喆)
MolecularcharacterizationofAgeratumyellowveinChinavirusinfectingSonchusarvensisinYunnan,Chinaandassociatedsatellites
Zhao Liling, Zhong Jing, Zhang Xiaoyun, Li Tingting, Ding Ming
(InstituteofBiotechnologyandGermplasmResources,YunnanAcademyofAgriculturalSciences;KeyLaboratoryofAgriculturalBiotechnologyofYunnanProvince,Kunming650223,China)
Weeds are crucial intermediate hosts of begomoviruses and frequently harbor multiple begomoviruses and associated satellites.Sonchusarvensis, a common weed in Honghe,Yunnan Province, showed symptoms typical of begomovirus infection in 2014. Begomoviruses associated withS.arvensiswas studied using cloning, sequencing and bioinformatic analysis. Two full-length begomoviruses, two betasatellites and one alphasatellite were isolated from one sample. Sequence analysis showed that these begomovirus sequences shared the highest identity (99% or 96%) with Ageratum yellow vein China virus (AYVCNV), confirming both of them as an isolate of AYVCNV. These two betasatellite sequences shared 97% identity with Malvastrum yellow vein betasatellite (MaYVB), confirming all of them as isolates of MaYVB. The alphasatellite sequence shared 86.3% identity with Tomato yellow leaf curl China alphasatellite (TYLCCNA), confirming it as an isolate of TYLCCNA. This is the first report of begomovirus disease complex associated withS.arvensisin China.
begomoviruses; alphasatellite; betasatellite;Sonchusarvensis
S 436.43
: ADOI: 10.3969/j.issn.0529-1542.2017.05.010
2016-10-13
: 2017-01-07
云南省应用基础研究青年项目(2014FD067);国家自然科学基金(31260421)
* 通信作者 E-mail:mingd73@163.com
