藏绵羊FAM134B基因克隆与表达分析
2017-09-27李文丽杜晓华高景波申超超
李文丽,杜晓华,刘 霞,,高景波,申超超,张 超,刘 伟
(1.甘肃农业大学 生命科学技术学院,甘肃 兰州 730070; 2.甘肃省草食动物生物技术重点实验室,甘肃 兰州 730070; 3.甘肃农业大学 动物医学院,甘肃 兰州 730070)
藏绵羊FAM134B基因克隆与表达分析
李文丽1,2,杜晓华2,3,刘 霞1,2,*,高景波1,2,申超超1,2,张 超1,2,刘 伟1,2
(1.甘肃农业大学 生命科学技术学院,甘肃 兰州 730070; 2.甘肃省草食动物生物技术重点实验室,甘肃 兰州 730070; 3.甘肃农业大学 动物医学院,甘肃 兰州 730070)
采用RT-PCR技术克隆了藏绵羊FAM134B(family with sequence similarity 134, member B)基因,利用生物学软件进行相关生物信息学分析,同时用实时荧光定量PCR(real-time fluorescence quantitative PCR, qRT-PCR)研究组织表达水平。克隆获得FAM134B基因全长1 144 bp (GenBank登录号: KX580309),开放阅读框1 071 bp,编码356个氨基酸。系统进化树表明,藏绵羊FAM134B和绵羊、山羊关系较近。藏绵羊FAM134B基因编码蛋白的分子式为C1717H2696N448O572S12,分子量为39 151.72 u,理论等电点(pI)为4.49,消光系数为24 450,不稳定系数为43,疏水指数为80.31,平均亲水性为-0.422,属不稳定可溶性酸性蛋白质。二级结构主要是α-螺旋(45.51%)、无规卷曲(41.85%)和延伸链(12.64%),属于混合类蛋白质。亚细胞定位和功能分析结果显示,FAM134B编码的蛋白在内质网、细胞质膜概率分别为30.4%和21.7%,主要在蛋白质运输和结合(77.3%)方面起作用。系统发育树表明,藏绵羊FAM134B氨基酸序列与绵羊、山羊同源性非常高。qRT-PCR结果表明,FAM134B基因网胃中表达最高,十二指肠表达量最低。研究结果可为进一步研究藏绵羊FAM134B基因结构和生理功能及其遗传特性提供理论依据。
藏绵羊;FAM134B基因;CDS区克隆;生物信息学分析;表达
FAM134B(family with sequence similarity 134,member B)是最近发现的一种功能基因,其具体的功能还不是太清楚。Kurth等[1]研究发现FAM134B基因调控自主神经元以及疼痛感知神经元。此外,FAM134B基因在人食道鳞状细胞癌变机理方面有着非常重要的意义[2]。高尔基体在蛋白质加工修饰方面起着重要作用,FAM134B基因缺失会导致出现新的高尔基体蛋白影响高尔基体的功能和脂质的合成[2]。袁章琴等[3]研究FAM134B基因极有可能与脂肪沉积相关,推测可以通过调控FAM134B的表达来调控脂肪的沉积。藏绵羊(Tibetan sheep)主要分布于我国青藏高原及与其毗邻的川、滇、甘等高寒地区,具有良好的肉用价值。朱武政等[4]研究发现,FAM134B基因在山羊肌内脂肪沉积方面起着重要作用,这为肉质品质改良奠定了理论基础。目前,国外针对FAM134B基因的研究尚处于起步阶段,且主要集中在人、鼠上,国内也仅见对猪、牦牛、黑山羊FAM134B基因研究的相关报道[3-5],对于高原特色动物藏绵羊FAM134B基因的研究尚未见报道。
本研究旨在克隆藏绵羊FAM134BCDS区,分析基因结构特点,探索藏绵羊FAM134B基因结构和生理功能,丰富藏绵羊FAM134B基因的遗传信息数据,进一步揭示藏绵羊FAM134B基因的遗传特性。
1 材料与方法
1.1 材料
从甘肃省甘南藏族自治州合作市采集藏绵羊大脑枕部、脑干、直肠、松果体、脾脏、肝脏、结肠、十二指肠、盲肠、结肠、网胃、回肠,将其快速切下,放入装有RNA冻存液小管中,再迅速放入液氮中,拿到实验室-70 ℃保存。
1.2 主要试剂
组织RNA冻存液、RNA提取试剂盒、DNA胶回收试剂盒购自生工生物工程(上海)股份有限公司;RNase-Free ddH2O、TaKaRa反转录试剂盒、DL2000 DNA marker和pGM-T克隆载体试剂盒购自北京天根生化科技公司; IPTG 及 X-Gal 购自 Amersco公司;Trans-5α感受态细胞购自北京全式金生物技术有限公司;TaKaRaTaq酶购自北京百泰克生物技术公司。
1.3 方法
1.3.1 藏绵羊大脑枕部组织总RNA提取及RT-PCR反应
用RNA试剂盒提取藏绵羊大脑枕部组织总RNA,用紫外分光光度计与1%琼脂糖凝胶电泳进行质量检测。按反转录试剂盒说明书反转录为cDNA,用1%琼脂糖凝胶电泳检测,-70 ℃保存。
1.3.2 引物设计
根据GenBank中绵羊FAM134B基因序列(登录号:XM_004017080.1),用Primer3.0在线软件设计特异性引物,预计扩增大小1 100 bp;引物序列如表1所示:
1.3.3 PCR扩增
总反应体系25 μL:上下游引物各1 μL,RNase-Free ddH2O 12 μL,Taq酶10 μL,cDNA模板1 μL。PCR反应条件:94 ℃预变性4 min;94 ℃变性30 s,59 ℃退火30 s,72 ℃延伸65 s,35个循环;72 ℃终延伸10 min,4 ℃保存。PCR扩增产物用1%的琼脂糖凝胶电泳检测。
1.3.4 目的片段TA克隆与鉴定
用胶回收试剂盒回收目的片段,在4 ℃条件下过夜连接 pGM-T克隆载体;连接产物转化至Trans-5α感受态细胞,培养数小时后挑取白色阳性单克隆菌落,再进行菌液PCR 鉴定。扩增产物用1%的琼脂糖凝胶电泳检测,筛选阳性菌液送金斯瑞股份有限公司进行测序。
表1引物信息
Table1Primer information

目的基因Targetgene引物序列(5'~3')Primersequence片段Fragment/bp用途PurposeFAM134BF:GCAGAAATGCCTGAAGGTGA1144扩增FAM134BcDNA片段R:TTGGTAGTTTGTGCTCTGTCFAM134BcDNAfragmentamplificationFAM134BF:CACAGAGAAGAAGTCAGCAG171实时荧光定量PCRR:TTCAAAGTCATCGCCCTCCTqRT-PCRGAPDHF:GCAAGTTCCACGGCACAG118实时荧光定量PCRR:TCAGCACCAGCATCACCCqRT-PCR
GAPDH: 3-磷酸甘油醛脱氢酶
GAPDH: Glyceraldehyde 3-phosphate dehydrogenas
1.3.5 序列生物信息学分析
使用ProtParam(http://web.expasy.org/protparam/)[6]、NCBI的ORF Finder[7]分析进行基因阅读开放框分析,用Protfun(http://www. cbs.dtu.dk/services/ProtFun/)在线软件对FAM134B基因的功能进行预测分析[8]用PredictProtein和(http://www.predictprotein.org)[9]、SWISS-MODEL(http://swissmodel.expasy.org/)[10]等分析软件与Lasergene7.1软件包中的
Editseq软件[10]和protean软件[11],PHD软件[12]分析FAM134B的理化性质以及一级结构、二级结构、三级结构进行预测分析;利用Graph Pad Prism5软件[13]分析荧光定量PCR结果;使用Lasergene7.1软件中的MegAlign软件[14]进行同源性分析及构建系统进化树;使用在线亚细胞定位工具PSORT II Prediction(http://psort.hgc.jp/ form.html)[15]预测蛋白质的亚细胞定位。
1.3.6 藏绵羊FAM134B基因mRNA在不同组织的表达分析
选3-磷酸甘油醛脱氢酶 (glyceraldehyde 3-phosphate dehydrogenase,GAPDH)为内参基因,再根据藏绵羊FAM134BcDNA片段,设计一对荧光引物设计内参基因引物(表1)。20 μL PCR 扩增体系包括:Taq酶10 μL,上下游引物各1 μL,cDNA 1.5 μL,ddH2O 7.8 μL,采用ABI荧光定量PCR仪进行定量分析。荧光定量PCR反应条件如下:95 ℃预变性5 min;95 ℃变性15 s,60 ℃退火20 s,72 ℃延伸5 min,40个循环。
2 结果与分析
2.1 藏绵羊FAM134B基因总RNA检测
用1%琼脂糖凝胶电泳检测藏绵羊大脑枕部组织总RNA,得到两条带,分别为28S rRNA、18S rRNA,并且28S的亮度是18S的2倍,5S rRNA很弱,几乎看不到(图1);所测定的D260/D280为1.8~2.0,无蛋白质和DNA污染,RNA完整性较好。
用1%琼脂糖凝胶电泳检测藏绵羊大脑枕部RT-PCR扩增产物,出现单一目的DNA条带,条带清晰明亮(图2),紫外灯下切胶回收,用于克隆测序。将克隆后的阳性菌液[16]用1%琼脂糖凝胶电泳检测(图2),送公司测序,获得藏绵羊FAM134B基因CDS区序列,提交至GenBank(登录号:KX580309)。
2.2藏绵羊FAM134B基因CDS区核苷酸序列、编码氨基酸序列以及开放阅读框分析
参考Kozak法则[17],利用NCBI的ORF Finder 分析藏绵羊基因CDS序列,得到长1 144 bp的ORF,起始密码子ATG位于22 bp处,终止密码子TAG位于1 092 bp处。通过软件分析得到A=28.94%,G=24.74%,T=23.53%,C=22.78%。共编码356个氨基酸残基(图3)。

图1 藏绵羊大脑枕部组织总RNA琼脂糖凝胶电泳Fig.1 Agarose gel check of total RNA from Tibetan sheep occipital lobe of brain
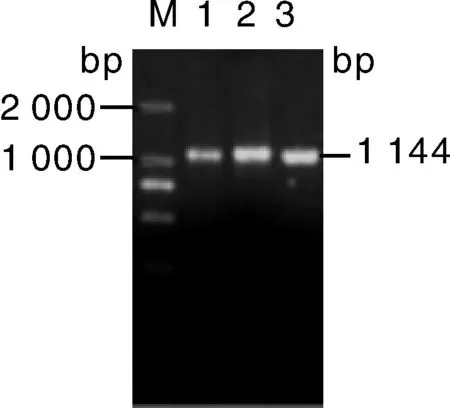
M, DL2 000 DNA marker; 1, PCR扩增产物; 2, DNA纯化回收; 3, 菌液PCRM, DL2 000 DNA marker; 1, The PCR amplicons; 2, Purification recycling DNA; 3, The amplicons of bacteria liquid PCR图2 藏绵羊FAM134B基因RT-PCR扩增产物琼脂糖凝胶电泳Fig.2 Agarose gel check of RT-PCR amplicons of FAM134B gene in Tibetan sheep
2.3藏绵羊FAM134B基因编码蛋白的理化性质分析
用Prot Param软件和DNA star软件包中的Edit Sequence软件预测蛋白质的理化性质,主要包括氨基酸组成、分子式、相对分子质量、等电点、消光系数、半衰期、疏水性等[18]。结果表明:
藏绵羊FAM134B编码的蛋白由356个氨基酸残基组成,分子式为C1717H2696N448O572S12,分子量为39 151.6 u,理论等电点(pI)为4.49,说明该蛋白为酸性蛋白质。在氨基酸残基中Leu(12.64%)、和Ser(11.24%)的频率较高,极性氨基酸占28.37%,疏水性氨基酸占31.74%,带电氨基酸占31.46%,其中包括 17.13%的酸性氨基酸及9.27%的碱性氨基酸(表2)。预测其消光系数(γ=280 nm)为24 450,不稳定系数为43,属不稳定类蛋白质[19]。在哺乳动物网织红细胞内的半衰期为30 h[20],平均亲水性为-0.422,疏水指数为80.31,属可溶性蛋白[21]。
2.4藏绵羊基因编码蛋白的二级与三级结构预测
预测分析蛋白质的二级结构有助于认识蛋白的空间结构[22]。使用PHD软件预测藏绵羊基因编码蛋白的二级结构,结果表明,藏绵羊FAM134B主要是由α-螺旋(alpha helix)和延伸链(extended strand)无规卷曲(random coil)构成(图4),其中45.51%的氨基酸构成α-螺旋,41.85%的氨基酸构成无规卷曲,12.64%构成延伸链,因此藏绵羊基因编码的蛋白可归为混合类-蛋白质[23]。采用SWISS-MODEL软件对藏绵羊编码的蛋白质建模获得部分三级结构模型(图4),从部分模型中可以看出,其三级结构中主要由α-螺旋、无规卷曲和延伸链构成,跟二级结构预测结果基本一致。

图3 藏绵羊FAM134B基因CDS区的核苷酸和编码蛋白的氨基酸序列Fig.3 Nucleotide sequence and amino acid sequence in the CDS region of Tibetan sheep FAM134B gene
表2藏绵羊FAM134B基因编码蛋白的氨基酸组成
Table2Composition of amino acid coded by Tibetan sheepFAM134Bgene
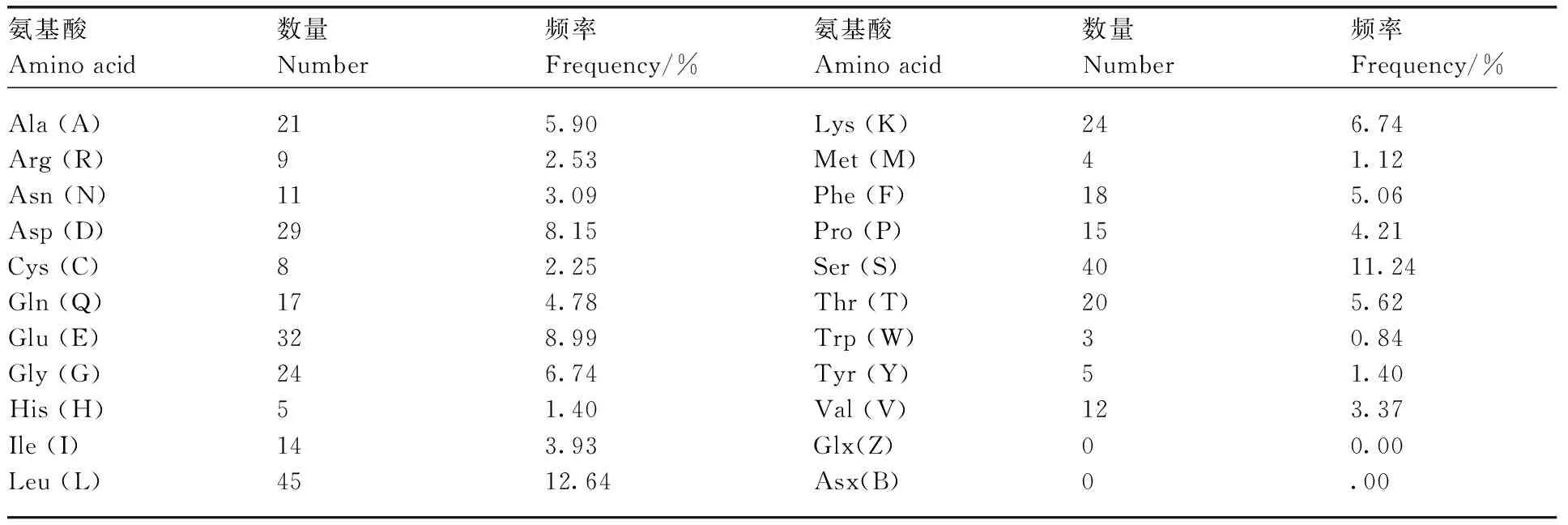
氨基酸Aminoacid数量Number频率Frequency/%氨基酸Aminoacid数量Number频率Frequency/%Ala(A)Arg(R)Asn(N)Asp(D)Cys(C)Gln(Q)Glu(E)Gly(G)His(H)Ile(I)Leu(L)21911298173224514455.902.533.098.152.254.788.996.741.403.9312.64Lys(K)Met(M)Phe(F)Pro(P)Ser(S)Thr(T)Trp(W)Tyr(Y)Val(V)Glx(Z)Asx(B)244181540203512006.741.125.064.2111.245.620.841.403.370.00.00
2.5藏绵羊FAM134B亚细胞定位以及功能预测分析
利用PSORT II Prediction软件对FAM134B的亚细胞定位进行预测分析,结果表明,FAM134B编码的蛋白在内质网、细胞质膜、空泡、细胞核、高尔基体、细胞骨架和线粒体中分别占30.4%、21.7%、17.4%、17.4%、4.3%、4.3%和4.3%。在内质网中分布最多,其次是细胞质膜。利用Protfun2.2软件预测FAM134B功能分类,结果显示,该蛋白在运输与结合过程、电压激活离子通道、嘌呤和嘧啶、信号转导分子、离子通道、辅因子的生物合成、阳离子通道、转录因子和转录调控因子发挥功能的可能性分别为0.773、0.279、0.331、0.205、0.169、0.279、0.146、0.219和0.111(表3),前三种明显高于其他预测功能,由此可推测FAM134B基因编码的蛋白在物质运输与结合过程、辅因子的生物合成等过程中起到离子通道载体和信号转导及转录调控等重要作用,尤其是协助蛋白质的组装运输等功能。
2.6藏绵羊FAM134B蛋白同源性及系统发育分析
利用MegAlign软件对藏绵羊和其他物种的FAM134B核酸序列和氨基酸序列进行对比(表4),结果表明,藏绵羊FAM134B氨基酸序列与普通绵羊完全一致,与山羊具有较高同源性,与人类最远,说明藏绵羊FAM134B在哺乳动物之间具有高度保守性;根据藏绵羊和其他动物FAM134B 氨基酸序列,使用MegAlign 软件,采用邻近法构建分子进化树。结果表明,藏绵羊与绵羊FAM134B 聚为一类,距离较近,与人进化关系较远(图5)。
2.7藏绵羊FAM134B基因在不同组织中mRNA表达分析
如图6所示,FAM134B基因表达量从高到低依次是:网胃、回肠、盲肠、大脑枕叶、脑干、结肠、松果体、肝脏、直肠、脾脏、十二指肠。表明FAM134B基因在各个组织中的表达有差异,说明FAM134B的转录有一定的组织特异性,在网胃中最高,在脾脏和十二指肠中表达量最低,其他组织中表达量居中。

图中黑色的线表示α-螺旋,深灰色的线表示延伸链,浅灰色的线代表无规则卷曲The black line represents α-helix, the dark grey line represents extended strand and the light grey line represents random coil图4 FAM134B氨基酸序列二级结构、三级结构预测Fig.4 The predicted secondary structure and a part of putative tertiary structure of amino acid sequence of FAM134B
表3FAM134B功能分析结果
Table3Analysis of FAM134B function
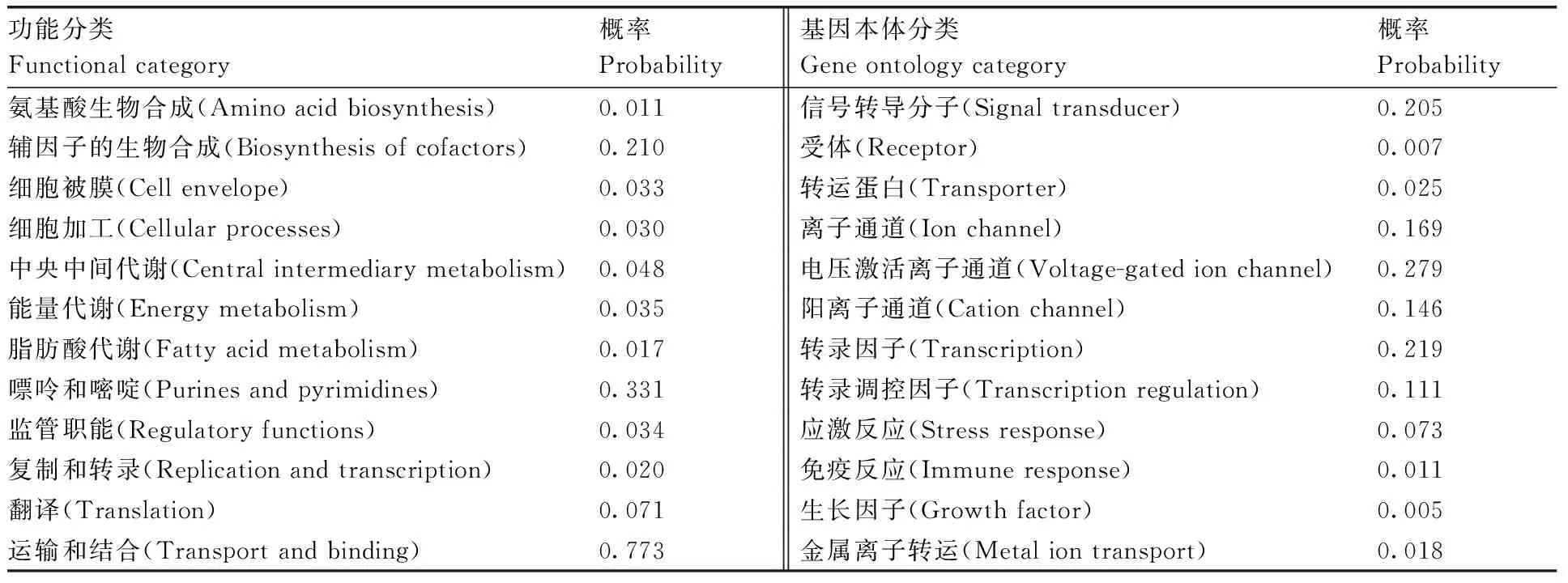
功能分类Functionalcategory概率Probability基因本体分类Geneontologycategory概率Probability氨基酸生物合成(Aminoacidbiosynthesis)0.011信号转导分子(Signaltransducer)0.205辅因子的生物合成(Biosynthesisofcofactors)0.210受体(Receptor)0.007细胞被膜(Cellenvelope)0.033转运蛋白(Transporter)0.025细胞加工(Cellularprocesses)0.030离子通道(Ionchannel)0.169中央中间代谢(Centralintermediarymetabolism)0.048电压激活离子通道(Voltage-gatedionchannel)0.279能量代谢(Energymetabolism)0.035阳离子通道(Cationchannel)0.146脂肪酸代谢(Fattyacidmetabolism)0.017转录因子(Transcription)0.219嘌呤和嘧啶(Purinesandpyrimidines)0.331转录调控因子(Transcriptionregulation)0.111监管职能(Regulatoryfunctions)0.034应激反应(Stressresponse)0.073复制和转录(Replicationandtranscription)0.020免疫反应(Immuneresponse)0.011翻译(Translation)0.071生长因子(Growthfactor)0.005运输和结合(Transportandbinding)0.773金属离子转运(Metaliontransport)0.018
表4藏绵羊和其他物种间FAM134B基因序列和氨基酸序列一致性比较
Table4Identities of nucleotide and amino acid sequences of FAM134B between Tibetan sheep and other species
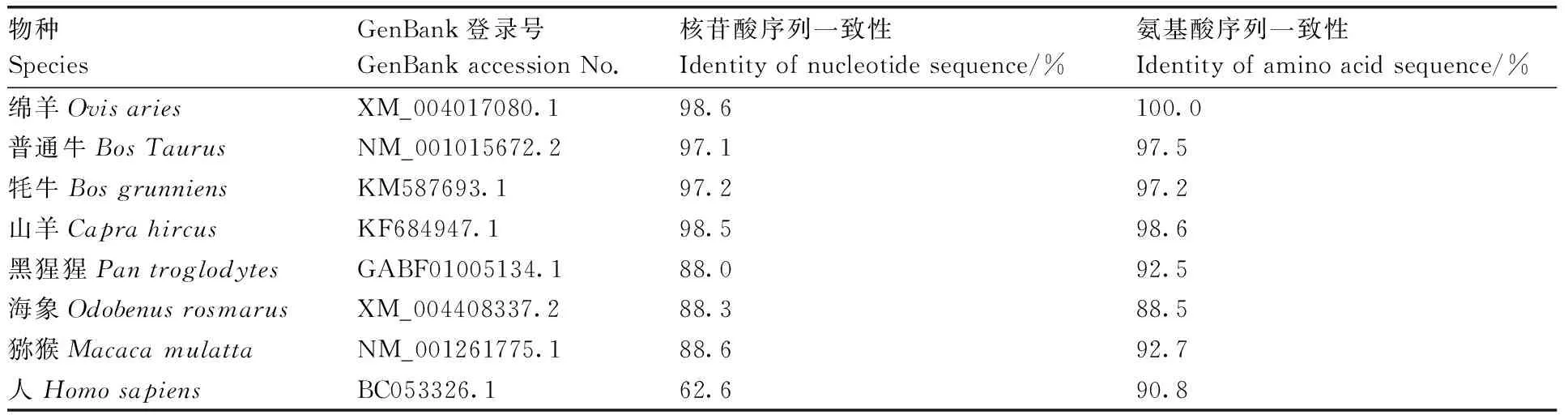
物种SpeciesGenBank登录号GenBankaccessionNo.核苷酸序列一致性Identityofnucleotidesequence/%氨基酸序列一致性Identityofaminoacidsequence/%绵羊OvisariesXM_004017080.198.6100.0普通牛BosTaurusNM_001015672.297.197.5牦牛BosgrunniensKM587693.197.297.2山羊CaprahircusKF684947.198.598.6黑猩猩PantroglodytesGABF01005134.188.092.5海象OdobenusrosmarusXM_004408337.288.388.5猕猴MacacamulattaNM_001261775.188.692.7人HomosapiensBC053326.162.690.8
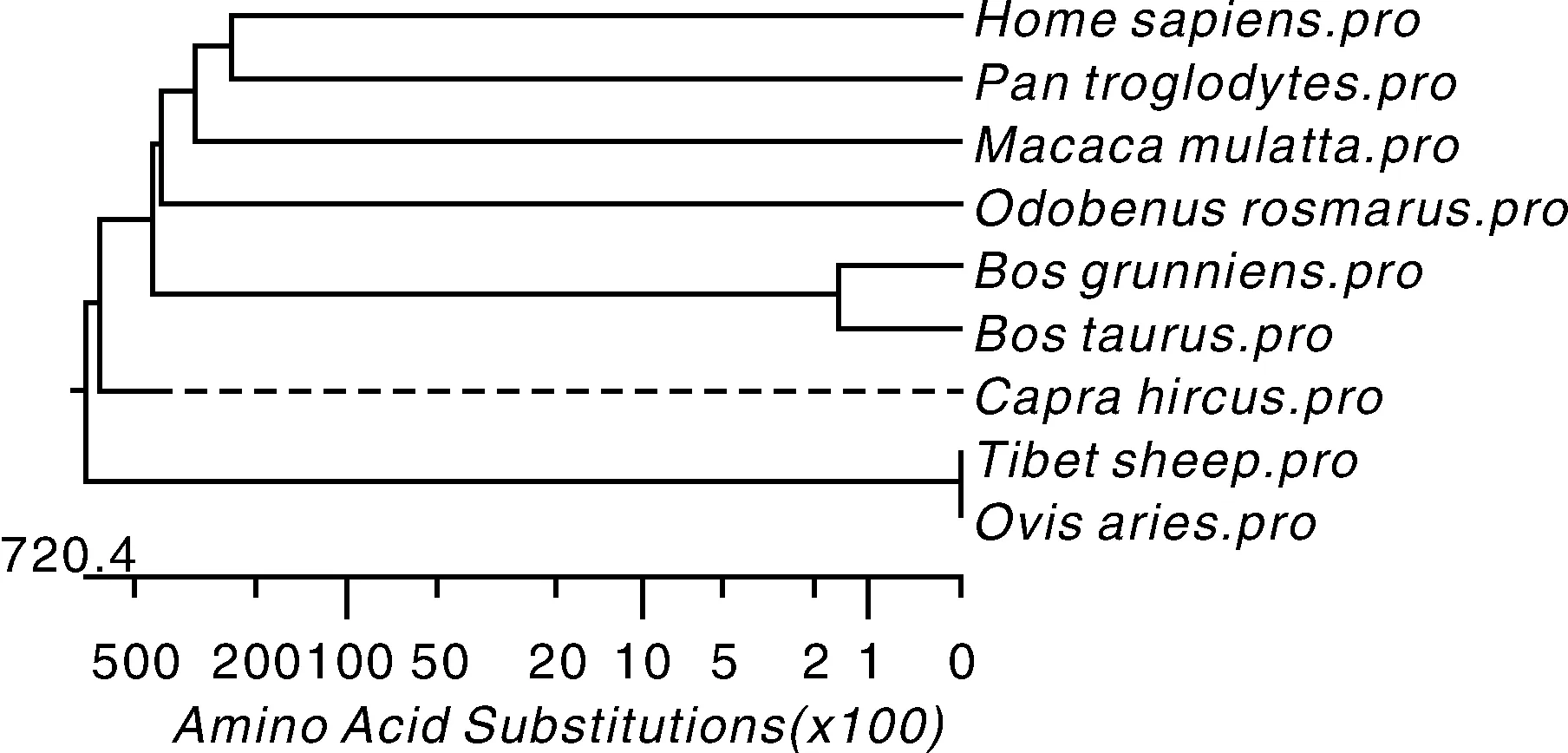
图5 FAM134B氨基酸序列系统进化树Fig.5 Phylogenetic tree of amino acid sequences of FAM134B

图6 FAM134B基因在不同组织中的表达Fig.6 Relative expression of FAM134B mRNA in different tissues
3 讨论
目前,国内外对FAM134B基因生理功能的研究相对较少,仅在人、小鼠、牦牛、黑山羊上有研究,绵羊FAM134B基因的核酸序列已有报道,但是在高原特色动物藏绵羊上没有报道。本试验采用RT-PCR获得了藏绵羊FAM134B基因的CDS区核苷酸序列和氨基酸序列,并对藏绵羊FAM134B不同组织基因的相对表达量进行了研究。用生物信息学方法预测和分析FAM134B基因编码产物,为更一步研究藏绵羊FAM134B基因的遗传特性奠定了理论基础。
蛋白质半衰期与其稳定性有关。贾浩等[24]的研究认为,一般来说,半衰期长则蛋白质稳定性高。本试验表明,FAM134B基因编码的蛋白具有较长的半衰期,但却属于不稳定蛋白。预测的FAM134B的二级结构是以无规卷曲和α-螺旋和延伸链为主,无规卷曲和α-螺旋各占一半,结构决定功能,推测这种结构可能会影响该蛋白在物质运输或离子通道中以及信号转导中的作用,具体功能有待后续试验验证。
藏绵羊FAM134B基因亚细胞定位和功能预测结果显示,此蛋白在运输与结合过程中发挥功能的可能性为77.3%,作为电压激活离子通道离子通道、和阳离子通道发挥功能的可能性分别为27.9%、16.9%、14.6%,这个结果和曾国敏等[5]研究的牦牛FAM134B基因功能完全一致,推测可能与牦牛亲缘关系较近有关,可推测此蛋白在物质运输方面起主要作用。
物种间亲缘关系的远近用相似性或者同源性来反映。藏绵羊与绵羊、山羊核酸序列一致性分别为98.6%,98.5%;氨基酸序列达到100%,与山羊达到98.6%,与绵羊氨基酸序列同源性为100%。上述结果与绵羊的动物分类学和氨基酸序列的系统发育得到的观点是一致的,说明藏绵羊FAM134B在生物进化中相对比较保守。
基因组织表达分析有助于研究转录活性[25],在研究不同组织分子调控时,需要考虑的一个重要因素是mRNA表达[26],藏绵羊FAM134B基因在各个组织中都有表达,说明FAM134B的转录有一定的组织特异性,这有可能与在不同组织中表达的功能相关。通过荧光定量PCR分析发现,藏绵羊FAM134B基因相对表达量从高到低依次是:网胃、回肠、盲肠、大脑枕叶、脑干、结肠、松果体、肝脏、直肠、脾脏、十二指肠。在网胃和回肠中表达量相对高于其他组织,在十二指肠和脾脏中表达量最低。网胃中表达量最高,而朱武政等[4]推测该基因可能与骨骼肌含量有关,肌肉中骨骼肌含量很高,肌肉组织是肌内脂肪沉积最重要的组织[27],网胃结构包括胃壁肌肉故推测可能控制脂肪沉积,这与本试验结果一致。大脑和小肠、肝脏表达量也很高,而脂质代谢主要在肝脏和小肠中进行,推测该基因也可能参与神经系统和脂质代谢的调控[28]。
由于FAM134B基因发现时间很短,故国内外有关该基因功能的报道很少。本试验用多种生物信息学软件对藏绵羊FAM134B编码产物进行预测和分析,为揭示FAM134B基因结构和生理功能及遗传特性奠定理论基础。
[1] KURTH I, PAMMINGER T, HENNINGS J C, et al. Mutations inFAM134B, encoding a newly identified Golgi protein, cause severe sensory and autonomic neuropathy[J].NatureGenetics, 2009, 41(11):1179-1181.
[2] TANG W K, CHUI C H, FATIMA S, et al. Oncogenic properties of a novel geneJK-1 located in chromosome 5p and its overexpression in human esophageal squamous cell carcinoma[J].InternationalJournalofMolecularMedicine, 2007, 19(6): 915- 923.
[3] 袁章琴,任阳,汪以真.猪新基因FAM134B的克隆及稳定干扰肌内前体脂肪细胞阳性克隆的筛选[J].中国畜牧杂志,2013, 49(9): 10-14. YUAN Z Q, REN Y, WANG Y Z. The new gene cloning of pig FAM134B and stable interference intramuscular preadipocytes positive clones screening[J].ChineseJournalofAnimalScience, 2013, 49(9):10-14. (in Chinese with English abstract)
[4] 朱武政,江明锋,唐璐,等.山羊FAM134B基因克隆、序列分析及其mRNA表达与肌内脂肪含量的相关性[J]. 农业生物技术报, 2014,22(10) :1277-1285. ZHU W Z, JIANG M F, TANG L, et al. Cloning, sequence analysis of FAM134B and correlation analysis between its mRNA expression and intramuscular fat content in goat (Caprahirus)[J].JournalofAgriculturalBiotechnology, 2014,22(10) :1277-1285. (in Chinese with English abstract)
[5] 曾国敏,杜晓华,杨阳,等.牦牛FAM134B基因CDS区克隆与生物信息学分析[J].华北农学报,2015,30(6):77-83. ZENG G M, DU X H, YANG Y, et al. Cloning and bioinformatics analysis on CDS ofFAM134Bgene in yak[J].ActaAgriculturaeBoreali-Sinica, 2015, 30(6):77-83. (in Chinese with English abstract)
[6] 季舒涵,昝林森,王洪宝,等.秦川牛A-FABP基因的生物信息学分析[J].西北农林科技大学学报(自然科学版), 2010, 38(6):77-81. JI S H, ZAN L S, WANG H B, et al. Biological information analysis ofA-FABPgene in Qinchuan cattle[J].JournalofNorthwestA&FUniversity(NaturalScienceEdition), 2010, 38(6):77-81. (in Chinese with English abstract)
[7] SAMBOOK J, FRITSCH E F, MANLATIS T. Molecular cloning: A laboratory manual[M]. 2nd ed. New York: Cold Spring Harbor Laboratory, 1989.
[8] 罗轶.鸡FATP1基因cDNA的克隆、组织表达及其生物信息学分析[D].雅安:四川农业大学, 2008. LUO Y. cDNA cloning, tissue expression and bioinformatic analysis of fatty acid transport protein 1 in chicken[D].Ya’an: Sichuan Agricultural University, 2008. (in Chinese with English abstract)
[9] 孙雪婧, 杜晓华, 杨孝朴, 等. 牦牛CYGB基因CDS区克隆与生物信息学分析[J].中国农业科学,2014,47(13):2690-2698. SUN X J, DU X H, YANG X P, et al. Cloning and bioinformatics analysis on CDS ofCYGBgene in yak[J].ScientiaAgriculturaSinica, 2014, 47(13):2690-2698. (in Chinese with English abstract)
[10] 李盛杰,杜晓华,罗玉柱,等.天祝白牦牛NGB基因的克隆及生物信息学分析[J]. 畜牧兽医学报, 2013, 44(3): 395-398. LI S J, DU X H, LUO Y Z, et al. Cloning and bioinformatics analysis ofNGBgene of Tianzhu white yak[J].ChineseJournalofAnimalandVeterinarySciences, 2013, 44(3): 395-398. (in Chinese with English abstract)
[11] 魏琳琳,高晋生,王晋霖,等. 绵羊LPIN1基因的克隆和 mRNA 表达研究[J].畜牧兽医学报, 2013, 44(9): 1371-1379. WEI L L, GAO J S, WANG J L, et al. Study on the cloning and ontogenetic mRNA expression of ovineLP1N1 gene[J].ActaVeterinariaetZootechnicaSinica, 2013, 44(9): 1371-1379. (in Chinese with English abstract)
[12] 陶士珩. 生物信息学[M]. 1版.北京:科学出版社, 2007: 177-179.
[13] 石宁宁,杜晓华,罗玉柱,等. 甘南牦牛NGB基因克隆及序列分析[J].西北农林科技大学学报(自然科学版), 2013, 41(4): 1-7. SHI N N, DU X H, LUO Y Z, et al. Cloning and sequence analysis ofNGBgene in Gannan yak[J].JournalofNorthwestA&FUniversity(NaturalScienceEdition), 2013, 41(4):1-7. (in Chinese with English abstract)
[14] 孙伟,李达, 苏锐,等.绵羊YAP1基因全长cDNA克隆及生物信息学分析[J].中国农业科学, 2013, 46(8): 1725-1735. SUN W, LI D, SU R, et al. Cloning and bioinformatics analysis of full-length cDNA sequence ofYAP1 gene in sheep[J].ScientiaAgriculturaSinica, 2013, 46(8): 1725-1735. (in Chinese with English abstract)
[15] JENSEN L J, GUTA R, BLOM N, et al. Prediction of human protein function from post-translational modifications and localization features[J].JournalofMolecularBiology, 2002, 319:1257-1265.
[16] 陈书霞,王晓武,房玉林.单菌落PCR法直接快速鉴定重组克隆[J].微生物学通报,2006, 33(3):52-56. CHEN S X, WANG X W, FANG Y L. Rapid characterization of recombination clone by PCR screening of individual bacterial colonies[J].Microbiology, 2006, 33(3):52-56. (in Chinese with English abstract)
[17] KOZAK M. Structural features in eukaryotic RNAs that modulate the initiation of translation[J].BiologicalChemistry, 1991, 266(30): 19867-19870.
[18] KYTE J, DOOLITTLE R F. A simple method for displaying the hydropathic character of a protein[J].JournalofMolecularBiology,1982,157(1):105-132.
[19] GURUPRASAD K, REDDY B V B, PANDIT M W. Correlation between stability of a protein and its dipeptide composition: a novel approach for predicting in vivo stability of a protein from its primary sequence[J].ProteinEngineering, 1990, 4(2): 155-161.
[20] KYTE J, DOOLITTLE R F. A simple method for displaying the hydropathic character of a protein[J].JournalofMolecularBiology, 1982, 157:105-132.
[21] 刘芳,格日力.藏羚羊组蛋白去乙酰化酶1基因编码区的克隆与序列分析[J].兽类学报,2011,31(4):396-340. LIU F, GE R L. Cloning and sequencing of HDAC1 coding cDNA sequence from Tibetan antelope (Pantholopshodgsonii)[J].ActaTheriologicaSinica, 2011, 31(4):396-340. (in Chinese with English abstract)
[22] 徐飞,成述儒,罗玉柱.绵羊DRB1 基因生物信息学分析[J].生物技术通报,2011(1):113-118. XU F, CHENG S R, LUO Y Z. Bioinformatics analysis of sheepDRB1 gene[J].BiotechnologyBulletin,2011(1):113-118. (in Chinese with English abstract)
[23] ROST B, SANDER C. Combining evolutionary information and neural networks to predict protein secondary strucure[J].Proteins,1994,19(1) :55 -72.
[24] 贾浩,张小白,宋晓峰.人类胞内蛋白半衰期与其亚细胞定位的相关性研究[J].计算机与应用化学,2011,28(4) : 411-414. JIA H, ZHANG X B, SONG X F. Relationship between intracellular protein half-life and subcellular localization in human cells[J].ComputersandAppliedChemistry,2011,28(4) : 411-414.(in Chinese with English abstract)
[25] BENNETT M K, SEO Y K, DATTA S, et al. Selective binding of sterol regulatory element- binding protein isoforms and co-regulatory proteins to promoters for lipid metabolic genes in liver[J].JournalofBiologicalChemistry, 2008, 283(23): 15628-15637.
[26] GRAUGNARD D E, PIANTONI P, BIONAZ M, et al. Adipogenic and energy metabolism gene networks in longissimus lumborum during rapid post-weaning growth in Angus and Angus × Simmental cattle fed high-starch or low-starch diets[J].BMCGenomics, 2009,10:142.
[27] DA COSTA A S, PIRES V M, FONTES C M, et al. Expression of genes controlling fat deposition in two genetically diverse beef cattle breeds fed high or low silage diets[J].BMCVeterinaryResearch, 2013, 9(1): 1-16.
[28] XU H, XU G, WANG D, et al. Molecular cloning and tissue distribution of the phosphotyrosine interaction domain containing 1(PID1) gene in Tianfu goat[J].Gene, 2013, 515(1):71-77.
(责任编辑张 韵)
MolecularcloningandexpressionanalysisofFAM134BgeneinTibetansheep
LI Wenli1,2, DU Xiaohua2,3, LIU Xia1,2,*, GAO Jingbo1,2, SHEN Chaochao1,2, ZHANG Chao1,2, LIU Wei1,2
(1.CollegeofLifeScienceandTechnology,GansuAgriculturalUniversity,Lanzhou730070,China; 2.GansuKeyLaboratoryofHerbivorousAnimalBiotechnology,Lanzhou730070,China; 3.CollegeofVeterinaryMedicine,GansuAgriculturalUniversity,Lanzhou730070,China)
The gene ofFAM134B(family with sequence similarity 134, member B) was cloned by RT-PCR. The bioinformatics analysis was carried out by using biological software. The expression level ofFAM134Bwas also studied by real-time fluorescence quantitative PCR. The full length ofFAM134Bgene was 1 144 bp (GenBank accession number: KX580309), open reading frame 1 071 bp, encoding 356 amino acids. The phylogenetic tree showed that the Tibetan sheepFAM134Bhad a close relationship with the sheep and goat. The molecular formula of FAM134B protein in Tibetan sheep was C1717H2696N448O572S12, its molecular weight was 39151.72 u, the theory isoelectric point was 4.49, the extinction coefficient was 24 450. The instability index was 43, the aliphatic index was 80.31, and the grand average hydropathicity was -0.422. It was unstable and soluble acidic protein. The secondary structure of FAM134B was mainly composed of α-helices and random coil and extended chain, with α-helices was 45.51%, random coil was 41.85% and extension chains was 12.64%, which belongs to mixed type of protein. The results of subcellular localization and function showed that the frequencies of FAM134B protein in endoplasmic reticulum and cytoplasmic membrane were 30.4% and 21.7%. It mainly plays a role in protein transport and binding(77.3%). Phylogenetic tree showed that the amino acid sequence of FAM134B showed high homology with sheep and goat. qRT-PCR results showed that the expression ofFAM134Bgene was the highest in the stomach and lowest in the duodenum. The results can provide a theoretical basis for further study on the structure and physiological functions and genetic characteristics ofFAM134Bgene.
Tibetan sheep;FAM134Bgene; CDS cloning; bioinformatics; expression
S826
:A
:1004-1524(2017)09-1474-08
李文丽,杜晓华,刘霞,等. 藏绵羊FAM134B基因克隆与表达分析[J].浙江农业学报,2017,29(9): 1474-1481.
10.3969/j.issn.1004-1524.2017.09.08
2016-12-26
农业部畜禽遗传资源与种质创新重点实验室开放课题资助(nzdsys2015-1)
李文丽(1991—),女,甘肃会宁人,硕士研究生,从事生物化学与分子生物学研究工作。E-mail: 1406523498@qq.com
*通信作者,刘霞,E-mail:liux@gsau.edu.cn
