基于DNA条形码技术鉴别5种列当属药用植物△
2017-09-21韩奇亨汤健王晓琴李旻辉
韩奇亨,汤健,王晓琴,李旻辉,3,4,5*
(1.包头医学院,内蒙古 包头 014060;2.内蒙古医科大学 药学院,内蒙古 呼和浩特 010110;3.内蒙古自治区中医药研究所,内蒙古 呼和浩特 010110;4.内蒙古自治区特色药用植物培育与保护工程技术研究中心,内蒙古 包头 014060;5.内蒙古自治区特色道地药材资源保护与利用重点实验室,内蒙古 包头 014060)
基于DNA条形码技术鉴别5种列当属药用植物△
韩奇亨1,汤健1,王晓琴2*,李旻辉1,3,4,5*
(1.包头医学院,内蒙古 包头 014060;2.内蒙古医科大学 药学院,内蒙古 呼和浩特 010110;3.内蒙古自治区中医药研究所,内蒙古 呼和浩特 010110;4.内蒙古自治区特色药用植物培育与保护工程技术研究中心,内蒙古 包头 014060;5.内蒙古自治区特色道地药材资源保护与利用重点实验室,内蒙古 包头 014060)
目的:建立一种快速、准确、标准化的5种列当属药用植物的DNA条形码鉴别方法。方法:采集5种列当属药用植物,所有样品进行总DNA提取,PCR扩增,并对扩增产物进行测序得到相应的序列,测序结果使用CONTIG、BioEdit、MEGA6.0软件进行分析,构建系统发育树。结果:测得5种列当属植物的ITS、rbcL、trnL-F和matK全长序列共200条,4种序列长度范围在364~1240 bp,其中ITS、trnL-F和matK可将其鉴别出来。结论:rbcL序列无法有效鉴别5种列当属药用植物,trnL-F与ITS序列能够成功鉴别,可作为列当属植物的分子鉴别方法。
分子鉴别;DNA 条形码;列当属;
分子标记方法起源于1987年[1],具有灵敏度高、准确性好、客观性强等优势,但分子标记法在重复性和稳定性上并不尽如人意[2]。此外,由于其操作的复杂程度,也限制了分子标记法的推广应用。DNA条形码概念于2003年被加拿大Guelph 大学Hebert 等人首次正式提出[3],因其具有对物种进行快速、准确鉴别的特点而备受外界关注。使用核基因序列与叶绿体基因序列搭配对植物进行鉴别的方法因可靠性较高被国内外广泛采用。
列当属植物在我国共分布有25 种,为多年生、二年生或一年生肉质寄生草本,其中16种列当属植物的寄主有明确记载,多为蒿属植物。列当属植物多生于沙丘、山坡及沟边草地上,生长环境分布在海拔850~4000 m。列当属主要活性成分为苯乙醇苷类和多糖,其具有抗疲劳,改善脑组织循环,增强免疫力的药理作用,在传统医学中主要被用作强筋壮骨,补肾助阳,常被作为肉苁蓉的代用品。25 种列当属植物中有6 种在蒙、藏、傈僳、彝的民族医药中作为民族药应用,占总数的24%[4-6]。
列当(紫花列当)Orobanchecoerulescens、黄花列当O.pycnostachya、弯管列当O.cernua、毛药列当O.ombrochares属于列当属列当组,常作为蒙药使用。列当(紫花列当)、黄花列当、弯管列当、毛药列当以全草入药,由于这几种列当经炮制切片后外形、颜色十分相近,且显微鉴别也较难区分,故本实验尝试采用DNA条形码技术进行鉴别[6-8]。实验中选用4种常用DNA条形码引物序列对列当(紫花列当),黄花列当,弯管列当、毛药列当和分枝列当O.aegyptiaca进行分子鉴别,用以比较不同候选片段对此5种植物的鉴别能力,其中分枝列当属于列当属小苞组本,用于考察这4种序列对列当属属内组间的鉴别能力,从而筛选可鉴别该属植物的优选DNA条形码序列,为该属植物的分子鉴别提供参考依据。
1 材料与方法
1.1 材料
本实验选用的5种列当属植物采自内蒙古自治区阿拉善盟、巴彦淖尔市,其中列当(紫花列当)13株,黄花列当4株,弯管列当29株,分枝列当3株,毛药列当1株,具体样品信息见表1。样品由内蒙古医科大学王晓琴教授采集并鉴定,凭据标本保存于内蒙古医科大学生药教研室。
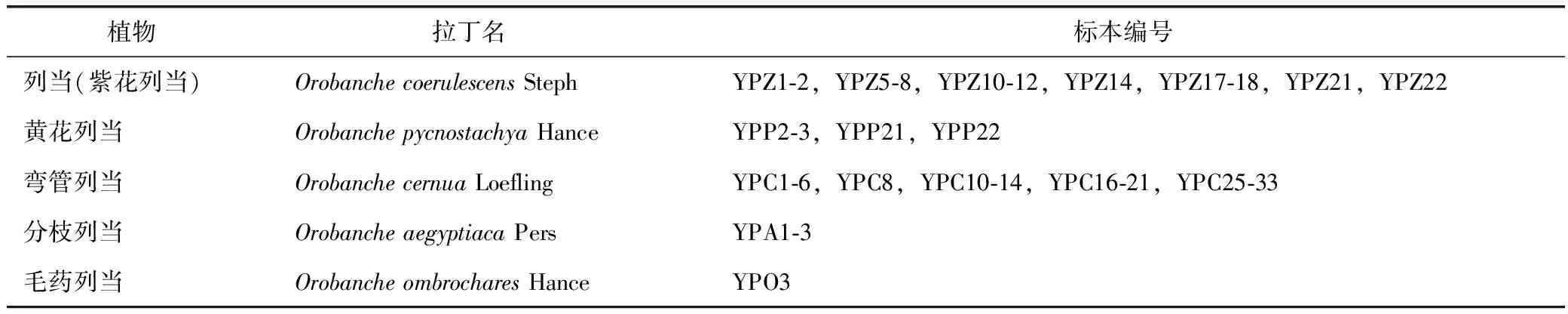
表1 材料信息表
1.2 试剂
DP305-02植物基因组DNA提取试剂盒(天根公司),1×TAE缓冲液、琼脂糖(Promega公司),2× Taq DNA Master Mix (Takara公司),2 000 bp DNA Markar (Takara公司),巯基乙醇(Amresco公司),核酸染料(赛百盛公司),三氯甲烷﹑无水乙醇﹑异丙醇均为国产分析纯。
1.3 仪器
Arktik-5020型PCR仪(美国Thermo),LABOFUGE 400R型低温冷冻干燥仪(美国Thermo),5000 plus型紫外凝胶成像分析仪(赛智ChampGel),掌上离心机(德国IKA miniG),手持移液器(德国Eppendorf),MM400型混合型球磨仪(德国Retsch)。
2 方法
2.1 总DNA 的提取
取适量(约100 mg)的植物样品干燥的茎,液氮研磨成细粉后,使用试剂盒法提取,获得总DNA,于-20 ℃保存备用。
2.2 引物设计
选择目前中药鉴别中常用的通用引物[9],详情见表2。
2.3 DNA 扩增
4种引物片段PCR扩增体系为40.0 μL,包含:2×Taq DNA Master Mix(Takara 公司)20.0 μL,正向、反向引物各1.0 μL,DNA模板1.0 μL,ddH2O 17 μL。

表2 引物信息
反应条件:
①ITS (1F/4R)片段
预变性温度:94 ℃,5 min;变性温度:94 ℃,1 min;退火条件:45 ℃,45 s;复性条件:72 ℃,1 min;35个循环;延伸条件72 ℃,7 min,最后4 ℃保存。
②rbcL (1F/724R)片段:
预变性条件:94 ℃,4 min;变性温度:94 ℃,30 s;退火条件:52 ℃,45 s;复性条件:72 ℃,1 min;35个循环;延伸条件72 ℃,7 min;最后4 ℃保存。
③trnL-F(trnL-F c/trnL-F f)片段:
预变性条件:94 ℃,5 min;变性温度:94 ℃,1 min;退火条件:50 ℃,45 s;复性条件:72 ℃,1 min;35个循环;延伸条件72 ℃,7 min;最后4 ℃保存。
④matK(AF/8R)片段:
预变性条件:94 ℃,1 min;变性温度:94 ℃,50 s;退火条件:50 ℃,45 s;复性条件:72 ℃,1 min;35个循环;延伸条件72 ℃,7 min;最后4 ℃保存。
扩增结束后使用1%琼脂糖凝胶电泳进行检测,并使用凝胶成像系统分析。
2.4 测序
经过PCR 扩增后,PCR 原产物由生工生物工程(上海)股份有限公司测序部北京分部测序,各样品均采用正向和反向测序,以保证测序的准确性。
2.5 序列分析
将5种列当药材分别进行rbcL,ITS,trnL-F,matK 4个序列的比对分析。用CONTIG软件将DNA序列排序后,应用BioEdit 分析软件进行分析处理,结合Mega 5.0分析软件中的NJ(Neighbor-joining)工具构建系统进化树,并以Bootstrap 作1000次可信度分析。
3 结果
3.1 评价4种DNA条形码序列
从5个不同物种中获得rbcL、ITS、trnL-F、matK序列各50条,共200条。引物通用性﹑PCR 扩增成功率以及序列扩增成功率,皆100%(表3),将获得的序列进行CONTIG软件和BioEdit分析软件对比分析,发现trnL-F变异位点228个,信息位点118个;rbcL变异位点47个,信息位点30个;matK变异位点279个,信息位点159个;ITS变异位点171个,信息位点146个。
3.2 5种列当属植物经典分类学的探讨
通过利用Mega 6.0软件,以NJ(Neighbor-joining)法构建系统进化树,从系统进化树(图1、图2、图3、图4)及序列分析可知,4种片段中所选择的ITS、trnL-F与matK片段效果较好,能够鉴别出5种列当属植物。而rbcL的鉴别能力相对较差,无法将5种列当属植物鉴别开来。
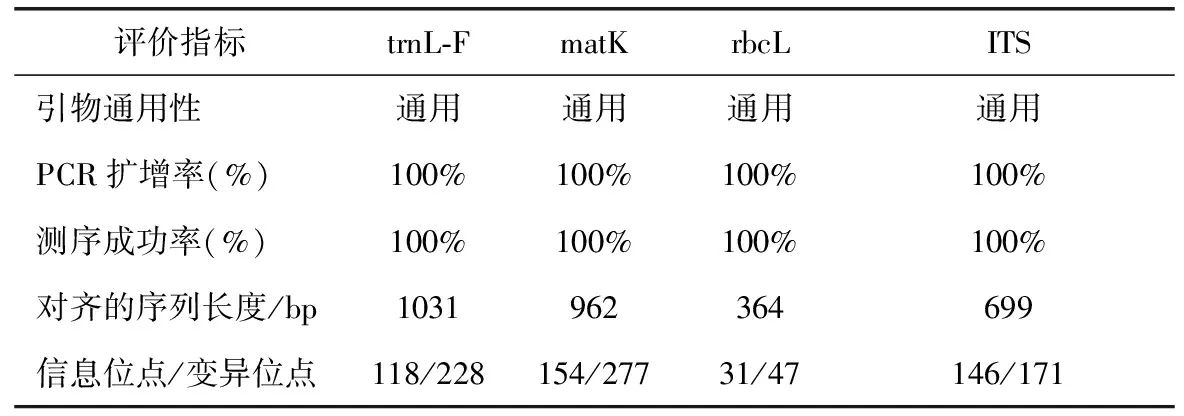
表3 评价4种DNA通用引物
3.3 trnL-F序列数据分析
通过对trnL-F序列进行分析对比之后,本实验发现248个变异位点,197个信息位点,通过这些信息位点的变化可以将这5种列当科植物很好的鉴别出来(表4),比如弯管列当与其他4种有两个独特的位点(17 bp:A或48 bp:A),都分别可看作一个特征位点。其他种的特征位点有:黄花列当(53 bp:T或556 bp:A),分枝列当(203 bp:G或205 bp:T),毛药列当(307 bp:T,447 bp:G或504 bp:G),列当(紫花列当)(447 bp:T)。
3.4 matK序列数据分析
通过对matK序列进行分析对比以后,本试验发现变异位点236个,信息位点147个,通过这些信息位点的变化可以将这5种列当科植物较好的鉴别出来(表5),比如毛药列当(19 bp:C或63 bp:G),分枝列当(20 bp:G,32 bp:A,88 bp:A,350 bp:G或398 bp:T),弯管列当(204 bp:A,371 bp:T或689 bp:C),列当(紫花列当)(350 bp:C或398 bp:A),黄花列当(914 bp:G)。此外在所获得序列的同一位点中,不同种列当碱基变异可能在2种以上,如350 bp,389 bp,914 bp等处。
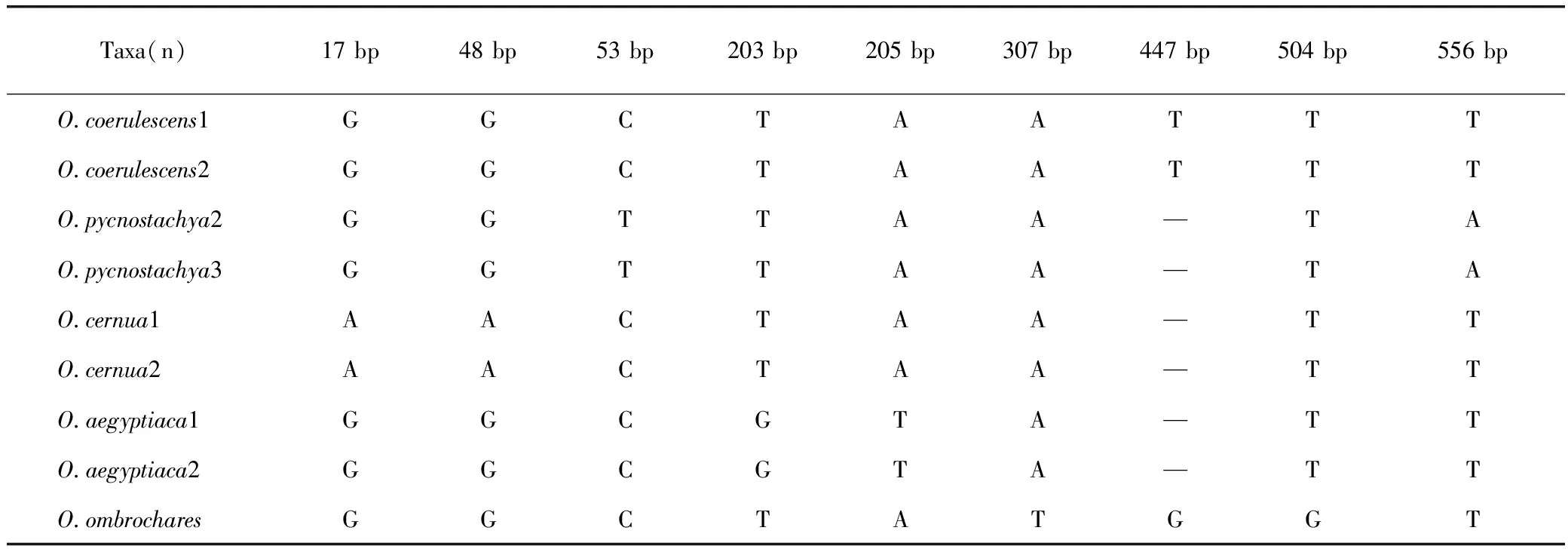
表4 5种列当属植物trnL-F序列信息位点
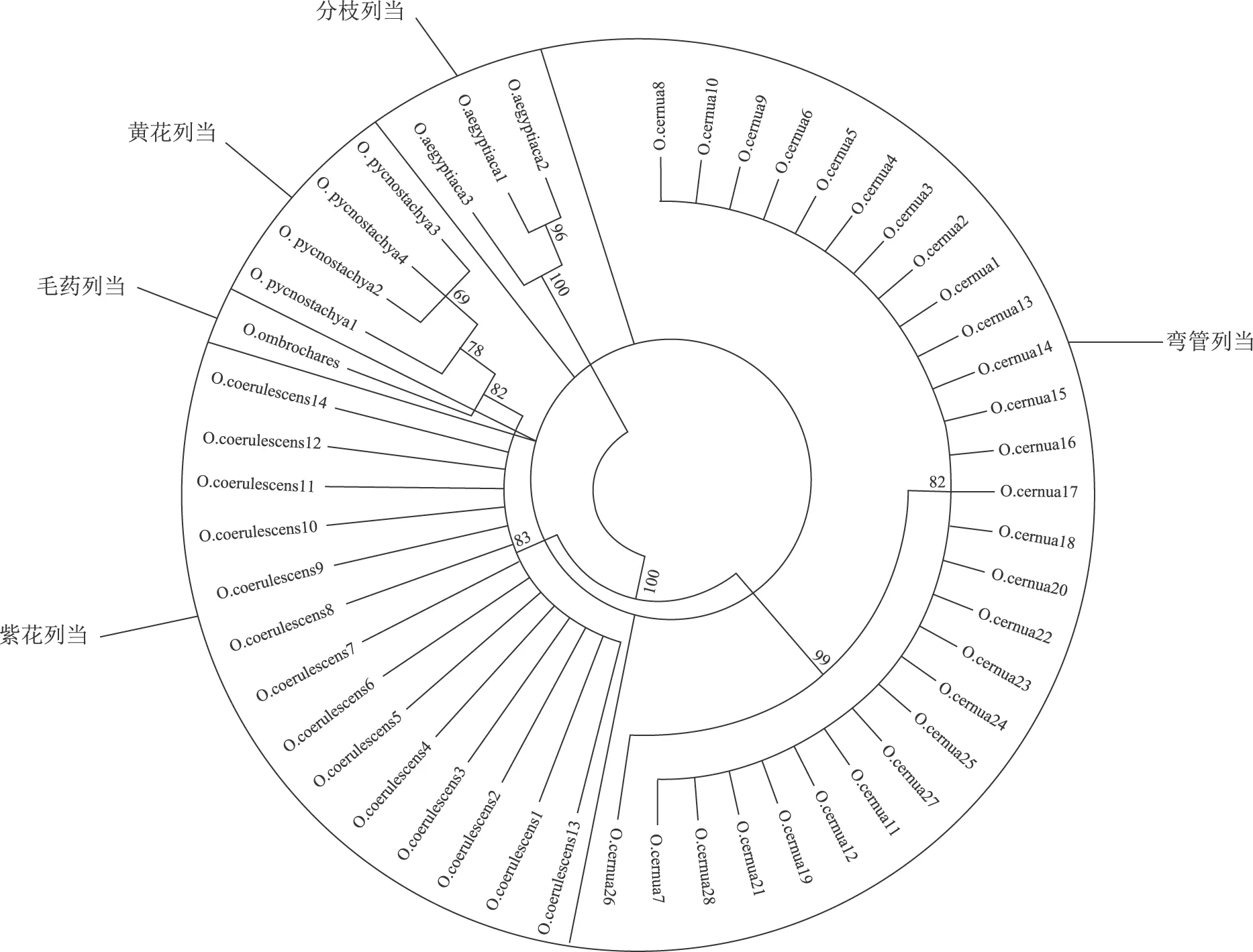
图1 基于trnL-F 序列的5 种列当科植物NJ 系统进化树
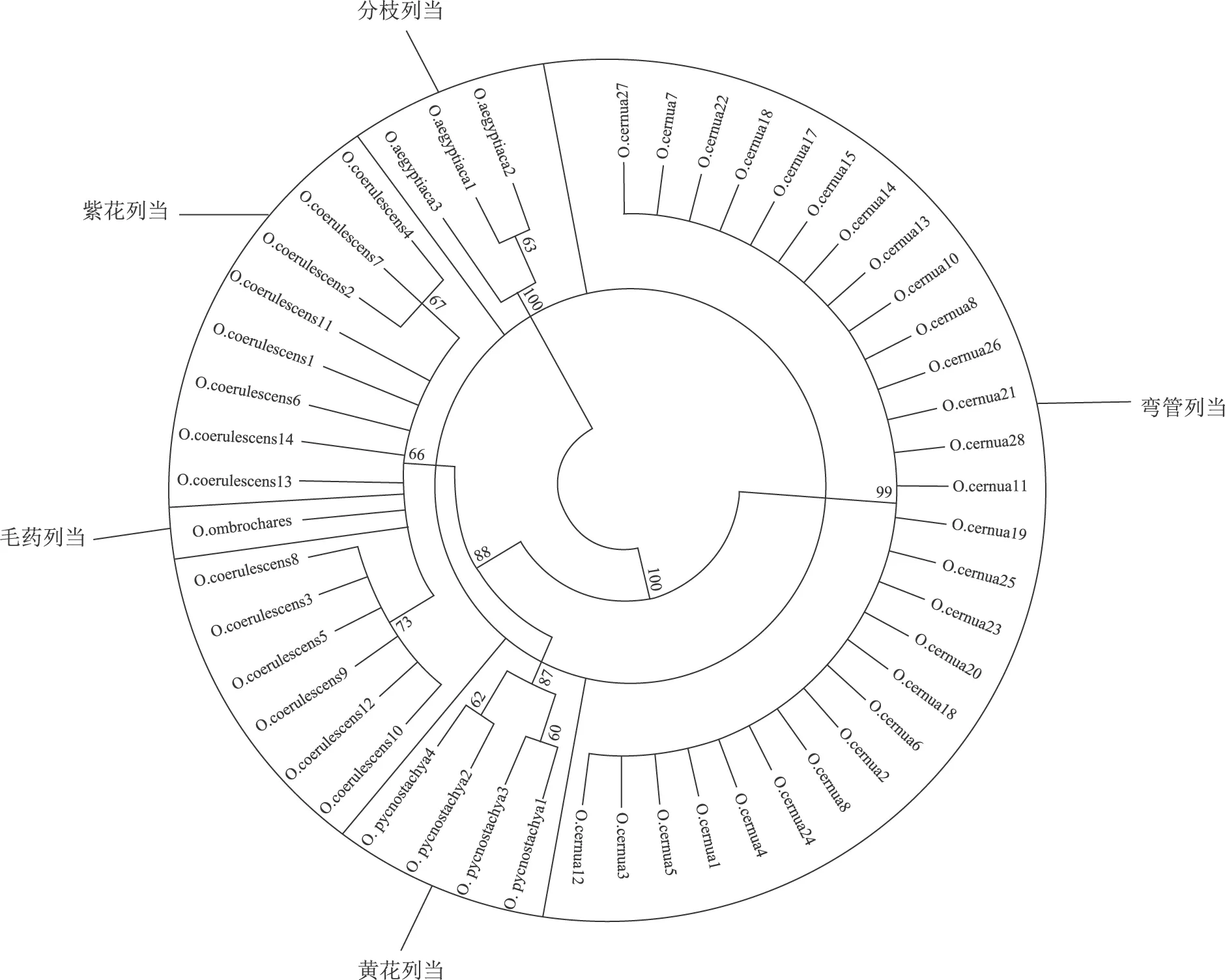
图2 基于matK 序列的5 种列当科植物NJ 系统进化树
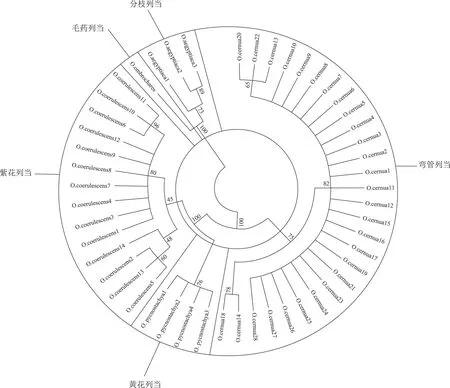
图3 基于ITS 序列的5 种列当科植物NJ 系统进化树
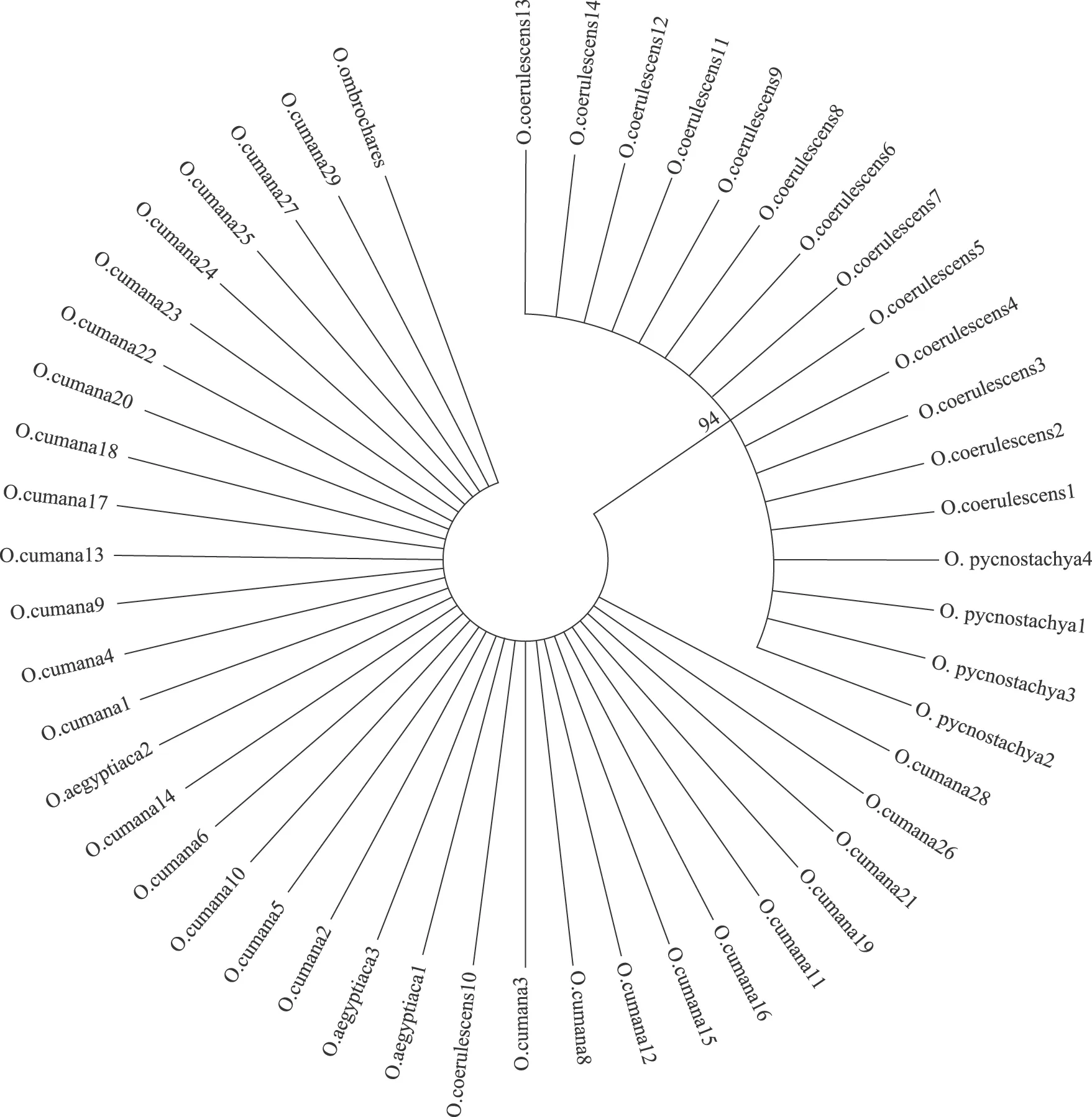
图4 基于rbcL 序列的5 种列当科植物NJ 系统进化树

表5 5种列当属植物matK序列信息位点
3.5 ITS序列数据分析
通过对ITS序列进行分析对比之后,共现171个变异位点,146个信息位点,通过这些信息位点的变化可以将这5种列当科植物很好的鉴别出来(表6),比如分枝列当(46 bp:T或311 bp:A),黄花列当(184 bp:T或299 bp:T),列当(紫花列当)(237 bp:G或451 bp:T),毛药列当(390 bp:C或396 bp:A),弯管列当(470 bp:G或595 bp:A)。

表6 5种列当属植物ITS序列信息位点
4 讨论
综合上述实验及数据处理结果可知,trnL-F、matK序列和ITS序列都可将本实验的物种鉴别出来。 rbcL序列在实验的扩增、测序环节成功率高,但通过CONTIG软件和BioEdit软件分析,使用Mega 6.0软件建树后发现,5种列当属植物的rbcL序列变异位点少,信息位点仅31个,且缺乏区分5种植物的特征位点,所构建的NJ树也显示,rbcL序列无法将5种列当植物区分出来,因此不适合此5种列当属药材鉴别。对于ITS序列和trnL-F、matK序列,trnL-F序列特征位点最多,其次为ITS序列,这两种序列鉴别能力最强,而matK序列区分毛药列当与列当(紫花列当)能力较trnL-F序列、ITS序列差。从各类DNA条形码技术的报道和相关文献来看,rbcL 序列适合用于科级的鉴别,matK 序列则通常用于鉴别属级的植物,而ITS 序列和trnL-F 序列更适合鉴别种级的不同个体[10],本实验的结果显然印证了此类观点。
对于matK序列,一般认为其通用性相对较差,就本实验结果,matK序列可考虑与其他序列进行搭配,如本实验中的ITS序列、trnL-F序列,从而增加对此5种列当属植物鉴别的准确性。对于ITS序列,本实验虽然选择了ITS1和ITS4组合作为引物,但在实验的初期,选用的引物组合为常用的ITS4与ITS5,经过反复的实验,发现ITS4与ITS5通用引物组合的PCR成功率较低,故而改变ITS序列的组合,选用了的ITS1与ITS4组合,PCR效果较好。由于此组合多被用于真菌类生物的PCR实验[11-12],故实验易受真菌干扰,对实验材料的选择及实验操作有一定要求。但ITS序列的鉴别能力突出,可被用作鉴别的特征位点丰富,能很好鉴别目标植物,且在不同生境中分布的种内植物间,特征位点保守且稳定,而在种间却变异较大[13],故可作为此5种植物最理想的鉴别片段之一。对于trnL-F序列,本实验中选取的5种列当属植物之间差异大,碱基缺失、变异和可用作于鉴别的特征信息位点丰富,实验要求相对较低,因此也可作为此5种植物的理想鉴别片段。
随着我国民族医药的发展以及保健品市场的快速壮大,列当属药用植物的使用越来越多,越来越广泛,但市场上列当属植物的药材鱼龙混杂,以次充好的情况时有发生。到目前为止,对其鉴别一直沿用经典的性状鉴别、显微鉴别及理化鉴别方法。本次实验的结果表明:DNA条形码技术这一高效、准确的分子鉴别方法可在分子水平上解决这些问题,其实用性强、准确度高的特点可在中药以及民族药的鉴别和替代品的规范与发掘等方面发挥重要的作用,在药材的正本清源工作中提供有力的技术保障,并将药材的鉴别领入一个新的阶段。
[1] 黄映萍.DNA分子标记研究进展[J].中山大学研究生学刊(自然科学.医学版),2010,(2):27-36.
[2] 何希荣.中药材的鉴别[J].中国实验方剂学杂志,2008,14(4):41.
[3] 程希婷,王爱民,顾志峰.DNA条形码研究进展[J].基因组学与应用生物学,2011,30(6):748-758.
[4] 中国科学院中国植物志编辑委员会.中国植物志:第69卷[M].北京:科学出版社,1999[5].许国青,王晓琴,文迪.列当科药用植物研究进展[J].中国民族医药杂志,2012,10(18):35-38.
[6] 白贞芳,刘勇,王晓琴.列当属、肉苁蓉属和草苁蓉属植物传统药物学调查[J].中国中药杂志,2014,39(23):4548-4552.
[7] 周谧,张继.黄花列当的生药学研究[J].药物分析杂志,2007,27(11):1719-1721.
[8] 杨来秀,王素巍,王晓琴,等.列当的生药鉴别[J].华西药学杂志,2015,30(1):54-55.
[9] 陈士林,姚辉,宋经元,等.基于DNA barcoding(条形码)技术的中药材鉴定[J].世界科学技术—中医药现代化,2007,9(3):7-12.
[10]闫化学,于杰.DNA条形码技术在植物中的研究现状[J].植物学报,2010,45(1):102-108.
[11]曹永军,程萍,喻国辉.利用ITS1和ITS4通用引物扩增香蕉枯萎病菌核酸片段鉴定其生理小种[J].热带作物学报,2010,31(7):1098-1102.
[12]郭鹏豪,刘秀丽,崔颖鹏.真菌通用引物Its1和Its4在丝状真菌鉴定中的价值评价[J].中国微生态学杂志,2013,25(8):922-924.
[13]刘建全,陈之端,廖志新.“藏茵陈”原植物及其混淆种类的ITS序列比较[J].药学学报,2001,36(1):67-70.
StudyonIdentificationofFiveSpeciesofGenusOrobanchewithDNABarcodingTechnology
HAN Qiheng1,TANGJian1,WANGXiaoqin2*,LIMinhui1,3,4,5*
(1.BaotouMedicalCollege,Baotou014060,China;2.SchoolofPharmacy,InnerMongoliaMedicalUniversity,Hohhot010110,China;3.InnerMongoliaAutonomousRegionAcademyofChineseMedicine,Hohhot010110,China;4.InnerMongoliaResearchCenterofCharacteristicMedicinalPlantsCultivationandProtectionEngineeringTechnology,Baotou014060,China;5.InnerMongoliaKeyLaboratoryofCharacteristicTraditionalChineseMedicinalResourcesProtectionandutilize,Baotou014060,China)
Objective:To establish a rapid,accurate and standardized method for identifying five species from genusOrobanchewith DNA barcoding technology.Methods:Total DNA of 5 kinds of medicinal plants from genusOrobanchewere extracted.ITS,rbcL,trnL-F and matK gene sequence amplified by PCR.PCR product was directly sequenced,and the results were aligned and analyzed with different software (CONTIG、BioEdit、MEGA6.0) to construct the phylogeny trees.Results:The 200 complete ITS,rbcL,trnL-F and matK sequences of 5 species from genusOrobanchehave been obtained,and the length of 4 sequences region varied from 364 to 1240 bp.They were distinguished by trnL-F,ITS or matK sequence.Conclusion:RbcL sequence was not suitable to identify 5 species from the genusOrobanche,but ITS and trnL-F region could be a pretty good marker to distinguish these species,and it will be a potential molecular identification method for identifyingOrobancheplants.
Molecular identification;DNA barcoding;OrobancheL.
国家自然科学基金项目(81260615)
] 王晓琴,教授,研究方向:中药资源学及生药学;E-mail:nywangxiaoqin@163.com; 李旻辉,教授,研究方向:中蒙药资源保护与开发利用、分子生药学;Tel:(0471)6262232,E-mail:li_minhui@aliyun.com
10.13313/j.issn.1673-4890.2017.7.004
2016-10-08)
*[
