饲喂发酵棉粕肉牛牛粪中细菌数量及多样性研究
2017-09-06梁敏张文举张凡凡陈宁尹君亮
梁敏,张文举,张凡凡,陈宁,尹君亮
(1.石河子大学动物科技学院,新疆石河子 832003;2.新疆农垦科学院畜牧兽医研究所,新疆石河子 832000)
饲喂发酵棉粕肉牛牛粪中细菌数量及多样性研究
梁敏1,张文举1,张凡凡1,陈宁2,尹君亮2
(1.石河子大学动物科技学院,新疆石河子 832003;2.新疆农垦科学院畜牧兽医研究所,新疆石河子 832000)
【目的】研究饲喂发酵棉粕对安格斯肉牛牛粪中细菌数量及多样性的影响。【方法】采用平板计数法计算细菌的数量;采用PCR技术对提出的DNA进行细菌16Sr DNA基因V6-V8区扩增,将PCR产物进行变性梯度凝胶电泳(DGGE)。得到指纹图谱特异性条带切胶回收并进行克隆测序,通过BLAST鉴定细菌菌种。【结果】(1)试验前期,C组和T组总细菌、大肠杆菌和乳酸菌差异显著(P<0.05);C组和T组金黄色葡萄球菌差异不显著(P>0.05)。试验后期,C组和T组总细菌差异不显著(P>0.05);C和T组大肠杆菌、金黄色葡萄球菌和乳酸菌差异显著(P<0.05)。(2)牛粪中存在的菌种有沙门氏菌(Salmonella)、瘤胃球菌(Ruminococcus)、乳酸菌(Lactobacillus)、毛螺旋菌(Lachnospiraceae)、志贺菌(Shigella)、梭菌(Clostridium)、大肠杆菌(Escherichiacoli)、肠杆菌(Enterobacter)和肠球菌(Enterococcus),而其中乳酸菌、大肠杆菌可能是牛粪中的优势菌群。【结论】试验前期和后期总细菌、大肠杆菌、金黄色葡萄菌的数量有明显变化,牛粪中存在多种细菌表现了牛粪中微生物菌群的多样性。
发酵棉粕;细菌;数量;DGGE;多样性
0 引 言
【研究意义】蛋白质是维持生命机体所必需的营养物质。畜牧业中常采用谷物、饼粕、草类等原料配制动物日粮来满足动物对蛋白质的需求[1-2]。目前随着畜牧业生产的发展,蛋白饲料紧缺问题逐渐凸显,原有常规饲料亦然不能满足目前快速发展的需求,所以如何合理开发和利用其它非常规蛋白质饲料,对我国畜牧业的发展起到至关重要的作用。棉籽因其产量高、蛋白含量丰富、代谢能高等,因此开发其副产物不仅可减少资源浪费,且为有效解决我国饲料紧缺问题提供了可能[3-4]。【前人研究进展】棉粕经过发酵后,其表面附着的微生物利用底物大量繁殖,不仅提高了自身菌体蛋白的含量,且有益于家畜的有效利用[5]。然而动物肠道中寄生着种类繁多和数量巨大的微生物。在正常情况下,互相之间以稳定的比例关系与宿主肠道相互依存又相互影响,形成动态的微生态平衡。而饲料源是否发酵可能会影响家畜肠道菌群的稳定性,从而影响家畜的吸收利用水平[6-8]。嗜酸乳酸杆菌能够调整肠道菌群平衡,其分泌的抗生物素类物质嗜酸乳菌素、嗜酸杆菌素、乳酸菌素,对肠道致病菌产生的拮抗作用,抑制肠道不良微生物的增殖。枯草芽孢杆菌能促进有益厌氧菌生长,并产生乳酸等有机酸类,降低肠道pH值,间接抑制其它致病菌生长。提高免疫球蛋白和抗体水平,增强细胞免疫和体液免疫功能。其合成的α-淀粉酶、蛋白酶、脂肪酶、纤维素酶等酶类,在消化道中与动物体内的消化酶类一同发挥作用[9]。刘忠元[10]用热带假丝酵母发酵玉米秸秆粉,结果发现玉米秸秆粉中的粗纤维含量下降,粗蛋白含量提高,可以提高秸秆的饲用价值。【本研究切入点】嗜酸乳酸杆菌、枯草芽孢杆菌和热带假丝酵母均可以作为发酵棉粕的微生物制剂,但是三种菌混合发酵的棉粕对于肉牛的作用还需要进一步的研究。而且目前发酵棉粕在肉牛上的应用还比较欠缺,有关发酵棉粕日粮对肉牛肠道微生物影响方面的报道也比较少[11-12],应该引起足够重视。肠道正常菌群与肠道免疫系统的形成和功能、肠上皮细胞的生长有密切关系,并可以阻挡病原微生物的入侵[13-14]。据研究报道,粪中微生物与结肠微生物间具有99%的相似性[15],因此可利用粪样来反映肠道微生物的菌群情况。传统微生物群落结构多采取分离鉴定的方法进行研究,而对于复杂体系且不能分离培养的微生物很难进行群落研究。采用PCR和变性梯度凝胶电泳(DGGE)分子指纹图谱技术对于难以分离的菌种可以进行快速有效的检测[16-18]。【拟解决的关键问题】对发酵棉粕饲喂肉牛粪样中的主要细菌含量进行研究,并采用PCR-DGGE技术探讨其粪样中细菌多样性,以期揭示发酵棉粕饲喂对肉牛肠道微生物菌群数量和多样性特征规律,为健康的肉牛养殖技术提供相关理论基础。
1 材料与方法
1.1 材 料
新疆西部波尔多牧业有限责任公司选取10月龄左右,健康无疾病,初始体重差异不显著(259.11±35.62) kg的黑安格斯肉牛18头,随机分成两组(每组9头),即试验(Treatment, T)和对照(Control, C)处理。试验期从2016年10月8至11月18日,共42 d,预饲期10 d,正饲期32 d。T处理添加6 %的发酵棉粕,C处理添加6 %的未发酵棉粕。发酵棉粕和未发酵棉粕购于新疆天康饲料添加剂公司生产的成品。发酵使用的菌种是嗜酸乳杆菌(Lactobacillusacidophilus)、枯草芽孢杆菌(Bacillussubtilis)以及热带假丝酵母菌(Candidatropicalis)。列出2个处理与精料混合再与基础日粮制成TMR,混匀后饲喂,每天分别于06:00和18:00饲喂,任其自由采食,自由饮水。牛舍温度控制与室外气温相同(10℃),自然光照。表1,表2

表1 发酵棉粕营养水平

表2 基础日粮组成及营养水平
注:1、精料成分为玉米51%、麸皮24%、豆粕18%、尿素1.5%、食盐1%、碳酸氢钙2.5%、添加剂2%;2、除代谢能为计算值外其余指标为实测值
Note: 1. Feed ingredients corn 51%, wheat bran 24%, soybean meal 18%, urea 1.5%, salt 1%, calcium hydrogen carbonate 2.5%, additive 2%. In addition to the metabolic energy for the calculation of the remaining indicators for the measured value
1.2 方 法
1.2.1 样品采集
正饲期的第16 d(试验前期)和第32 d(试验后期),分别在肉牛的直肠取粪样5 g左右,置于灭菌的螺口管中,每头肉牛的粪样采取3份,迅速放入液氮保存,用于细菌DNA的提取和平板计数使用。
1.2.2 细菌平板培养
采用传统平板计数法进行计数[19],其中总细菌采用NA培养基,好氧培养,培养温度37℃;大肠杆菌采用LB培养基,好氧培养,培养温度37℃;金黄色葡萄球菌采用BP培养基,好氧培养,培养温度37℃;乳酸菌采用MER培养基,好氧培养,培养温度37℃。在恒温箱培养2~3 d的时间,即可进行菌落计数。其中梯度稀释总细菌和大肠杆菌选择10-5、10-6、10-7三个稀释梯度;其中梯度稀释金黄色葡萄球菌和乳酸菌选择10-5、10-6、10-7和10-8四个稀释梯度。计数结果10为底数的对数表示。
1.2.3 样品总细菌DNA提取
从灭菌的螺口管中称取0.5 mL的粪便样本至2 mL离心管中,并置于冰上。粪样中细菌总DNA的提取方法按照提取试剂盒(TIANamp Stool DNA Kit)说明进行提取。
1.2.4 细菌16Sr DNA 基因V6-V8区PCR扩增
选用细菌通用引物对细菌的16Sr DNA V6-V8区域进行PCR扩增,扩增体系为50 μL:2 μL模板DNA,2 μL上游引物(U968-GC),2 μL下游引物(L1401),PCR Mix预混液 25 μL,dd H2O 19 μL。引物序列为,U968-GC:5’-CGC CCG GGG CGC GCC CCG GGC GGG GCG GGG GCA CGG GGG GAA CGC GAA GAA CCT TAC-3’;L1401:5’-CGG TGT GTA CAA GAC CC-3’。扩增程序:95℃预变性10 min,(95℃变性45 s,56℃褪火45 s,72℃延伸45 s)33个循环,最终72℃延伸10 min。PCR产物用1.2%的琼脂糖凝胶电泳检测,最后于-20℃保存。
1.2.5 变性梯度凝胶电泳
DGGE采用Dcode突变检测系统(Bio-Rad)及双梯度法进行。聚丙烯酰胺凝胶的浓度为8%(丙烯酰胺∶甲叉双丙烯酰胺=37.5∶1质量比),变性梯度为40%~60%,其中40%浓度的胶(低浓度)为100%储存液4.4 mL+0%储存液6.6 mL,60%浓度的胶(高浓度)为100%储存液6.6 mL+0%储存液4.4 mL,最后将两种浓度的胶加入专用注射器缓缓注入电泳玻璃板中间,然后快速注入浓缩胶(0%储存液6 mL+20%过硫酸铵60 μL+四甲基乙二胺6 μL),插入梳子,待胶半干时缓缓拔下梳子,点入PCR产物,放置与电泳槽中(50×TAE缓冲液,预热至60℃)200 V电压预10 min,然后85 V电压跑14 h。最后用溴化乙锭(EB)进行染色30 min、dd H2O脱色3次,脱色完毕后采用凝胶成像仪进行成像拍照保存,用于后期分析。
1.2.6 回收条带及测序
在紫外灯下用无菌刀片从胶上切下有差异和无差异的条带,将切下的条带放入对应编号的灭菌的1.5 mL离心管中,用去离子水漂洗两次后加入20 μL TE溶液,4 ℃过夜。将回收的DNA用不带GC夹子的U968/L1401扩增,扩增条件同上。扩增产物经1.2%的琼脂糖凝胶电泳后成像,切下明亮条带进行回收、纯化(采用PCR回收纯化试剂盒)。连接T载体(采用T-载体PCR产物克隆试剂盒)后送至北京六合华大基因公司进行测序。测得的16S rDNA序列经过Genbank数据库进行比对。
1.3 数据处理
试验数据采用Excel 2013软件进行整理,利用SPSS17.0软件对分析结果进行统计分析,通过独立样本t检验方法进行显著性检验,试验结果以平均值±标准差(mean±SD)表示,P<0.05为差异显著,P>0.05为差异不显著。
2 结果与分析
2.1 发酵棉粕饲料对肉牛牛粪中细菌数量的影响
试验前期,C组和T组总细菌差异显著(P<0.05);C组和T组大肠杆菌差异显著(P<0.05);C组和T组金黄色葡萄球菌差异不显著(P>0.05);C组和T组乳酸菌差异显著(P<0.05)。试验后期,C组和T组总细菌差异不显著(P>0.05);C和T组大肠杆菌差异显著(P<0.05);C和T组金黄色葡萄球菌差异显著(P<0.05);C组和T组乳酸菌差异显著(P<0.05)。总细菌、大肠杆菌、金黄色葡萄球菌数量的变化体现了牛粪中微生物菌群数量上的多样性。
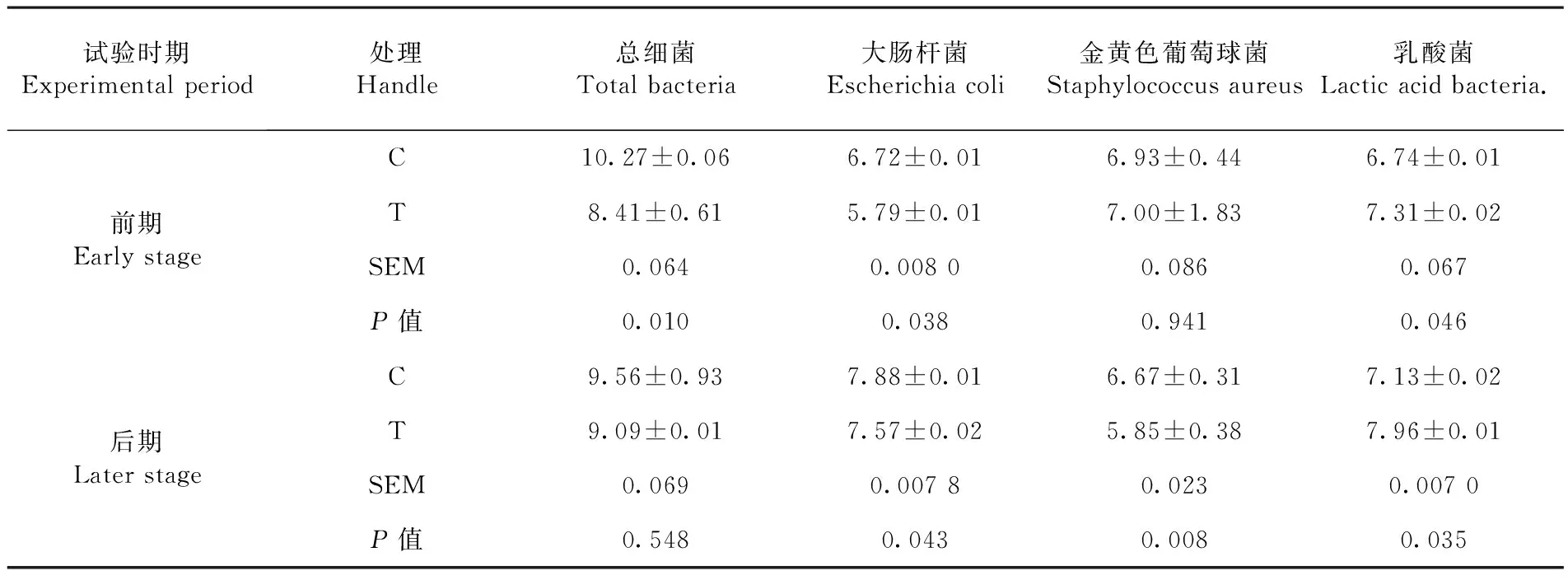
表3 不同时期发酵棉粕饲料下肉牛牛粪中细菌数量变化
注:表中T为试验组和C为对照组
Note: T in the table is the treatment group and C as the control group
2.2 细菌16S rDNA 基因V6-V8区PCR扩增
试验提取的总DNA,经过1%的琼脂凝胶电泳检测,基因组DNA长度在23 kp左右。PCR产物经1.2%琼脂糖凝胶电泳检测。结果显示,各泳道均得到清晰明亮、长度在500 bp且无非特异性扩增的条带,阴性对照无条带,符合目的基因片段,可用于后续的DGGE电泳。图1(a)
2.3 DGGE多样性
2.3.1 变性梯度凝胶电泳结果
通过DDGE技术从各个时期的样品中分离出来了不同数目的条带。4个泳道中不同条带数目,可以直接说明粪样中微生物菌群的多样性。DGGE图谱中每一个泳道代表一个样品,泳道中的每一个条带都有其自身的迁移速度和对应的光强。不同的迁移速度代表不同的细菌种类,条带的亮度代表着细菌种类的数量,亮度越高说明此种细菌的数量众多,可以鉴别出此样品中微生物菌群的优势菌。除了存在不同的条带还存在相同的条带,表明其属于同一种细菌,其明暗程度也表明了细菌的数量差异。图1(b)
2.3.2 DGGE指纹图谱
通过Quantity One软件对DGGE指纹图谱进行分析。不同泳道的聚类分析显示,不同泳道可分为3类,其中CB为一类,CA分为一类,TB和TA分为一类。不同泳道的相似性分析如图1(d)所示。其中相似性最小(56.4)的为CB和CA。相似性最大(72.0)的TB和TA。CB和TB的相似性为58.7。TB和CA的相似性为62.3。CB和TA的相似性为59.8。CA和TA的相似性为60.5。图1(c)

注:图中CB(Control Before)为对照组前期;TB(Treatment Before)为试验组前期;CA(Control After)为对照组后期;TA(Treatment After)为试验组后期
Note: the figure in CB (Control Before) as the control group in early; TB (Treatment Before) as the experimental group early; CA (Control After) as the control group later; TA (Treatment After) as the experimental group later
图1 16S rDNA 基因V6-V8区PCR扩增、DGGE指纹图谱、聚类分析及相似性矩阵
Fig.1 PCR amplification of 16S rDNA gene in V6-V8 region, DGGE fingerprinting, cluster analysis and similarity matrix
2.3.3 回收条带测序及序列
DGGE图谱主要条带的测序结果表明,从不同组别和时期的细菌DGGE图谱上共标记了11条特异性条带,将检测出的每一个条带序列和NCBI数据库进行序列比对,得到同源性最高的菌株,分别是:1为Salmonellaentericasp.,2为Ruminococcussp.,3为Lactobacillussp.,4为Lactobacillussp.,5为Lachnospiraceaesp.,6为Shigellasp.,7为Clostirdiumsp.,8为Escherichiacolisp.,9为Escherichiacolisp.,10为Enterobactersp.,11为Enterococcussp.,且同源性均超过了96%。
试验DGGE图谱主要条带切胶测序,粪中细菌种类较为丰富。CB中条带最亮的为4、9、11,表明乳酸菌、大肠杆菌、肠球菌可能是其优势菌群;TB中条带最亮的为4、5、8、9,表明乳酸菌、大肠杆菌可能是其优势菌群;CA中条带最亮的为3、8、9,表明乳酸菌、大肠杆菌可能是其优势菌群;TA中条带最亮的为3、8、9,表明乳酸菌、大肠杆菌可能是其优势菌群。CB、TB、CA、TA四组中有两个相同菌属,包括2个乳酸菌和2个大肠杆菌。表4
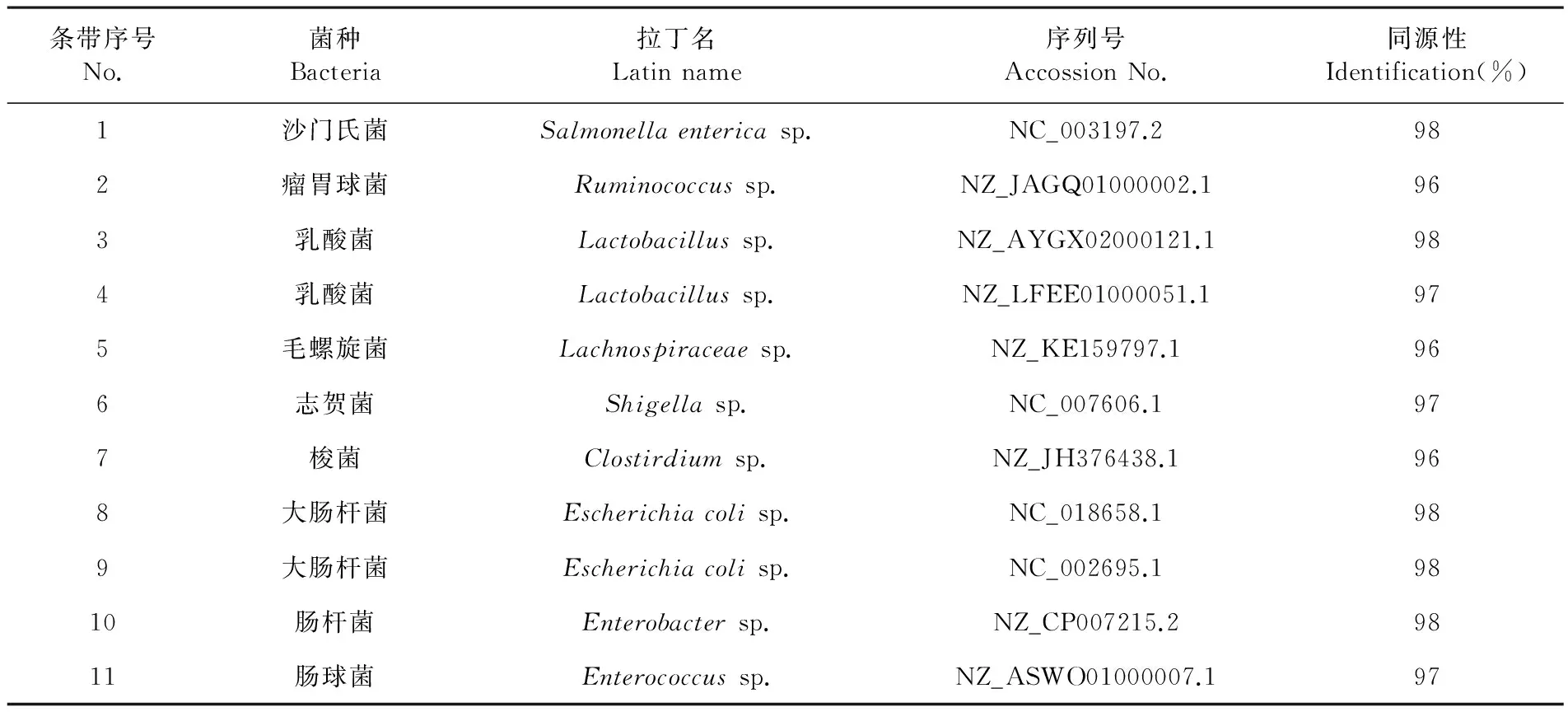
表4 DGGE图谱中条带的基因片段序列比对
3 讨 论
3.1 牛粪中细菌数量差异性的影响
牛粪中菌群的数量采用传统的平板计数方法。此方法虽然利用不同类型的培养基经过适时的培养即可得到相应的菌落数量,实行起来简单,但是在后期进行菌落计数的时候,由于肉眼无法清除的识别每个菌落的数量,可能造成菌落计数结果偏低[20-22]。
试验前期和试验后期上,金黄色葡萄球菌含量均成降低的趋势,金黄色葡萄球菌在健康动物的机体内正常繁殖具有其一定的作用,但如果失去平衡的话,将会引起动物机体的紊乱严重的话会导致动物生病,例如金黄色葡萄球菌可以产生肠毒素,肠毒素是一种耐热的外毒素,也不受胰蛋白酶的影响,可引起急性胃肠炎。但是乳酸菌的含量在试验前期和试验后期是成上升趋势的,乳酸菌在动物体内能发挥许多的生理功能。大量研究资料表明,乳酸菌能促进动物生长,调节胃肠道正常菌群、维持微生态平衡,从而改善胃肠道功能;提高食物消化率和生物效价;降低血清胆固醇,控制内毒素;抑制肠道内腐败菌生长;提高机体免疫力等[23]。试验结果表明,在安格斯肉牛上饲喂发酵棉粕,在一定程度上改变了其肠道微生物细菌数量的变化,可能减少某些致病细菌的数量,增加有益菌的数量。
反刍动物的肠道中寄居着大量的微生物,微生物菌群的数量及整个微生物区系的组成受多种生理因素、饲料组成和环境因素等影响[24]。粪样中的微生物与结肠中的微生物有99%的相似性,因此想了解反刍动物肠道微生物的数量及多样性可以通过粪中的微生物反推肠道微生物的数量及多样性。饲喂同种饲料且相同品种的牛,其粪中微生物区系不完全相同,由于动物自身生理上存在个体差异,故在试验结果中,不能排除个体差异对不同细菌数量造成的影响。
3.2 牛粪中细菌多样性的影响
不同提取DNA的方法会直接影响细菌多样性的结果,因此提取纯度高,杂带少的DNA是进行变性凝胶电泳成功的首要条件。试验采用TIANamp Stool DNA Kit,该试剂盒操作方法简单、提取所需时间短且去除抑制物、杂质蛋白及细胞中其他有机化合物的效果很好,被广泛应用于动物粪便DNA的提取。此方法可获得纯度高,质量稳定的DNA片段,可满足后续变性凝胶电泳的试验。
微生物相似性及多样性在DGGE指纹图谱中体现为条带的有无及条带亮度的变化。由于DGGE技术只能分辨数量占超过总细菌1 %的细菌种类[25],所以指纹图谱中的条带一定程度上应理解为优势细菌,某一条带的缺失并不一定是其不存在,而可能是数量上处于劣势,在图谱中不能得以体现。PCR-DGGE是一种快速可靠的比较细菌群落的方法,DGGE条带再经切胶和测序,有助于人们了解微生物多样性。试验结果中没有测序得出嗜酸乳酸杆菌、枯草芽孢杆菌和热带假丝酵母菌等菌的序列,其原因可能是由于在切胶回收条带的环节人为误差造成的,故在试验结果中,不能排除个人操作技术的影响。
对DGGE指纹图谱进行分析,不同泳道的聚类分析划分为3类,其中CB、CA各为一类,TB和TA归为一类。说明饲喂未发酵棉粕的消化道菌群中细菌的数量以及分布上存在较大差别。结合微生物技术的结果来看,试验前期和试验后期上C组总细菌和乳酸菌数量都有减少的趋势,大肠杆菌数量有增加的趋势,金黄色葡萄球菌数量变化不大;试验前期和试验后期上T组总细菌和乳酸菌数量变化不明显,大肠杆菌数量有增加的趋势,金黄色葡萄球菌数量有减少的趋势。CB、CA各为一类,很可能是因为C组总细菌数量和其中各种菌的数量在试验前期和后期有不同变化的原因造成的。
微生物菌群非常依赖日粮分解产物作为最终代谢底物,不同种类细菌所需底物和生长条件不同[26]。聂存喜等[27]研究用不同微生物发酵的多种小分子代谢产物含量会因菌种而异。棉粕使用嗜酸乳酸杆菌、枯草芽孢杆菌以及热带假丝酵母菌三种复合菌种发酵,可能改变其产生的代谢产物的含量,而且不同细菌生长所需的营养物质也不同,因此改变了肠道细菌多样性,具体的影响机制还需进一步深入研究。
4 结 论
4.1 饲喂发酵棉粕,牛粪中细菌的数量有所改变。试验前期,添加发酵棉粕显著降低总细菌和大肠杆菌数量,提高乳酸菌数量。试验后期,添加发酵棉粕显著降低大肠杆菌、金黄色葡萄球菌数量,显著提高乳酸菌数量。
4.2 饲喂发酵棉粕,通过测序检测到牛粪含有大量的微生物,测序得到11种细菌,分别所属瘤胃球菌、乳酸菌、大肠杆菌、梭菌等8个菌属。
4.3 饲喂未发酵棉粕组初期与末期(CB与CA)前后DGGE指纹图谱聚类分析划分为2类,而饲喂发酵棉粕初期与末期(TB与TA)前后DGGE指纹图谱聚类分析划分为1类。
References)
[1] 何涛. 棉籽粕的发酵脱毒及其在肉仔鸡中的应用研究[D]. 北京:中国农业科学院硕士学位论文, 2008.
HE Tao. (2008).StudiesonBio-detoxificationofCottonseedMealandItsApplicationinBroilerDiets[D]. Master Dissertation.Chinese Academy of Agricultural Sciences, Beijing. (in Chinese)
[2]郭书贤, 王冬梅, 梁运祥. 微生物发酵棉籽饼粕脱毒与利用研究进展[J]. 中国酿造, 2009, 28(1): 4-10.
GUO Shu-xian, WANG Dong-mei, LIANG Yun-xiang. (2009). Research progress on microbial detoxification and utilization of cottonseed cake [J].ChinaBrewing, 28(1):4-10. (in Chinese)
[3]Zhang, W. J., Xu, S. H., Sun, J. Y., & Xia, Y. (2007). Development of a microbial fermentation process for detoxification of gossypol in cottonseed meal.AnimalFeedScience&Technology, 135(2): 176-186.
[4]ZHANG, Wen-ju, Zi-rong, Jian-yi, & YANG. (2006). Effect of selected fungi on the reduction of gossypol levels and nutritional value during solid substrate fermentation of cottonseed meal.JournalofZhejiangUniversity-ScienceB(Biomedicine&Biotechnology) , 7(9): 690-669.
[5]邱良伟. 发酵对棉粕营养特性及其在肉鸡中的应用研究[D]. 合肥:安徽农业大学硕士学位论文, 2012.
QIU Liang-wei. (2012).Effectofdifferentfermentationprocessonthenutritionalcharacteristicsofcottonseedmeal[D]. Master Dissertation. Anhui Agricultural University, Hefei. (in Chinese)
[6]张国民, 张国栋. 用发酵棉粕代替豆粕饲喂犊牛效果试验[J]. 新疆畜牧业, 2004,(2): 18-20.
ZHANG Guo-min, ZHANG Guo-dong. (2004).Using fermented cottonseed meal instead of soybean meal fed calves effect test [J].XinjiangAnimalHusbandry, (2):18-20. (in Chinese)
[7]闫理东, 张文举, 聂存喜, 等. 发酵棉粕对黄羽肉鸡生产性能和屠宰性能的影响[J]. 石河子大学学报(自然科学版), 2012, 30(2): 171-176.
YAN Li-dong, ZHANG Wen-ju, NIE Cun-xi, et al. (2012). Effect of Fermented Cottonseed Meal on Growth Performance and Slaughter Performance in Yellow-Feathered Broilers [J].JournalofShiheziUniversity(NaturalScience) , 30(2):171-176. (in Chinese)
[8]汤江武, 吴逸飞, 孙宏, 等. 发酵棉粕对肉鸡生长性能、血清生化指标及免疫功能的影响[J]. 中国畜牧杂志, 2011, 47(5): 29-34.
TANG Jiang-wu, WU Yi-fei, SUN Hong, et al. (2011). Effects of Fermented Cottonseed Meal on Growth Performance, Serum Biochemical Parameters and Immunity of Broilers [J].ChineseJournalofAnimalScience, 47(5):29-34. (in Chinese)
[9]高学文,姚仕义,Huong Pham, 等. 基因工程菌枯草芽孢杆菌GEB3产生的脂肽类抗生素及其生物活性研究[J]. 中国农业科学,2003,(12):1 496-1 501.
GAO Xue-wen, YAO Shi-yi, Huong Pham, et al. (2003). Lipopeptide Antibiotics Produced by the Engineered StrainBacillussubtilisGEB3 and Detection of Its Bioactivity [J].ScientiaAgriculturalSinica, (12):1,496-1,501. (in Chinese)
[10]刘忠元. 热带假丝酵母和黑曲霉发酵玉米秸秆的条件优化[D]. 长春: 吉林大学硕士学位论文, 2007.
LIU Zhong-yuan. (2007).OptimizingtheFermentationConditionofMaizeStoverbyCandidatropicalisandAspergillusniger[D]. Master Dissertation. Jilin University, Changchun. (in Chinese)
[11]秦金胜, 禚梅, 许衡, 等. 发酵棉粕和普通棉粕替代豆粕对猪生长性能的影响[J]. 新疆农业大学学报, 2010, 33(6): 496-501.
QIN Jin-sheng, ZHUO Mei, XU Heng, et al. (2010). Effect of Common Cottonseed Meal and Fermented Cottonseed Meal Substituted for Soybean Meal on the Growth Performance of Pigs [J].JournalofXinjiangAgriculturalUniversity, 33(6):196-501. (in Chinese)
[12]丁超,和玉丹,郑云林.发酵棉粕替代豆粕对生长猪生长性能的影响[J].饲料工业,2010,31(24): 47-48.
DING Chao, HE Yu-dan, ZHENG Yun-lin. (2010). Effect of Fermented Cottonseed Meal Substituted for Soybean Meal on the Growth Performance of Pigs [J].FeedIndustry, 31(24): 47-48.(in Chinese)
[13] Lamont, R. F., Sobel, J. D., Akins, R. A., Hassan, S. S., Chaiworapongsa, T., & Kusanovic, J. P., et al. (2011). The vaginal microbiome: new information about genital tract flora using molecular based techniques.BjoganInternationalJournalofObstetrics&Gynaecology, 118(5): 533.
[14] Wang, Y., Ametaj, B. N., Ambrose, D. J., & G?nzle, M. G. (2013). Characterisation of the bacterial microbiota of the vagina of dairy cows and isolation of pediocin-producing pediococcus acidilactici.BMCMicrobiology, 13(1): 1-11.
[15]图雅. 虎粪细菌区系16S rDNA动态[D]. 南京:南京农业大学博士学位论文,2002.
TU Ya. (2002).DynamicofBacterial16SrDNAofTiger'sFecalFlora[D]. PhD Dissertation. Nanjing Agricultural University, Nanjing. (in Chinese)
[16]潘康成,陈正礼,崔恒敏,等.利用ERIC-PCR和PCR-DGGE技术分析喂服枯草芽孢杆菌肉鸡肠道菌群的多样性[J].动物营养学报,2010,22(4): 985-991.
PAN Kang-cheng, CHEN Zheng-li, CUI Heng-min, et al. (2010). Analysis of Intestinal Microflora Diversity in Broilers Treated with Bacillus subtilis by ERIC-PCR and PCR-DGGE [J].ChineseJournalofAnimalNutrition, 22(4): 985-991. (in Chinese)
[17]吴利, 余育和, 冯伟松. PCR-DGGE技术在环境微型生物群落研究中的应用 [J]. 合肥师范学院学报, 2010, 28(6): 103-108.
WU Li, YU Yu-he, FENG Wei-song. (2010). Application of PCR-DGGE in Study of Microbial Community [J].JournalofHefeiNormalUniversity, 28(6):103-108. (in Chinese)
[18]Zoetendal, E. G,. Akkermans, A. D. L, Vos W. M. D. (1998).Temperature Gradient Gel Electrophoresis Analysis of 16S rRNA from Human Fecal Samples Reveals Stable and Host-Specific Communities of Active Bacteria.Applied&EnvironmentalMicrobiology, 4(10): 3,854-3,859.
[19]陈天寿. 微生物培养基的制造与应用[M]. 中国农业出版社, 1995.
CHEN Tian-shou. (1995).ManufactureandApplicationofMicrobialCultureMedium[M]. Beijing: China Agriculture Press. (in Chinese)
[20]Amann, R. I., Ludwing, W., Schleifer, K. H. (1995). Phylogenetic identification and in situ detection of individual microbial cells without cultivation.MicrobiologicalReviews, 59(1): 143-169.
[21]Allen, M. J., Edberg, S. C., & Reasoner, D. J. (2004). Heterotrophic plate count bacteria--what is their significance in drinking water.InternationalJournalofFoodMicrobiology, 92(3): 265-274.
[22]Kell, D. B., Kaprelyants, A. S., Weichart, D. H., Harwood, C. R., & Barer, M. R. (1998). Viability and activity in readily culturable bacteria: a review and discussion of the practical issues.AntonievanLeeuwenhoek,73(2): 169-187.
[23]赵红霞, 詹勇, 许梓荣. 乳酸菌的研究及其应用[J]. 江西饲料, 2003,(1): 9-12.
ZHAO Hong-xia, ZHAN Yong, XU Zi-rong. (2003).Research and application of lactic acid bacteria [J].JiangxiFeed, (1):9-12. (in Chinese)
[24]张心壮, 孟庆翔, 崔振亮,等. 利用PCR-DGGE技术研究不同代乳料对奶公犊粪样细菌区系的影响[J]. 中国畜牧杂志, 2013, 49(5): 27-30.
ZHANG Xin-zhuang, MENG Qing-xiang, CUI Zhen-liang, et al. (2013).Study on different effects of milk replacer on calf fecal bacterial flora by the technology of PCR-DGGE [J].ChineseJournalofAnimalScience, 49(5):27-30. (in Chinese)
[25]吴高锋, 李文刚, 高卫科, 等. PCR-DGGE的原理及在动物肠道菌群分析中的应用[J]. 中国畜牧兽医, 2008, 35(6): 37-39.
WU Gao-feng, LI Wen-gang, GAO Wei-ke, et al. (2008). The principle of PCR-DGGE and its application in the analysis of intestinal flora in animals [J].ChinaAnimalHusbandry&VeterinaryMedicine, 35(6):37-39. (in Chinese)
[26]胡平. 不同形态玉米日粮及日龄对鹅肠道微生物区系的影响[D]. 扬州:扬州大学硕士学位论文, 2010.
HU Ping. (2010).EffectsofCornFeedFormsandAgeonIntestinalMicrofloraofGeese[D]. Master Dissertation. Yangzhou University, Yanzhou. (in Chinese)
[27]聂存喜, 张文举, 闫理东,等.棉籽粕源发酵蛋白质饲料的代谢产物研究[J]. 动物营养学报, 2012, 24(8): 1 602-1 609.
NIE Cun-xi, ZHANG Wen-ju, YAN Li-dong, et al. (2012). A Study of Metabolites of Protein Feed Fermented with Cottonseed Meal Mixed Substrates [J].ChineseJournalofAnimalNutrition, 24(8):1,602-1,609. (in Chinese)
Study of Bacterial Number and Diversity in Manure of Beef Cattle Fed by Fermented Cottonseed Meal
LIANG Min1, ZHANG Wen-ju1, ZHANG Fan-fan1, CHEN Ning1, YIN Jun-liang2
(1. College of Animal Science and Technology, Shihezi University, Shihezi Xinjiang 832003, China; 2. Research Institute of Animal Husbandry and Veterinary Medicine of Xinjiang Academy of Agricultural and Reclamation Sciences, Shihezi Xinjiang 832000, China)
【Objective】 To study the effect of feeding fermented cottonseed meal on the number of bacteria and diversity in Angus beef cattle manure.【Method】The number of bacteria was calculated by plate counting method, V6-V8 DNA gene amplification of bacterial 16S rDNA was performed by PCR technique. The PCR products were subjected to denaturing gradient gel electrophoresis (DGGE). The specific bands of fingerprint were obtained and sequenced, and bacterial strains were identified by BLAST.【Result】(1) In early experiment, there were significant differences in total bacteria,EscherichiacoliandLactobacillusbetween group C and group T (P< 0.05), but no significant difference inStaphylococcusaureusbetween them (P> 0.05). In late experiment, there was no significant difference in total bacteria between group C and group T (P> 0.05), butEscherichiacoli,StaphylococcusaureusandLactobacilluswere significantly different (P< 0.05). (2) Bacteria found in cow dung were:Salmonella,Ruminococcus,Lactobacillus,Lachnospiraceae,Shigella,Clostridium,Escherichiacoli,EnterobacterandEnterococcus, and among them,LactobacillusandEscherichiacolimight be the dominant bacteria in cow manure. 【Conclusion】Total bacteria,EscherichiacoliandStaphylococcusaureushave obvious change in the number of early and late experiment period. There are many kinds of bacteria in cow dung, which reflect the diversity of microbial flora in cow manure.
fermented cottonseed meal; bacteria; number; DGGE; diversity
ZHANG Wen-ju (1966- ), male, native place: Shanxi. Professor, research field: The development and utilization of feed resources. (E-mail) zhangwj1022@sina.com
10.6048/j.issn.1001-4330.2017.07.019
2017-04-12
新疆兵团重大科技项目(2014AA001)
梁敏(1992- ),女,新疆乌鲁木齐人,硕士研究生,研究方向为饲料资源的开发与利用,(E-mail)10214969502@qq.com
张文举(1966- ),男,陕西临潼人,教授,博士生导师,研究方向为饲料资源的开发与利用,(E-mail)zhangwj1022@sina.com
S816
A
1001-4330(2017)07-1323-09
Supported by: Major Science and Technology R&D Program of XPCC (2014AA001)
