基于SRAP标记的黄瓜霜霉菌遗传多样性分析
2017-08-09白玲玲刘行风陈政瑜周莹莹张艳菊
白玲玲, 刘行风, 陈政瑜, 周莹莹, 刘 丽, 张艳菊*, 张 奥, 杨 森
(1. 东北农业大学农学院, 哈尔滨 150030; 2. 黑龙江省哈尔滨市阿城区种子服务中心, 哈尔滨 150039)
基于SRAP标记的黄瓜霜霉菌遗传多样性分析
白玲玲1, 刘行风1, 陈政瑜1, 周莹莹2, 刘 丽1, 张艳菊1*, 张 奥1, 杨 森1
(1. 东北农业大学农学院, 哈尔滨 150030; 2. 黑龙江省哈尔滨市阿城区种子服务中心, 哈尔滨 150039)
本研究对来自黑龙江、辽宁、河北、山东、江苏5个省12个黄瓜主产区的77个黄瓜霜霉菌菌株,采用SRAP分子标记进行了遗传多样性分析。从35对SRAP引物中筛选出10对引物,共产生9 554条扩增条带,其中9 132条表现多态性,占95.6%。基于SRAP分子标记,77个黄瓜霜霉菌菌株的遗传距离为0.60~1.00,表明黄瓜霜霉菌具有丰富的遗传多样性。通过聚类分析,77个黄瓜霜霉菌菌株聚类为8个类群,并且来自相同地区的菌株大多数聚集于同一类群中,表明黄瓜霜霉菌群体的遗传多样性与其地理来源密切相关。
黄瓜霜霉病; 分子标记; SRAP; 群体遗传
由藻物界卵菌门古巴假霜霉菌Pseudoperonosporacubensis(Berkeley & Curtis) Rostovzev侵染引起的霜霉病是黄瓜生产上的重要叶部病害。该病害广泛分布于世界各地,在适宜条件下,传播流行速度极快,一到两周时间即可造成叶片全部枯死,对黄瓜生产威胁极大[1-2]。
目前,国内外学者利用不同的分子标记技术对黄瓜霜霉菌遗传多样性开展了相应的研究。Wang等[3]通过对rDNA-ITS序列进行研究认为,黄瓜霜霉菌在种内保持高度的遗传一致性,而种间遗传存在差异且与亲缘关系有密切的关系。Sarris等[4]利用AFLP对来自捷克、克里特岛、法国和荷兰的黄瓜霜霉菌菌株进行了遗传多样性研究,发现霜霉菌菌株被划分为2个独立的类群,一个类群为捷克、法国和荷兰的所有菌株;另一个类群为克里特岛的菌株,分析表明,菌株遗传多样性与其地理起源、寄主品种、致病型和对杀菌剂抗性有关。Quesada-Ocampo等[5]通过对黄瓜霜霉菌的群体遗传结构分析,构建了全球第一个黄瓜霜霉菌群体遗传结构图。Polat等[6]采用ISSR和SRAP分子标记对以色列、捷克和土耳其的87个黄瓜霜霉菌菌株进行分析,证实菌株间具有显著的遗传多样性,采自土耳其和捷克的菌株表现出一致的遗传背景,而以色列的菌株则明显不同,认为可能是由于菌株迁移或有性生殖现象引起的。在中国,刘艳玲等[7]对中国12个城市的34株黄瓜霜霉菌的rDNA-ITS序列进行SNP分析表明,菌株来源与地理区域存在一定的相关性,菌株间在遗传上存在较大的差异。张艳菊等[8]利用27个RAPD随机引物对18株黄瓜霜霉菌进行全基因组DNA的PCR扩增,获得RAPD标记共230个,发现各菌株之间的相似性系数较低,在基因组DNA 水平上存在差异,具有丰富的遗传多样性。
相关序列扩增多态性(sequence-related amplified polymorphism, SRAP)是一种基于PCR的分子标记技术[9],具有快速、可靠、高效、低成本、无需知道基因组的序列信息等优点[10],已经成功应用于遗传多样性分析、遗传图谱的构建、重要性状的基因定位等方面的研究。Tang等[11]采用ISSR和SRAP分子标记,对中国的34株栽培木耳Auriculariaauricula的遗传多样性进行了分析,认为SRAP的分类更细,内容更丰富。Liu等[12]采用RAPD、ISSR和SRAP三种标记对香菇Lentinulaedodes的遗传多样性进行了分析,成功转化为SCAR标记。Zhang等[13]利用ISSR和SRAP分子标记对金顶侧耳Pleurotuscitrinopileatus进行了遗传多样性分析,并建立了其系统发育进化树,利用SRAP分子标记将菌株分为了6个类群,表现出较高的遗传多样性水平。Zhang等[14]利用SRAP标记分析了猪苓Polyporusumbellatus的遗传多样性。Ma等[15]对中国东北的13个地区的松口蘑Tricholomamatsutake进行了遗传多样性鉴定,发现菌株间的遗传距离与地理隔离具有极高的正相关性。
本文利用SRAP分子标记对采自我国5个省12个黄瓜主产区的黄瓜霜霉菌群体进行遗传多样性研究,了解霜霉菌遗传多样性和演化关系,为有效防治黄瓜霜霉病提供理论依据。
1 材料与方法
1.1 供试菌株
2014年至2015年,在黄瓜霜霉病盛发期,从黑龙江、辽宁、山东、河北、江苏5个省12个黄瓜主产区的温室大棚中采集77个黄瓜霜霉病样品(表1),用于SRAP分析。
1.2 试验方法
1.2.1 DNA提取
黄瓜霜霉菌基因组DNA的提取采用CTAB方法[15],稍加改良。DNA的纯度与浓度使用紫外分光光度计(DU600,Beckman,美国)检测后,置于-20℃保存待用。
1.2.2 SRAP反应体系和反应程序
PCR反应总体积为20 μL,其中10×PCR Buffer (含20 mmol/L Mg2+) 2.0 μL,dNTPs(各2.5 mmol/L) 2 μL,Taq酶(5U/μL)0.3 μL,ddH2O 12.7 μL,上下游混合引物(10 μmol/L)1 μL, 模板DNA(50 ng/μL)2 μL。反应程序为:94℃预变性30 s;94℃变性1 min,35℃退火30 s,72℃扩展1 min,进行3个循环;然后94℃变性1 min,50℃退火1 min,72℃扩展1 min,进行35个循环;72℃延伸10 min;4℃保存。
1.2.3 SRAP引物筛选
7个SRAP上游引物和5个SRAP下游引物(表2)由上海生物工程有限公司合成,随机组合成35对引物,选取3个模板DNA分别进行PCR扩增,筛选扩增条带清晰,重复性和多态性显著的引物,用于本研究黄瓜霜霉菌遗传多样性分析。
1.2.4 SRAP分析
在PCR扩增产物中加入2 μL加样缓冲液,用8%的非变性聚丙烯酰胺凝胶检测。凝胶大小为180 cm×120 cm×1 mm,电泳缓冲液为1×TBE,每个点样孔加2.0 μL样品,180V恒压下电泳约1.0 h。电泳结束后,进行银染。先将胶放入固定液中,轻摇7~8 min;倒掉固定液,加入渗透液,轻摇10 min左右;倒掉渗透液,蒸馏水水洗30 s;倒掉蒸馏水后,加入显色液,轻摇至DNA条带显出为止;倒掉显色液,用蒸馏水洗1次;倒掉蒸馏水,加入终止液终止反应;统计带型并照相。
1.2.5 数据分析
在PAGE胶上同一位置,出现条带的记为“1”,无条带的记为“0”,生成“0”和“1”组成的原始矩阵。利用软件NTSYS-PC(ver. 2.1)中的SIMQUAL程序计算菌株间相似系数(similarity coefficient),获得相似系数矩阵。利用其中的SAHN程序和UPGMA方法进行聚类分析,通过Tree Plot模块建立聚类分析树状图。
表1 黄瓜霜霉菌菌株名称及来源
Table 1 Name ofPseudoperonosporacubensisisolates and their origins

编号Number菌株名称Isolatecode年份Year来源Origin编号Number菌株名称Isolatecode年份Year来源Origin1Pc-hlj12014黑龙江省,哈尔滨市40Pc-hb32014河北省,沧州市2Pc-hlj22014黑龙江省,哈尔滨市41Pc-hb42014河北省,廊坊市3Pc-hlj32014黑龙江省,哈尔滨市42Pc-hb52014河北省,廊坊市4Pc-hlj42014黑龙江省,哈尔滨市43Pc-hb82015河北省,沧州市5Pc-hlj52014黑龙江省,哈尔滨市44Pc-hb92015河北省,沧州市6Pc-hlj62014黑龙江省,哈尔滨市45Pc-hb102015河北省,沧州市7Pc-hlj72014黑龙江省,哈尔滨市46Pc-hb112015河北省,沧州市8Pc-hlj82014黑龙江省,哈尔滨市47Pc-hb122015河北省,沧州市9Pc-hlj92014黑龙江省,哈尔滨市48Pc-hb132015河北省,沧州市10Pc-hlj112014黑龙江省,哈尔滨市49Pc-hb162015河北省,石家庄市11Pc-hlj122014黑龙江省,哈尔滨市50Pc-hb172015河北省,石家庄市12Pc-hlj132015黑龙江省,哈尔滨市51Pc-hb182015河北省,石家庄市13Pc-hlj142015黑龙江省,哈尔滨市52Pc-hb192015河北省,石家庄市14Pc-hlj152015黑龙江省,哈尔滨市53Pc-hb202015河北省,石家庄市15Pc-hlj212014黑龙江省,哈尔滨市54Pc-hb212015河北省,石家庄市16Pc-hlj222014黑龙江省,哈尔滨市55Pc-hb222015河北省,石家庄市17Pc-hlj232014黑龙江省,哈尔滨市56Pc-hb232015河北省,石家庄市18Pc-hlj242014黑龙江省,哈尔滨市57Pc-sd12014山东省,寿光市19Pc-hlj252014黑龙江省,哈尔滨市58Pc-sd22014山东省,寿光市20Pc-hlj272014黑龙江省,哈尔滨市59Pc-sd52015山东省,寿光市21Pc-hlj282014黑龙江省,哈尔滨市60Pc-sd62015山东省,寿光市22Pc-ln12014辽宁省,沈阳市61Pc-sd72015山东省,寿光市23Pc-ln22014辽宁省,沈阳市62Pc-sd82015山东省,寿光市24Pc-ln62015辽宁省,沈阳市63Pc-sd92015山东省,寿光市25Pc-ln72015辽宁省,沈阳市64Pc-sd102015山东省,寿光市26Pc-ln82015辽宁省,沈阳市65Pc-sd112015山东省,寿光市27Pc-ln102015辽宁省,沈阳市66Pc-sd122015山东省,寿光市28Pc-ln272015辽宁省,盘锦市67Pc-sd132015山东省,寿光市29Pc-ln282015辽宁省,盘锦市68Pc-sd142014山东省,临沂市30Pc-ln312015辽宁省,盘锦市69Pc-sd152014山东省,临沂市31Pc-ln332015辽宁省,凌源市70Pc-sd172014山东省,临沂市32Pc-ln342015辽宁省,凌源市71Pc-sd192014山东省,临沂市33Pc-ln352015辽宁省,凌源市72Pc-sd202014山东省,临沂市34Pc-ln362015辽宁省,凌源市73Pc-sd222015山东省,泰安市35Pc-ln372015辽宁省,凌源市74Pc-sd272015山东省,泰安市36Pc-ln412014辽宁省,鞍山市75Pc-sd282015山东省,泰安市37Pc-ln422014辽宁省,鞍山市76Pc-js12014江苏省,连云港市38Pc-hb12014河北省,沧州市77Pc-js22014江苏省,连云港市39Pc-hb22014河北省,沧州市
2 结果与分析
2.1 黄瓜霜霉菌基因组DNA的提取
所提取的黄瓜霜霉菌基因组DNA经1%(w/v)琼脂糖凝胶电泳检测(图1),显示条带清晰完整,无拖尾现象,DNA无降解,可满足SRAP分子标记研究。
2.2 黄瓜霜霉菌SRAP-PCR结果
采用上、下游引物自由组合的35对引物对霜霉菌菌株进行PCR扩增,从中筛选出10对多态性好、带型清晰的引物组合(表3)。利用这10对引物对77个黄瓜霜霉菌菌株进行SRAP分析,总共扩增出条带9 554条,大小为100~5 000 bp不等,其中9 132条为多态性条带,多态性比率为95.6%。引物组合Me9/Em14扩增出的条带数最多,达1 406条;引物组合Me2/Em4扩增出的条带数最少。每个SRAP引物对扩增出的平均条带数为955条(表3)。
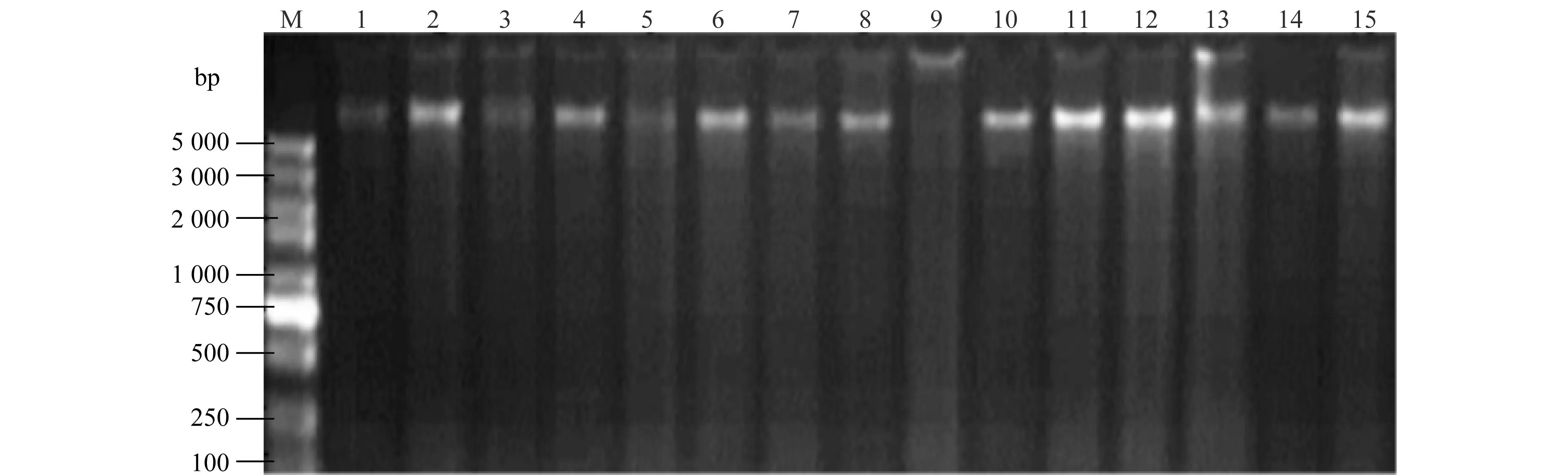
图1 1%琼脂糖凝胶电泳检测部分黄瓜霜霉菌基因组DNAFig.1 Detection of genomic DNA of some Pseudoperonospora cubensis isolates based on ethidium bromide-stained 1% agarose gel
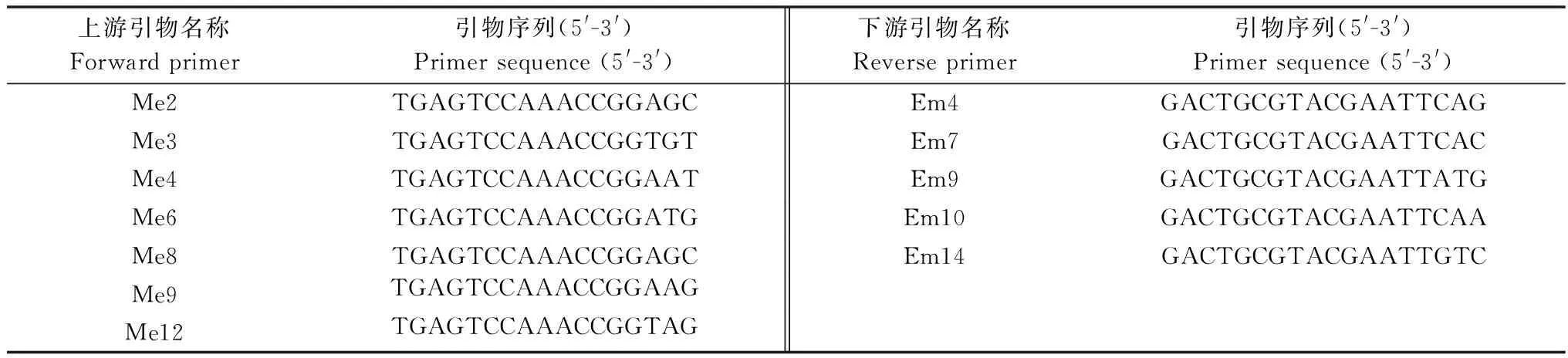
上游引物名称Forwardprimer引物序列(5'-3')Primersequence(5'-3')下游引物名称Reverseprimer引物序列(5'-3')Primersequence(5'-3')Me2TGAGTCCAAACCGGAGCEm4GACTGCGTACGAATTCAGMe3TGAGTCCAAACCGGTGTEm7GACTGCGTACGAATTCACMe4TGAGTCCAAACCGGAATEm9GACTGCGTACGAATTATGMe6TGAGTCCAAACCGGATGEm10GACTGCGTACGAATTCAAMe8TGAGTCCAAACCGGAGCEm14GACTGCGTACGAATTGTCMe9TGAGTCCAAACCGGAAGMe12TGAGTCCAAACCGGTAG
表3 10对引物组合对77个黄瓜霜霉菌菌株SRAP扩增结果
Table 3 SRAP-PCR amplification of 77 isolates ofPseudoperonosporacubensisusing 10 primer pairs

引物编号Primercode引物组合Primercombination总条带数/条Totalofband多态性条带数目/条Polymorphicband多态性比率/%PercentageofpolymorphicbandSRAP-1Me8/Em10692692100SRAP-2Me8/Em1457850086.5SRAP-3Me2/Em4569569100SRAP-4Me2/Em998190492.2SRAP-5Me4/Em1485658868.7SRAP-6Me6/Em4895895100SRAP-7Me6/Em711301130100SRAP-8Me12/Em1012211221100SRAP-9Me3/Em412271227100SRAP-10Me9/Em1414061406100
2.3 SRAP聚类分析
利用软件NTSYS-PC(ver. 2.1)对供试的77个黄瓜霜霉菌菌株基因组DNA的SRAP-PCR扩增条带的数据进行统计分析,获得这些菌株间的聚类分析树状图(图2)。由图可见,黄瓜霜霉菌群体遗传多样性比较丰富,遗传分化较大,菌株间的相似系数在0.60~1.00之间,在相似系数为0.73时(图2),所有菌株被分为8个类群,第一类群为黑龙江的所有菌株共21个,占总菌株数的27.3%;第二类群为江苏的2个菌株(Pc-js1和Pc-js2),占总菌株数的2.6%;第三类群为辽宁的16个菌株,占总菌株数20.8%;第四类群为河北的所有菌株(19个)和山东的部分菌株(10个),共29株,占总菌株数的37.7%;第五类群为山东的2个菌株(Pc-sd1和Pc-sd5),第六类群为山东菌株Pc-sd8和Pc-sd9;第七类群为山东菌株Pc-sd19;第八类群为4个山东菌株Pc-sd2、Pc-sd13、Pc-sd12和Pc-sd17。
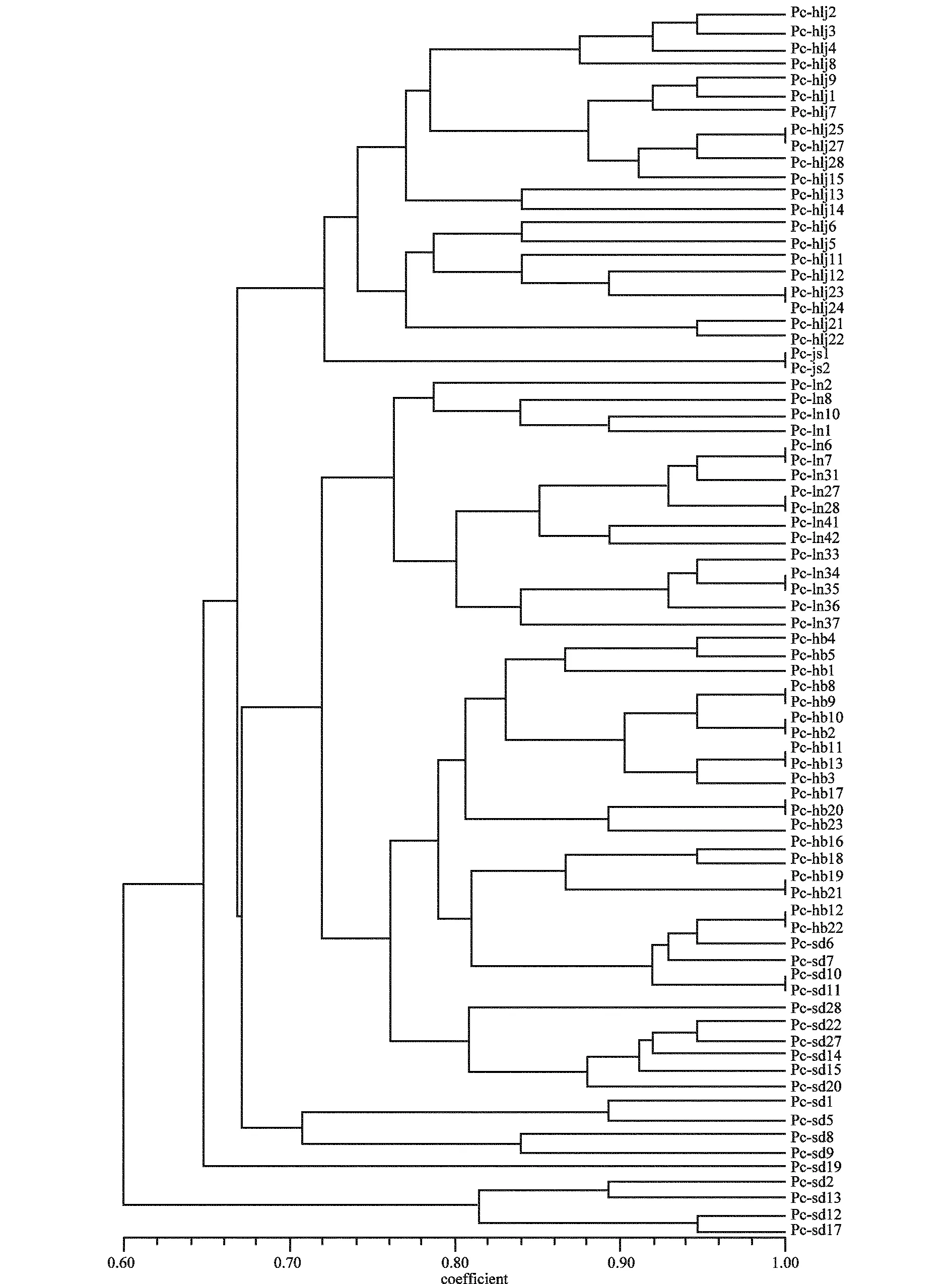
图2 77株黄瓜霜霉菌菌株基于SRAP标记的聚类分析图Fig.2 Dendrogram of 77 isolates of Pseudoperonospora cubensis based on analysis of genetic similarity of the pathogenic populations using SRAP markers
3 讨论
SRAP分子标记技术是利用一对设计独特的引物扩增基因阅读框区域(opening read frame, ORF),上游引物特异性扩增外显子区域,下游引物特异性扩增内含子及启动子区域。本研究对5个省12个黄瓜主产区的77个黄瓜霜霉菌菌株的SRAP进行了研究,从分子水平上揭示出黄瓜霜霉菌群体遗传多样性比较丰富。聚类结果表明,来自黑龙江、辽宁、江苏和河北的所有菌株分别聚类于同一类群内,来自山东的菌株则聚类于5个不同的类群。此研究结果一方面表明基于SRAP分析的黄瓜霜霉菌群体遗传多样性与其地理来源存在密切的相关性,同一地区来源的菌株归于相同的类群;另一方面也可以看出,来自山东的菌株具有较高的遗传多样性。
突变和有性生殖被认为是病菌发生变异的主要原因。由于病菌发生变异,产生新的基因型,从而使菌株表现丰富的遗传多样性。2012年Zhang等[17]在黑龙江、吉林、辽宁、河北、山东、宁夏、陕西和青海省发现了卵孢子的存在,并证实越冬后的卵孢子翌年仍具有活性,可以作为黄瓜霜霉病的初侵染源。2013年Cohen等[18]报道来自我国山东、北京和黑龙江的菌株同时存在两种交配型A1和A2,进一步证实在这些地区病菌在自然条件下发生有性生殖。Zhang等和Cohen等的研究均在山东省发现病菌存在有性生殖,表明进行有性生殖以及异宗配合的病菌,由于发生了遗传物质的重组,因此比不进行有性生殖的病菌具有更大的变异性,产生具有更强适应性、更多样化的新侵染性后代。这些新的后代往往具有相对强的侵染性和复杂的毒力结构,并逐渐取代适应性和侵染性较弱的种群,成为优势种群,使病害发生更加严重。张艳菊等[19]于2015年报道,相比于黑龙江、吉林、辽宁、北京等省份采集的菌株,来自山东省的黄瓜霜霉菌菌株对杀菌剂嘧菌酯的抗性水平最高,其原因可能是因为长期大量使用嘧菌酯使菌株产生了抗药性,这也从另一方面解释了来自山东的病菌具有较高遗传多样性的原因。
迄今为止,教科书及多数学者对黄瓜霜霉病的初侵染源还未形成定论,认为由于北方高寒地区冬季没有黄瓜种植,推测这一地区的初侵染源可能是由发病较早的地区随季风吹来的孢子囊释放游动孢子侵染所致。但是,Zhang等[17]的研究结果表明黑龙江、吉林、辽宁、河北、山东、宁夏、陕西和青海省等地黄瓜霜霉菌均发现存在卵孢子,并可作为黄瓜霜霉病发生的初侵染源。Cohen等[18]的报道也有力地支持了Zhang等[17]的结论。本研究通过对黄瓜霜霉菌遗传多样性的研究,发现同一地区菌株相似性较高,而不同地区菌株间相似性较低,从分子水平证实黄瓜霜霉菌与地理来源存在相关性。
[1] Lebeda A, Cohen Y. Cucurbit downy mildew (Pseudoperonosporacubensis)-biology, ecology, epidemiology, host-pathogen interaction and control [J]. European Journal Plant Pathology, 2011, 129(2): 157-192.
[2] 刘艳玲, 张艳菊, 蔡宁, 等. 黄瓜霜霉病病原与抗病性研究进展[J]. 东北农业大学学报, 2009, 40(4): 127-131.
[3] Wang Na, Ma Yajun, Yang Cuiyun, et al. rDNA-ITS sequence analysis of pathogens of cucumber downy mildew and cucumber powdery mildew [J]. Frontiers of Agriculture in China, 2008, 2(3): 317-320.
[4] Sarris P, Abdelhalim M, Kitner M, et al. Molecular polymorphisms between populations ofPseudoperonosporacubensisfrom Greece and the Czech Republic and the phytopathological and phylogenetic implications [J]. Plant Pathology, 2009, 58(5): 933-943.
[5] Quesada-Ocampo L M, Granke L L, Olsen J, et al. The genetic structure ofPseudoperonosporacubensispopulations [J]. Plant Disease, 2012, 96(10): 1459-1470.
[6] Polat L, Baysal Ö, Mercati F, et al. Characterization ofPseudoperonosporacubensisisolates from Europe and Asia using ISSR and SRAP molecular markers [J]. European Journal of Plant Pathology, 2014, 139(3): 641-653.
[7] 刘艳玲, 张艳菊, 秦智伟, 等. 黄瓜霜霉菌卵孢子形成及rDNA-ITS序列分析[C]∥彭友良,朱有勇.中国植物病理学会2009年学术年会论文集. 北京:中国农业科学技术出版社,2009.
[8] 张艳菊, 张宏宇, 秦智伟, 等. 黄瓜霜霉菌毒性及分子多态性分析[J]. 东北农业大学学报, 2010(2): 25-30.
[9] Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging inBrassica[J]. Theoretical and Applied Genetics, 2001, 103: 455-461.
[10]陈碧云, 伍晓明, 陆光远, 等. 甘蓝型油菜花瓣缺失基因的图谱定位[J]. 遗传, 2006, 28(6): 707-712.
[11]Tang Lihua, Xiao Yang, Li Li, et al. Analysis of genetic diversity among ChineseAuriculariaauricularcultivars using combined ISSR and SRAP markers [J]. Current Microbiology, 2010, 61(2): 132-140.
[12]Liu Jingyu, Ying Zenghe, Liu Fang, et al. Evaluation of the use of SCAR markers for screening genetic diversity ofLentinulaedodesstrains [J]. Current Microbiology,2012,64(4): 317-325.
[13]Zhang Qiusheng, Xu Binglian, Liu Linde, et al. Analysis of genetic diversity among ChinesePleurotuscitrinopileatusSinger cultivars using two molecular marker systems (ISSRs and SRAPs) and morphological traits [J]. World Journal of Microbiology and Biotechnology, 2012, 28(5): 2237-2248.
[14]Zhang Y, Kang Y, Qin Y, et al. Genetic diversity of endangeredPolyporusumbellatusfrom China assessed using a sequence-related amplified polymorphism technique [J]. Genetics and Molecular Research, 2012, 11(4): 4121-4129.
[15]Ma Dalong, Yang Guoting, Mu Liqiang, et al. Application of SRAP in the genetic diversity ofTricholomamatsutakein northeastern China [J]. African Journal of Biotechnology, 2010, 9(38): 6244-6250.
[16]王关林, 方宏筠. 植物基因工程[M]. 北京: 科学出版社, 2009.
[17]Zhang Yanju, Pu Zijing, Qin Zhiwei, et al. A study on the overwintering of cucumber downy mildew oospores in China [J]. Journal of Phytopathology, 2012, 160(9): 469-474.
[18]Cohen Y, Rubin A E, Liu X L, et al. First report on the occurrence of A2 mating type of the cucurbit downy mildew agentPseudoperonosporacubensisin China [J]. Plant Disease, 2013, 97(4): 559.
[19]张艳菊, 王莹, 党悦嘉, 等.黄瓜霜霉菌对嘧菌酯抗药性的研究[J]. 东北农业大学学报, 2015(4): 23-28.
(责任编辑:杨明丽)
会 讯
第二十四届国际果树病毒及嫁接传播病害研究理事会(International Council for the Study of Virus and other Graft Transmissible Diseases of Fruit Crops,ICVF)于2017年6月4-9日在希腊瑟萨洛尼基召开。来自世界各地的近100位学者参加了会议。中国大陆有两人参加了此次会议,分别是华中农业大学的王国平教授以及中国农业科学院植物保护研究所的李世访研究员。该次大会包括40个口头报告以及37个墙报,内容涉及植物病毒、类病毒和植原体等方面。其中6位学者作了特邀大会报告,题目分别为:
1、希腊果树的栽培生产现状及展望
2、希腊果树病毒研究概况介绍
3、侵染果树的类病毒:从接种到高通量测序(NGS)
4、NGS技术在植物病毒研究中的应用以及对果树无病毒种苗调运产生的影响
5、果树植原体病害
6、通过抑制寄主植物DICER的表达打破植物的防御系统
此次大会的一个显著特点就是NGS技术在果树病毒及果树病毒病害研究中的广泛应用。近乎一半的报告不论是口头报告还是墙报中都提到了NGS技术,说明该技术在研究果树病毒上的应用空间还很大。会议期间参观了希腊瑟萨洛尼基附近的果园,考察了李痘病毒和桃潜隐花叶类病毒的发生分布以及危害状况。会后对ICVF的学术委员会进行了改选,新增委员5人。经过投票确定,第25届ICVF会议将于2020年在荷兰召开。
(李世访,中国农业科学院植物保护研究所 北京 100193)
Genetic diversity ofPseudoperonosporacubensisbased on SRAP markers
Bai Lingling1, Liu Xingfeng1, Chen Zhengyu1, Zhou Yingying2, Liu Li1, Zhang Yanju1, Zhang Ao1, Yang Sen1
(1.CollegeofAgriculture,NortheastAgriculturalUniversity,Harbin150030,China; 2.SeedServiceCenterofAchengDistrictinHarbin,HeilongjiangProvince,Harbin150039,China)
The genetic diversity ofPseudoperonosporacubensisisolates sampled from diseased cucumber plants in 12 major cucumber-producing regions of 5 provinces, including Heilongjiang, Liaoning, Hebei, Shandong, and Jiangsu in China, was analyzed by sequence-related amplified polymorphism (SRAP). Ten out of 35 SRAP primers used in the present study produced genomic DNA polymorphism in 77P.cubensisisolates, amplifying a total of 9 554 bands, 9 132 of which were polymorphic, accounting for 95.6%. The genetic distance of the 77P.cubensisisolates ranged from 0.60 to 1.00 based on analyses of the SRAP markers. The results showed that theP.cubensispopulations presented a high genetic diversity and 77 isolates could be classified into 8 groups. Most of the isolates from the same area were clustered in the same group by using the UPGMA method, suggesting that the genetic diversity ofP.cubensisisolates was related to their geographical origins.
cucumber downy mildew; molecular marker; SRAP; population genetics
2017-02-19
2017-04-05
国家自然科学基金 (31171792);黑龙江省自然科学基金重点项目(ZD2016003)
S 436.421.11
A
10.3969/j.issn.0529-1542.2017.04.027
* 通信作者 E-mail: zhangyanju1968@163.com
