5种仓储豆象的分子鉴定和检测
2017-08-09刘昌燕焦春海万正煌
刘昌燕, 李 莉, 焦春海, 万正煌
(湖北省农业科学院粮食作物研究所, 粮食作物种质创新与遗传改良湖北省重点实验室, 武汉 430064)
5种仓储豆象的分子鉴定和检测
刘昌燕, 李 莉, 焦春海, 万正煌*
(湖北省农业科学院粮食作物研究所, 粮食作物种质创新与遗传改良湖北省重点实验室, 武汉 430064)
本研究以在检疫中经常被截获的5种豆象为研究对象,通过对GenBank中5种豆象mtDNA COⅠ序列的查询,并进行序列比对,共设计了30对引物,通过筛选获得5对能分别鉴定5种豆象的特异引物,并用50、25、10、5、1、0.5、0.1、0.05 ng/μL 8个不同浓度系列的豆象DNA模板来检测灵敏度,结果表明,豌豆象和四纹豆象特异引物对的灵敏度为0.1 ng/μL,蚕豆象、菜豆象和绿豆象特异引物对的灵敏度为0.5 ng/μL。该方法可用于快速准确地鉴定5种豆象,对于豆象类害虫的检测监测具有重要意义。
豆象科; mtDNA COⅠ基因; 特异引物; 分子鉴定
豆象是豆象科昆虫的总称,分布于除南极之外的各大陆,尤其以亚洲的热带区以及中美和南美地区数量最多[1]。豆象科昆虫目前全世界记录约1 400种,分属于58个属[2]。该科昆虫大约有84%的种类取食豆科植物种子,成为栽培豆科植物的一类重要害虫[3]。1997年浙江宁海曾发生蚕豆象大规模为害,为害率高的达到80%以上,经济损失严重[4]。绿豆象严重为害绿豆、豇豆、鹰嘴豆、蚕豆和豌豆等多种豆类[5-6],主要在仓内蛀食储藏的豆粒,造成的损失达30%以上[7]。我国共记载有18种储藏物中豆象科昆虫,其中绿豆象Callosobruchuschinensis、蚕豆象Bruchusrufimanus和豌豆象Bruchuspisorum为我国主要的储藏豆类害虫;而菜豆象Acanthoscelidesobtectus和四纹豆象Callosobruchusmaculatus为我国规定禁止入境的检疫性害虫[8]。
目前口岸检验检疫部门对豆象科昆虫的鉴定主要以成虫的外部形态特征作为鉴定依据,但本研究涉及的5种豆象个体微小(约2~5 mm),且寄主范围、生活习性和种间外部形态极为相似,给种类鉴定工作带来了极大的困难[1,9]。同时,豆象能够以卵、幼虫、蛹、成虫随寄主果实和运输工具等进行远距离人为传播,且主要以卵、幼虫、蛹等非成虫形态为主,需将其饲养至成虫才能进行种类鉴定,整个检疫鉴定过程长达数周,不适应口岸快速检验检疫的要求,且容易混淆误检,无法适应实际工作的需要。我国在进口货物上截获的豆象不单单存在于豆类上,在玉米、小麦、棉花等货物中也存在,极大地增加了检疫的难度[2]。因此,寻找一种正确快速的鉴定方法对口岸检验检疫具有十分重要的指导意义。
分子标记是实现分子鉴定的重要技术手段,目前已应用于昆虫近缘种、地理种群的鉴别。细胞色素C氧化酶含有13个亚基,其中细胞色素氧化酶亚基Ⅰ(COⅠ)是细胞色素氧化酶的重要组成部分,编码该亚基的基因片段不同位置的进化速率有所不同,通过选取该基因的适宜基因片段进行测序和序列分析,进而在DNA水平上对物种进行正确地识别鉴定,已成为生物分类学研究的发展方向[10-11]。相比于其他分子标记技术,由于其具有统一性和标准性而成为生物分类学研究中应用最为广泛的分子手段之一[12-13]。目前,COⅠ基因已用于鳞翅目、膜翅目、半翅目等物种鉴定,并对基于形态学的物种分类进行了验证或修正[14-15]。
本研究以在检疫中经常被截获的5种豆象为研究对象,设计了以COⅠ为目标基因的特异性引物并对其特异性进行验证,旨在建立豆象类害虫快速分子鉴定技术,为有效截阻豆象类害虫的传入及进一步扩散,保障我国豆类安全生产提供技术保障。
1 材料与方法
1.1 供试虫源
本研究所用的昆虫样品、来源和寄主见表1。所有虫源样品均用体式显微镜进行鉴定,以确定种类。用于分子生物学试验的豆象标本用100%乙醇浸泡并保存于-20℃冰箱。
表1 5种豆象来源及寄主
Table 1 Sources and hosts of five bean weevils

昆虫样品Sample来源Source寄主Host绿豆象Callosobruchuschinensis北京门头沟绿豆Vignaradiata豌豆象Bruchuspisorum湖北武汉豌豆Pisumsativum蚕豆象Bruchusrufimanus湖北武汉蚕豆Viciafaba四纹豆象Callosobruchusmaculatus重庆永川绿豆Vignaradiata菜豆象Acanthoscelidesobtectus云南曲靖菜豆Phaseolusvulgaris
1.2 主要试剂
TaqDNA聚合酶、蛋白酶K、DL1000 DNA Marker、Tris、EDTA、SDS均购自TaKaRa公司。胶回收试剂盒购自天根生化科技有限公司,其他生化试剂均为国产分析纯。
1.3 基因组DNA的提取
豆象基因组DNA的提取参照Sunnuck等[16]的盐析法并稍作改进。将单头豆象于1.5 mL离心管中研磨粉碎,加入300 μL TENS (50 mmol/L Tris, pH 7.5, 400 mmol/L NaCl, 20 mmol/L EDTA, 0.5% SDS)和100 μg/mL蛋白酶K,于37℃水浴4 h,加入85 μL 5 mol/L NaCl剧烈振荡15 s;14 000 r/min离心5 min, 取上清液,加入等体积无水乙醇沉淀DNA,12 000 r/min离心5 min, 用70%乙醇洗涤沉淀2次,沉淀充分干燥后,加入30 μL蒸馏水溶解DNA,-20℃保存备用。
1.4 特异引物对的设计
通过GenBank中登录的豌豆象(EF570097)、四纹豆象(AY625417)、蚕豆象(AY997313)、菜豆象(KF157281)和绿豆象(AY625416)的mtDNA CO Ⅰ序列,用DNAMAN软件对目标种的mtDNA CO Ⅰ序列进行比对分析,根据分析结果,针对目的基因序列人工设计特异引物,并用Primer 5对引物进行评估,检查引物Tm值、GC%、错配、二聚体和发夹结构等,并用GenBank中的BLAST程序检查同源序列[17]。本研究共设计了30对引物,通过筛选获得能分别特异鉴定5种豆象的特异引物5对(表2),引物筛选的原则是所有引物在所设计的PCR反应体系和反应条件下能特异地鉴定出某一种豆象。引物由武汉擎科生物技术有限公司合成。
1.5 PCR扩增
分别以单头豆象的基因组DNA为模板,用表2的特异性引物进行PCR扩增,以水作阴性对照。PCR反应体系(25 μL):10×buffer(含Mg2+)2.5 μL、2.5 mmol/L dNTPs 2.5 μL、10 μmol/L引物(上、下游引物)各1.0 μL、2.5 U/μLTaq酶 0.2 μL、基因组DNA 1 μL,ddH2O 16.8 μL。PCR反应条件:95℃预变性5 min;95℃变性30 s、54℃退火30 s、72℃延伸1 min,35个循环;72℃ 10 min;4℃保存。取5 μL PCR产物进行1.2%琼脂糖凝胶电泳检测,溴化乙锭(EB)染色,在凝胶成像系统中观察并成像分析。
表2 引物序列及扩增产物
Table 2 Primer sequences and amplification products

引物 Primer豆象种类 Species序列5'-3' Sequence目标片段长度/bp LengthBp1Bp2Cm1Cm2Br1Br2Ao1Ao2Cc1Cc2豌豆象Bruchuspisorum四纹豆象Callosobruchusmaculatus蚕豆象Bruchusrufimanus菜豆象Acanthoscelidesobtectus绿豆象CallosobruchuschinensisAGTTGGGGGTTTAACTGGTGTATGTTAAACCTGTAAATAATGGGGGTCCCTACTGGAATTAAAGTAAAAGTAACTCCGGTAAAAAGGAGTGGGAGGATTGACTGGTGTTAGTTAATCCTGTAAATAATGAACATGATAGCTATCGGATTAACCTAAAAATATAGTAATAAATTGGGAGTACCAACCGGAATCAAAGTAAGTGACACCAGTAAAAAGAGGA166276167432274
1.6 PCR产物回收测序
PCR产物采用天根琼脂糖凝胶回收试剂盒进行切胶回收纯化,回收纯化后的PCR产物由武汉擎科生物科技有限公司进行测序。
1.7 特异引物对的灵敏度检测
供试豆象的基因组DNA用蒸馏水进行系列稀释,得到基因组DNA浓度分别为:50、25、10、5、1、0.5、0.1、0.05 ng/μL。分别取每种稀释液1 μL为模板(水为阴性对照),用上述5对特异性引物分别对5种豆象的基因组DNA进行PCR扩增,PCR产物在1.2%琼脂糖凝胶电泳上检测。
2 结果与分析
2.1 引物特异性验证
用设计的特异性引物对所有供试豆象的基因组DNA进行PCR扩增,其中Bp1/Bp2仅在豌豆象基因组DNA扩增出166 bp特异性条带(图1);Cm1/Cm2仅在四纹豆象基因组DNA中扩增出276 bp特异性条带(图2);Br1/Br2仅在蚕豆象基因组DNA中扩增出167 bp特异性条带(图3);Ao1/Ao2仅在菜豆象中扩增出432 bp特异性条带(图4);Cc1/Cc2仅在绿豆象中扩增出274 bp特异性条带(图5),其他豆象基因组DNA及水对照均无扩增片段。表明设计的引物在供试豆象范围内具有特异性。
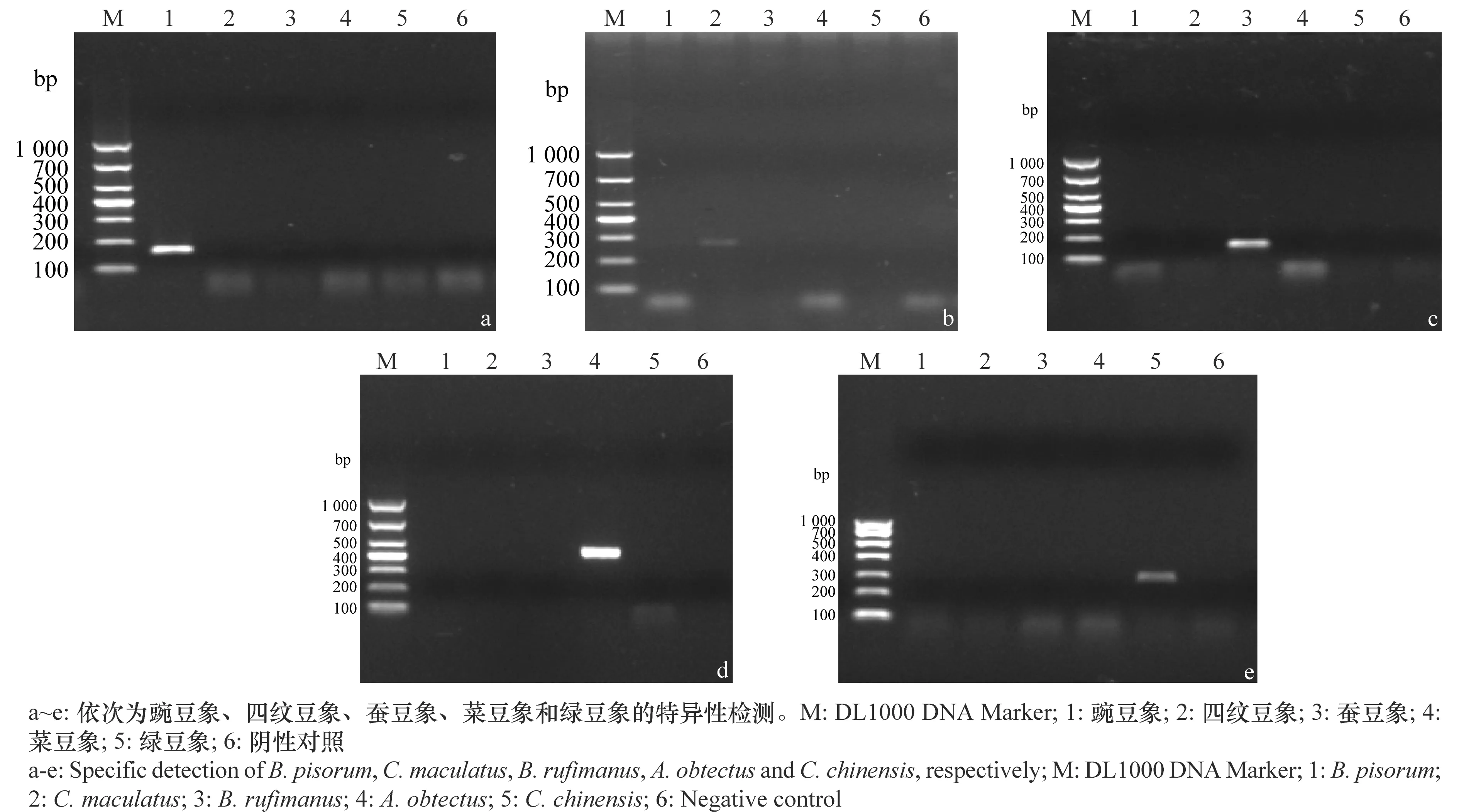
图1 5种豆象的特异性检测Fig.1 Detection of five bean weevils with specific primers
2.2 PCR产物测序验证
分别对扩增出的PCR产物进行测序和序列分析,对产物的特异性进行进一步确认。经测序表明,豌豆象特异引物对扩增的PCR产物与原序列有5个碱基的差异,四纹豆象特异引物对扩增的PCR产物序列与原序列有4个碱基的差异,蚕豆象特异引物对扩增的PCR产物与原序列有9个碱基的差异,菜豆象特异引物对扩增的PCR产物与原序列有6个碱基的差异,绿豆象特异引物对扩增的PCR产物与原序列没有碱基的差异,造成这些差异的原因可能是测序的差异或者不同地理种群间序列存在差异。
2.3 特异引物灵敏度检测
PCR方法的检测灵敏度分别用50、25、10、5、1、0.5、0.1、0.05 ng/μL等8种不同浓度的豆象DNA为模板来确定。由图2可知,当模板浓度为0.05 ng/μL时,豌豆象和四纹豆象特异引物对均没有扩增出特异性条带,当模板浓度为0.1 ng/μL及以上时,均产生特异性条带,而水对照即CK无扩增片段(图2)。由此可知,应用本试验的PCR鉴定方法可以检测到浓度为0.1 ng/μL的豌豆象和四纹豆象基因组DNA,灵敏度非常高。从图8~10可以看出,蚕豆象、菜豆象和绿豆象特异引物对的灵敏度为0.5 ng/μL。
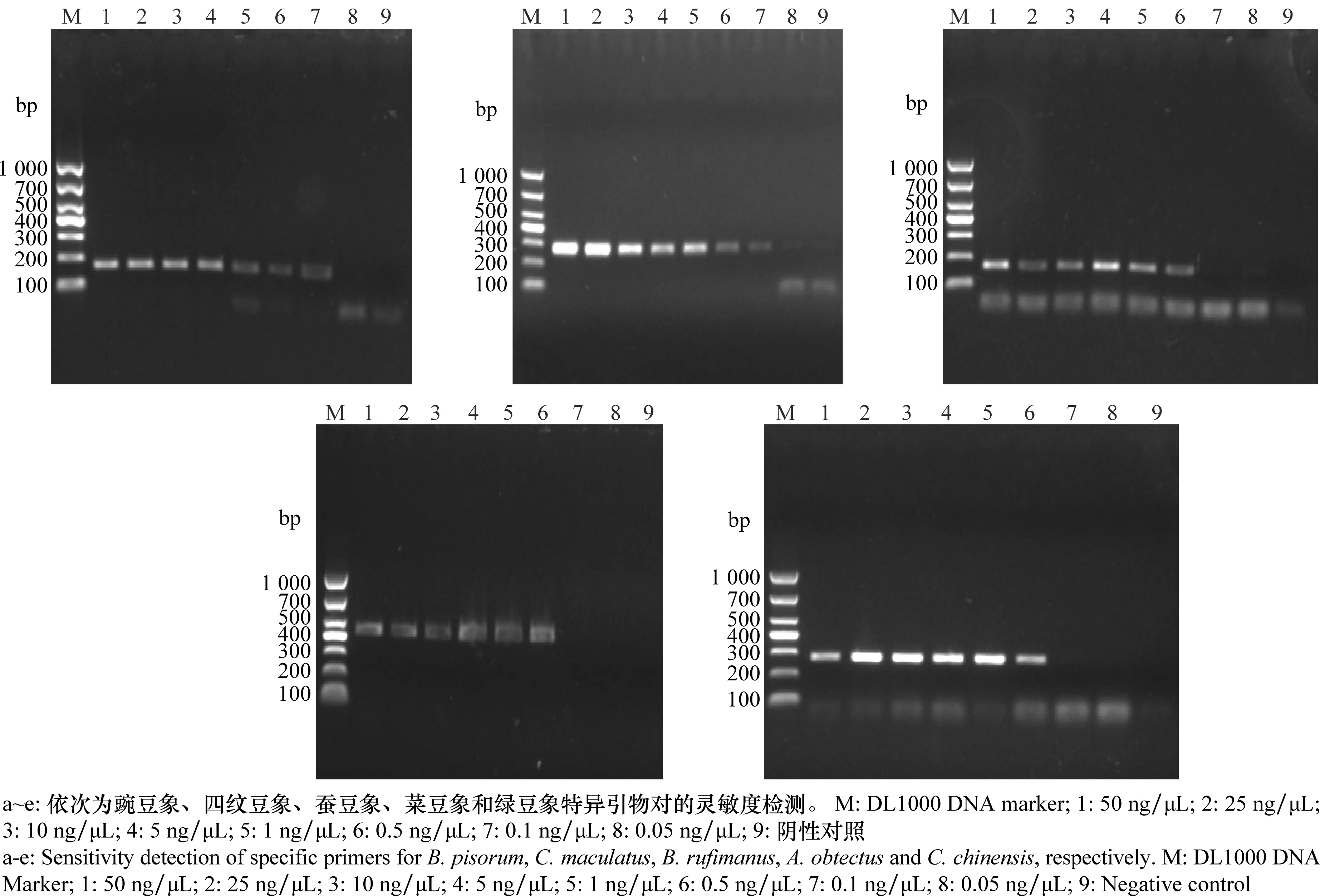
图2 特异引物的灵敏度检测Fig.2 Sensitivity detection of specific primers for five bean weevils
3 讨论
现代分子生物学技术尤其是分子标记技术为研究生物之间的进化关系开辟了新手段。PCR的广泛应用以及基因测序的精确和简便使分子系统分析达到空前的水平。许多研究者利用RFLP或RAPD标记分析物种或地理种群的遗传多态性,并据此推导其亲缘关系。如顾耕等[18]采用RAPD技术对生活在辣椒、番茄和烟草上的棉铃虫进行了寄主种群遗传分化的研究。魏晓棠等[19]利用AFLP技术对我国4个地理种群的棉褐带卷蛾进行遗传结构分析,为不同地理种群的棉褐带卷蛾的分类鉴定和遗传变异分析提供参考依据。但RFLP需要筛选大量的限制酶,而RAPD需要筛选大量随机引物。以核酸序列测定为基础,扩增单一的稳定条带,实现豆象的分子鉴定具有方便、稳定和可靠的优点。顾杰等[20-21]结合PCR和酶切技术,从分子水平对鹰嘴豆象和四纹豆象进行了快速检测,鉴定灵敏度达10 ng/μL,并检测了四纹豆象4个地理种群的Cytb和COⅠ基因序列,对不同地理种群间遗传结构多态性进行了研究。本研究通过序列搜索比对,并对设计的相关引物进行筛选鉴定,获得5对能特异鉴定5种豆象的特异性引物,灵敏度达0.5 ng/μL以上,且该方法具有不受豆象发育阶段和环境条件的影响,对于非成虫态的样品及形态结构不完整的成虫样品,均能从基因序列上获得准确、可靠的鉴定信息等优点。该技术操作简便快捷,缩短了检测时间,提高了工作效率。因此,该技术可以用于豆象的检测鉴定工作,并在检疫性害虫的检疫检验与检测监测中具有重要应用价值。
[1] 张生芳, 刘永平, 武增强. 中国储藏物甲虫[M]. 北京: 中国农业出版社, 1998.
[2] 喻梅, 谢令德, 徐广文. 六种检疫性储粮豆象防治研究进展[J]. 粮食储藏, 2006(6): 9-12.
[3] Johnson C D.Biosystematics of the Arizona, California, and Oregon species of the seed beetle genusAcanthoscelidesSchilsky (Coleoptera: Bruchidae)[M]. University of California Publications in Entomology, 1970, 59: 1-116.
[4] 叶秀亦, 钱永谦, 谢时秀, 等. 宁海地区蚕豆象发生危害及其防治[J]. 植物保护, 1997, 23(5): 31-32.
[5] 刘昌燕, 李莉, 陈宏伟, 等. 绿豆象在不同豆类上的生长发育研究[J]. 湖北农业科学, 2015, 54(22): 5610-5617.
[6] 刘昌燕, 万正煌, 仲建锋, 等. 绿豆象的辐照致死效应研究[J]. 湖北农业科学, 2014, 53(9): 4607-4610.
[7] 仲建锋, 万正煌, 李莉, 等. 低温和高温对仓储绿豆象的防治效果[J]. 中国农业科学, 2013, 46(1): 54-59.
[8] 刘昌燕, 焦春海, 仲建锋, 等. 食用豆虫害研究进展[J]. 湖北农业科学, 2014, 53(24): 5908-5912.
[9] 陈新, 魏春艳, 任炳忠, 等. 豆象属昆虫检疫重要性概述[J]. 植物检疫, 2013, 27(1): 63-67.
[10]Hebert P D N, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes [J]. Proceedings of the Royal Society B: Biological Sciences, 2003, 270(1512): 313-321.
[11]Floyd R, Lima J, deWaard J, et al. Common goals: policy implications of DNA barcoding as a protocol for identification of arthropod pests [J]. Biological Invasions, 2010, 12(9): 2947-2954.
[12]Shufran K A, Puterka G J. DNA barcoding to identify all life stages of holocyclic cereal aphids (Hemiptera: Aphididae) on wheat and other Poaceae [J]. Annals of the Entomological Society of America, 2011, 104(1): 39-42.
[13]Mehrdad H, Daniel H J, John M B, et al. DNA barcodes distinguish species of tropical Lepidoptera [J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(4): 968-971.
[14]刘慎思, 张桂芬, 万方浩. 基于mtDNA COⅠ基因的离腹寡毛实蝇属常见种DNA条形码识别和系统发育分析[J]. 昆虫学报, 2014, 57(3): 343-355.
[15]Acs Z, Challis R J, Bihari P, et al.Phylogeny and DNA barcoding of inquiline oak gallwasps (Hymenoptera: Cynipidae) of the Western Palaearctic [J]. Molecular Phylogenetics and Evolution, 2010, 55(1): 210-225.
[16]Sunnuck P, Hales D F.Numerous transposed sequences of mitochondrial cytochrome oxidase I-II in aphids of the genusSitobion(Hemiptera: Aphididae)[J]. Molecular Biology and Evolution, 1996, 13(3): 510-524.
[17]Muraji M, Nakahara S.Discrimination among pest species ofBactrocera(Diptera: Tephritidae) based on PCR-RFLP of the mitochondrial DNA[J]. Applied Entomology and Zoology, 2002, 37(3): 437-446.
[18]顾耕, 张迎春, 王思芳. 棉铃虫不同寄主种群遗传分化的RAPD分析[J]. 华东昆虫学报, 2002, 11(2): 30-34.
[19]魏晓棠, 张成标, 张京宣, 等. 基于AFLP方法对不同地理种群棉褐带卷蛾遗传分化的研究[J]. 应用昆虫学报, 2015, 50(3): 686-692.
[20]顾杰, 王莉萍, 毛雅琴, 等. 鹰嘴豆象与四纹豆象的分子检测技术[J]. 植物保护学报, 2010, 37(3): 211-216.
[21]顾杰, 毛雅琴, 王莉萍, 等. 四纹豆象不同地理种群的遗传分化[J]. 昆虫学报, 2009, 52(12): 1349-1355.
(责任编辑:杨明丽)
Molecular identification and detection of five bean weevils
Liu Changyan, Li Li, Jiao Chunhai, Wan Zhenghuang
(InstituteofFoodCrops,HubeiAcademyofAgriculturalSciences,HubeiKeyLaboratoryofFoodCropGermplasmandGeneticImprovement,Wuhan430064,China)
Molecular identification of five bean weevils that were frequently intercepted from customs was studied in this paper. Based on the mtDNA COⅠ gene sequences ofCallosobruchuschinensis,Bruchusrufimanus,B.pisorum,AcanthoscelidesobtectusandC.maculatusfrom GenBank, 30 pairs of primers were designed, and the five bean weevils can be specifically identified by PCR amplification with five pairs of specific primers. Moreover, the sensitivity of PCR was detected at 50,25,10,5,1,0.5,0.1 and 0.05 ng/μL genomic DNA ofC.chinensis,B.rufimanus,B.pisorum,A.obtectusandC.maculatus,respectively.The results showed that the sensitivity forB.pisorumandC.maculatuswas 0.1 ng/μL, while 0.5 ng/μL for the other three. The method provides a rapid and reliable tool for identification of the five bean weevils, which could be applied in detecting and monitoring of bean weevil pests.
Bruchidae; mtDNA COⅠ gene; specific primers; molecular identification
2016-09-30
2017-01-10
国家现代农业产业技术体系建设专项(CARS-09); 科技部国际合作重点项目(2011DFB31620); 湖北省农业科技创新中心资助项目(2016-620-000-001-006)
S 379
A
10.3969/j.issn.0529-1542.2017.04.022
* 通信作者 E-mail: zhwan168@163.com
