微卫星技术在NIH小鼠群体遗传结构分析中的应用
2017-08-04王锡岩李晓晖闵凡贵岳秉飞
郭 羽,王 洪,魏 杰,王锡岩,李晓晖,闵凡贵,岳秉飞*
(1.北京天坛生物制品股份有限公司,北京 100176; 2.中国食品药品检定研究院,北京 100050; 3.广东省实验动物监测所,广东省实验动物重点实验室,广州 510663)
微卫星技术在NIH小鼠群体遗传结构分析中的应用
郭 羽1#,王 洪2#,魏 杰2,王锡岩1,李晓晖1,闵凡贵3,岳秉飞2*
(1.北京天坛生物制品股份有限公司,北京 100176; 2.中国食品药品检定研究院,北京 100050; 3.广东省实验动物监测所,广东省实验动物重点实验室,广州 510663)
目的 应用微卫星DNA技术对A单位NIH小鼠和B单位NIH小鼠的遗传结构进行对比分析。方法 利用30个微卫星位点对2个NIH小鼠种群进行PCR扩增和STR扫描,借助统计软件Popgene 1.32进行群体遗传结构分析。结果 A单位NIH群体获得74个等位基因型,平均杂合度为0.3108,多态性信息含量为0.2637;B单位NIH群体获得76个等位基因型,平均杂合度为0.3257,多态性信息含量为0.2777。群体间比较显示,群体间分化系数为0.3932,遗传一致性指数0.3971,遗传距离为0.9235,群体差异显著。结论 两个NIH小鼠群体间存在严重的遗传分化,形成了两个不同的封闭群群体。
封闭群;NIH小鼠;微卫星DNA;遗传结构
N:NIH小鼠(SW),叫作NIH小鼠,也叫作NIH (BXS)小鼠,于1936年Walter Reed Army Institute of Research选取12只瑞士种小鼠育种,起初使用近交方式交配(BXS)繁殖传代,后又封闭繁育达十年以上培育而成。NIH小鼠成为有近交历史的封闭群动物,具有遗传稳定,个体差异较小的优点。NIH(SW)为屏障环境生活的小鼠,体型小,产仔多,生长繁殖快,雄性好斗,饲养管理方便,对环境的适应性和对疾病的抵抗力强。1977年WHO建议各国使用NIH小鼠作为百日咳菌苗和破伤风检定用标准动物,结合我国以KM小鼠检定百日咳时出现较大结果差异的问题,卫生部防疫司决定引进NIH小鼠。1980年7月2日由北京生物制品研究所引入该种小鼠,在国内繁育并推广使用。1991年又从中国药品生物制品检定所(现已更名为:中国食品药品检定研究院)引进400对NIH小鼠更新血缘,两个NIH(H-2q型)群体生产至今。为降低近交系数,每群500对NIH小鼠,严格按照封闭群生产繁殖原则进行独立保种,闭锁繁育。
NIH小鼠是我国用量较大的小鼠之一,我国各地区的NIH小鼠种群并不多,主要存在于国家实验动物种子中心和部分实验动物生产企业,以及生物制药企业。NIH小鼠广泛应用于药理学、毒理学、细菌学等领域,以及药品、生物制品的生产与检定工作中,还可用于制备单克隆抗体。NIH小鼠由于对病毒和细菌比较敏感,中国药典上规定:百日咳疫苗原液效价测定、百日咳疫苗原液毒性检查、乙型脑炎灭活疫苗(Vero细胞)免疫原性检查、重组乙型肝炎疫苗(CHO细胞)效价测定,以上四项试验必须使用NIH小鼠进行[1]。那么,封闭群小鼠的群体遗传质量必然决定应用NIH小鼠的疫苗检定工作的稳定性和有效性。
此前,我们测定了A单位NIH小鼠群体的血液生理生化指标和脏器系数,我们发现测定结果与B单位的NIH小鼠群体的一些指标存在一定的差异,研究结果表明A单位NIH小鼠已形成群体自身特有的生物学特性参数[2]。本研究将应用微卫星DNA技术比较分析A单位NIH群体和B单位NIH群体的遗传结构。
1 材料和方法
1.1 实验动物
随机选取A单位NIH小鼠30只,8周龄,SPF级,雌雄各半【SCXK(京)2015-0012】【SYXK(京)2011-0008】。随机选取B单位NIH小鼠30只,8周龄,SPF级,雌雄各半【SCXK(京)2014-0013】【SYXK(京)2011-0008】。
1.2 试剂和仪器设备
Taq酶、50 bp DNA marker、dNTP、琼脂糖均购自Takara公司;其余试剂均为国产。日本Biospec-mini紫外分光光度计,Mettler GB303电子天平,Bio-Rad MyCycler型PCR仪,Bio-Rad Model 3000Xi型电泳仪,Gel Logic 212PRO 紫外与可见光凝胶分析装置。
1.3 DNA提取
动物安乐死后,取尾部100 mg,-20℃冷冻保存备用。将100 mg组织加入到含有蛋白酶K的抽提缓冲液中消化过夜,酚/氯仿法提取DNA,经紫外分光光度计检测吸光度A260/A280值在1.8 ~ 2.0之间,样品合格。取适量稀释成50~100 ng/μL作为DNA模板,4℃保存备用。
1.4 PCR扩增和STR扫描
30对微卫星引物分别以FAM、HEX、TAMRA进行荧光标记,由北京美吉桑格生物医药有限公司合成[3]。
A单位的30份NIH小鼠样本基因组DNA和B单位的30份封闭群NIH小鼠样本基因组DNA作为PCR扩增模板,PCR反应体系为20 μL,其中:10× buffer 2 μL,上下游引物(100 pmol/μL)各1 μL,4× dNTP 100 μmol/L 1.2 μL,Taq酶0.2 μL,50 ~ 100 ng 基因组DNA 1 μL,纯水(ddH2O)13.6 μL。扩增条件:94℃预变性5 min, 94℃变性30 s,退火温度30 s,72℃延伸30 s,35个循环,接72℃继续延伸8 min,扩增产物4℃保存。
取扩增产物5 μL经1.5%的琼脂糖凝胶电泳150 V,30 min,拍照成相。PCR扩增产物以1∶3∶5 混合。取1 μL进行STR扫描检测。
1.5 STR扫描结果的判读
利用GeneMapperv4.0软件,进行结果分析。扫描结果出现两种波形:一种为纯合基因型,只有一个主波;另一种为杂合基因型,有两个主波。根据软件读出波峰处的扩增产物的bp数。由GeneMapperv4.0软件读出该群体30个样本在每个微卫星位点的扩增片断大小。每个位点的等位基因根据扩增片断从小到大顺序排列记录为a,b,c,d等。将所有样本的每个微卫星位点的基因型以ab,bb等形式输入Popgene 1.32软件的数据文件以供统计分析。
1.6 数据统计分析
运用Popgene 1.32软件,分析得到在每个微卫星位点上30个样本的等位基因数、有效等位基因数、香隆指数和平均杂合度。利用Cervus 3.0软件计算群体平均多态信息含量(polymorphism information content,PIC)。按照Nei(1978)氏法计算两个单位NIH小鼠群体的遗传一致性指数和遗传距离[4]。
2 结果
2.1 A单位NIH小鼠群体的测定结果
A单位NIH小鼠微卫星测定结果见表1。在30个微卫星位点共检测出74个等位基因,各基因座等位基因数1 ~ 5个不等,其中5个基因座(D5Mit48、D10Mit12、D15Mit5、D12Nds11、D14Mit3)呈现单态性,只有1个等位基因,平均等位基因数为2.4667。平均杂合度为0.3108。Hardy-Weinberg遗传平衡P值中,有17个位点P> 0.05。利用Cervus 3.0软件统计得到A单位NIH群体的30个微卫星位点的平均多态性信息含量(PIC)为0.2637。剔除5个单态性位点,剩余25个位点的平均多态信息含量(PIC)为0.3165。
注:P> 0.05表示该位点处于遗传平衡;P< 0.01 表示该位点处于遗传分化。
Note.P> 0.05 indicates the genetic equilibrium in the loci;P< 0.01 indicates the genetic differentiation in the loci.
2.2 B单位NIH小鼠群体的测定结果
B单位NIH小鼠微卫星测定结果见表2。在30个微卫星位点共检测出76个等位基因,各基因座等位基因数1~6个不等,其中5个基因座(D10Mit12、D15Mit5、D12Nds11、D13Mit3、D14Mit3)呈现单态性,只有1个等位基因,平均等位基因数为2.5333。平均杂合度为0.3257,Hardy-Weinberg遗传平衡P值中,有20个位点P>0.05。利用Cervus 3.0软件统计得到B单位NIH群体的30个微卫星位点的平均多态性信息含量(PIC)为0.2777。剔除5个单态性位点,剩余25个位点的平均多态信息含量(PIC)为0.3365。
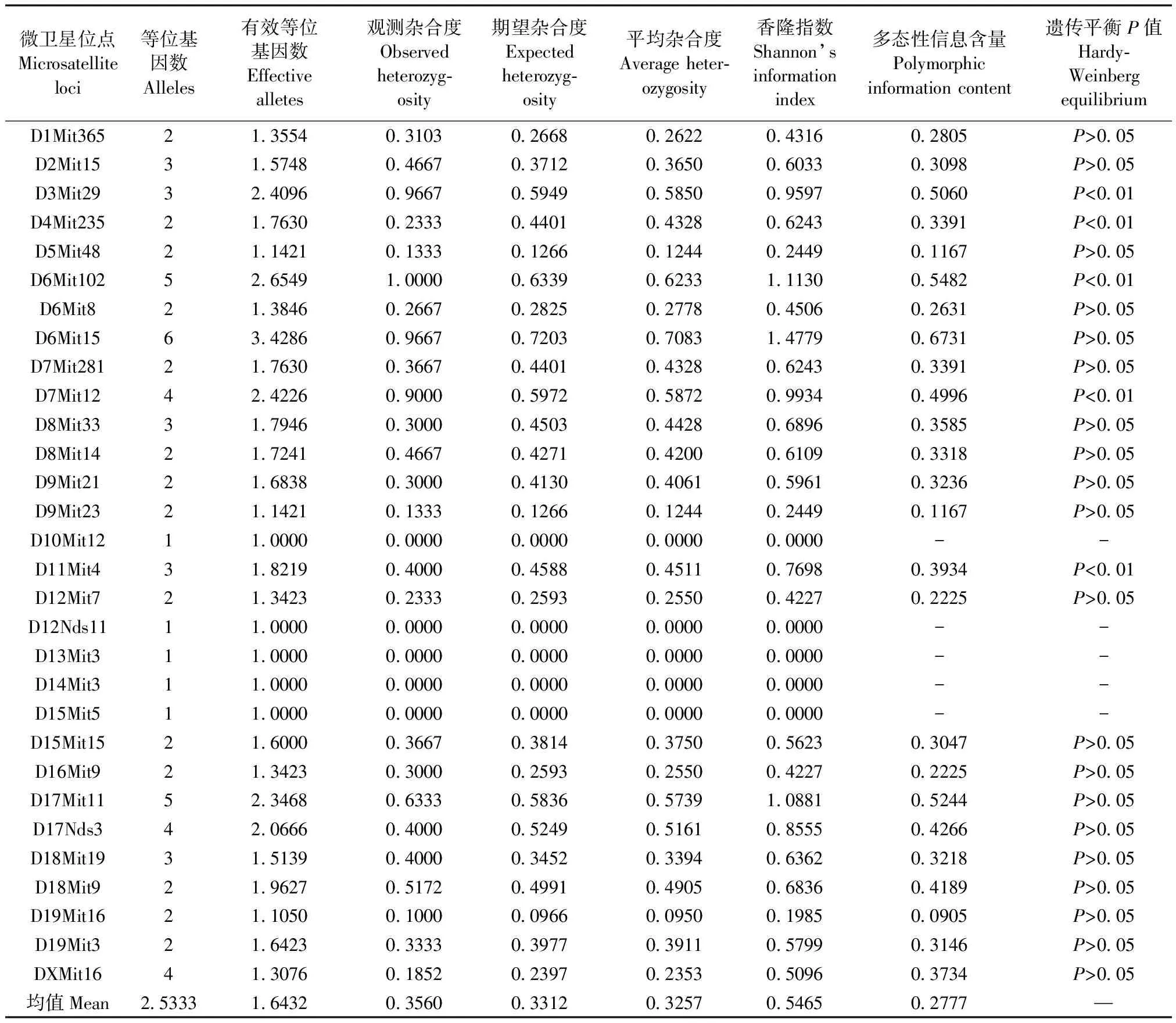
表2 B单位NIH小鼠在30个微卫星位点上的基因频率、平均杂合度、多态性信息含量Tab.2 Gene frequency, average heterozygosity and polymorphism information content of 30 microsatellite loci in the NIH mice from Unit B
注:P> 0.05表示该位点处于遗传平衡;P< 0.01 表示该位点处于遗传分化。
Note.P> 0.05 indicates the genetic equilibrium in the loci;P< 0.01 indicates the genetic differentiation in the loci.
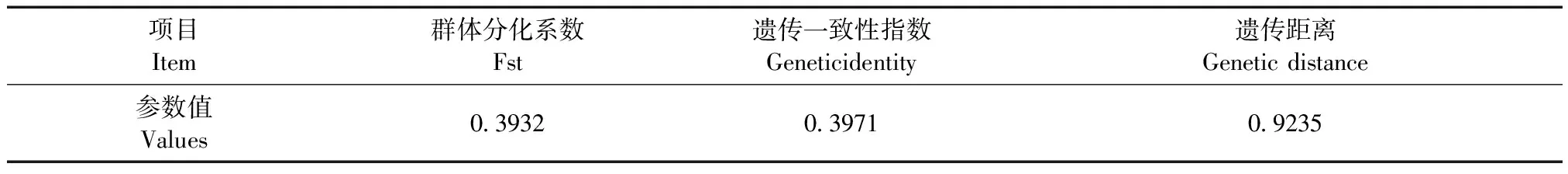
表3 两个NIH小鼠群体的遗传参数Tab.3 Genetic parameters of the two NIH mice populations
2.3 两个群体遗传相似性比较
将A单位NIH小鼠微卫星检测结果与B单位的结果进行比较,发现等位基因数、平均杂合度、PIC等相关指标有一定的差异,B单位的结果略高于A单位的结果。两个群体的PIC值均位于0.25 ~ 0.5,属于中等程度的多态性。在测定的30个位点中,两个群体均有5个单态性位点,其中4个位点(D10Mit12、D12Nds11、D14Mit3、D15Mit5)一致。
进一步分析两个群体的群间分化系数(F-statistics,Fst)、遗传一致性指数和遗传距离,结果见表3,群间分化系数为0.3932,遗传一致性指数为0.3971,遗传距离为0.9235。
3 讨论
3.1 平均杂合度、多态性信息含量和遗传平衡
杂合度或群体平衡状态是群体遗传结构分析最直接和最有效的方法,杂合度值越高所在群体的遗传多样性就越丰富,较理想的封闭群体的杂合度值应介于0.5 ~ 0.7之间。本研究中A单位NIH小鼠群体的平均杂合度是0.3108,B单位的NIH小鼠群体的平均杂合度是0.3257。B单位的杂合度略高于A单位的杂合度。显然,两个群体的杂合度都没有达到0.5 ~ 0.7之间,都不是理想的封闭群小鼠群体。多态性信息含量是表示微卫星座位多态性高低的一个指标。当微卫星座位PIC > 0.5时,表明该遗传标记具有高度的可提供遗传信息性,为高度多态性座位;当 0.25 < PIC < 0.5时,表明该遗传标记能够较为合理的提供遗传信息,为中度多态性座位; 当 PIC < 0.25时,表明该遗传标记可提供的遗传信息较差,为低度多态性座位。本研究中A单位NIH小鼠群体的平均多态性信息含量为0.2637,B单位的NIH小鼠群体的平均多态性信息含量为0.2777。两个群体的多态性信息含量均介于0.25 ~ 0.5之间,为中度多态性群体,但是两个群体的PIC值均濒临0.25,已经出现趋于低度多态性群体的趋势。本研究结果提示保种机构有必要采取有效措施提高群体的平均杂合度和多态性信息含量。本研究中应用的微卫星方法曾用于封闭群KM小鼠群体的遗传结构分析[3],得到上海KM小鼠群体的平均杂合度为0.5342,平均多态性信息含量为0.4735。说明上海KM小鼠群体是较理想的封闭群小鼠群体,也说明该方法用于封闭群小鼠群体遗传结构分析的有效性。
Hardy-Weinberg遗传平衡检验结果显示,A单位NIH小鼠有10个位点出现遗传分化(P< 0.01),高于B单位NIH小鼠,后者仅有5个位点(P< 0.01)。B单位NIH小鼠群体的遗传多态性更为丰富。在各机构封闭繁育过程中,由于群体规模的限制,造成基因多态性的递减,逐步形成了不同NIH小鼠群体的平均杂合度、多态性信息含量和遗传平衡状态不完全一致的状况。
3.2 群间分化系数、遗传一致性指数和遗传距离
群间分化系数Fst是测定群体间遗传分化的主要参数,Fst < 0.05时,表示群体间有轻度遗传分化;0.05 ≤ Fst < 0.15时,表示群体间有中度遗传分化;0.15 ≤ Fst < 0.25时,表示群体间有中重度遗传分化;Fst ≥ 0.25时,表示群体间有重度遗传分化,群体差异显著[5]。本研究中两个NIH小鼠群体的群间分化系数为0.3932,大于0.25,表明两个群体间产生了重度的遗传分化,两个群体差异显著。遗传一致性指数与遗传距离是反映群体遗传相似性的两个指标,两者呈负对数关系,其中,遗传距离为显性函数,且当遗传距离大于0.2时,认为两个群体为同属不同种;在0.03 ~ 0.2之间时为同属同种[6]。本研究中两个NIH小鼠群体的遗传一致性指数偏低,为0.3971;遗传距离超过了0.2,为0.9235,表明这两个群体已经不属于同一品(种)系的动物。依据本研究应用的30个微卫星位点提供的多态性信息可初步判定,该两个群体NIH小鼠间存在严重的遗传分化,形成了两个不同的封闭群群体。基于此,本机构将保种的NIH小鼠命名为A单位NIH小鼠,符合遗传学要求。
3.3 生化标记检测法与微卫星DNA检测法的比较
几年前,魏杰等[7]分析了同样两个封闭群NIH小鼠群体基于14个生化标记位点的多态性差异,显示群间分化系数和遗传距离均非常小,遗传一致性指数高。与本研究中应用30个微卫星位点得到的群体遗传结构参数大相径庭。主要原因有两个方面:一方面是因为两种方法的测定时间不同,存在群体遗传结构发生巨大变化的可能。另一方面,生化标记方法与微卫星DNA方法选取的位点数量和涵盖的染色体数目都有很大的差异,导致两种方法得到不同的结果。通过两种评价方法获得的研究结果均可以为封闭群动物的保种和繁育提供必要的参考。
综上,本研究结果提示,源于不同保种机构的封闭群NIH小鼠的群体遗传结构特征存在一定差异,应为两个NIH小鼠群体补充新的血缘,提高封闭群小鼠的群体杂合度,改善封闭群小鼠的遗传质量。同时,保种机构有必要定期进行遗传质量监测,建立本机构的基础数据库,从而为其繁育与应用提供有益的参考。
致谢:本论文的研究内容得到了首都医科大学陈振文教授的指导,特此表示感谢!
[1] 国家药典委员会编,中华人民共和国药典2015版,中国医药科技出版社。
[2] 郭羽,魏杰,王锡岩,等. Tiantanbio: NIH小鼠血液生理生化和脏器指标的测定分析[J]. 实验动物科学,2016, 33(6): 45-49.
[3] 王洪,杜小燕,徐平,等. 上海KM小鼠种子群体遗传状况分析[J],中国比较医学杂志,2014,24(12): 27-32.
[4] Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals [J]. Genetics,1978,89(3): 583-590.
[5] Ballous F, Lugon-Moulin N. The estimation of population differentiation with microsatellite markers[J]. Mol Ecol, 2002, 11(2): 155-165.
[6] Thorpe JP. The molecular dock hypothesis: biochemical evolution, genetic differentiation and systematics[J]. Annu Rev Ecol Syst, 1982, 13: 139-168.
[7] 魏杰, 王洪, 李芳芳, 等. 两个封闭群NIH小鼠群体的遗传监测结果的比较分析[J]. 中国比较医学杂志, 2015, 25(5): 33-36.
Application of microsatellite technology in the genetic structure analysisof NIH mice
GUO Yu1#, WANG Hong2#, WEI Jie2, WANG Xi-yan1, LI Xiao-hui1, MIN Fan-gui3, YUE Bing-fei2*
(1.Beijing Tiantan Biological Product Co. Ltd., Beijing 100176, China; 2.National Institutes for Food AndDrug Control, Beijing 100050; 3.Guangdong Laboratory Animals Monitoring Institute, Guangdong Provincial KeyLaboratory of Laboratory Animals, Guangzhou 510663)
Objective To compare and analyze the genetic structure of NIH mice bred in Unites A and B, using microsatellite technology. Methods Thirty SPF 8-week old outbred NIH mice (half male and half female) of each population were randomly chosen from the Units A and B, respectively. PCR amplification and STR scan were performed to determine the genetic characteristics of two outbred populations using microsatellite loci, and the population genetic structure was analyzed with statistical software Popgene 1.32. Results In the NIH mouse population form the Unit A, 74 alleles were obtained, with an average heterozygosity of 0.3108 and polymorphism information content of 0.2637. In the NIH mouse population from the Unit B, 76 alleles were obtained, with an average heterozygosity of 0.3257 and polymorphism information content of 0.2777. The inter-population comparison showed that genetic differentiation coefficient Fst was 0.3932, the genetic identity was 0.3971, and the genetic distance was 0.9235. The population difference was significant. Conclusions There is serious genetic differentiation between the two NIH mice populations, resulting in the formation of two different closed populations.
Outbred population; NIH mice; Microsatellites DNA; Genetic structure
郭羽(1980-),男,工程师,大学,研究方向:比较医学研究;王洪(1977-),女,副研究员,硕士,研究方向:实验动物质量控制。#共同第一作者
岳秉飞(1960-),男,研究员,博士,研究方向:动物遗传学。Email: y6784@126.com
研究报告
R-33
A
1671-7856(2017) 07-0087-06
10.3969.j.issn.1671-7856. 2017.07.016
2016-12-06
