猫科动物DNA条形码及线粒体假基因对物种鉴定的影响
2017-07-31蔡延森李佳凌赵矫张亮
蔡延森, 李佳凌, 赵矫, 张亮
(1.西南医科大学医学细胞生物学与遗传学教研室,四川泸州646000; 2. 西南医科大学形态学研究室,四川泸州646000;3. 成都大熊猫繁育研究基地,四川省濒危野生动物保护生物学重点实验室,成都610081)
猫科动物DNA条形码及线粒体假基因对物种鉴定的影响
蔡延森1, 李佳凌2, 赵矫1, 张亮3*
(1.西南医科大学医学细胞生物学与遗传学教研室,四川泸州646000; 2. 西南医科大学形态学研究室,四川泸州646000;3. 成都大熊猫繁育研究基地,四川省濒危野生动物保护生物学重点实验室,成都610081)
在多种动物类群中,基于线粒体细胞色素c氧化酶亚基Ⅰ(COⅠ)基因的DNA条形码是一种高效的物种鉴别手段,然而猫科Felidae动物中广泛存在的线粒体假基因可能影响DNA条形码的有效性。本研究共涉及猫科动物12属25种119个样本。采用3对条形码通用引物对6属11种29个猫科动物样本进行了扩增及测序。结果3个样本扩增失败,8个样本得到假基因,18个样本获得了条形码序列。结合另外93条猫科动物条形码序列(源自BOLD Systems),采用Kimura 2-parameter模型计算遗传距离,构建Neighbor-Joining(NJ)树。结果显示,遗传距离种内为0%~8.1%,平均0.8%;种间为1.4%~13.1%,平均8.7%;属间为8.2%~21.8%,平均15.1%。NJ树显示,除3个种外,其余物种均以极高的置信度(99%)形成单系分支。而假基因序列有些可以单独形成分支,有些夹杂在COⅠ序列形成的分支中,对物种鉴定产生干扰。
COⅠ; 猫科; 线粒体假基因
近年来,DNA条形码作为一种分子级别的物种鉴定手段,已经在多种昆虫、鱼类、鸟类、哺乳类等动物中显示了其准确、高效、便捷的特点(Hebertetal.,2003,2004;蔡延森等,2009,2014;Caietal.,2011,2017;Kressetal.,2015;Lietal.,2017)。在动物中,常用的DNA条形码是一段标准化的线粒体基因片段,即细胞色素c氧化酶亚基Ⅰ(COⅠ)基因片段(Hebertetal.,2003)。该段序列带有丰富的物种信息,适用PCR扩增的通用引物,极少出现插入或缺失,并且具有母系遗传、无内含子、重组受限等特点(Brownetal.,1982)。这些特点使得条形码序列的测序及序列分析较方便快捷,进而准确地显示出样本的种属信息。这种快捷准确的物种鉴定技术,对于检验检疫、动物制品质量控制、生物多样性保护、圈养动物管理等各方面工作有重要意义。
线粒体假基因(nuclear mitochondrial pseudogenes,numts)是起源于线粒体,后来整合到核基因组中并失去功能的DNA序列(Lopezetal.,1994)。用条形码通用引物从总基因组DNA中扩增COⅠ基因片段时,可能得到numts序列而并非mtDNA上的目的基因片段(周志军等,2014)。虽然numts并不是普遍存在于所有物种中,但猫科Felidae动物核基因组中存在着大段的numts序列,例如家猫线粒体全基因组约17 kb,其核基因组中就有高达7.9 kb的numts(Lopezetal.,1994)。这不仅会增加物种鉴定的难度,甚至可能会导致错误的鉴定结论。
本研究对25种猫科动物的样本进行了分析,包括国家Ⅰ级重点保护动物——东北虎Pantheratigrisaltaica、华南虎P.tigrisamoyensis、雪豹P.uncia、金钱豹P.pardus,国家Ⅱ级保护动物亚洲金猫Catopumatemminckii、兔狲Otocolobusmanul、猞猁Lynxlynx、欧洲野猫Felissilvestrissilvestris、荒漠猫F.bieti,以及列入IUCN濒危物种红色名录的狮子P.leo、美洲狮Pumaconcolor、美洲豹P.onca、猎豹Acinonyxjubatus等,并对其中出现较高频率numts的现象进行了分析,探究DNA条形码对猫科动物物种鉴定的可靠性。
1 实验材料与方法
1.1实验材料
本研究共涉及猫科动物12属25种119个样本,其中6属11种29个样本为本实验室提供(表1)。所有样本均通过非损伤取样获得,采集自猫科动物的粪便,从中获取毛发后提取毛囊DNA,然后保存于4 ℃冰箱中备用或95%乙醇中-80 ℃长期保存。其余序列下载自The Barcode of Life Data Systems(BOLD Systems,http://www.boldsystems.org)。在BOLD猫科动物条形码目录中下载了全部323个序列(截止2017年3月),删除非COⅠ基因及长度小于500 bp的COⅠ序列,最后剩下93条序列参与分析(含numts)。

表1 样本信息Table 1 Sample information
注:*扩增得到假基因,☆扩增失败。
Notes:*nuclear mitochondrial pseudogene was obtained,☆amplification was failed.
1.2DNA提取、PCR扩增及测序
DNA提取采用QIAamp DNA Stool Mini Kit (Qiagen,Hilden,Germany)。PCR扩增共使用了3对引物,首先使用脊椎动物COⅠ通用引物对LCOI1490:5’-GGTCAACAAATCATAAAGATATTGG-3’,HCOI2198:5’-TAAACTTCAGGGTGACCAAAAAATCA-3’。如果扩增失败或测序结果为套峰,则替换引物重新扩增,使用引物对VF1:5’-TTCTCAACCAACCACAAAGACATTGG-3’,VR1:5’-TAGACTTCTGGGTGGCCAAAGAATCA-3’;或Zlf04:5’-TCTCAACTAAYCAYAAAGAYATYGG-3’,Zlr04:5’-TAAACTTCrGGGTGACCAAArAATCA-3’。每25 μL PCR反应体系包含:约25 ng DNA模板,2.5 mM MgCl2,1×PCR buffer(TaKaRa,中国大连),1.0 U Taq DNA聚合酶(TaKaRa,中国大连),上下游引物各0.5 μM, 每种dNTP各50 μM。PCR反应参数:94 ℃预变性5 min;94 ℃ 45 s,51 ℃ 45 s,72 ℃ 45 s,共30个循环;72℃最后延伸10 min;4 ℃保存。PCR产物用1%的琼脂糖凝胶电泳检测,用凝胶回收试剂盒(E.Z.N.A. Gel Extraction Kit)回收约700 bp目的片段,直接双向测序。
1.3序列分析
将测序所获正、反向序列导入DNAman,EditSeq等软件进行拼接,使用MEGA 6与同源序列进行比对。由于猫科动物中numts出现的频率大大高于其他物种,而COⅠ假基因大多存在提前的终止密码子或带有插入/缺失导致的移码突变,所有序列还逐一进行了手工校正并分析其蛋白序列,根据是否存在终止密码子、移码突变进而鉴定扩增所得为COⅠ基因片段或numts。最后用Kimura 2-parameter(K2P)模型计算遗传距离,并构建Neighbor-Joining(NJ)树。
线粒体DNA的核转移时间计算方法采用Li等(1981)的计算公式:δ=(μ1+μ2)t。其中t为分歧时间,δ为序列之间遗传差异,μ1和μ2分别为线粒体DNA和numts每年每个核苷酸替换速率,μ1=1.7×10-8,μ2=4.7×10-9。
2 结果
本研究共提供了29个猫科动物个体的新样本,其中3个样本(jdPTA01、jdPPA827、jdPPA829)扩增失败,更换引物后扩增依然失败或测序结果为套峰,无法读取序列。8个样本测序结果最初为套峰,更换引物重新扩增测序后,得到的序列有明显numt特征,存在碱基插入/缺失或终止密码子。其余18个样本经过1~3次扩增,得到660 bp左右清晰无套峰的片段,序列用MEGA 6比对分析无插入/缺失、终止密码子,通过NCBI中Blast相似性检索,确定为COⅠ序列。
2.1遗传距离分析
DNA条形码序列的K2P遗传距离在猫科动物的种内差异普遍较高,最低为0,最高达8.1%,平均为0.8%;同属种间遗传距离为1.4%~13.1%,平均为8.7%;同科属间遗传距离为8.2%~21.8%,平均为15.1%。种内、同属种间、同科属间的遗传距离分布频率见图1(numts序列未计入)。
Hebert等(2004)提出DNA条形码划分物种的10×规则,即种间遗传差异要高于种内遗传差异10倍。野猫Felissilvestris及其圈养亚种家猫F.s.catus的种内个体间差异最大达到了10.3%,超过平均值(0.8%)12.8倍,不符合Hebert的10×规则。
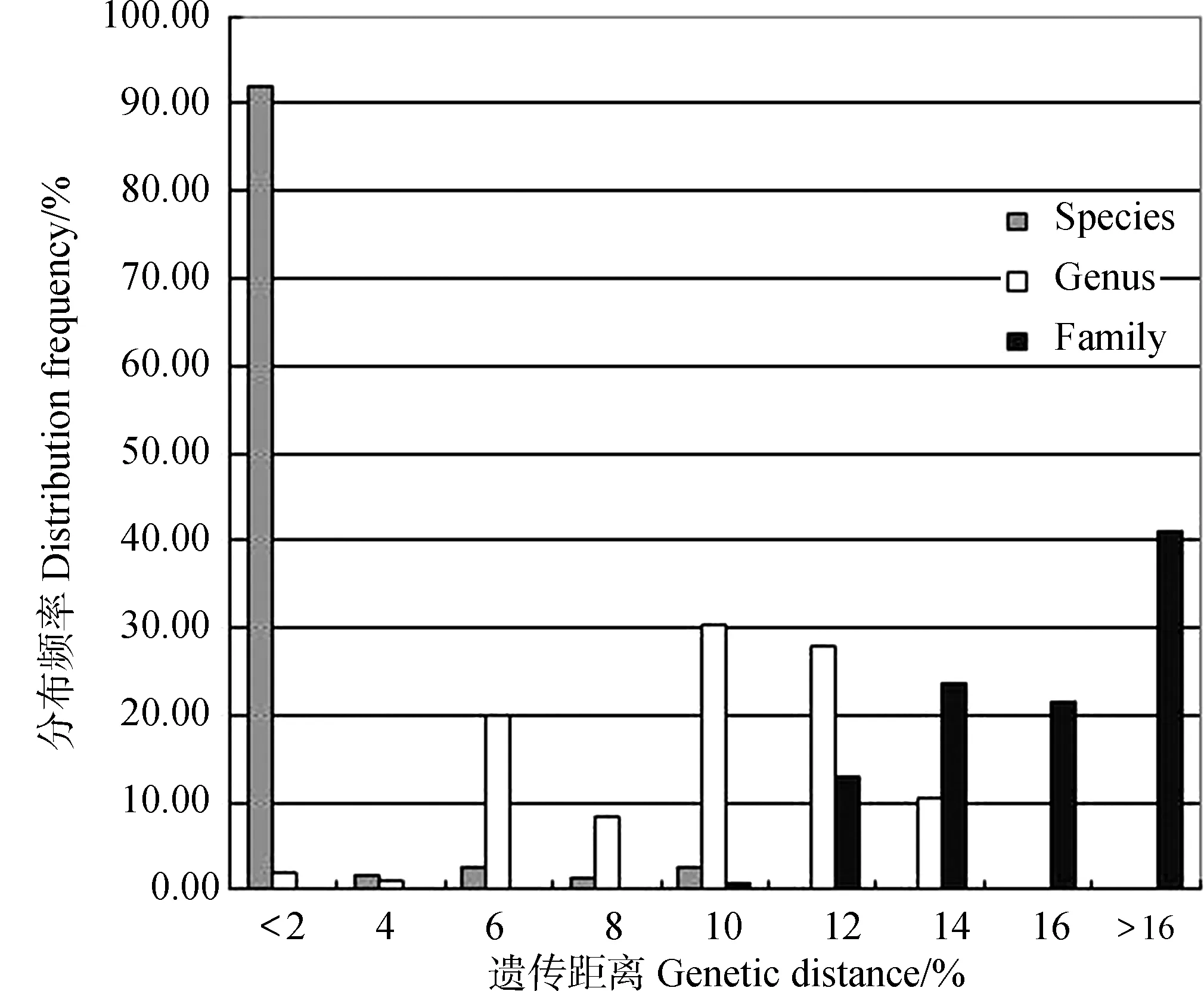
图1 种内、同属种间、同科属间的遗传距离分布频率Fig. 1 Distribution of genetic distances within species, genus and family of Felidae
2.2聚类分析
本研究中有16个种含多个样本参与分析。MEGA 6构建NJ树结果显示,其中13个种以极高的置信度(99%)各自形成单系分支,表明这些种可被DNA条形码准确鉴定(图2),包括猎豹、狞猫Caracalcaracal、亚洲金猫、猞猁、加拿大猞猁L.canadensis、短尾猫L.rufus、兔狲、狮子、老虎P.tigris、金钱豹、雪豹、扁头豹猫Prionailurusplaniceps、美洲狮。另外3个种没有形成单系,分别有2~4个分支,包括豹猫属Prionailurus的豹猫P.bengalensis,渔猫P.viverrinus和野猫(F.silvestris和F.s.catus)。其余9个种只包含1个样本参与分析,所有物种没有共享序列,在NJ树上物种没有重叠。
在豹猫属中,物种的种内平均差异为1.1%~5.3%,种间平均差异为4.6%~6.3%,物种之间没有共享序列。表明虽然存在较大的种内分化,但基于聚类分析,DNA条形码依然能够对它们进行有效的物种鉴别。
在NJ树上,野猫大部分个体聚在一起形成一个大支(Felissilvestris和F.s.catus),该群体内遗传差异平均为0.3%(0~1.6%),其余部分序列分别形成了3个小分支(F.s.catus1、F.s.catus2和F.s.catus3)。这3个小分支同大分支的平均遗传差异分别为7.9%、3.6%、5.3%,分别有4、1、9处碱基位点突变导致氨基酸发生改变。在NCBI中用Blast相似性检索,这3个分支中的序列同家猫线粒体假基因全序列的相似度分别达到99%、96%、95%。
2.3假基因的分析
在狮子、老虎、金钱豹、荒漠猫4个物种中检测出带有终止密码子的序列,为明显的numts特征。对这些numts序列进行分析发现:狮子、老虎、金钱豹3个物种的numts以87%的置信度聚在一起,群体内遗传差异为0.5%~1.4%。根据Li等(1981)对mtDNA和numts的分歧时间计算方法,可以得知狮子、老虎、金钱豹的核转移事件发生的时间分别为约220万年前、350万年前、270万年前。
荒漠猫假基因序列(图2,NJ树上F.bietinumts)同野猫2个个体的“COⅠ序列”(图2,NJ树上F.s.catus1)以100%的置信度聚为一支,群体内遗传差异仅为0.5%~0.8%;而野猫F.s.catus1这一支中的2个序列同野猫其他个体之间的遗传差异高达6.9%~10.3%,因此推测这2个序列极有可能是numts(GBMA5696-13|Feliscatus|COⅠ-5P|JX426133和GBMIN43334-14|Feliscatus|COⅠ-5P|KF771218)。
3 讨论
本研究中猫科动物种内和种间的平均遗传差异分别为0.8%和8.7%,NJ树上大部分物种都以极高置信度形成单系,这些数据表明DNA条形码在很大程度上能够对猫科动物实现快速、准确的鉴定。个别物种NJ树上没有形成单系且种内遗传差异较大,可能有两方面的原因。
一方面,可能是进化方面的原因:新近分化的物种存在不完全谱系分选现象;近源物种的种间杂交导致基因渗透;由于地理/生殖隔离导致较大的种内遗传差异、多亚种等。这些因素都可能导致DNA条形码对物种的鉴别出现困难甚至失败。没有形成单系的豹猫和渔猫都是豹猫属的近源物种。其中,豹猫至少含8个亚种(Mukherjeeetal.,2016);渔猫虽然没有亚种划分,但该物种的栖息地碎片化,长期处于地理/生殖隔离状态(Mukherjeeetal.,2016)。此外,全世界广泛分布的野猫有22个亚种(Wozencraft,2005),在NJ树上有4个分支。可见,正是这些进化方面的原因导致了这些物种的种内分化。
另一方面的原因是numts同COⅠ共扩增的现象,会对物种鉴定造成干扰、错误,甚至产生不存在的“新种”等。在NJ树上,狮子、老虎、金钱豹3个物种看似有多个分支,种内绝大部分个体形成一个大支,另一些个体则形成几个小分支。但实际上,这几个小分支里面所包含的都是numts序列。如果将numts误认为是DNA条形码对其进行分析,则会得出错误的结论。
误把numts当COⅠ的情况可能是野猫鉴定失败的主要原因。在NJ树上,F.s.catus1这支野猫和荒漠猫的numts以100%的置信度聚在一起,二者间的遗传差异仅为0.5%~0.8%,并且这支中的个体同种内其他个体的差异最高达10.3%(平均7.9%)。虽然在F.s.catus1中没有检测到终止密码子、插入/缺失,但推测它极有可能是numts:首先,猫科动物中不同物种的numts通常聚为一支(如:狮子、老虎、金钱豹),而它同荒漠猫numts也高度相似聚在一起;其次,该序列在GenBank中Blast,同家猫numts序列相似度为99%;最后,根据Li等(1981)的方法计算mtDNA发生核转移的时间,发现该序列发生核转移的时间约350万年前,该时间同狮子、老虎、金钱豹的numts发生时间相似(220万年~350万年)。这些证据都从侧面暗示了它是numts。
综上,DNA条形码数据库中已有序列的准确性对基于该技术的物种鉴定至关重要,而通用引物从总基因组DNA中扩增时,可能得到numts序列而并非COⅠ目的基因片段,这一现象应该得到重视。研究者如果不能正确区分numts和mtDNA,把错误的序列关联物种信息并存入数据库,则会导致后续的各项工作出现混乱和错误。本研究表明该问题在猫科动物中普遍存在,其他种类的动物中是否也存在大量numts,对条形码技术形成类似的干扰影响,有待进一步研究发现。
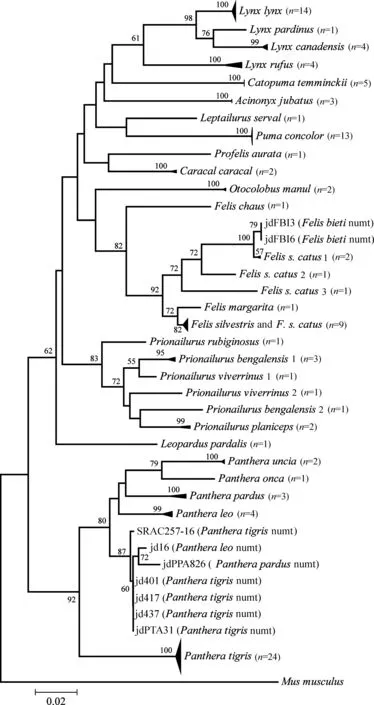
图2 猫科动物25种119个样本的COⅠ及numts序列所构建的NJ树Fig. 2 NJ tree of 119 COⅠ and numts sequences in 25 species of Felidae
蔡延森, 刘芹, 王译伟, 等. 2014. 四川宜宾产桃花水母的线粒体COⅠ序列片段分析[J]. 泸州医学院学报, (1): 79-82.
蔡延森, 张修月, 岳碧松, 等. 2009. 我国8种猛禽的DNA条形码技术研究[J]. 四川动物, 28(3): 334-340.
周志军, 尚娜, 常岩林, 等. 2014. DNA条形码揭示日本纺织娘(Mecopodaniponensis)个体内和个体间的序列变异[J]. 生物技术通报, (5): 129-136.
Brown WM, Prager EM, Wang A,etal. 1982. Mitochondrial DNA sequences of primates: tempo and mode of evolution[J]. Journal of Molecular Evolution, 18(4): 225-239.
Cai YS, Zhang L, Wang Y,etal. 2016. Identification of deer species (Cervidae, Cetartiodactyla) in China using mitochondrial cytochrome c oxidase subunit Ⅰ (mtDNACOⅠ)[J]. Mitochondrial DNA Part A: the Journal of DNA Mappong Sequencing, and Analysis, 27(6): 4240-4243.
Cai YS, Zhang L, Shen FJ,etal. 2011. DNA barcoding of 18 species of Bovida[J]. Chinese Science Bulletin, 56(2): 164-168.
Hebert PDN, Cywinska A, Ball SL,etal. 2003. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society B-Biological Sciences, 270: 313-321.
Hebert PDN, Stoeckle MY, Zemlak TS,etal. 2004. Identification of birds through DNA barcodes[J]. PLoS Biology, 2(10): e312.
Li J, Cui Y, Jiang J,etal. 2017. Applying DNA barcoding to conservation practice: a case study of endangered birds and large mammals in China[J]. Biodiversity & Conservation, 26(3): 653-668.
Li WH, Gojobori T, Nei M. 1981. Pseudogenes as a paradigm of neutral evolution[J]. Nature, 292(5820): 237-239.
Lopez JV, Yuhki N, Masuda R,etal. 1994. Numt, a recent transfer and tandem amplification of mitochondrial DNA to the nuclear genome of the domestic cat[J]. Journal of Molecular Evolution, 39(2): 174-190.
Mukherjee S, Appel A, Duckworth JW,etal. 2016.Prionailurusviverrinus[DB/OL]. The IUCN Red List of Threatened Species 2016:e.T18150A50662615.
Wozencraft WC. 2005. Mammal species of the world: a taxonomic and geographic reference (3rd Ed.)[M]. Baltimore: Johns Hopkins University Press: 536-537.
DNABarcodinginFelidae,andtheEffectsofNuclearMitochondrialPseudogenesonSpeciesIdentification
CAI Yansen1, LI Jialing2, ZHAO Jiao1, ZHANG Liang3*
(1. Department of Medical Cell Biology and Genetics, Southwest Medical University, Luzhou, Sichuan Province 646000, China;2. Department of Morphology, Southwest Medical University, Luzhou, Sichuan Province 646000, China; 3. Key Laboratory of Conservation Biology on Endangered Wildlife, Chengdu Research Base of Giant Panda Breeding, Chengdu 610081, China)
In a variety of animal species, DNA barcoding based on mitochondrial cytochrome c oxidase subunit Ⅰ (COⅠ) gene is an efficient tool for species identification. However, the presence of large mitochondrial pseudogenes in felids may affect the effectiveness of DNA barcoding. In this study, 119 felid samples from 25 species of 12 genera were analyzed.3 pairs of universal primers were used to amplify and sequence theCOⅠ genes of 29 felid samples that belonged to 11 species from 6 genera. The results showed that the amplification was failed in 3 samples, nuclear mitochondrial psedogenes (numts) were obtained in 8 samples, and barcode sequences were derived in 18 samples. Combined with the other 93 felid barcode sequences (derived from BOLD Systems), Kimura 2-parameter model was used to calculate the genetic distance and to construct Neighbor-Joining (NJ) tree. The genetic distance within species were 0%-8.1%, with an average of 0.8%; the interspecific genetic distance were 1.4%-13.1%, with an average of 8.7%; and the genetic distance within family were 8.2%-21.8%, with an average of about 15.1%. The NJ tree showed that most of the species formed a monophyletic clade with high confidence (99%), except 3 species; some of the numts sequences could form a clade on NJ tree, and the others were inserted in the branches of theCOⅠ sequences, which may compromise the species identification.
COⅠ; Felidae; nuclear mitochondrial pseudogenes
2017-03-13接受日期:2017-06-15
四川省教育厅科研项目(12ZA242)
蔡延森(1982—), 男, 硕士, 讲师, E-mail:cys_cys@aliyun.com
*通信作者Corresponding author, E-mail:zliang@panda.org.cn
10.11984/j.issn.1000-7083.20170079
Q959.8
: A
: 1000-7083(2017)04-0425-06
