性成熟前猪睾丸组织中印记基因DNA甲基化状态分析
2017-06-06白利鹏李丹婷曹随忠沈留红余树民
陈 曦,杨 芳,李 捷,白利鹏,李丹婷,曹随忠,沈留红,余树民
(四川农业大学 动物医学院,四川 成都 611130)
性成熟前猪睾丸组织中印记基因DNA甲基化状态分析
陈 曦,杨 芳,李 捷,白利鹏,李丹婷,曹随忠,沈留红,余树民*
(四川农业大学 动物医学院,四川 成都 611130)
为探求性成熟前猪睾丸中印记基因DNA甲基化状态,以1、2、3月龄三元杂交猪睾丸实质组织为材料,在利用MeDIP-seq(甲基化DNA免疫沉淀测序技术)建立性成熟前猪睾丸全基因组DNA甲基化图谱的基础上,比对得到了8个已证实的猪印记基因甲基化状态。结果显示,8个印记基因DIRAS3、IGF2、IGF2R、MEST、NAP1L5、NNAT、PEG10、PLAGL1,在性成熟前猪睾丸组织中的甲基化水平均不高,表现为“非甲基化”或“部分甲基化”;NNAT、PLAGL1启动子甲基化水平在1~3月龄间保持增长,NAP1L5、IGF2R启动子甲基化水平在1~2月龄下降,2~3月龄增长,DIRAS3、IGF2、MEST、PEG10 promoter甲基化水平在1~2月龄增长,2~3月龄下降;图谱中仅比对到4个基因的gene body甲基化状态,IGF2R、MESTgene body甲基化水平在1~3月龄保持下降,PLAGL1、PEG10 gene body甲基化水平在1~2月龄增长,2~3月龄下降。推测这8个印记基因在性成熟前猪睾丸发育过程中均具有一定活性,且DNA甲基化对印记基因发挥一定的调控作用。
DNA甲基化;印记基因;睾丸;猪;性成熟前
睾丸是雄性哺乳动物产生精子和雄激素的器官,在雄性生殖系统中扮演着重要的角色。猪不仅是畜牧业的重要经济动物,还是人类疾病研究的重要模型动物。在性成熟前猪睾丸组织中,精子发生尚未完全启动,生精细胞数量很少,各类睾丸功能体细胞是该阶段的主要细胞类型。睾丸体细胞增殖分化最终形成性成熟后精子发生所必需的微环境,是睾丸结构塑造和雄性生殖力维持的基础。DNA甲基化 (DNA methylation) 作为一种重要的表观遗传修饰,在睾丸发育过程中基因组DNA甲基化也发挥着不可忽视的作用。Liang等[1]在小鼠睾丸中发现大量发育阶段特异性甲基化的位点,并且这些位点在胚胎及新生期甲基化,而在成年后呈现非甲基化,提示基因组DNA的阶段性去甲基化在睾丸发育中发挥作用。另外,Takashima等[2]证实,任意一种DNA甲基转移酶在小鼠中异常表达均会引起精子发生缺陷而导致雄性不育。由此可知,正确的DNA甲基化模式对于睾丸正常发育是必须的,而异常的甲基化状态则可导致睾丸功能异常和精子发生阻滞。
在哺乳动物配子或合子发生期间,等位基因受到不对称的表观遗传修饰,导致基因表达具有亲本选择性,这一现象被称作基因组印记 (genomic imprinting),这类基因即为印记基因[3]。目前关于印记基因的研究多围绕人和小鼠展开,且着重于探究印记基因与胚胎发育的关系,而DNA甲基化是控制基因印记的重要机制之一[4],因此,研究印记基因甲基化状态可为研究发育调控机制提供依据,但猪性成熟前睾丸中印记基因的甲基化状态的相关研究还未见报道。
结合高通量测序的甲基化DNA免疫沉淀技术 (MeDIP-seq) 是检测全基因组DNA甲基化简单高效的方法之一[5]。本试验在利用MeDIP-seq对性成熟前 (1月龄、2月龄、3月龄) 猪睾丸甲基化状态进行全基因组检测的基础上,挑选出8个已证实的猪印记基因:DIRAS3、IGF2、IGF2R、MEST、NAP1L5、NNAT、PEG10、PLAGL1,分析这些印记基因的甲基化状态,旨在揭示猪性成熟前睾丸中印记基因的甲基化模式,为深入研究DNA甲基化和印记基因对睾丸发育的调控机制提供理论依据。
1 材料与方法
1.1 试验动物
所有试验动物均来自四川省崇州市某规模化猪场,为三元杂交猪(皮特兰×长白猪×大白猪),接受正常免疫程序及良好饲喂。试验动物基本情况如表1。
1.2 样品采集与DNA的抽提
本试验严格按照全国《实验动物管理条例》规定,采集睾丸组织样品,-80 ℃保存备用。按照Universal Genomic DNA Extraction Kit Ver.3.0 (Takara Co. Ltd)说明书,从睾丸实质组织样品中提取基因组DNA。
1.3 MeDIP-seq
基因组DNA使用Bioruptor超声破碎仪打断成200~800 bp的片段,取800 ng DNA按照Genomic DNA Sample Prep Kit (Illumina) 说明书,进行末端修复、加A尾、连接测序接头,经琼脂糖凝胶电泳筛选出连接成功的DNA片段用于后续实验。
经上述处理后的DNA样品参考Down等[6]方法进行MeDIP操作。甲基化DNA免疫沉淀抗体为BioMagTM磁珠 (QIAGEN) 耦合的小鼠抗5′-甲基胞嘧啶单抗,非特异性的小鼠IgG抗体作为阴性对照。使用MinElute PCR Purification Kit(QIAGEN)对免疫沉淀的DNA进行洗脱和纯化,进行14个循环的PCR扩增 (使用Genomic DNA Sample Prep Kit中的单端PCR引物),最后由2%琼脂糖凝胶电泳筛选300~900 bp的DNA片段,并利用MinElute柱纯化。
表1 试验动物基本信息
Table 1 Basic information of experimental animals

分组Group年龄(性别)Age(Sex)数量Number父本来源Maleparent母本来源MaternalparentT11月龄Onemonthofage(♂)3皮特兰Pietrain二元杂交母猪aTwo⁃waycrosssowaT22月龄Twomonthsofage(♂)3皮特兰Pietrain二元杂交母猪bTwo⁃waycrosssowbT33月龄Threemonthsofage(♂)3皮特兰Pietrain二元杂交母猪cTwo⁃waycrosssowc
甲基化免疫沉淀后的DNA使用荧光定量PCR (qPCR) 进行质控,检测甲基化区域的富集情况。DNA用洗脱液等份稀释至5 ng·μL-1,取1 μL用于qPCR。H19启动子区域的特异性甲基化位点作为阳性对照,GAPDH启动子区域的非甲基化位点作为阴性对照,引物序列见表2。质控合格的DNA测序文库送至上海康成测序,使用Illumina HiSeq 2000测序平台。
1.4 数据处理
测序reads由Illumina HiSeq 2000生成收集,采用Off-Line Basecaller软件 (OLB V1.8) 进行测序图像分析以及碱基检出和转换。通过Solexa CHASTITY质量过滤后,使用Bowtie软件[7](V2.1.0) 将clean reads与猪基因组 (UCSC susScr3) 进行比对。猪印记基因信息从Geneimprint印记基因数据库 (www.geneimprint.com) 下载得到,与测序数据比对。猪基因组CpG岛位置及RefSeq基因位置信息从UCSC Genome Browser下载得到,转录起始位点上游700 bp到其下游200 bp的区域被定义为启动子 (promoter),转录起始位点下游2 000 bp到转录终止位点的区域被定义为gene body。比对后reads由HTSeq软件[8]收集并计算甲基化得分,基因组某一特定区域甲基化水平的计算公式及判定规则[9]:甲基化得分=(CpG length/1000) × (mapped reads/kb),得分低于15.15 reads·kb-1的定义为“非甲基化(unmethylated)”;15.15~152.27 reads·kb-1的定义为“部分甲基化 (partially methylated)”;高于152.27 reads·kb-1则定义为“高甲基化(highlymethylated)”。
表2 qPCR引物序列
Table 2 Primers for qPCR

引物Primer大小Size/bp序列SequenceH1979F:5’TGAATGCTGCGATAGGAGG3’R:5’CCGTAATCCACGGTAAACACT3’GAPDH71F:5’CTCTGCTCCCTCCCCGTT3’R:5’TCCACTCCGCCAGGCTTTA3’
2 结果与分析
2.1 MeDIP质控结果
使用qPCR对甲基化区域的富集情况进行质控。H19是猪的印记基因,其promoter区域表现特异性甲基化,作为阳性对照;看家基因GAPDH的promoter区域表现非甲基化,作为阴性对照。从图1可看出,MeDIP特异地富集甲基化的DNA区域 (H19 promoter),而非甲基化区域 (GAPDHpromoter) 富集极少,说明本试验MeDIP抗体富集效率高、特异性好,得到的甲基化数据真实可靠。
2.2 印记基因的比对
MeDIP-seq共获得19.04 Gb原始数据,经过滤约有超过190 M的clean reads可用于后续分析。为了得到全基因组DNA甲基化图谱,我们将MeDIP-seq所获clean reads比对到猪基因组上 (UCSC susScr3),9个样品的平均比对率为82.94%。以上结果表明,本试验MeDIP-seq的测序数据质量理想。通过对猪性成熟前3个发育时间点 (1、2、3月龄,即T1、T2、T3) 睾丸全基因组CpG岛甲基化水平的检测,本试验成功构建了性成熟前猪睾丸全基因组DNA甲基化图谱。
将Geneimprint印记基因数据库 (www.geneimprint.com) 上收录的共计30个已证实的猪印记基因比对到甲基化图谱上,成功比对到8个印记基因,分别为DIRAS3 (DIRAS family, GTP-binding RAS-like 3)、IGF2 (insulin-like growth factor 2)、IGF2R(insulin-like growth factor type II receptor)、MEST(mesoderm-specific transcript)、NAP1L5 (nudeosome assembly protein l-like 5)、NNAT(neuronatin)、PEG10 (patemal expression gene 10)以及PLAGL1 (pleiomorphic adenoma gene-like 1)。

T1、T2、T3分别代表1、2、3月龄三个组别的睾丸样品;Rep1、Rep2、Rep3代表每组的3个生物学重复T1, T2 and T3 represent the testis tissue samples of 1, 2 and 3 months old pigs, respectively; Rep1, Rep2 and Rep3 represent the three biological replicates of each group图1 MeDIP质控结果Fig.1 Quality assessment of MeDIP
2.3 性成熟前猪睾丸中印记基因甲基化状态
本研究检测了这8个印记基因的promoter和gene body两种基因元件在性成熟前猪睾丸组织中的甲基化状态,并分析了其甲基化水平在性成熟前的3个发育时间点 (T1、T2、T3) 之间的变化特征,本次MeDIP-seq只检测到了其中4个基因gene body区域的甲基化状态。如表3所示,在性成熟前猪睾丸组织中,8个印记基因均处于较低的甲基化水平,即“非甲基化”和“部分甲基化”的状态。DIRAS3、NAP1L5、NNAT的promoter甲基化状态在3个时间点上均为“部分甲基化”,甲基化水平相较于其他5个基因的promoter较高。在检测到gene body区域甲基化的4个印记基因中,IGR2R、PEG10、PLAGL1的gene body甲基化状态在3个时间点上均为“部分甲基化”,而MEST的gene body甲基化状态在3个时间点上均为“非甲基化”。其中,IGR2Rgene body的甲基化水平高于其promoter的甲基化水平。
表3 八个猪印记基因在性成熟前猪睾丸中的甲基化状态
Table 3 The DNA methylation status of 8 imprinted genes in prepubertal porcine testis

GenenamePromoterT1T2T3GenebodyT1T2T3DIRAS3+++///IGF2—++///IGF2R——++++MEST—++———NAP1L5+++///NNAT+++///PEG10++—+++PLAGL1—+++++
“—”表示甲基化状态为“非甲基化”;“+”表示甲基化状态为“部分甲基化”;“++”表示甲基化状态为“高甲基化”;“/”表示未从全基因组甲基化图谱中未比对到该基因gene body的甲基化状态。
“—” represents “Unmethylated”, “+” represents “Partially methylated”, and “++” represents “Highly methylated” for the methylation status; “/” represents that the gene body methylation status of this gene had not been mapped in this profile.
观察8个印记基因promoter甲基化水平在T1、T2、T3之间的变化发现 (图2),NNAT、PLAGL1 promoter甲基化水平在T1到T3之间表现出持续的增长;NAP1L5、IGF2Rpromoter甲基化水平在T1到T2之间增长,而在T2到T3之间下降;DIRAS3、IGF2、MEST、PEG10 promoter甲基化水平在T1到T2之间下降,在T2到T3之间增长。观察4个印记基因gene body甲基化水平在T1、T2、T3之间的变化发现 (图3),IGF2R、MESTgene body甲基化水平在T1到T3之表现出持续的降低;PLAGL1、PEG10 gene body甲基化水平在T1到T2之间增长,在T2到T3之间下降。

图2 八个猪印记基因Promoter的甲基化水平在性成熟前3个时间点间的变化Fig.2 The change of methylation levels of 8 porcine imprinted gene promoters during the three time points of prepuberty
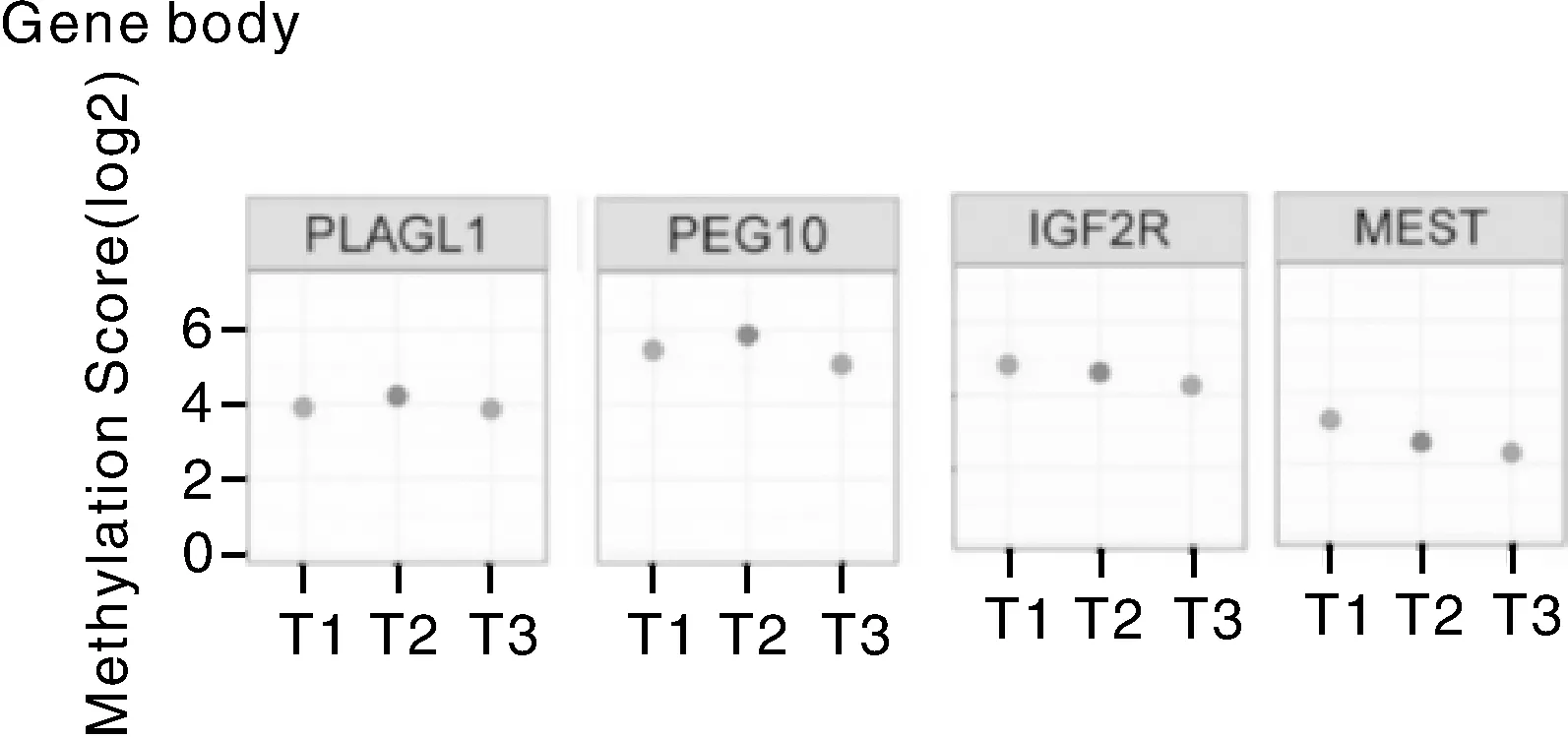
图3 4个猪印记基因Gene body的甲基化水平在性成熟前3个时间点间的变化Fig.3 The change of methylation levels of 4 porcine imprinted gene bodies during the three time points of prepuberty
3 讨论
DNA甲基化是建立和维持基因组表观遗传状态的基础,也是基因组印记的关键机制[4],被形象地比喻为基因表达的开关[10]。在哺乳动物中,通常认为promoter区域的甲基化抑制基因表达[11],而发生在gene body区域的甲基化作用则较为复杂,但有研究认为其对基因表达有一定的正向作用[12]。印记基因在哺乳动物胚胎发育以及细胞增殖过程中发挥着重要的作用,研究显示印记基因表达具有一定的时空特异性[13-14],进而调控器官发育。尽管目前已有大量文献报道印记基因方面的研究[15],但多围绕人和小鼠,着重印记基因与胚胎发育的关系,而对猪已知的印记基因与睾丸发育之间的关系鲜有报道,在性成熟前猪睾丸中的研究则更是空白。本试验在通过MeDIP-seq建立性成熟前猪睾丸全基因组DNA甲基化图谱的基础上,从中分析得到了已知的8个猪印记基因,DIRAS3、IGF2、IGF2R、MEST、NAP1L5、NNAT、PEG10、PLAGL1的甲基化状态,这8个基因在性成熟前猪睾丸组织中的甲基化水平均不高,呈现“非甲基化”或“部分甲基化”,提示这8个印记基因在睾丸发育过程中均具有一定活性。此外,根据“营养冲突学说”,父源表达的印记基因促进生长,而母源表达的基因抑制生长[16],这8个印记基因除DIRAS3和IGF2R外,均为母系印记、父源表达,这也提示上述印记基因在性成熟前猪睾丸的发育过程中可能发挥一定的调控作用。此外,在本研究构建的全基因组DNA甲基化图谱中只比对到了IGF2R、MEST、PEG10及PLAGL14这4个基因的gene body甲基化状态,比较这4个基因promoter和gene body区域甲基化状态的差异,我们发现除MEST基因gene body甲基化水平略低于promoter,其他3个基因gene body甲基化水平均高于promoter (表3、图2、图3)。上述结果印证了功能基因的promoter区域大多维持在较低的甲基化水平[11];而gene body相较于promoter具有更高的甲基化水平,则可能表明gene body较高的甲基化状态更支持基因的表达。
NNAT、PLAGL1 promoter的甲基化水平在1~3月龄间增长 (图2)。NNAT编码的蛋白在脑发育及神经系统结构的形成和维持中发挥重要作用[17],但它与睾丸发育之间的关系未见报道,在本研究中同其他基因相比,其promoter甲基化水平相对较高。PLAGL1可以诱导细胞周期阻滞和细胞凋亡,是肿瘤抑制基因[18]。在小鼠中,PLAGL1上游有一个差异甲基化CpG岛,可能是该基因的印记控制中心 (ICR),该ICR在精子中处于非甲基化状态而在卵子中甲基化[19]。在本研究中发现PLAGL1 promoter甲基化在1月龄“非甲基化”,在2、3月龄“部分甲基化”,但总的来看甲基化水平低;此外还发现PLAGL1 gene body表现“部分甲基化”状态,其甲基化水平略高于promoter甲基化水平,以上结果提示在猪中,PLAGL1的ICR可能也位于该基因上游。
NAP1L5、IGF2Rpromoter甲基化水平在1~2月龄下降,在2~3月龄增长 (图2)。NAP1L5的具体功能尚不明确,但其编码蛋白与核小体组装蛋白具有同源性,且可能含有组蛋白结合位点[20]。研究显示IGF2R在猪中为父系印记、母源表达[21],但根据印记基因数据库收录信息来看,IGF2R在猪中表现为逃脱印记。IGF2R是肿瘤抑制基因,编码清道夫受体,调控IGF2的生物活性,并对IGF起负反馈调节[21]。在小鼠中,IGF2R也为父系印记,其可能的印记机制是IGF2R第二个内含子中的CpG岛在卵子上甲基化而在精子上无甲基化[22]。在本研究中,IGR2Rgene body表现“部分甲基化”状态,且其甲基化水平高于promoter的甲基化水平,其可能产生的具体影响有待进一步的探究。
DIRAS3、IGF2、MEST、PEG10 promoter甲基化水平在1~2月龄增长,在2~3月龄下降 (图2)。DIRAS3在印记基因数据库中收录为表达两个亲源等位基因,但有研究表明其在猪中为母系印记[23]。DIRAS3是肿瘤抑制基因,不仅抑制生长,而且降低繁殖功能[24]。尽管DIRAS3 promoter甲基化为“部分甲基化”状态,但整体来看其甲基化水平是8个基因中最高的,提示该基因的功能在性成熟前猪睾丸发育中可能受到抑制。此外,有研究发现DIRAS3基因在成年猪睾丸组织中表现为非甲基化[25],而在本研究中DIRAS3在性成熟前猪睾丸组织中甲基化水平较高,这就表明DIRAS3在猪睾丸发育中过程中可能呈现去甲基化的趋势,且在性成熟后的猪睾丸中发挥一定的生理功能。MEST与脑的发育以及母性行为有关,且在雄激素丰富的组织中表达量非常高[26]。睾丸是产生雄激素的主要器官,MESTpromoter甲基化水平在1到2月龄间上升,到3月龄时又下降,这可能与睾丸间质细胞的增殖阶段有关。睾丸间质细胞负责合成并分泌睾酮,其增殖高峰分别在1月龄和3月龄左右出现[27],这就与MESTpromoter甲基化水平的变化相吻合。1~2月龄间质细胞增殖速度降低,产生的睾酮减少,MEST表达可能降低,其promoter甲基化水平相对增长;同理,到3月龄间质细胞又进入增殖高峰,MESTpromoter甲基化水平也就相对下降。此外,MESTgene body为“非甲基化”状态,且甲基化水平呈下降趋势,提示其gene body的“非甲基化”可能与该基因的表达相关。IGF2在猪中具有部分印记效应,是调控猪脂肪沉积的主要因子之一,PEG10主要调控胎盘的发育,但它们与睾丸发育之间的关系尚不明确[28]。
综上所述,本研究对性成熟前猪睾丸组织中DIRAS3、IGF2、IGF2R、MEST、NAP1L5、NNAT、PEG10、PLAGL1这8个印记基因的甲基化状态及在该发育阶段甲基化水平的变化特征进行了检测和分析。结果显示,8个印记基因甲基化水平均不高,且在1、2、3月龄之间甲基化水平均有一定的变化,提示印记基因甲基化状态可能对性成熟前猪睾丸发育有一定的调控作用,但其调控的具体机制以及DNA甲基化在该发育阶段对印记基因表达产生的具体影响仍有待进一步的研究。
[1] LIANG P, SONG F, GHOSH S, et al. Genome-wide survey reveals dynamic widespread tissue-specific changes in DNA methylation during development[J].BMCGenomics, 2011, 12(1): 1447-1448.
[2] TAKASHIMA S, TAKEHASHU M, LEE J, et al. Abnormal DNA methyltransferase expression in mouse germline stem cells results in spermatogenic defects1[J].BiologyofReproduction, 2009, 81(1): 155-164.
[3] FERGUSON-SMITH A C. Imprinting and the epigenetic asymmetry between parental genomes[J].Science, 2001, 293(5532): 1086-1089.
[4] KANEDA M, OKANO M, HATA K, et al. Essential role for de novo DNA methyltransferase Dnmt3a in paternal and maternal imprinting[J].Nature, 2004, 429(6994): 900-903.
[5] JACINTO F V, BALLESTAR E, ESTELLER M. Methyl-DNA immunoprecipitation (MeDIP): hunting down the DNA methylome[J].Biotechniques, 2008, 44(1): 35-39.
[6] DOWN T A, RAKYAN V K, TURNER D J, et al. A Bayesian deconvolution strategy for immunoprecipitation-based FNA methylome analysis[J].NatureBiotechnology, 2008, 26(7): 779-785.
[7] LANGMEAD B, SALZBERG S L, LANGMEAD B, et al. Fast gapped-read alignment with Bowtie 2[J].NatureMethods, 2012, 9(4): 357-359.
[8] ANDERS S, PYL P T, HUBER W. HTSeq—a Python framework to work with high-throughput sequencing data[J].Bioinformatics, 2015, 31(2): 166-169.
[9] MAUNAKEA A K, NAGARAJAN R P, BILENKY M, et al. Conserved role of intragenic DNA methylation in regulating alternative promoters[J].Nature, 2010, 466(7303): 253-257.
[10] JAENISCH R, BIRD A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals[J].NatureGenetics, 2003, 33(S2): 245-254.
[11] MICHAEL W, INES H, STADLER M B, et al. Distribution, silencing potential and evolutionary impact of promoter DNA methylation in the human genome[J].NatureGenetics, 2007, 39(4): 457-466.
[12] DAUDI J, CONLEY A B, YI S V, et al. On the presence and role of human gene-body DNA methylation[J].Oncotarget, 2012, 3(4): 462-474.
[13] GUILLEMOT F, CASPARY T, TILGHMAN S M, et al. Genomic imprinting of mash2, a mouse gene required for trophoblast development[J].NatureGenetics, 1995, 9(3): 235-242.
[14] WANG Y, JOH K, MASUKO S, et al. The mouse Murr1 gene is imprinted in the adult brain, presumably due to transcriptional interference by the antisense-oriented U2af1-rs1 gene[J].MolecularandCellularBiology, 2004, 24(1): 270-279.
[15] 陈秀莉, 马利兵. 哺乳动物基因组印记的研究进展[J]. 生物技术通报, 2015, 31(1): 46-50. CHEN X L, MA L B. Research progress in genomic imprinting in mammals[J].BiotechnologyBulletin, 2015, 31(1):46-50. (in Chinese with English abstract)
[16] CONSTANT F, GUUILLOMOT M, HEYMAN Y, et al. Large offspring or large placenta syndrome? Morphometric analysis of late gestation bovine placentomes from somatic nuclear transfer pregnancies complicated by hydrallantois[J].BiologyofReproduction, 2006, 75(1): 122-130.
[17] GU T, SU X, ZHOU Q, et al. Molecular characterization of the neuronatin gene in the porcine placenta[J].PlosOne, 2012, 7(8): e43325-e43325.
[18] ENLUND F, PERSSON F, STENMEN G. Molecular analyses of the candidate tumor suppressor gene, PLAGL1, in benign and malignant salivary gland tumors[J].EuropeanJournalofOralSciences, 2004, 112(6): 545-547.
[19] ARIMA T, DREWELL R A, ARNEY K L, et al. A conserved imprinting control region at the HYMAI/ZAC domain is implicated in transient neonatal diabetes mellitus[J].HumanMolecularGenetics, 2001, 10(14): 1475-1483.
[20] 顾婷, 赵书红, 李长春. 猪NAP1L5基因克隆、序列鉴定及在不同妊娠时期胎盘中的差异表达分析[J]. 畜牧兽医学报, 2012, 43(2): 175-179. GU T, ZHAO S H, LI C C. Cloning, sequence, identification and expression analysis ofNAP1L5 in porcine placenta at different gestation stages[J].ActaVeterinariaetZootechnicaSinica, 2012, 43(2): 175-179. (in Chinese with English abstract)
[21] 李莉, 陈预明, 白银山, 等. 猪IGF2R基因多态性及其遗传印记[J]. 中国农业科学, 2010, 43(10): 2156-2161. LI L, CHEN Y M, BAI Y S, et al. Studies on SNP and genomic imprinting of the IGF2R gene in swine[J].ScientiaAgriculturaSinica, 2010, 43(10): 2156-2161. (in Chinese with English abstract)
[22] STOGER R, KUBICJA P, LIU C G, et al. Maternal-specific methylation of the imprinted mouse Igf2r locus identifies the expressed locus as carrying the imprinting signal[J].Cell, 1993, 73(1): 61-71.
[23] CHENG H C, ZHANG F W, DENG C Y. NNAT and DIRAS3 genes are paternally expressed in pigs[J].GeneticsSelectionEvolution, 2007, 39(5): 599-607.
[24] YU Y, LUO R, LU Z, et al. Biochemistry and biology of ARHI(DIRAS3), an imprinted tumor suppressor gene whose expression is lost in ovarian and breast cancers[J].MethodsinEnzymology, 2006, 407: 455-468.
[25] 程焕臣.猪印记基因的分离、印记鉴定及甲基化分析[D].武汉:华中农业大学,2008. CHENG H C. Isolation, imprinting identification and methylation analysis of imprinted genes in pigs[D]. Wuhan: Huazhong Agricultural University, 2008. (in Chinese with English abstract)
[26] ISLES A R, DAVIES W, WILKINSON L S. Genomic imprinting and the social brain[J].PhilosophicalTransactionsoftheRoyalSocietyBBiologicalSciences, 2006, 361(1476): 2229-2237.
[27] STRAATEN H W V, WENSING C J. Leydig cell development in the testis of the pig[J].BiologyofReproduction, 1978, 18(1): 86-93.
[28] BISCHOFF S R, TSAI S, HARDISON N, et al. Characterization of conserved and nonconserved imprinted genes in swine[J].BiologyofReproduction, 2014, 81(5): 906-920.
(责任编辑 张 韵)
DNA methylation status of imprinted genes in prepubertal porcine testis
CHEN Xi, YANG Fang, LI Jie, BAI Lipeng, LI Danting, CAO Suizhong, SHEN Liuhong, YU Shumin*
(CollegeofVeterinaryMedicine,SichuanAgriculturalUniversity,Chengdu611130,China)
To determine the DNA methylation status of imprinted genes in the prepubertal porcine testis, we chose the testis tissues of 1, 2 and 3 months old pigs, and detected the methylation levels of eight imprinted porcine genes on the basis of the genome-wide DNA methylation profile of prepubertal porcine testis, using the method of MeDIP-seq. The results demonstrated thatDIRAS3,IGF2,IGF2R,MEST,NAP1L5,NNAT,PEG10 andPLAGL1, all these eight imprinted genes showed low methylation levels with “unmethylated” or “partially methylated” status. The methylation levels ofNNATandPLAGL1 promoters increased from one month old (T1) to three months old (T3); the methylation levels ofNAP1L5 andIGF2Rpromoters decreased from T1 to two months old (T2) but increased from T2 to T3; while the methylation levels ofDIRAS3,IGF2,MESTandPEG10 promoters increased from T1 to T2 but decreased from T2 to T3. Besides, we only aligned 4 gene bodies from the methylation profile, and the methylation levels ofIGF2RandMESTgene bodies decreased from T1 to T3, while the methylation levels ofPLAGL1 andPEG10 gene bodies increased from T1 to T2 but decreased from T2 to T3. It can be postulated from the results that these eight imprinted genes may be active during the development of prepubertal porcine testis, and DNA methylation may play a role in regulation of imprinted genes.
DNA methylation; imprinted genes; testis; pigs; prepuberty
http://www.zjnyxb.cn陈曦,杨芳,李捷,等. 性成熟前猪睾丸组织中印记基因DNA甲基化状态分析[J].浙江农业学报,2017,29(5): 722-728.
10.3969/j.issn.1004-1524.2017.05.06
2017-01-21
国家自然科学基金 (31172379)
陈曦(1993—),女,四川遂宁人,硕士,研究方向为动物生殖内分泌与细胞工程。E-mail: chenxi1313@foxmail.com
*通信作者,余树民,E-mail: yayushumin@163.com
S828.3
A
1004-1524(2017)05-0722-07
浙江农业学报ActaAgriculturaeZhejiangensis, 2017,29(5): 722-728
