重庆市2011—2016年甲型H1N1流感病毒分离株NA基因变异分析
2017-05-18陈爽叶盛凌华喻臻唐云
陈爽 叶盛 凌华 喻臻 唐云
400042 重庆市疾病预防控制中心微生物检测所
·论著·
重庆市2011—2016年甲型H1N1流感病毒分离株NA基因变异分析
陈爽 叶盛 凌华 喻臻 唐云
400042 重庆市疾病预防控制中心微生物检测所
目的 研究重庆市2011—2016年甲型H1N1流感病毒株神经氨酸酶(NA)基因特征,为甲型H1N1流感病毒感染的监测和预防提供试验依据。方法 按分离时间及地点的不同选取43株甲型H1N1流感病毒,经RT-PCR扩增病毒NA基因片段并进行序列测定,进行核苷酸及氨基酸序列分析。结果 基于NA基因的进化树显示,2011—2016年的甲型H1N1流感病毒与疫苗株关系均较近。43株病毒之间的NA基因同源性较高,与疫苗株A/California/07/2009(H1N1)的核苷酸同源性为97.9%~99.4%,氨基酸同源性为96.8%~98.9%。研究中大部分甲型H1N1病毒NA蛋白上都具有疫苗株NA蛋白具有的8个糖基化位点,但也有部分毒株增加42位(NQS)的糖基化位点,或者丢失386位的糖基化位点。结论 甲型H1N1病毒的NA序列变异情况逐年增加,但总体上来说,重庆地区大部分甲型H1N1病毒仍对达菲敏感;同时应加强耐药性监测,为制定应对甲型H1N1流感流行措施提供参考。
流行性感冒病毒(Influenza virus,简称流感病毒),属正粘病毒科(Orthomyxoviridae),按核衣壳蛋白(Nucleocapsid protein, NP)和基质蛋白(Matrix protein, MP)不同,分为甲(A)、乙(B)、丙(C)3 型。其中,甲型流感病毒[Influenza A(H1N1) virus]因其表面的血凝素(Hemagglutin, HA)有16个亚型,神经氨酸酶(Neuraminidase, NA)有9个亚型,变异、重组非常频繁,在引起人类流感流行史上有十分重要的意义。HA基因是流感病毒主要抗原基因,其编码的HA蛋白具有免疫原性,能使人体产生保护性抗体;而NA 基因编码的NA蛋白是其表面另一种重要的糖蛋白,是NA抑制剂的主要作用点,NA抑制剂也成为WHO推荐预防和治疗甲型H1N1流感的首选药物,NA催化活性位点、抗原决定簇及糖基化位点的变化均会导致NA抑制剂敏感性发生改变,产生耐药现象[1]。
在对重庆地区的甲型H1N1流感病毒HA基因进行研究后,发现该病毒的HA基因已经累积了一定的变异[2];而重庆地区的甲型H1N1流感病毒的NA基因相关研究暂未见报道。因此,依托重庆本地的流感监测网络,对本地区2011—2016年甲型H1N1流感病毒的NA基因特征进行分析,以了解其遗传进化特征和变异情况,及时发现具有流行病学意义的耐药株,为我市甲型H1N1流感病毒流行的防治和预测提供理论依据。
1 材料与方法

图1 重庆市2011年1月至2016年6月流感监测情况Fig.1 Surveillance of influenza in Chongqing from Jan. 2011 to Jun. 2016
1.1 病毒分离 2011年1月至2016年6月,对重庆市疾控中心流感网络实验室各个哨点医院的样品,以及重庆市各区县流感暴发样本,根据流行性感冒诊断标准[3],进行核酸检测和病毒分离。将分离到的流感病毒株中的甲型H1N1病毒株,按不同采样地点和分离时间,选取代表毒株。
1.2 目的基因扩增 将选取的病毒分离株,用德国QIAGEN公司全自动核酸提取仪QIAxtractor及配套的RNA核酸提取试剂QIAxTractor Reagent Pack VX提取病毒核酸,用德国Qiagen公司一步法RT-PCR试剂盒OneStep RT-PCR Kit和上海英骏公司合成的WHO发布的甲型H1N1流感病毒全基因组测序引物[4]中NA片段引物,完成病毒NA基因核酸的PCR扩增,经美国Invitrogen公司E-Gel© 琼脂糖凝胶电泳系统电泳后,观察到目的条带确认目的片段扩增成功。
1.3 基因测序与序列分析 含有目的片段的PCR反应产物直接送大连宝生物和上海英骏公司进行基因测序和拼接。测序所得序列采用BIOEDIT 7.0软件进行序列比对和剪切;采用MEGA 6.0基于Neighbour-joining(NJ)法生成系统进化树,进行序列分析和氨基酸位点差异分析,以及进行核苷酸和氨基酸序列差异分析;使用NetNGlyc 1.0 Server进行糖基化位点分析。
2 结果
2.1 重庆市2011年1月至2016年6月流感监测病原学分析 从2011年1月至2016年6月间,重庆5家国家级流感网络实验室共检测疑似流感标本26 218份,其中3 568份标本核酸阳性,阳性率为13.61%。其中678份标本核酸检测结果为甲型H1N1,占标本总数的2.59%。从时间分布上看,甲型H1N1在2011年1—3月,2013年1—6月、10—12月出现流行高峰,为当季优势毒株。在2011年7—9月,2012年10—12月,2013年7—9月,2014年1—3月,2016年1—6月有散在毒株出现(图1)。共分离得到甲型H1N1病毒289株。本研究按病毒对应的原始标本的采集时间和采集地点的不同,从中选取了43株甲型H1N1病毒,扩增NA基因并测序,经过拼接和整理后得到其NA基因组序列信息。

注:Bootstrap method,Bootstrap value:1000图2 基于NA基因的系统进化树Note: Bootstrap method,Bootstrap value:1000Fig.2 Phylogenetic analysis of NA gene sequences
2.2 基因进化分析 经与WHO推荐疫苗株A/California/07/2009(H1N1)(以下简称疫苗株)NA基因序列(Genbank ID: KU933487.1)比对,43株2011年至2016年毒株均未出现核苷酸插入与缺失。将上述43株病毒的NA基因序列与Genbank数据库中不同年代、不同分离地点的甲型H1N1流感病毒的NA序列对齐剪切后生成系统进化树(图2)。结果显示, 2009年、2011年至2016年的甲型H1N1流感病毒与疫苗株关系均较近,相同年份的毒株聚集在一个小分枝下,不同年份的毒株按年聚集在一起,在大分枝交叉分布。从2009年开始,越早分离的病毒在进化树中的位置离疫苗株相对更近,后分离得到的病毒在进化树中的位置离疫苗株相对更远,如:2009年与2011年的病毒比2013年和2016年的病毒离疫苗株更近。
2.3 核苷酸及氨基酸差异情况 43株分离株的NA基因的核苷酸同源性为96.8%~100%,氨基酸同源性为95.8%~100.0%;与疫苗株的核苷酸同源性为97.9%~99.4%,氨基酸同源性为96.8%~98.9%(表1)。

表1 2011—2016年甲型H1N1流感病毒株NA基因核苷酸及氨基酸同源性

表2 2011—2016年甲型H1N1毒株相对疫苗株的NA氨基酸位点变化情况

续表2 2011—2016年甲型H1N1毒株相对疫苗株NA氨基酸位点变化情况
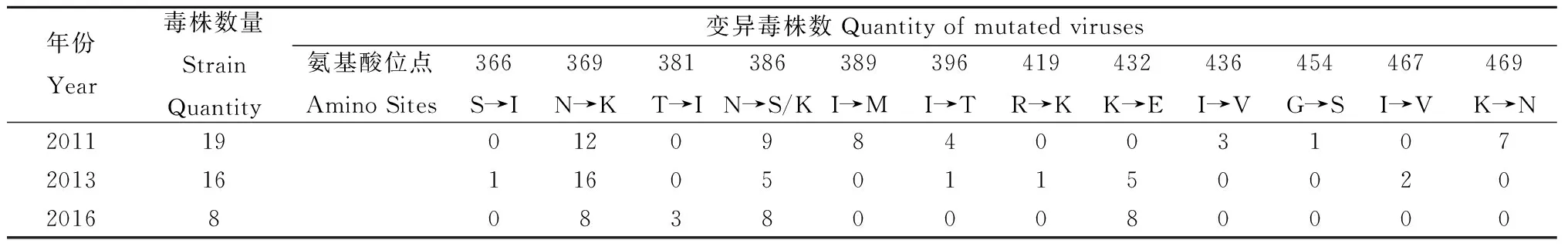
续表2 2011—2016年甲型H1N1毒株相对疫苗株NA氨基酸位点变化情况
2.4 氨基酸变异情况 2011—2016年间的43株甲型H1N1分离株与疫苗株的NA氨基酸序列仅存在一个共同的差异位点:N248D(按N1排序,以下出现的氨基酸序列均为N1排序)。2011年的19株序列与疫苗株相比,全部出现V106I变异的情况;2013年的16株序列与疫苗株相比,全部出现N44S、N200S、V241I、N248D、N369K变异;2016年的8株序列与疫苗株相比,全部出现N44S、N200S、V241I、N248D、V264I、N270K、I321V、N369K、N386K、K432E变异。除此之外,各年度还有部分毒株相对于疫苗株出现氨基酸变异的情况(表2)。
2.4.1 酶活性位点变异:NA 蛋白的酶催化活性位点位于4聚体每个亚单位球状头部的表面,其相关位点的氨基酸组成高度保守。NA 蛋白的酶活性位点包括8 个催化位点,分别是R118、D151、R152、R225、E277、R293、R368、Y402和11个辅助位点E119、R156、W179、S180、D199、I223、E228、H275、E278、N295、E425[5,6]。本次研究的毒株中,A/重庆渝中/SWL11297/2013(H1)出现D151E变异,A/重庆巴南/SWL1677/2013(H1)出现H275Y变异,此两株毒株出现了位于NA蛋白的催化位点或辅助位点的变异,且A/重庆巴南/SWL1677/2013(H1)出现H275Y的变异,提示此毒株是达菲耐药株[7,8]。
2.4.2 抗原决定簇变异:通常认为流感病毒NA蛋白的抗原决定簇有7个氨基酸区域,分别为:152、198~200、328~334、337~344、364~367、396~399、431~434(按N1排序)[9,10],国内近年也有报道,认为140~157可能是抗原决定簇位点[11]。本次研究中D151E,N200S,I365T,S366I,I396T和K432E变异位于抗原决定簇区域内,可能会影响NA蛋白的抗原性。
2.5 糖基化位点分析 经NetNGlyc 1.0 Server分析,本次研究中的大部分甲型H1N1病毒,其NA蛋白上大都具有疫苗株的NA蛋白上具有的8个糖基化位点,即分别位于50、58、63、68、88、146、235和386的糖基化位点。除此以外,2011年有10株(10/19),2013年有16株(16/16),2016年有8株(8/8)病毒的NA蛋白,由于发生了N44S的氨基酸残基改变,从42位起形成了NQS的结构,从而使在NA蛋白上增加了位于42位的糖基化位点(图3)。而2011年有9株(9/19),2013年有5株(5/16),2016年有8株(8/8)毒株,由于发生了N386S/K的氨基酸残基改变,因而丢失了386位的糖基化位点。

注:“□”:潜在糖基化位点;“Ca”:疫苗株A/California/07/2009(H1N1);“14”:“A/重庆巴南/SWL178/2011(H1)”;“28”:“A/重庆巴南/SWL1677/2013(H1)”;“38”:“A/重庆渝中/SWL310/2016(H1)”图3 疫苗株与3个病毒分离株NA基因编码氨基酸序列对比Note:“□”: Potential glycosylation sites; “Ca”: A/California/07/2009(H1N1); “14”:A/ChongqingBN/SWL178/2011(H1);“28”:A/ChongqingBN /SWL1677/2013(H1); “38”:“A/ChongqingYZ /SWL310/2016(H1)Fig.3 Comparison of amino acid residues of NA protein between the vaccine strain and three isolated viruses
3 讨论
2009年暴发于墨西哥、美国的新型甲型流感,其病原为重组新型流感病毒[12],该病毒2009年6月进入重庆并本土化[13,14]。2011开始至2016年6月,甲型H1N1流感病毒在重庆出现两次流行高峰。本研究对2011—2016年两次流行高峰期内分离的43株甲型H1N1流感病毒的NA 基因进行序列测定,与WHO推荐的疫苗株A/California/07/2009(H1N1)对比,进行了进化特征、同源性及相关功能位点分析。
系统进化分析表明,重庆分离株和国内不同年度分离的毒株间存在的年代差异大于地域差异,毒株分离年代相隔越远,毒株亲缘差异越大。核苷酸和氨基酸变异情况对比显示,43株病毒NA基因同源性不低于96.8%,氨基酸同源性不低于95.8%;与疫苗株核苷酸同源性在97.9%以上,氨基酸同源性在96.8%以上,但总体说来所有毒株之间同源性均较高。
本次研究中甲型H1N1流感病毒氨基酸位点变异随着年份增加,出现了旧的变异位点消失,新的变异位点增加,且持续存在的变异位点的变异频率也在改变。但是总体上来说,随着年份的增加,所有分离毒株均出现变异位点数量增加,这说明因氨基酸序列改变而发生的抗原性变化逐年积累的可能性较大。
糖基化在稳定蛋白的三维结构和其被水解以及阻碍抗体的识别中起到重要作用。糖基化位点的改变或增减会对病毒的抗原性和生物特性带来一定的影响;特别是当糖基化位点的改变发生在抗原决定簇时,可能会造成病毒抗原性改变[15,16]。本次研究中的大部分甲型H1N1病毒株和疫苗株一样,在NA蛋白上具有8个稳定的糖基化位点;并且部分分离的毒株增加了位于42位的糖基化位点,或者丢失了位于386位的糖基化位点。相较于疫苗株,糖基化位点未发生较大的改变。这也和山东[17]、南京[18]等分离到的甲型H1N1流感病毒的情况相同。
NA 蛋白是流感病毒包膜上一种重要的糖蛋白,能水解唾液酸,防止成熟病毒自身凝集,促使新产生的病毒颗粒从感染细胞释放,是目前NA抑制剂的重要作用靶点。本研究选择的43株甲型H1N1流感病毒,除了A/重庆渝中/SWL11297/2013(H1)出现D151E变异和A/重庆巴南/SWL1677/2013(H1)出现H275Y变异外,其余病毒NA蛋白酶催化活性位点和辅助位点均未发生改变。D151E位于酶催化活性位点,其改变可能会影响病毒对药物的敏感性,需要进一步进行验证。而H275Y的氨基酸突变已被证实会产生达菲耐药[7,8,19],可对上述两株病毒再进行耐药性检测。
综上所述,本次研究提示,2011—2016年重庆市甲型H1N1流感病毒与疫苗株A/California/07/2009(H1N1)相比,变异程度在逐年积累,但总体上甲型H1N1流行株与疫苗株仍保持较高的同源性,普遍对达菲敏感。但随着时间推移,相对于疫苗株,2013年和2016年的甲型H1N1病毒已经积累了更多的变异,提示应当持续监测甲型H1N1流感病毒,加强耐药性监测,可以及时准确发现抗原漂移株与耐药株,掌握甲型H1N1流感病毒的流行与进化规律,预测其发生发展情况,为制定应对甲型H1N1流感流行措施提供参考,为科学有序地防控甲型H1N1流感提供依据。
[1] Gubareva LV, Kaiser L, Hayden FG. Influenza virus neuraminidase inhibitors[J]. Lancet, 2000, 355(9206):827-835. doi: 10.1016/S0140-6736(99)11433-8.
[2] 叶盛, 喻臻, 陈爽,等. 重庆市2009~2014年甲型H1N1流感病毒分离株HA基因变异分析[J]. 重庆医学, 2016, 45(30):4226-4229. doi:10.3969/j.issn.1671-8348.2016.30.016.
[3] WS285—2008,流行性感冒诊断标准[S]. 北京:人民卫生出版社,2008.
[4] World Health Organization. Sequencing primers and protocol. [DB/OL](2016-11-22). http://www.who.int/csr/resources/publications/swineflu/sequencing_primers/en/.
[5] Colman PM, Hoyne PA, Lawrence MC. Sequence and structure alignment of paramyxovirus hemagglutinin-neuraminidase with influenza virus neuraminidase[J]. J Virol, 1993, 67(6):2972-2980.
[6] Colman PM. Influenza virus neuraminidase: structure, antibodies, and inhibitors[J]. Protein Sci, 1994, 3(10):1687-1696. doi: 10.1002/pro.5560031007.
[7] Nguyen HT, Sheu TG, Mishin VP, et al. Assessment of pandemic and seasonal influenza A (H1N1) virus susceptibility to neuraminidase inhibitors in three enzyme activity inhibition assays[J]. Antimicrob Agents Chemother, 2010, 54(9):3671-3677. doi: 10.1128/AAC.00581-10.
[8] Nguyen HT, Fry AM, Gubareva LV. Neuraminidase inhibitor resistance in influenza viruses and laboratory testing methods[J]. Antivir Ther, 2012, 17(1 Pt B):159-173. doi: 10.3851/IMP2067.
[9] Colman PM, Varghese JN, Laver WG. Structure of the catalytic and antigenic sites in influenza virus neuraminidase[J]. Nature, 1983, 303(5912):41-44. doi: 10.1038/303041a0.
[10] 田疆, 周经姣, 陈艺韵,等. 甲型H1N1流感病毒神经氨酸酶基因遗传进化分析[J]. 中山大学学报(医学科学版), 2010, 31(2):207-212. doi:10.13471/j.cnki.j.sun. yat-sen.univ(med. sci). 2010.0034.
[11] 黄平,俞守义,柯昌文.逐步预测和统计学筛选人H5N1毒株神经氨酸酶蛋白B细胞表位[J].科学通报,2008,53(22):2748-2753.doi: 10.3321/j.issn:0023-074X.2008.22.012.
[12] Smith GJ, Vijaykrishna D, Bahl J, et al. Origins and evolutionary genomics of the 2009 swine-origin H1N1 influenza A epidemic[J]. Nature, 2009, 459(7250):1122-1125. doi: 10.1038/nature08182.
[13] 王伟, 潘明, 常国辉,等. 中国内地首例确诊甲型H1N1流感病例的实验室检测[J]. 病毒学报, 2009(s1):4-7. doi: 10.13242/j.cnki.bingduxuebao.001986.
[14] 李勤, 肖达勇, 龙江,等. 重庆市甲型H1N1流感流行特征及趋势分析[J]. 现代预防医学, 2010, 37(21):4003-4006.
[15] Schulze IT. Effects of glycosylation on the properties and functions of influenza virus hemagglutinin[J]. J Infect Dis, 1997, 176(s1):S24-S28. doi: 10.1086/514170.
[16] Abe Y, Takashita E, Sugawara K, et al. Effect of the addition of oligosaccharides on the biological activities and antigenicity of influenza A/H3N2 virus hemagglutinin[J]. J Virol, 2004,78(18): 9605-9611. doi: 10.1128/JVI.78.18.9605-9611.2004.
[17] 刘倜, 林艺, 王爽,等. 甲型H1N1流感病毒神经氨酸酶基因进化分析[J]. 中国公共卫生, 2011, 27(1):20-22. doi:10.11847/zgggws2011-27-01-10.
[18] 何敏, 石利民, 王燕, 等. 2013年南京市甲型H1N1流感病毒NA基因的进化分析[J]. 南京医科大学学报(自然科学版), 2014,34(12): 1783-1788. doi: 10.7655/NYDXBNS20141248.
[19] Hurt AC, Ernest J, Deng YM, et al. Emergence and spread of oseltamivir-resistant A(H1N1) influenza viruses in Oceania, South East Asia and South Africa[J]. Antiviral Res, 2009,83(1): 90-93. doi: 10.1016/j.antiviral.2009.03.003.
(本文编辑:吕新军)
Analysis of NA gene variation of influenza A(H1N1) virus isolated in Chongqing from 2011 to 2016
ChenShuang,YeSheng,LingHua,YuZhen,TangYun
DepartmentofMicrobeDetection,ChongqingCenterforDiseaseControlandPrevention,Chongqing400042,China
YeSheng,Email:infcqcdc@163.com
Objective To research the gene characteristics of neuraminidase(NA) gene of influenza A(H1N1) virus of Chongqing from 2011 to 2016. Methods Forty-three H1N1 influenza virus strains isolated from 2011 to 2016 were chosen according to the different time and places of isolation. NA genetic sections were amplified by RT-PCR and sequenced, and then the sequences of nucleic and amino acid were analyzed. Results Phylogenetic analysis indicated that the relationship between H1N1 viruses from 2011 to 2016 and the vaccine strain was close to each other. Compared with the vaccine strain A/California/07/2009(H1N1), the homologies in nucleotide and amino acid sequences for NA gene were 97.9%-99.4% and 96.8%-98.9%, respectively. Most of the strains had the same eight potential glycosylation sites as the vaccine strains, and some strains got new potential glycosylation sites at NA protein site 42, while some lost their potential glycosylation sites at NA protein site 386. Conclusions The changes of H1N1 NA gene had increased year by year, but H1N1 viruses in Chongqing area were still sensitive to Daffy. And monitoring the drug resistant viruses should be strengthened, in order to provide measures against H1N1 viruses.
H1N1; Influenza Virus; Sequence analysis; NA gene
叶盛,Email: infcqcdc@163.com
10.3760/cma.j.issn.1003-9279.2017.02.003
甲型H1N1;流感病毒;序列分析;NA基因
2016-1 1-22)
