青岛市2015年成人散发腹泻病例诺如病毒感染分子流行病学特征
2017-05-16赵丹苏志磊张凤史晓燕汪照国
赵丹 苏志磊 张凤 史晓燕 汪照国
266033 青岛市疾病预防控制中心
·论著·
青岛市2015年成人散发腹泻病例诺如病毒感染分子流行病学特征
赵丹 苏志磊 张凤 史晓燕 汪照国
266033 青岛市疾病预防控制中心
目的 了解青岛市2015年成人散发腹泻病例中诺如病毒(Norovirus, NoV)感染的分子流行病学特征。方法 收集2015年5月至12月间青岛市成人散发腹泻病例粪便标本409份,Real-time RT-PCR进行NoV检测,应用RT-PCR扩增NoV ORF1和ORF2部分基因片段,并进行序列测定和系统进化分析。结果 NoV检测阳性率为18.1%(74/409),其中GI 10份,GII 64份。53份GII被成功测序,分成4个基因型,分别是GII.Pe-GII.4(26/53)、GII.P17-GII.17(19/53)、GII.P12-GII.3(7/53)和GII.P16-GII.13(1/53)。GII.Pe-GII.4全部为GII.4-Sydney-2012变异株。2015年5月至8月,GII.4 只占 5.3%(1/19),GII.17是最主要的流行株(68.4%,13/19)。而2015年9月至12月,GII.17减少至10.9%(6/55),GII.4成为最主要的流行株,增加至45.5%(25/55)。结论 NoV是成人病毒性腹泻的重要病原体。GII.4-Sydney-2012和新变异株GII.P17-GII.17是2015年青岛市成人散发腹泻病例中NoV的主要流行株。
诺如病毒(Norovirus, NoV)属于杯状病毒科诺如病毒属,是引起非细菌性胃肠炎暴发和散发的重要病原体之一,全球20%的急性胃肠炎是由NoV引起的,导致至少90%的病毒性胃肠炎和50%的食源性胃肠炎暴发[1]。NoV是RNA病毒,基因组大小7.5~7.7 kb,包括3个开放阅读框架(ORF)。ORF1编码非结构蛋白,包括RNA聚合酶(RNA-dependent RNA polymerase, RdRp)、核苷三磷酸酶(Nucleoside triphosphatases, NTPases)和蛋白酶(Protease)等。ORF2 和ORF3分别编码主要衣壳(Capsid)蛋白VP1 和小衣壳蛋白VP2。根据VP1核苷酸序列差异,分为6个基因组(GI—GVI),GI、GII和 GIV组毒株可感染人类,其中GI有8个基因型(GI.l—GI.8),GII至少有21个基因型(GII.1—GII.21),包含了大部分可感染人类的毒株。NoV通过两个机制发生变异,包括突变和同源重组。自然重组事件在NoV中非常普遍,而最常见的重组发生在VP1基因上游的ORF1-ORF2重叠区[2]。GII.4是近年来全球NoV最主要流行基因型。2014—2015年冬春季,中国、日本、美国、韩国等国家相继报道发现GII.17新变异株[3,4,5]。为了解青岛市NoV基因特征,2015年开展了青岛市散发腹泻病例NoV监测和基因型分析,以明确本地区NoV分子流行病学特征,为今后NoV疫情防控工作提供科学依据。
1 材料与方法
1.1 标本 收集2015年5—12月青岛市第八人民医院和城阳区人民医院急性腹泻(每日腹泻稀便或水样便≥3次)散发住院病例粪便标本409份,-70℃冻存。采集过程均告知患者并征得同意。
1.2 标本处理和核酸提取 冻存标本溶解,加入0.9 ml生理盐水至1.5 ml EP管中,加入0.1 g固体粪便标本或0.1 ml液体粪便标本,置于漩涡振荡器混匀,室温下3 000 rpm离心5 min,吸取上清液提取核酸。利用罗氏自动核酸提取仪LC 2.0,试剂采用罗氏MagNA Pure LC Total Nucleic Acid Isolation Kit(Roche),提取方法按照试剂盒说明书。
1.3 NoV检测 采用Real-time RT-PCR检测,试剂盒采用上海之江NoV GI/GII核酸测定试剂盒,操作方法按照试剂盒说明书,仪器为罗氏LC480II型荧光定量PCR仪。
1.4 序列扩增 参考文献引物[6-8],扩增VP1、RdRp基因片段和ORF1-ORF2重叠区。引物G1SKF/G1SKR: CTGCCCGAATTYGTAAATGA/CCAACCCARCCATTRTACA和G2SKF/G2SKR: CNTGGGAGGGCGATCGCAA/CCRCCNGCATRHCCRTTRTACAT,分别用来扩增GI和GII VP1基因的5′端(即ORF2的C区)[6];引物P290H/289H: GATTACTCCAGGTGGGACTCCAC/TGACGATTTCATCATCACCATA,用来扩增GII RdRp 基因的3′端(即ORF1的A 区)[7]。引物NV33/G2SKR: TACCACTATGATGCAGATTA/CCRCCNGCATRHCCRTTRTACAT,用来扩增GII ORF1-ORF2重叠区[8]。扩增程序如下:50℃ 30 min;94℃ 2 min;94℃ 30 s,58℃ 30 s,72℃ 1 min(1 min/kb),40个循环; 72℃ 7 min。扩增试剂盒采用TaKaRa One-Step RT-PCR Kit(TaKaRa,大连,中国),仪器为AB-96 PCR扩增仪。
1.5 序列测定和分型 RT-PCR产物经过1.5 %琼脂糖凝胶电泳鉴定,阳性产物送青岛擎科梓熙生物技术有限公司测序。测序结果应用Sequencher软件进行序列拼接,以原始峰图为准进行矫正。基因分型通过Nov自动分型工具[9](http://www.rivm.nl/mpf/norovirus/typingtool)和NCBI GenBank nucleotide BLAST(http://blast.ncbi.nlm.nih.gov/ ) 进行。
1.6 序列分析 通过MegAlign软件,采用Clustal W方法进行同源性分析。应用Mega 6.0软件进行序列比对及系统进化分析,系统进化树绘制采用Neighbour-joining方法,Kimura-2-Parameter模式进行,Bootstrap值进行1000次重复运算,参考株均来自于GenBank。
2 结果
2.1 流行特征 对409份粪便标本进行Real-time RT-PCR定性检测,得到阳性标本74份,阳性率18.1 %,其中GI 10份,GII 64份。阳性标本在监测期的每个月都可以检测到,7月、8月和9月的阳性率分别为6.4%、5.6%和6.0%,阳性率较低,但仍有流行,而5月、6月、10月、11月和12月的阳性率分别为22.2%、24.0%、29.2%、37.0%和50.0%。阳性标本中男性患者44例,女性患者30例,男女性别比3∶2。NoV在不同性别间检出差异无统计学意义(χ2=2.24,P=0.134>0.05)。阳性样本来自不同年龄组,NoV在4个年龄段均有检出,主要集中在21~40岁,占54.1%,不同年龄段检出率差异具有统计学意义(χ2=8.77,P=0.032<0.05),见表1。

表1 青岛市2015年5—12月NoV病例年龄和性别分布
2.2 基因型分析 8个GI毒株可成功扩增出VP1片段,分为3个基因型,分别是GI.2(2/74, 2.7%)、GI.3(3/74, 4.1%)、GI.6(3/74, 4.1%)。53个GII毒株可成功扩增出RdRp和VP1片段,分为4个基因型,分别是GII.P17-GII.17(19/74, 25.7%)、GII.Pe-GII.4(26/74, 35.1%)、GII.P12-GII.3(7/74, 9.5%)、GII.P16-GII.13(1/74, 1.4%)。最主要两个基因型是GII.P17-GII.17和GII.Pe-GII.4。GII.Pe-GII.4均属于GII.4-Sydney-2012。2015年9月之前,青岛散发NoV病例中GII.P17-GII.17占据绝对优势(13/19, 68.4%),GII.Pe-GII.4检出较少(1/19, 5.3%)。2015年9月开始,GII.Pe-GII.4成为主要流行株(25/55, 45.5%),GII.P17-GII.17降为第三位(6/55, 10.9%),仅次于GII.P12-GII.3(7/55, 12.7%)。
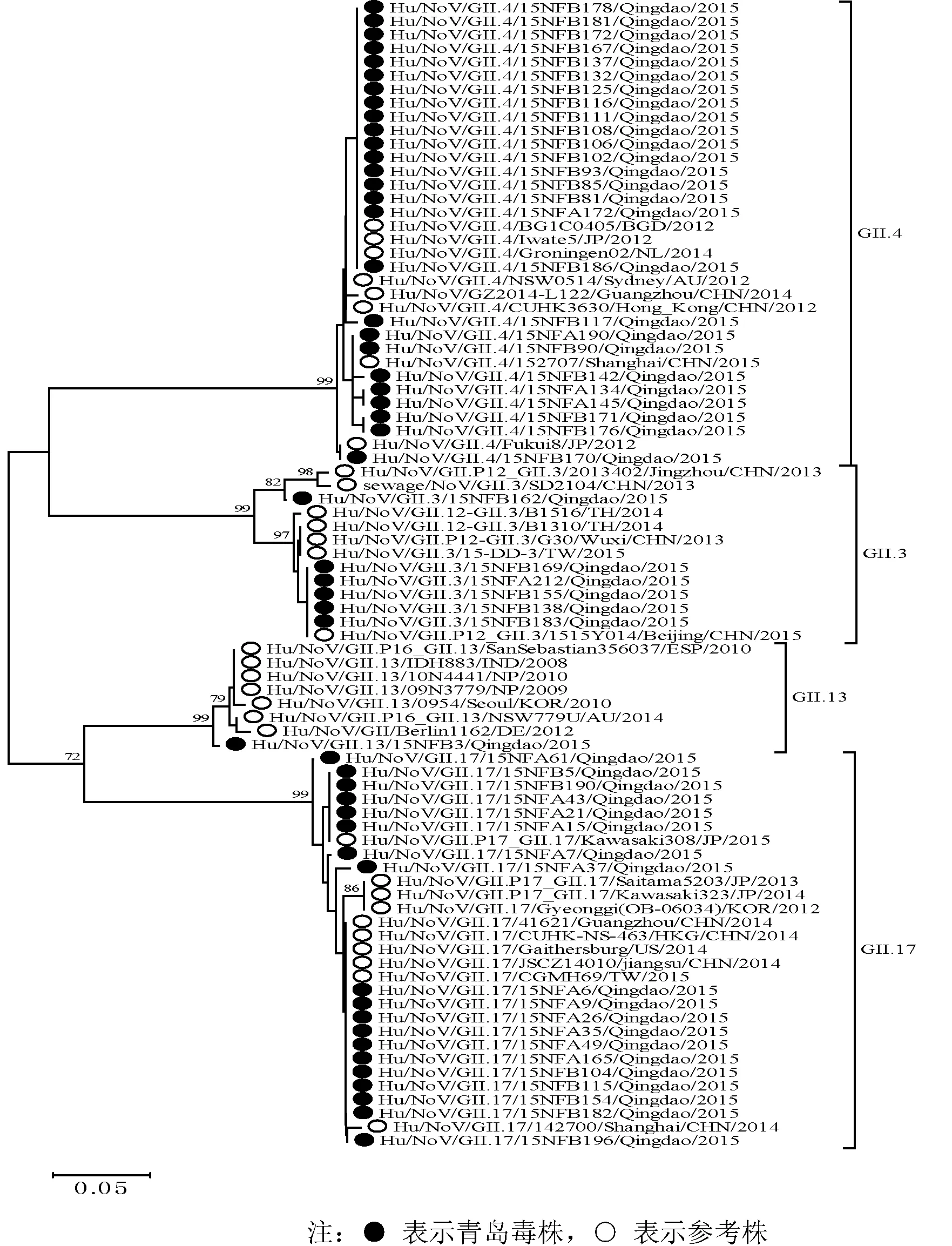
注:● 青岛毒株,○ 参考株图1 青岛市2015年5—12月NoV GII亚型VP1系统进化分析Note: ● indicates Qingdao virus strains,○ indicates reference strainsFig.1 Phylogenetic analysis of nucleotide sequences of VP1 genes of NoV GII strains in Qingdao from May to Dec in 2015
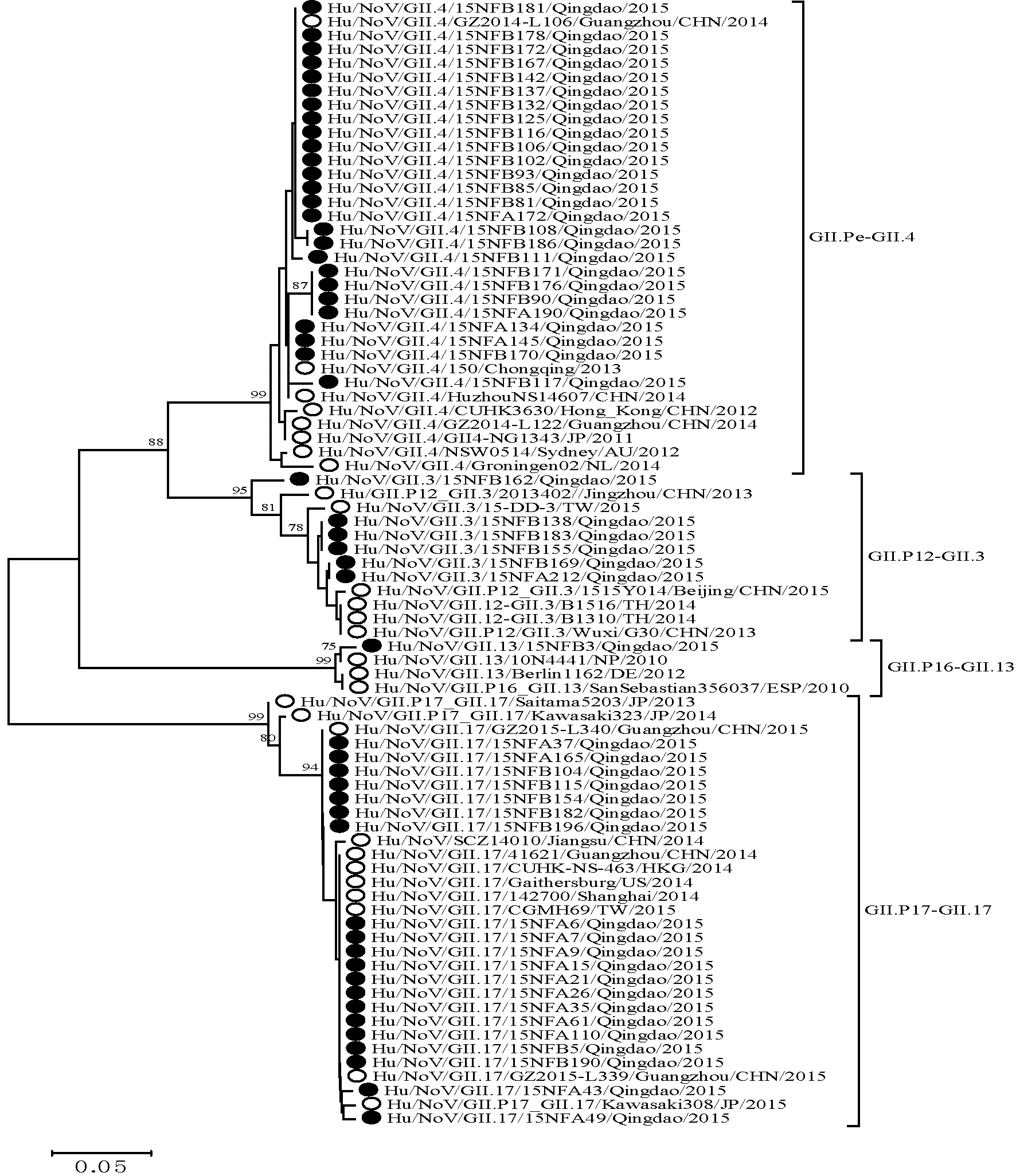
注:●青岛毒株,○ 参考株图2 青岛市2015年5—12月NoV GII亚型RdRp系统进化树Note: ● indicates Qingdao virus strains,○ indicates reference strainsFig.2 Phylogenetic analysis of nucleotide sequences of RdRp genes of NoV GII strains in Qingdao from May to Dec in 2015
2.3 同源性和系统进化分析 核苷酸序列比对表明,GII.17、GII.4、GII.3的VP1区核苷酸同源性分别为96.8%~100%、97.7%~99.7%、93.6%~99.4%。VP1和RdRp进化分析显示,本研究中GII 毒株分为4个亚型(图1, 2)。GII.17青岛株与全球2014—2015年新变异株GII.P17-GII.17在进化上非常接近,但在RdRp序列上与Kawasaki308/JP/2015株更接近,核苷酸同源性99.2%~99.6%,与Saitama5203/JP/2013和Kawasaki323/JP/2014的核苷酸同源性97.7 %~98.5 %。GII.4青岛株均属于GII.4-Sydney-2012,其中部分毒株在RdRp序列上与NSW0514/Sydney/AU/2012原型株的核苷酸同源性96.6%~97.0%,出现了一定程度的进化。大部分GII.3青岛株与2014—2015年GII.P12-GII.3流行参考株类似,例外的是毒株15NFB162/Qingdao/2015在VP1序列上与2013年流行参考株更接近,在RdRp序列上与其他GII.3毒株的核苷酸同源性85.2%~97.3%,有一定程度的差异。GII.13青岛株属于GII.P16-GII.13,与2008—2014年GII.P16-GII.13参考株的VP1核苷酸同源性97.2%~98.8%,在RdRp序列上与尼泊尔毒株10N4441/NP/2010更接近,核苷酸同源性为99.4%。
2.4 重组株的确认 测序得到的ORF1-ORF2重叠区序列与VP1和RdRp分型序列一致,可以确认青岛GII.3型毒株均属于GII.P12-GII.3重组型,GII.13型毒株属于GII.P16-GII.13重组型。
3 讨论
NoV是1968年在美国俄亥俄州诺瓦克地区的一所学校中暴发的急性胃肠炎患者粪便中发现的,并因此而得名。一直以来,NoV没有令人满意的动物模型且难以在体外培养。直到2016年8月,利用一种新的源自人干细胞的类肠道组织培养物终于成功地培养了人NoV[10]。NoV全年都可流行,夏季的阳性率较低,主要集中在冬春季。NoV感染主要集中在21~40岁年龄组人群。男性感染者的数量明显多于女性,似乎显示男性更易于感染NoV,苏州也有类似情况发生[11]。本研究中,成人腹泻散发病例NoV阳性率为18.1%,表明NoV是引起成人腹泻的重要原因。
青岛毒株中GI和GII型均有检出,主要是GII型,检出率由大到小依次是 GII.Pe-GII.4、GII.P17-GII.17、GII.P12-GII.3和GII.P16-GII.13。过去20年里,大约每2~4年GII.4就会发生一次大的变异,形成新的变异株。本研究中青岛GII.4均为GII.4-Sydney-2012。GII.4-Sydney-2012仍然是目前青岛最主要的NoV流行株,但是部分毒株与原型株相比,显示了一定的差异,这种差异是否又预示了一次大变异的开始值得关注。GII.17青岛株与同时期中国、日本、美国、韩国等国家的GII.17毒株非常相近。这种在2014年底被发现的新变异株,被NoroNet命名为GII.P17-GII.17,它的RdRp被认为是一种新的聚合酶,与以往的GII.17毒株有显著的不同[3]。GII.P12-GII.3似乎只在包括中国、日本、韩国、澳大利亚等的亚太地区出现[12]。往年研究显示GII.P12-GII.3仅次于最流行的GII.4[13],因此对GII.3持续关注非常重要。近年来,GII.13的检出也呈现上升趋势,德国、印度、尼泊尔、澳大利亚、新西兰、中国等均有报道[12,14]。GII.P16-GII.13在青岛市被检出,显示这种亚型也具有普遍的流行能力。这种小亚型是否会如同GII.17一样成为重要的流行株也值得关注。本研究中的所有亚型在世界范围内都能找到,因此需要更多的工作来研究它们的起源和传播。
值得注意的是,作为近年最主要流行型的GII.4-Sydney-2012在2015年5—8月并不占优势,检出率远远低于刚刚流行起来的GII.P17-GII.17。然而,在2015年9—12月,GII.4-Sydney-2012株成为最主要流行株。这种情况与GII.4-Sydney-2012株刚出现时情况不同。GII.4-Sydney-2012 株在2012年底被发现,随后在全球范围内持续流行,成为了最主要的NoV流行株。新变异株为何传播如此之快,什么样的生物差异能够改变流行趋势也是亟需解决的问题。本研究的监测时间短,因此GII.P17-GII.17是否会持续流行仍不确定,需要进一步的监测。
[1] Ahmed SM, Hall AJ, Robinson AE, et al. Global prevalence of norovirus in cases of gastroenteritis: a systematic review and meta-analysis[J]. Lancet Infect Dis, 2014, 14:725-730. doi: 10.1016/S1473-3099(14)70767-4.
[2] Stephanie MK, Christiane EW, Ian GG, et al. Advances in norovirus biology[J]. Cell Host Microbe, 2014, 15(6):668-680. doi:10.1016/j.chom.2014.05.015.
[3] Matsushima Y, Ishikawa M, Shimizu T, et al. Genetic analyses of GII.17 norovirus strains in diarrheal disease outbreaks from December 2014 to March 2015 in Japan reveala novel polymerase sequence and amino acid substitutions in the capsid region[J]. Euro Surveill, 2015, 20(26):21173. doi:10.2807/1560-7917.ES2015.20.26.21173.
[4] Gabriel IP, Kim YG. Genome of emerging norovirus GII.17, United States, 2014[J]. Emerg Infect Dis, 2015, 8:1477-1479. doi:10.3201/eid2108.150652.
[5] Chan MCW, Lee N, Hung TN, et al. Rapid emergence and predominance of a broadly recognizing and fast-evolving norovirus GII.17 variant in late 2014[J]. Nature Communications, 2015, 6:10061. doi: 10.1038/ncomms10061.
[6] Kojima S, Kageyama T, Fukushi S, et al. Genogroup-specific PCR primers for detection of Norwalk-like viruses[J]. J Virol Methods, 2002, 100(1-2):107-114. doi:10.1007/s11237-006-0014 -7.
[7] Jiang X, Huang PW, Zhong WM, et al. Design and evaluation of a primer pair that detects both Norwalk-and Sapporo-like caliciviruses by RT-PCR[J]. J Virol Methods, 1999, 83(1-2): 145-154. doi:10.1016/S0166-0934(99)00114-7.
[8] Kim JS, Kim HS, Hyun J, et al. Molecular epidemiology of human norovirus in Korea in 2013[J]. Biomed Res Int, 2015, 2015:468304. doi:10.1155/2015/468304.
[9] Kroneman A, Vennema H, Deforche K, et al. An automated genotyping tool for enteroviruses and noroviruses[J]. J Clin Virol, 2011, 51(2): 121-125. doi:10.1016/j.jcv.2011.03. 006.
[10] Khalil E, Sue EC, Kosuke M, et al. Replication of human noroviruses in stem cell-derived human enteroids[J]. Science, 2016, 353(6306):1387-1393. doi:10.1126/science.aaf5211.
[11] Fu JG, Ai J, Qi X, et al. Emergence of two novel norovirus genotype II.4 variants associated with viral gastroenteritis in China[J]. J Med Virol, 2014, 86(7):1226-1234. doi:10.1002/ jmv.23799.
[12] Lim KL, Hewitt J, Sitabkhan A, et al. A multi-site study of norovirus molecular epidemiology in Australia and New Zealand, 2013-2014[J]. PLoS One, 2016, 11(4):1-19. doi:10.1371/journal.pone.0145254.
[13] Jin M, Xie HP, Duan ZJ, et al. Emergence of the GII4/2006b variant and recombinant noroviruses in China[J]. J Med Virol, 2008, 80:1997-2004. doi:10.1002/jmv.21308
[14] Hoa-Tran TN, Nakagomi T, Sano D, et al. Molecular epidemiology of noroviruses detected in Nepalese children with acute diarrhea between 2005 and 2011: Increase and predominance of minor genotype GII.13[J]. Infect Genet Evol, 2015, 30:27-36. doi: 10.1016/j.meegid.2014.12. 003.
(本文编辑:吕新军)
Molecular epidemiology of Norovirus infection among sporadic hospitalized adults with diarrhea in Qingdao, 2015
ZhaoDan,SuZhilei,ZhangFeng,ShiXiaoyan,WangZhaoguo
QingdaoMunicipalCenterforDiseaseControlandPrevention,Qingdao266033,China
WangZhaoguo,Email:wzg-003@163.com
Objective To analyze the molecular epidemiology of Norovirus(NoV) infection among sporadic hospitalized adults with diarrhea in Qingdao, 2015. Methods Four hundred and nine stool samples were collected from hospitalized adults with diarrhea and detected by Real-time RT-PCR. For genotyping, ORF1 and ORF2 were partially amplified by RT-PCR and sequenced. Results 18.1%(74/409) of stool samples were positive for NoV genogroup I(GI) (10/74) and genogroup II(G II) (64/74). Fifty-three positive samples of GII were sequenced and divided into 4 genotypes, including GII.Pe-GII.4(26/53), GII.P17-GII.17(19/53), GII.P12-GII.3(7/53) and GII.P16-GII.13(1/53). All GII.4 strains were the variants of GII.4-Sydney-2012. From May to Aug in 2015, GII.4 only accounted for 5.3%(1/19) and GII.17 were the major epidemic strains(68.4%,13/19). But from Sept to Dec in 2015, GII.17 decreased substantially to 10.9%(6/55) and GII.4 became the most predominated strains (45.5%, 25/55). Conclusions NoV is an important pathogen responsible for viral diarrhea among adults in Qingdao. GII.4-Sydney-2012 and the newly variant GII.P17-GII.17 were the predominant epidemic strains in Qingdao, 2015.
Norovirus; Diarrhea; Genotype; Molecular epidemiology
汪照国,Email: wzg-003@163.com
10.3760/cma.j.issn.1003-9279.2017.02.004
诺如病毒;腹泻;基因型;分子流行病学
2016-09-26)
