基于生成对抗网络的乳腺癌病理图像可疑区域标记
2017-05-04刘海东杨小渝朱林忠
刘海东,杨小渝,朱林忠
1. 中国科学院计算机网络信息中心,北京 100190
2. 中国科学院大学计算机与控制学院,北京 100049
3. 北京大学肿瘤医院,北京 100142
引言
当前,乳腺癌成为女性人群中常见的一种癌症疾病,严重的影响了女性的生活。乳腺癌的诊断首先通过 X 射线成像来进行检查,初步确定病灶的性质,然后穿刺获得该组织结构,并将其染色制成样本,得到组织切片。通过对组织进行切片,然后由病理医生在显微镜下通过观察该切片诊断得出是否为乳腺癌。在临床医学中,病理图像分析是重要的癌症确诊方法。针对乳腺癌病理诊断过程,诺丁汉分级系统提出,病理切片中所呈现的三种组织细胞形态学特征,包括腺小管的构成,细胞核的多形态和核分裂象的数量是乳腺癌评估的关键指标。这是诊断癌症常用的比较准确的方法。现在越来越多的工作是将显微镜观察后的结果转变为数字病理图像,病理学家可以通过电脑来分析这些病理图像,进而得出诊断结果。
目前在临床医院,对于组织切片的常规分析仍然是一个艰苦的、漫长的过程,需要人工手动完成,而且需要具备长期经验的病理学家才能完成这个过程。由于染色剂浓度、成像模式以及在制作病理切片中人为操作的影响,严重的影响病理图像的质量。并且手动观察,标记产生的有标记的乳腺癌病理图像的数量有限,使得病理图像分析成为当前该领域的一大难题。
目前在医疗领域,给出详细的病理图像标记是十分昂贵的,如何利用这些少量的有标记 (仅标记该病理图像是否为乳腺癌) 的病理图像数据来提高病理图像识别的准确率以及采用弱监督学习的方式给出可疑区域是急需解决的问题。一旦这类任务得到解决,则会极大的提高病理专家诊断的速度,减少由于医生疲劳而造成的错误诊断。
为此本论文提出了一种基于弱监督学习的可疑区域标记网络。它是一种基于生成对抗网络 (Generative Adversarial Network, GAN) 的病理图像特征提取和可疑区域标记网络。该网络首先训练一个基于 GAN 的特征提取网络,其中该特征提取网络后接一个分类网络,通过利用有标记数据 (仅标记该病例图像是否为乳腺癌) 来完成分类网络的有监督训练。然后融合提取到的特征,给出最终的病理图像可疑区域标记。实验证明,该网络提取到的病理图像特征具有好的判别能力,经临床医生评估,得到的可疑区域标记结果初步符合病理学专家的直观,说明使用我们的方法提取到的特征具有比较好的判别性。
其中需要指出的是,本文提出的基于弱监督学习病理图像可疑区域标记方法,需要基于有标记的病理图像数据来训练分类网络,只有保证该分类网络的识别准确率足够高的情况下,才能保证该网络中的特征提取模块能够提取到具有判别力的特征,通过对提取到的特征进行融合,来给出病理图像的可疑区域标记结果。
1 相关工作
1.1 传统病理图像分类方法
传统病理图像分类方法的主要是乳腺癌病理专家通过经验手动设计特征提取方法,来完成病理图像特征提取进而识别出是否是乳腺癌。针对癌症图像自动诊断技术的研究已经超过了 40 年[1]。但是由于医疗图像的复杂性,癌症图像诊断仍然是一个巨大的挑战。例如 Kowal 等人[2]针对细胞核分割,比较和测试了多种不同的算法,这些实验均在 500 个图像数据上进行,数据包含两个类别,一个是良性肿瘤,一个是恶性肿瘤,最终的分割准确率达到 96% 到 100%。Filipczuk 等人[3]设计并实现了一个乳腺癌诊断系统,这个诊断系统针对细针活检得到的细胞病理图片进行分类,来辨别是肿瘤的类型,该系统在 737 张图像上进行实验,达到了 98% 的准确率。与以上两个工作相似,George 等人[4]也实现了一个系统,该系统是针对乳腺癌细针活检得到的图片进行细胞核分割。使用多种不同的机器学习方法,例如神经网络和支持向量机,在 92 张图片中,达到的准确率为 76% 到94%。Zhang 等人[5]提出了一种级联的方法,在第一层级联,作者希望解决简单的问题,难的问题交由第二级联来完成,在第二级联使用了更加复杂的模式分类系统来完成。他们在以色列理工学院提供的 361 张图像组成的数据集上,得到的 97% 的分类准确率。同一个作者的另一个工作[6],使用了集成分类器,在这个数据集上得到了 92% 的准确率。
直到现在,大多数的工作是在乳腺癌病理图像分析上进行的,使用的都是非常小的数据集,而且在科学界也很难拿到这些数据。为了解决这种情况,Spanhol 等人[7]提供了包含 7909 张乳腺癌病理图像数据集,在该研究中,作者评估了 6 种不同的纹理特征描述子和不同的分类器,进行了一系列的实验,准确率从 80% 到 85%,识别的准确率主要这取决于病理图像的放大倍数。[7]中得出的结果表明纹理特征可以为乳腺癌病理图像分类提供更好的特征表示。然而,现在许多机器学习方法的弱点在于强烈依赖于特征工程,LeCun 等人[8]认为机器学习算法应该更少的依赖于特征工程,算法可以从数据中自动对图像中的模式进行提取和组织。现有深度学习方法主要基于卷积神经网络[18](CNN) 来进行图像的特征提取和分类。
1.2 基于深度卷积网络的病理图像分类方法
基于卷积神经网络 (CNN) 的方法在图像特征提取和分类任务上取得了很好的效果。CNN 由 LeCun等人[9]提出,使用该技术在多个不同的模式识别问题上[10-11]上取得了很好结果。CNN 在大量有标记的数据集上取得了非常好的效果,例如 ImageNet 等数据集上。如图 1 所示,是典型的由 CNN 网络结构构成的 AlexNet 网络,由此网络来完成图像分类任务。
Hafemann 等人[12]的实验显示,在微观和宏观的图片上,CNN 提取得到的纹理特描述符的表征能力超过了传统的纹理描述符,进一步说明 CNN 在病理图像识别任务中取得了非常明显的效果。除此之外,使用传统方法提取病理图像中的特征需要大量的领域知识,而且根据这些领域知识得到的结果并不适用于其他的情况。在 BreaKHis 数据集上进行的一系列实验[7]显示,CNN 要比其他的使用了纹理描述符的机器学习算法的结果好。通过使用简单的融合规则,合并不同的 CNN 结构,例如最大池化,乘积,求和等操作后,得到的病理图像识别结果相比于[7]中的识别准确率,提高了 6%。
CNN 在医学图像领域同样得到了很大的关注,但是并没有得到全面的推广,阻碍其应用的主要原因是缺少大量的医学标记数据,因此便不能通过 CNN来得到好的特征,最终导致分类任务的准确率并不能得到极大的提升。近期针对 CNN 方法,Bayramoglu等人[24]提出的基于深度学习的放大倍数独立的乳腺癌病理图像分类器,准确率比传统的针对不同放大倍数分别训练的分类器的准确率高。Spanhol 等人[25]等人提出了基于 DeCAF 特征的乳腺癌病理图像分类算法,该算法利用预训练来初始化模型参数,最终得到目前最高的分类准确率。
1.3 基于生成对抗网络的病理图像分类方法
近期生成对抗网络 (Generative Adversarial Network, GAN)[14]的提取在拟合数据分布方面取得了很好的效果。基于 GAN 方法分别训练一个生成器和一个判别器,判别器用于判别数据来自于真实训练数据还是生成器生成的数据。通过采用博弈训练的方式来使生成器生成符合真实训练数据分布的数据。但是这种方法无法生成具有标记的数据,Mirza 等人[16]提出的条件生成对抗网络用于生成指定标记的图像,这样便给扩充样本量提供条件。通过增加样本数据可以防止模型过拟合,提高模型的识别准确率。Radford等人[26]提出的深度卷积生成对抗网络,其中将 GAN用于图像特征提取,实现了半监督的图像识别,但是并没有说明具体细节,而且只是用到了简单的图像识别场景。前面提到大大多数基于 GAN 的图像生成方法,只能生成小分辨率的图像,而对于高分辨率的图像,Denton 等人[17]使用拉普拉斯金字塔,将卷机神经网络结构引入到拉普拉斯金字塔方法中,在每一层使用[14]中提到的 GAN 来训练生成图像,逐层生成高质量的图像,最终得到高分辨率的可用于扩充训练数据的有效图像。
1.4 物体检测算法
传统的物体检测的研究重点在于特征提取和分类器。最早的工作是由 Viola 和 Jones[30]提出的,他们的工作中使用类似 Haar 的特征提取方法和增强的分类器对滑动窗口上的区域进行分类来检测是否存在物体。[31]中的算法提出使用 HOG 特征提取和线性 SVM来完成行人检测。DPM 算法[32]提出了不可变图模型,将隐 SVM 作为滑动窗口分类器。选择性搜索算法[33]采用空间金字塔特征和 SIFT 特征[34],并引入核SVM 完成分类。Regionlet 方法[35]在 HOG 特征和其他特征上学习一个增强的分类器。卷积层可以应用在任意大小的图像上,并能产生按比例大小的特征图。在 Overfeat 方法[36]中,在卷积特征图后接全连接网络,实现分类,定位和检测。在基于 SPP 网络的方法[37]中,对建议区域的卷积特征图进行池化操作,将得到的特征送入全连接网络,给出最终的分类结果。与[37]中的工作类似,同期还有几个工作[38-41],使用了SPP 网络的特征池化,采用相同的全局特征共享和区域多层感知机作为区域分类器,在最终的检测准确率上得到提升。在 Fast R-CNN[38]方法中,通过兴趣区域 (Region-of-Interest, ROI) 网络层实现端到端的共享卷积层的微调。在 Faster R-CNN[39],共享特征被用来得到建议区域,这样就减少了产生建议区域的负担。[40]中的方法不需要建议区域,而使用 SPPnet[37]中预定义的区域。基于多区域的方法[41],提取多个尺度下的图像特征,然后进行池化,训练读个模型然后进行集成,给出最终的分类结果。
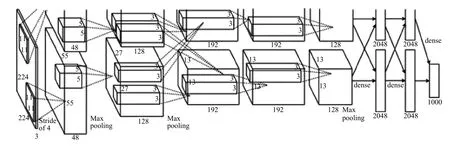
图1 AlexNet 网络结构Fig. 1 Network structure of AlexNet
2 一种基于生成对抗网络的病理图像特征提取与可疑区域标记模型
本节将阐述基于生成对抗网络提出的乳腺癌病理图像可疑区域标记网络。其中,2.1 节具体介绍生成对抗网络的基本概念和主要思想。2.2 节介绍我们提出的可疑区域标记网络的结构和参数设置。2.3 节介绍模型训练方法。
2.1 生成对抗网络 (Generative Adversarial Network,GAN) 概述
生成对抗网络 (GAN) 在拟合数据分布具有很强的能力,通过该模型可以以对抗的方式学习到数据中隐藏的抽象特征,进而利用这些特征可以提高模型的识别准确率。GAN 通过训练两个不同的网络:一个生成网络 (又称生成器)G,和一个判别式网络 (又称判别器)D。生成模型G捕捉数据的分布,判别模型D是一个二分类器,估计一个样本来自于训练数据(而非生成数据) 的概率。G和D为一般的非线性映射函数,可采用全连接神经网络、卷积神经网络等实现。通过不断调整G和D,直到D不能数据区分出来为止。在训练过程中,需要:(1) 优化G,使得它尽可能的让D混淆,(2) 优化D,使得它尽可能的区分出数据来自于真实数据分布还是生成器。D和G进行博弈的过程按照公式1最小化最大化优化公式:
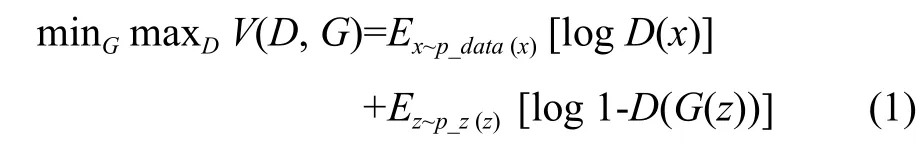
其中:x是真实的图像数据,z是从均匀分布采样得到的噪声数据。
这里假设G采用的 FCN (全连接神经网络),这个网络用来将随机噪声向量z通过一系列的转化,生成图像G(z)。D采用 CNN 和全连接网络构成,用来评估输入的图像是否来自于原有的训练数据集中,这个网络便可以区分输入的图像是真实的还是合成的。这两个网络是同时训练的,具体的优化公式如下:
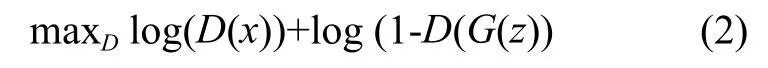
对于生成器G,希望优化如下公式:
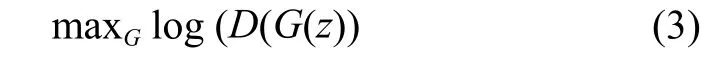
公式 2 和公式 3 是求解公式 1 得到的两个优化子问题,通过优化公式 2 和公式 3 便可以完成模型的训练。判别器D尽可能去辨别此时的病理图像从哪里产生,而G网络负责产生图像病理,与此同时,G网络尽可能产生更加真实的病理图像。
最终利用判别器D中的特征提取模块作为病理图像特征提取模块,提取病理图像的显著判别力的特征,以此增加乳腺癌病理图像识别网络的鲁棒性和泛化性能。
2.2 基于 GAN 的病理图像可疑区域标记网络的架构
如图 2 所示,为基于 GAN 的病理图像可疑区域标记网络。该对抗分类网络主要由 GAN 和基于 CNN的分类网络这两部分构成,最终通过融合多个卷积层的输出特征图来给出病理图像的可疑区域标记结果。相对于传统的基于 CNN 的分类网络,我们提出的对抗分类网络的主要特点在于两部分共用了特征提取网络实现了图像中有判别力的特征提取。该方法的机理在于:(1) 通过对抗分类网络 GAN 中生成器所生成的病理图像数据为判别特征提取网络的训练提供了更多的数据,防止了判别网络的过拟合,进而使得判别特征提取网络具有更好的通用性,使用少量的病理图像数据训练得到的特征提取网络就可以从病理图像中提取更具判别力的特征用于分类任务。(2) 通过共用特征提取网络,可以使分类网络充分利用基于 GAN 训练得到的特征提取网络来完成病理图像的特征提取,进而进行可疑区域标记。
该网络使用 GAN 来完成高维图像数据分布的建模。在该网络内引入了基于 CNN 的特征提取模块。通过对抗方式训练 GAN,也同时完成了图像特征提取模块的训练。从图中可以看到,该模型共分为三个部分:第一部分为图中左下部分,该模块完成了病理图像特征提取和 GAN 的对抗训练;第二部分为图中右上部分,该模块完成了有监督的病理图像分类任务。第三部分通过融合 CNN 的输出特征图来得到病理图像可疑区域的标记结果。
2.2.1 基于对抗训练的特征提取网络
在病理图像分类任务中,如何提取病理图像中丰富的纹理特征是十分关键的。好的特征很大程度决定病理图像分类的准确率。如图 3 (a) 所示,该特征提取网络的输入是乳腺癌病理图像,该图像逐层通过该网络,在每一层分别提取图像中的纹理特征,不断提取图像的高层抽象特征,用于最终的分类任务。
该网络第一层由 Inception 模块构成,紧接着是5×5 的卷积层,然后是 3×3 的最大池化层。通过层叠该结构,最终构成了特征提取网络。该网络最主要是使用了 GoogleNet[19]中的 Inception 模块,该模块分别使用了 1×1,3×3 和 5×5 的卷积核,分别从多个不同的尺度来提取图像中的纹理特征。

图2 病理图像可疑区域标记网络的结构图,该结构图共包含三部分:一部分为左下方的基于GAN的特征提取模块,第二部分为右上方的基于CNN的分类网络模块(其中,Uniform samples是指从均匀分布中采样得到的100维样本数据,Dense是指全连接层,Conv指卷积层,relu(rectified linear unit)是指修正线性单元,一种激活函数,Maxpool最大池化层,Global Average Pooling是指全局平均池化层,ADD是一种层与层相加的运算操作),第三部分为中间的特征融合层,实现了最终的病理图像可疑区域标记Fig. 2 The structure diagram of pathological image suspicious region labeling network. The structure diagram contains three parts:one part is the left bottom GAN-based feature extraction module, and the second part is the upper right CNN-based classi fi cation network module (where Uniform samples refer to uniformly sampled 100-dimensional sample data, Dense refers to the fully connected layer, Conv refers to the convolutional layer, relurefers to a recti fi ed linear unit, an activation function, Maxpoolrefers to maximization pooling layer, GlobalAveragePooling refers to the global Average pooling layer, ADD is a layer-to-layer addition operation. and the third part is the middle feature fusion layer, which labels the fi nal pathological image suspicious region.
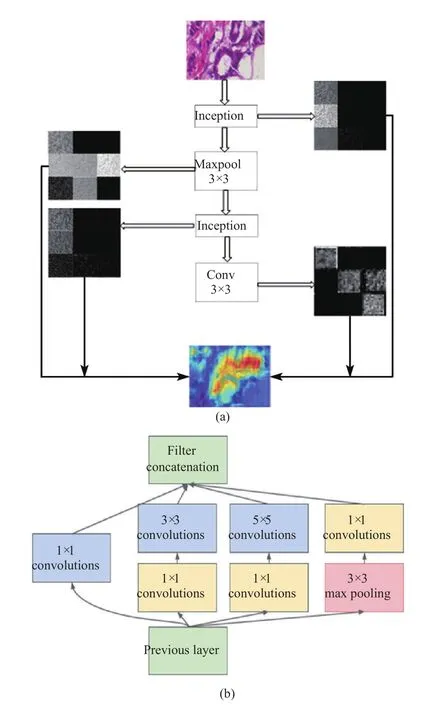
图3 特征提取模块, (a) 表示基于 GAN 的特征提取模块以及每层提取到的特征,最终通过融合各层的特征图得到可疑区域标记结果 (b) 表示 Inception 网络模块,其中Maxpool 表示最大池化层,Conv 表示卷积网络层,Filter concatenation 表示多特征融合层Fig. 3 Suspicious region labeling module,(a) indicates the feature extraction module and the features extracted at each layer. Finally, the suspicious regionlabeling result is obtained by fusing the feature maps of each layer. (b) indicates the Inception network module, where Maxpool represents the maximization pooling layer, Conv represents the convolutional network layer,and Concat represents multiple features Fusion layer
2.2.2 分类网络
如图 2 中所示,分类网络主要由分支全连接网络来完成,其采用对抗特征提取网络的输出作为输入,然后对特征进行多次非线性映射。采用多个卷积神经网络对之前得到的特征进一步抽象,最终使用全局平均池化层给出乳腺癌病理图像分类结果。
2.2.3 可疑区域标记
在第二阶段完成了有监督的病理图像分类网络的训练。在该阶段尽可能保证该分类模型具有较高的分类准确率。这样便使得卷积层提取到的特征更具有判别性,然后通过融合多个网络层的输出特征图,给出最终的病理图像可疑区域标记结果。
2.3 网络的训练过程
对抗分类网络主要包括图 2 中左下方所示的基于生成对抗的特征提取网络,以及图中右上方的基于CNN 的分类网络。因此对抗分类网络的训练主要包含 2 个阶段,分别是基于生成对抗的特征提取网络的训练和基于 CNN 的分类网络的训练。训练数据主要来自于 BreaKHis[7]乳腺癌病理图像数据集,具体介绍在 4.2 部分。
2.3.1 基于生成对抗的特征提取网络的训练
该特征提取网络的训练与 GAN 的训练相似。该网络的输入包含两部分,分别是真实的乳腺癌病理图像和来自均匀分布采样结果。该 100 维样本首先通过全连接网络进行映射,得到 128 个大小为 16×16 的特征图,然后通过 strides 为 2 的上采样网络层完成特征图的上采样,最终得到的 128 个大小为 32×32 的特征图。然后紧跟着是激活层用来对上一层网络的输出做一个非线性变换。通过这样的上采样和非线性激活,最终得到的生成图像大小为 128×128×3。将生成的图像标记为 0,原始的输入图像标记为 1,然后使用这些有标记的数据来完成判别器的有监督训练。从图 2中可以看到,判别器是由特征提取层构成,这样在训练 GAN 的同时,也完成了图像特征提取网络参数的训练。由于采用 GAN 的对抗训练方式,因此称该特征提取网络为对抗特征提取网络。由于传统的 GAN包含生成器和判别器,因此训练的过程中采用随机梯度下降的算法。
在实际训练过程中,由于不能直接得到解析解,因此采用迭代的数值计算方式对模型进行训练。如算法1 中,直接在内循环中训练判别器D的计算量很大,而且在有限的数据集上训练,容易导致D出现过拟合现象。因此我们选择训练k(k的选择可以根据D的复杂度和数据量的大小决定,实验中k取 2) 次训练D,1 次训练生成器G。只要G的变换足够慢,D在每一轮完整的训练 (k次D训练和 1 次G训练) 后都维持在最优解的附近。通过多轮训练,最终实现 GAN 的训练。相应的对抗特征提取网络参数也以对抗的方式得到了训练。
2.3.2 分类网络训练
在通过以上阶段的训练,得到了稳定的对抗特征提取网络,在此训练阶段,固定该特征提取网络,只对分类网络进行单独的训练。输入的病理图像大小为128×128×3,通过图 3 中所示的特征提取网络后,得到的特征大小为 3×3×128。然后经过全局平局池化层将特征映射到 128 维,然后再使用卷积神经网络和全局平均池化层完成最终的分类任务。
算法 1:生成对抗网络批量随机梯度下降训练算法。对于判别器D训练的次数k是超参数,为了最小化计算量,实验中选择k=2

3 模型实现
3.1 基本硬件配置和实验环境
硬件配置:实验采用 NVIDIA® Tesla® K80 GPU计算集群,该集群共包含 13 个 GPU 卡,每个卡含有11G 显存以及 40 个 4.2GHz CPU 核心和 256G 内存。
本论文代码实现采用的编程环境为 Ubuntu 16.04,采用 python 作为编程语言。本论文中网络结构设计采用 Google 开源的 Tensorflow 深度学习框架进行对抗分类网络设计 (通过 python 代码实现),并进行实验。
3.2 乳腺癌病理图像数据集
本实验采用的数据集是国际乳腺癌病理图像数据集 BreaKHis[7],该数据集采集了 82 个人在 2014 年全年的病理图像数据,该数据集包含 4 个放大倍数下观测得到的病理图像数据,如下表 1 所示。
如图 4 所示,是放大倍数为 400X 乳腺癌病理图像,其中共分为 8 个病理组织切片图。
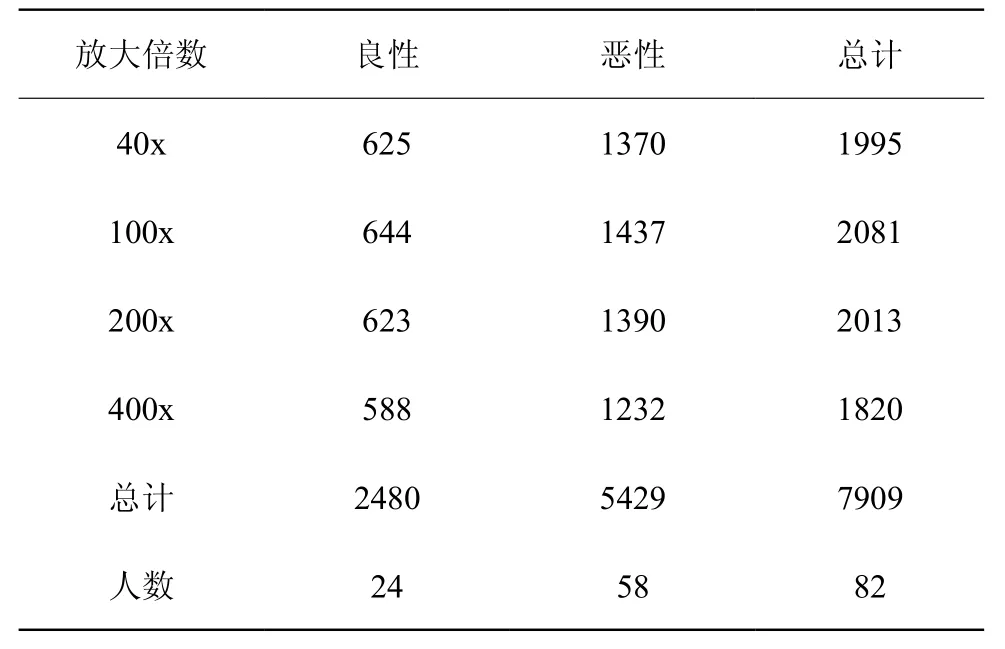
表1 病理图像数据集数据构成Table 1 Composition of Pathological Image Data Set
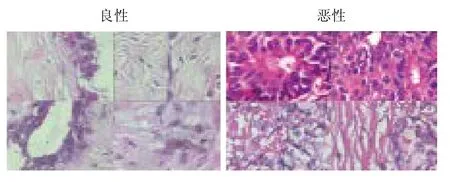
图4 400X 放大倍数的乳腺癌病理图像,来自于 BreaKHis数据集,其中展示了 2 种不同组织的乳腺病理切片图 (每种展示两张图片),类型分为良性肿瘤和恶性肿瘤Fig. 4 400X magni fi cation of breast cancer pathology images from the BreaKHis dataset showing mammary pathological sections of 2 different tissues (each showing two images), divided into benign and malignant tumors type
3.3 数据预处理
由于制作病理组织切片图的过程中,需要对组织进行染色处理,因此染色的不同也会造成病理图像的不同观测结果,不同区域具有明显的不均衡现象,因此需要对图像进行归一化处理,消除图像不均衡现象。为了提高模型的泛化性能,对病理图像进行了图像扩增,其中包括图像平移,图像旋转,图像镜像,图像亮度变化,裁剪和缩放。
4 验证
该实验部分主要论述提出的网络结构对于病理图像纹理特征的提取具有很好的效果。比近期提出的基于深度神经网络的方法[20]识别准确率高 3 个百分点。其中 4.1 节主要说明对抗特征提取网络得到很强的判别性。4.2 节介绍该网络在 BreaKHis[7]数据集的测试结果。
4.1 基于对抗特征提取网络,提取具有高判别性的深度图像特征
如图 3 (a) 所示,为主要的特征提取模块。该模块主要由 Inception 模块 (如图 3 (b) 所示) 和卷积模块构成。该模型主要从多个尺度来提取病理图像中的细节纹理特征,这些特征可用于病理图像识别网络来判断该病理图像是良性还是恶性。从图中可以看到,输入图像为乳腺癌病理图像,首先通过Inception 模块,图中截取了部分特征图用于显示,可以看出神经网络的前几层提取到的特征图包含的纹理特征占据整张图。随着网络的深入,模块提取到的特征更加抽象。
在对病理图像识别任务中,最重要的是图像中的纹理特征。如何从病理图像中更加有效的提取出纹理细节特征直接决定了病理图像的分类效果。图5是截取的由对抗分类网络中的特征提取网络的一些特征提取层的输出结果。从图中可以看到,采用生成对抗方式训练的特征提取网络可以提取到具有判别力的特征,而且对于病理图像中的纹理信息也得到了很完整的提取。从特征图看出,病理图像中很多细节纹理信息得到保留,同时特征图也提取到了对于可疑区域标记任务十分有用的特征。
从图 5 中可以看出,得到的特征图保留了病理图像中原有的纹理特征。且得到的特征图具有一定的可判别性,使用图 5 得到的特征图进一步融合得到如图6 的可疑区域,从图中可以看到,可疑区域准确的定位到细胞发生畸变的区域。由于没有明确的已标记的可疑区域数据,因此无法评价可疑区域的准确率,但通过与临床医生讨论,认为利用提取到的特征图得到的可疑区域基本符合病理学学家的直观判断,证明使用本论文提取到的特征具有较好的判别能力,用来找到一张病理图像中的可疑区域。
4.2 识别准确率对比实验

图5 特征提取网络层输出,(a) 为输入原始乳腺癌病理图像,从 (b)-(d) 依次为对抗特征提取网络由浅层到深层的输出特征图Fig. 5 Output of Feature extraction network layer, (a) Original breast cancer pathology image, and (b)-(d) are output feature maps from the shallow layer to the deep layer
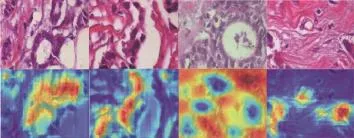
图6 可疑区域:融合不同网络层产生的特征图得到可疑区域,其中上面为原始图像,下面为相应的标记后的可疑区域图Fig. 6 Suspicious region: Fusing the feature maps generated by different network layers to obtain suspicious region, in which the top is the original image, and the following is the corresponding labeled suspicious region map.
由于最终的可疑区域标记是针对 400x 放大倍数的图片,因此针对放大倍数为 400x 的 1820 张训练数据集,进行了如表 2 中所示的 5 种划分方式,产生了5 种分类难度不同的数据集,来验证本论文提出的方法通过训练分类网络来得到高判别力的特征,其中数据集的5种划分方式,是按照[7]的作者提供的代码进行数据集的划分。
从表中可以看出,本论文提出的对抗分类网络在Fold1 数据集上取得了 85.23% 的准确率。在放大倍数为400x 的 5 种数据集上,达到的平均准确率为 83.8%。
本论文提出的以对抗方式训练的网络不仅可以利用少量的数据来完成特征提取网络的训练,而且得到的特征提取网络可以从病理图像中提取更具判别力的特征。由于好的病理图像识别结果,才能进一步保证特征提取模块提取到了具有判别力的特征。已有的病理图像识别方法大多对训练数据分片,通过数据增量来训练复杂的深度神经网络,这样便破环了原始图像的空间信息。而本文为了保留了原图的信息,并没有对图像进行切分,使得可以对全图进行可疑区域标记 (进行分片数据扩增的方法无法进行全图可疑区域标记)。
本文提出的网络在部分数据集上取得的分类效果比较好,但是仍然在一些数据集上不如已有分类方法的准确率高。近期提出的基于预训练的深度卷机网络分类器[25]在该数据集上得到了目前最高的准确率,但该方法仅提高了分类准确率,但并不能对病理图像中的可疑区域进行标记。
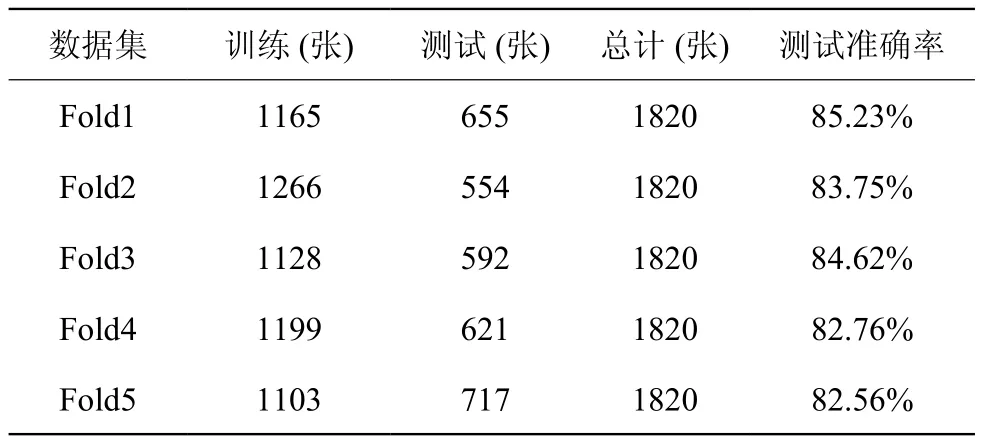
表2 放大倍数为 400x 的乳腺癌病理图像训练集与测试集划分以及模型测试结果,其中将 1820 张病理图像划分为 5个实验数据集 (不同实验数据集划分的识别难度不同, 划分代码按照数据提供者给出)Table 2 The division of Breast cancer pathological image training set and test set and model test results at a magni fi cation of 400x, of which 1820 pathological images are divided into 5 experimental data sets (different experimental data sets have different recognition dif fi culties, and the dividing code is given by data Providers)
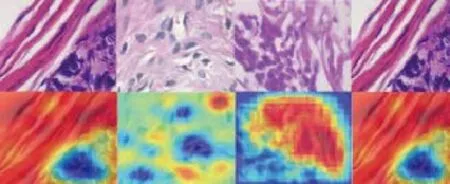
图7 基于对抗分类网络提取到的特征进行可疑区域标记结果,上面部分为原始的乳腺癌病理图像,下面部分是对应的可疑区域标记结果Fig. 7 Suspicious region labeling results. The top part is the original breast cancer pathology image, and the bottom part is the corresponding suspicious regionlabeling result.
5 讨论
本论文提出的基于 GAN 的乳腺癌病理图像可疑区域标记网络。我们不直接使用 GAN 生成乳腺癌病理图像,而是利用 GAN 中的对抗训练思想,来端到端的训练特征提取模型。实验证明以对抗方式训练提取得到的特征具有更好的判别能力。同时根据提取到的特征,对得到的特征图进行上采样,与原始输入图像融合,可以给出乳腺癌病理图像可疑区域的标记。如图 7 所示。对乳腺癌病理图像标记的结果并不理想,仍然存在一些区域不能给出准确的标记。
本文提出的对抗分类网络也同样存在可以改进的地方:(1) 由于考虑到硬件资源的限制,选择的输入图像的低分辨率作为输入 (尺度为 128*128),一定程度图像纹理信息提取不够精确,导致在部分病理图像可疑区域标记做的不够好。后面的实验可以通过增大输入图像的分辨率来进行实验,并调整训练过程中的模型参数以提高病理图像可疑区域标记的准确率。 (2) 由于数据只有两类标记,并没有给出已有可疑区域标记的病理图像数据,因此将来可以考虑融入已有可疑区域标记的图像数据,运用半监督的方式进一步提高病理图像可疑区域标记的准确率。
6 结论
本论文提出了一种新的乳腺癌病理图像可疑区域标记方法。该方法不需要训练数据具有可疑区域标记,而仅包含该肿瘤是良性还是恶性等弱标记即可。该方法将对抗神经网络应用到乳腺癌病理图像特征提取中,并将从图像中提取到的特征应用到图像可疑区域标记任务中,取得了很好的标记效果。该方法能够利用少量乳腺癌病理图像数据,采用有监督的方式训练分类器,即训练网络可以给出输入的乳腺癌病理图像是良性还是恶性。由于在模型训练过程中借鉴了 GAN 中的对抗训练方式,因此该模型中的特征提取网络提取到的特征更具有判别性,进而间接地给出准确的病理图像可疑区域标记结果。基于该方法,能够对乳腺癌病理图像进行可疑区域标记,帮助临床医学快速准确的定位乳腺癌病理图像中的可疑区域,加快诊断过程。本论文提出的对抗特征提取方式具有很好的泛化能力。基于本论文提出的乳腺癌病理图像特征提取和可疑区域标记,同样可以应用到其他类型癌症图像的分析任务中。并且,未来该方法还将拓展到CT 图像,核磁图像,以及更多的癌症类型任务。
[1]B. Stenkvist, S. Westman-Naeser, J. Holmquist, B.Nordin, E. Bengtsson, J. Vegelius, O. Eriksson, and C. H.Fox, “Computerized nuclear morphometry as an objective method for characterizing human cancer cell populations,”Cancer Research, vol. 38, no. 12, pp. 4688–4697, 1978.
[2]M.Kowal, P. Filipczuk, A. Obuchowicz, J. Korbicz,and R. Monczak,”Computer-aided diagnosis of breast cancer based on fi ne needle biopsy microscopic images,”Computers in Biology and Medicine, vol. 43, no. 10, pp.1563–1572, 2013.
[3]P. Filipczuk, T. Fevens, A. Krzy˙zak, and R. Monczak,“Computer-aided breast cancer diagnosis based on the analysis of cytological images of fine needle biopsies,”IEEE Transactions on Medical Imaging, vol. 32, no. 12,pp. 2169–2178, 2013.
[4]Y. M. George, H. L. Zayed, M. I. Roushdy, and B. M.Elbagoury, “Remote computer-aided breast cancer detection and diagnosis system based on cytological images,” IEEE Systems Journal, vol. 8, no. 3, pp. 949–964, 2014.
[5]Y. Zhang, B. Zhang, F. Coenen, and W. Lu, “Breast cancer diagnosis from biopsy images with highly reliable random subspace classifier ensembles,” Machine Vision and Applications, vol. 24, no. 7, pp. 1405 – 1420, 2013.
[6]Y. Zhang, B. Zhang, F. Coenen, J. Xiau, and W. Lu,“One-class kernel subspace ensemble for medical image classi fi cation,” EURASIP Journal on Advances in Signal Processing, vol. 2014, no. 17, pp. 1–13, 2014.
[7]F. Spanhol, L. S. Oliveira, C. Petitjean, and L. Heutte,“A dataset for breast cancer histopathological image classification,” IEEE Transactions of Biomedical Engineering, 2016.
[8 Y. LeCun, Y. Bengio, and G. Hinton, “Deep learning,”Nature, vol. 521, pp. 436–444, 2015.
[9]Y. LeCun, B. Boser, J. S. Denker, D. Henderson, R. E.Howard, W. Hubbard, and L. D. Jackel, “Backpropagation applied to handwritten zip code recognition,” Neural Computation, vol. 1, no. 4, pp. 541–551, 1989.
[10]A. Krizhevsky, I. Sutskever, and G. E. Hinton, “Imagenet classi fi cation with deep convolutional neural networks,” in Advances in Neural Information Processing Systems 25,2012, pp. 1097–1105.
[11]X. X. Niu and C. Y. Suen, “A novel hybrid cnn–svm classifier for recognizing handwritten digits,” Pattern Recognition, vol. 45, no. 13181325, 2012.
[12]L. G. Hafemann, L. S. Oliveira, and P. Cavalin, “Forest species recognition using deep convolutional neural networks,” in International Conference on Pattern Recognition, 2014, pp. 1103–1107.
[13]D. J Brenner and Eric J Hall. Computed tomographyłan increasing source of radiation exposure. N. Engl. J. Med.,357(22):2277–2284, 2007.
[14]I. Goodfellow, Jean Pouget-Abadie, Mehdi Mirza, Bing Xu, David Warde-Farley, SherjilOzair, Aaron Courville,and YoshuaBengio. Generative adversarial nets. In Advances in Neural Information Processing Systems,pages 2672–2680, 2014.
[15]N. Dong, T. Roger, P. Caroline, R. Su and S. Dinggang.Medical Image Synthesis with Context-Aware Generative Adversarial Networks. eprint arXiv:1612.05362, 2016.
[16]M. Mirza and S. Osindero. Conditional generative adversarial nets. CoRR, abs/1411.1784, 2014.
[17]Denton, Emily L, Chintala, Soumith, Szlam, Arthur,and Fergus, Rob. Deep generative image models using a laplacian pyramid of adversarial networks. In Cortes,C., Lawrence, N.D., Lee, D.D., Sugiyama, M., Garnett,R., and Garnett, R. (eds.), Advances in Neural Information Processing Systems 28, pp. 1486–1494. Curran Associates, Inc., 2015.
[18]Y. LeCun, B. Boser, J. S. Denker, D. Henderson,R. E.Howard, W. Hubbard, and L. D. Jackel.Backpropagationapplied to handwritten zip code recognition. Neural computation,1(4):541–551, 1989.
[19]Szegedy, C., Liu, W., Jia, Y., Sermanet, P., Reed, S.E., Anguelov, D., ... &Rabinovich, A. (2015). Going deeper with convolutions. computer vision and pattern recognition,, 1-9.
[20]Spanhol F A, Oliveira L S, Petitjean C, et al. Breast cancer histopathological image classi fi cation using Convolutional Neural Networks[C]. international symposium on neural networks, 2016: 2560-2567.
[21]S. Targ, D. Almeida, and K. Lyman. Resnet inresnet:Generalizing residual architectures. arXiv preprintarXiv:1603.08029, 2016.
[22]A. Krizhevsky, I. Sutskever, and G. E. Hinton.Imagenetclassification with deep convolutional neural networks. InNIPS, 2012.
[23]O. Russakovsky, J. Deng, H. Su, J. Krause, S. Satheesh,S.Ma, Z. Huang, A. Karpathy, A. Khosla, M. Bernstein,et al. Imagenet large scale visual recognition challenge.International Journal of Computer Vision, 115(3):211–252,2015.
[24]Bayramoglu N, Kannala J, Heikkilä J. Deep learning for magnification independent breast cancer histopathology image classification[C]// International Conference on Pattern Recognition. IEEE, 2017:2440-2445.
[25]Spanhol F A, Oliveira L S, Cavalin P R, et al. Deep features for breast cancer histopathological image classification[C]// IEEE International Conference on Systems, Man and Cybernetics. IEEE, 2017:1868-1873.
[26]Radford A, Metz L, Chintala S. Unsupervised Representation Learning with Deep Convolutional Generative Adversarial Networks[J]. Computer Science, 2015.
[27]Z Zhong, J Li Generative Adversarial Networks and Probabilistic Graph Models for Hyperspectral Image Classification arXiv preprint arXiv:1802.03495, 2018 -arxiv.org.
[28]He Z, Liu H, Wang Y, et al. Generative Adversarial Networks-Based Semi-Supervised Learning for Hyperspectral Image Classification[J]. Remote Sensing,2017, 9(10):1042.
[29]Guibas J T, Virdi T S, Li P S. Synthetic Medical Images from Dual Generative Adversarial Networks[J]. 2018.
[30]P. Viola and M. Jones, “Rapid object detection using a boosted cascade of simple features,” in CVPR, 2001.
[31]N. Dalal and B. Triggs, “Histograms of oriented gradients for human detection,” in CVPR, 2005.
[32]P. F. Felzenszwalb, R. B. Girshick, D. McAllester, and D.Ramanan, “Object detection with discriminatively trained partbased models,” TPAMI, 2010.
[33]J. R. Uijlings, K. E. van de Sande, T. Gevers, and A. W.Smeulders, “Selective search for object recognition,”IJCV,2013.
[34]D. G. Lowe, “Distinctive image features from scaleinvariant keypoints,” IJCV, 2004.
[35]X. Wang, M. Yang, S. Zhu, and Y. Lin, “Regionlets for generic object detection,” in ICCV, 2013.
[36]P. Sermanet, D. Eigen, X. Zhang, M. Mathieu, R.Fergus, and Y. LeCun, “Overfeat: Integrated recognition,localization anddetection using convolutional networks,”in ICLR, 2014.
[37]K. He, X. Zhang, S. Ren, and J. Sun, “Spatial pyramid pooling in deep convolutional networks for visual recognition,” inECCV, 2014.
[38]R. Girshick, “Fast R-CNN,” in ICCV, 2015.
[39]S. Ren, K. He, R. Girshick, and J. Sun, “Faster R-CNN:Towards real-time object detection with region proposal etworks,” in NIPS, 2015.
[40]K. Lenc and A. Vedaldi, “R-cnn minus r,” in BMVC, 2015.
[41]S. Gidaris and N. Komodakis, “Object detection via a multiregion& semantic segmentation-aware cnn model,”in ICCV,2015.
