基于红外光谱和随机森林的蕨麻产地鉴别
2017-04-25杨尚梅吉守祥
杨尚梅, 陈 颖, 吉守祥
(青海民族大学 a.化学化工学院; b.药学院,西宁 810007)
基于红外光谱和随机森林的蕨麻产地鉴别
杨尚梅a, 陈 颖a, 吉守祥b
(青海民族大学 a.化学化工学院; b.药学院,西宁 810007)

利用红外光谱与随机森林相结合的方法对不同产地蕨麻进行分类鉴别,测定了42个来自青海省不同产地的蕨麻样品的红外光谱。小波变换对红外光谱原始谱图数据进行了预处理,红外光谱数据压缩到原来的1/8,其分析精度与原始光谱数据基本相当。将42个样品划分为有30个样品的训练集和12个样品的测试集,建立随机森林预测蕨麻产地模型。使用内部交叉验证和外部数据进行验证,采用R语言实现随机森林算法, 并对模型的参数进行了优化。结果表明,所建立的判别模型中训练样本和测试样本判别正确率均为100%。建立的模型能够正确地对蕨麻样品快速进行产地鉴别,红外光谱法结合随机森林可作为中药材产域分类鉴别的一种新的尝试。
蕨麻; 红外光谱; 小波变换; 随机森林; R语言
0 引 言
蕨麻(PotentillaanserineL)为蔷薇科委陵菜属植物鹅绒委陵菜的根,中藏医常用药,又名戳玛、延寿果、人参果等,主产于青海及甘肃甘南等地区[1]。除常被作为营养进补药外,近些年又作为抗肿瘤用药而获得广泛应用[2]。已有色谱、光谱实验技术并结合化学计量学方法鉴别蕨麻质量的报道[3-4],到目前为止,多采用包括多元统计分析在内的单分类器模型鉴别方法[5-6]。本文针对光谱数据庞大,受算法局限,单分类器容易引发过度拟合,使所建模型精度有限,采用数据挖掘中分类器集成的随机森林方法,对不同产地蕨麻的红外光谱数据建模。由于随机森林算法对多线性不敏感,允许多达几千个解释变量,通过参数优化使所建模型精度高[7],成功地实现对青海蕨麻产地的鉴别。
1 实验部分
1.1 样品来源及处理
选用42个不同产地的蕨麻作为研究样品,它们分别采自青海玉树市、果洛州、海南州、西宁市及甘肃甘南等地,经青海省蕨麻研究中心李军乔教授鉴定为蔷薇科委陵菜属植物鹅绒委陵菜的根,即蕨麻。具体产地、样品数及分区见表1。
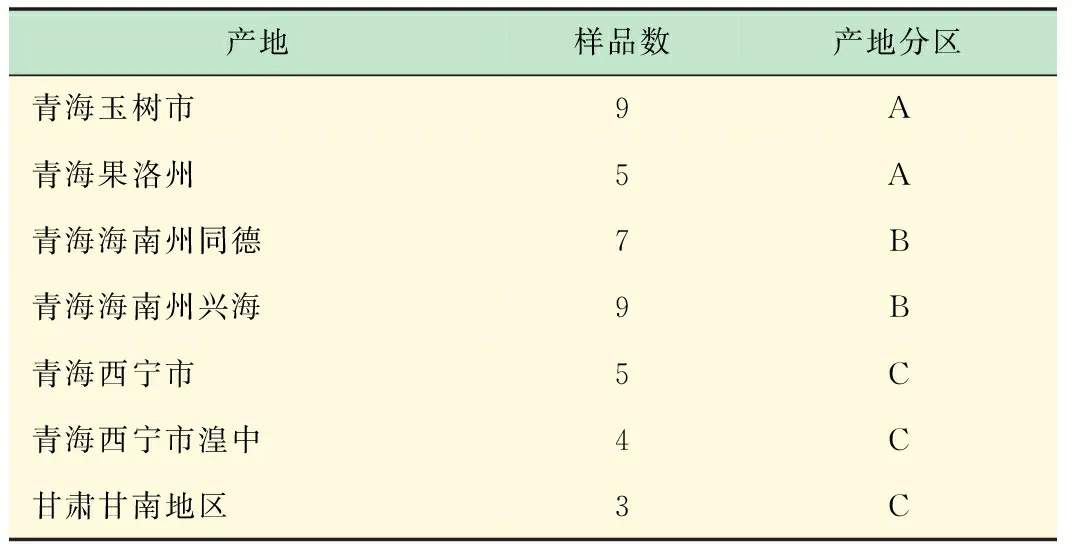
表1 样品来源及分区
采摘的鲜果按产地编号,经洗净晒干后,置于普通干燥箱内65 ℃以下干燥至恒重。制样时,取出用微型植物粉碎机粉碎,过100目筛后装袋密封备用。
1.2 实验仪器及药品
仪器:岛津IRPrestge-21型傅里叶变换红外光谱仪,分辨率0.5 cm-1,信噪比40 000∶1。新型高灵敏度DLATGS检测器。
药品:谱纯溴化钾,天津天光光学仪器有限公司出品。
1.3 不同产地蕨麻红外光谱的测定
按编号依次取已过100目筛的蕨麻粉末3 mg与150 mg光谱纯的溴化钾在研钵中混合均匀后压片,置于IRPrestge-21型傅里叶变换红外光谱仪样品池,按测定范围4 000~400 cm-1,信号累加16次扫描获取红外光谱(分辨率4 cm-1)。利用仪器自带IRsolution软件自动进行多点基线校正和平滑处理。每个样品平行测定3次,取其吸光度平均值作为该样品的红外光谱数据。
1.4 红外光谱的小波变换预处理
利用Matlab小波工具箱,通过小波母函数的选择,最高分解层次的确定以及采用启发式SURE(heursure)方法获取降噪和压缩阈值等步骤,通过编程实现红外光谱降噪和压缩[8]。用于本研究的红外光谱经压缩,数据长度由1 868压缩到241后,作为随机森林建模的输入数据。
2 随机森林算法建模
2.1 随机森林算法原理
随机森林算法是基于Bagging(Bootstrap aggregating)一种组合分类器算法发展而来[9-12]。影响随机森林分类预测能力的因素有:①森林中单棵树的强度,如果每一棵决策树的分类强度越大,则整体随机森林的分类性能越好;②森林中树之间的相关性,若树与树之间相关度越大,像似树与树之间技叶相互穿插越多,则随机森林的分类性能越差。
2.2 随机森林建模的计算机实现
本研究采用R软件平台下的扩展软件包randomForest建立红外光谱数据随机森林模型。调用randomForest即可以建立随机森林模型[13-15]。该函数预设置的核心参数有:mtry参数,表示树节点预选变量个数,决定单棵树性能;ntree参数,表示随机森林中树的数目,决定整片随机森林的性能和规模。
2.3 红外光谱随机森林建模
以42个蕨麻样品小波降噪压缩后的241个红外光谱数据及产地分区变量(REGION)与样品编号(No)作为随机森林建模的数据集(X42×243),分类变量为字符型变量。
随机选取30例样品作为训练集samp=sample(1∶42,30),余下的12例样品x=juema[-samp]作为测试集。建模时使用OOB(out-of-bag) 交叉验证算法能保证训练集与测试集独立,提高预测精度。
以训练集作为数据输入,用randomForest ( )函数默认的参数,即mtry=3,ntree=500时建模,在R平台调用该函数运行后,即可得到蕨麻红外光谱的随机森林模型juema.rf。模型的30个训练集回判全部判对,调用函数命令pred=predict(juema.rf,x),得出12个独立测试集的产地判定结果,其中有1例17号样品判错。初建的随机森林模型有待优化。
2.4 红外光谱随机森林的优化建模
随机森林模型优化是通过调整模型参数mtry和ntree实现的。可采用mtry从1~7逐一增加的方法,由基于OOB数据的模型误判率均值的大小确定模型最优节点变量数。结果当模型节点变量数为1时,模型误判率均值为0.022 276 41最低,因此,参数mtry选取为1。
利用R语言绘图函数plot,通过编程可得到模型误差Error与随机森林中树的数目trees的关系图(见图1),由该图可以确定参数ntree。从图1可以看出,当trees=450左右时,3类的分类误差最小(total),因此,参数ntree选取为450。
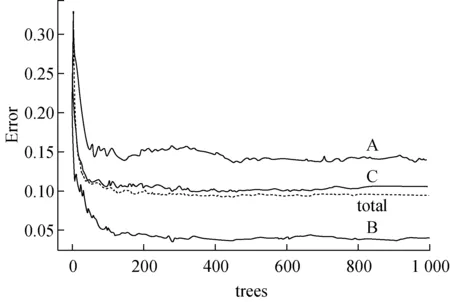
图1 随机森林模型误差与树数量关系图
以优化后的参数建立随机森林蕨麻产地鉴别模型,模型回判和独立测试集的产地归属判定结果正确率均达到100%。
3 结果与讨论
3.1 随机森林模型蕨麻产地鉴别结果
randomForest建模函数训练集和测试集的实际运行总的结果如下:
OOB estimate of error rate: 0%
Confusion matrix:
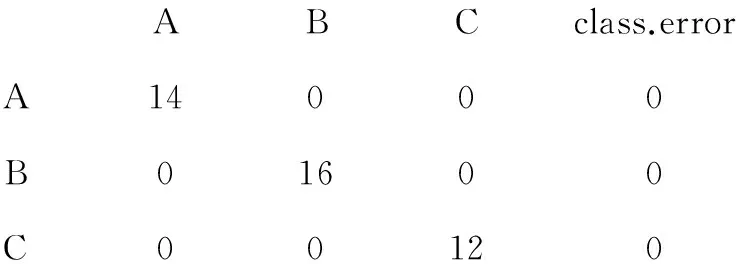
ABCclass.errorA14000B01600C00120
全部样品实现正确的地域分类,可见模型预测精度高,结果令人满意。青海蕨麻产地的上述分类与青海实际情况是一致的。地域划分为A类的青海玉树市与果洛州两地位置靠近,位于青海西南部,青藏高原东部,海拔、气候、土壤等生态环境相似,因而产品质量相似,且品质优良[5];地域划分为B类的青海海南州(同德与兴海县),位于青海湖之南,海拔、气候、土壤等生态环境与地域A存在较大的不同,蕨麻品质良好,自然归于另一类;地域划分为C类的西宁市与甘南地区,位于青海东部,青藏高原的东方门户,海拔、气候、土壤等生态环境与地域A、B存在较大的不同,品质也就有差异,它们划归于C类是合理的。
作为比较,同一数据集交叉验证下,采用单分类器的多元统计Fisher判别分析,误判达5例之多(3,11,17,29,39号样品),正确率88.1%。采用神经网络BP方法建模,随机选取30例为训练集,12例为独立检测集,神经网络参数调优后,预测误判也有2例(17,29号样品),正确率95.2%。可见随机森林建模精度高。
3.2 变量的重要性
图2是调用函数importance获得变量测算出的标准重要值MeanDecreaseAccuracy对241个蕨麻红外光谱波数变量位置的火柴杆图。从图中可以看出,前5个最大的标准重要值3.812 4 (波数位置X98,以下同)、3.080 0(X28)、2.979 6(X223)、2.971 9(X236)、2.848 6(X133)分布在红外光谱R—H(R=O,N,C)伸缩振动峰区至指纹区,揭示若仅取指纹区数据建模,这通常是部分红外光谱应用文献中的一种数据处理方法,必然会丢失部分光谱信息,影响建模精度。本研究虽然数据点由1 868个压缩至241个,但小波变换重构后的压缩数据,仍保留了原始光谱数据的信息,仍然是全谱建模,不会丢失光谱信息,能保证建模精度。

图2 特征波数重要性度量结果
3.3 随机森林判别模型
随机森林算法融合了Bagging算法和随机特征选取两大机器学习技术。大量的理论和实证研究都能够证明该算法建立的模型具有很高的预测准确率,模型结果对缺失数据、多元共线性和非平衡的数据稳健;而且在对数据进行分类的同时,还可以给出各个变量在分类过程中的重要性量度,该量度能够筛选出相对重要变量,从而加深对模型的理解。
4 结 语
实验测定了青海不同产地蕨麻的红外光谱。以R软件平台下的免费扩展软件包randomForest实现随机森林算法,建立了青海蕨麻产地鉴别模型。该模型对产地鉴别预测精度高,正确识别率达到100%,从而为蕨麻分类鉴别、质量控制提供了新的适用方法,也为鉴别其他中草药提供了思路。
随机森林建模方法对样本数据没有特定的要求,需要优化的参数少,模型稳定性好,适合光谱学大量样品数据建模。而且免费扩展软件包容易得到,因而建模方法易于推广,应用前景广阔。
[1] 刘 意,成 亮,延在昊,等.鹅绒委陵菜化学成分及药理作用研究进展[J].中草药,2015,46(8):159-166.
[2] 刘志军,白 瑶,郭丽霞,等.蕨麻的化学成分及药理活性研究进展[J].食品安全质量检测学报,2015,16(9):277-282.
[3] 侯陆星,蔡光明,张雅铭,等. 藏药蕨麻高效液相色谱指纹图谱研究[J]. 中南药学,2007,5(6):555-558.
[4] 夏 莲,孙志伟,李国梁,等. 藏药蕨麻多糖的光谱性质及单糖组成分析[J]. 天然产物研究与开发,2011,23(3): 453-457.
[5] 陈 颖,文 慧,谢久祥,等.青海及周围地区的蕨麻红外图谱的建立及计算机解析[J]. 云南师范大学学报(自然科学版),2014,34(4):65-70.
[6] 白 雁,张 威,王 星,等. 银黄颗粒剂的近红外光谱鉴别分析[J]. 实验室研究与探索,2010,29(6):22-23.
[7] 张晓明,王玉鑫,王 广,等. 基于Hadoop的网站入侵检测与分析系统设计[J]. 实验室研究与探索,2016,35(4):126-128.
[8] 刘明地,李 仲,吴启勋,等.枸杞产地的小波变换红外光谱的聚类分析鉴别[J].华中师范大学学报(自然科学版),2014,48(6):857-860.
[9] Breiman L. Random Forests[J]. Machine Learning, 2001,45:5-32.
[10] Diaz Uriarte R, Andres S A. Gene selection and classification of microarray data using random forest[J]. BMC Bioinform, 2006(7): 3-16.
[11] Prinzie A, Van Den Pdel D. Random forests for multiclassification: random multinomial logit[J]. Expert Systems with Applications,2008,34(3):1721-1732.
[12] Kurtanjek Z. Chemometric versus random forest predictors of ionic liquid toxicity[J]. Chemical and Engineering Quarterly, 2014,28(4): 459-463.
[13] Liaw A, Wiener M. Classification and regression by random forest[J]. Rnews, 2002,2(3): 18-22.
[14] Verikas A, Gelzinis A, Bacauskiene M. Mining data with random forests: A survey and results of new tests[J].Pattern Recognition, 2010, 44(2):330-349.
Identifying the Origin of Potentilla Anserine Based on Infrared Spectroscopy and Random Forest Method
YANGShangmeia,CHENYinga,JIShouxiangb
(a. College of Chemistry and Chemical Engineering; b. College of Pharmacy, Qinghai University for Nationalities, Xining 810007, China)
The infrared spectroscopy combining with random forest method was used in the identification of Potentilla anserine from different fields of Qinghai Province. Forty-two samples of Potentilla anserine from different fields of Qinghai province were surveyed by FTIR (Fourier transform infrared spectroscopy). The original data matrix of FTIR was pretreated with wavelet transform. The results showed that the infrared spectroscopy data were compressed to 1/8 of its original data, but the spectral information and analytical accuracy were not deteriorated. The 42 samples of Potentilla anserine were divided into 30 training samples and 12 validation samples. Random forest model was constructed by the training samples to predict the discrimination effect of identifying the origin of Potentilla anserine with internal cross validation and external validation sample. R language was adopted to achieve algorithm of random forest. Parameters of random forest model were optimized. The prediction accuracy of the proposed model was 100% for the training samples and 100% for the test samples. It can be concluded that the method is quite suitable for the fast discrimination of producing areas of Potentilla anserine. This infrared spectral analysis technology combined the random forest was proved to be a reliable and new practical method for the identification of geographical origin of Chinese medicine. The method in the present paper is very broad prospect of application.
Potentilla anserine; infrared spectroscopy; wavelet transform; random forest; R language
2016-06-27
国家自然科学基金资助项目(81160554)
杨尚梅(1976-),女,青海西宁人,硕士,讲师,现主要从事有机化学与分子光谱研究。
Tel.: 18797181523; E-mail: yangshm528@126.com
O 657.3
A
1006-7167(2017)03-0013-03
