基于线粒体COI基因序列的金乌贼群体遗传学研究❋
2017-04-10单斌斌刘淑德王晓梅高天翔韩志强张秀梅
单斌斌,宋 娜,刘淑德,涂 忠,王晓梅,高天翔,韩志强,张秀梅❋❋
(1.中国海洋大学水产学院,山东 青岛 266003; 2.山东省水生生物资源养护管理中心,山东 烟台 264003;3.中国水产科学研究院资源与环境研究中心,北京 100141; 4.中国水产科学研究院农业部渔业遥感科学观测实验站,北京 100141;5.浙江海洋大学水产学院,浙江 舟山 316022)
基于线粒体COI基因序列的金乌贼群体遗传学研究❋
单斌斌1,宋 娜1,刘淑德2,涂 忠2,王晓梅3, 4,高天翔5,韩志强5,张秀梅1❋❋
(1.中国海洋大学水产学院,山东 青岛 266003; 2.山东省水生生物资源养护管理中心,山东 烟台 264003;3.中国水产科学研究院资源与环境研究中心,北京 100141; 4.中国水产科学研究院农业部渔业遥感科学观测实验站,北京 100141;5.浙江海洋大学水产学院,浙江 舟山 316022)
利用线粒体COI基因片段序列比较分析了金乌贼(Sepiaesculenta)4个地理群体(乳山、青岛、日照和舟山)的遗传多样性和遗传结构。在4个群体117个样本的线粒体COI基因部分序列中,检测到27个单倍型。群体遗传多样性分析显示,4个群体的单倍型多样度为0.478~0.956,核苷酸多样性为0.001~0.006,舟山群体的遗传多样性远高于乳山、青岛和日照群体。群体间遗传分化指数显示,舟山群体与其他3个群体之间存在较大的遗传分化(0.186~0.292),而乳山、青岛和日照群体之间遗传分化较小(-0.007~0.039)。本研究结果暗示,增殖放流活动可能对山东省近海的金乌贼群体的遗传多样性产生了一定的影响。
金乌贼;COI基因;遗传多样性;遗传结构;增殖放流
金乌贼(Sepiaesculenta)隶属于乌贼目(Sepioidea)乌贼科(Sepiidae)乌贼属(Sepia),曾经广泛分布于俄罗斯远东海海域、中国沿海海域、日本的本州、四国、九州海域、朝鲜的西海岸、南海岸海域及菲律宾群岛海域[1]。金乌贼为一年生中下层洄游性头足类,游泳速度较慢,生长迅速,肉质鲜美且蛋白质含量丰富,具有较高的经济价值,曾为我国渔业捕捞的重要对象之一[2-4]。1980年代以来,随着捕捞压力的增大和产卵场环境恶化等因素,金乌贼资源量衰退明显[5]。为了修复和保护金乌贼资源,自1991年起,金乌贼作为一种珍贵的地方品种被列入山东省地方资源增殖计划,此后金乌贼渔业逐渐从捕捞开发型向资源增殖型转变。自2010年金乌贼苗种实现全人工繁育[6],山东省大力开展金乌贼增殖放流工作,放流量逐步提升。2016年,山东省投放胴长1.2 cm以上金乌贼苗种达125.2万头,同比2015年增加48.6%[7]。由于增殖放流的显著效果,山东省金乌贼年产量已由2007年的432 t逐步回升[8],增长到2012年1 864 t[9],近几年金乌贼年产量维持在1 000 t以上,资源增殖效果明显。
随着增殖放流在越来越多的海洋经济种类中得到应用,其对野生资源的遗传学影响也越来越引起研究者们的注意[10-11]。通常用于增殖放流的苗种的遗传多样性远小于该物种的野生群体的遗传多样性,这可能是由于参与繁殖的亲本数量较少、繁殖过程中发生近亲交配以及瓶颈效应等原因[12-13]。大量的放流苗种被投入自然海域,势必对野生群体造成遗传学影响。因此,为了弄清增殖放流对投放海域野生群体的影响,尤其当放流苗种与野生群体的遗传背景不同时,应对野生群体开展遗传学研究[14]。
迄今为止对金乌贼的研究主要集中在资源时空分布及胚胎发育、繁殖习性、生长发育、摄食行为等生物学方面[15-18]。金乌贼群体遗传多样性及遗传分化等方面的研究仅见Zheng对日本海域的4个群体以及中国海域的1个群体进行了微卫星分析[19]。
线粒体DNA(Mitochondrial DNA, mtDNA)具有结构简单、母系遗传、进化速率快、几乎不发生重组等特点,作为一种重要的分子遗传标记而被广泛应用于水产生物的系统发育、生物地理学和保护生物学研究,是分子生物学研究中的一个热门领域[20]。其中,细胞色素氧化还原酶I(COI)基因具有进化速率适中的特点,在头足类群体遗传学研究中得到广泛的应用[21-23]。本研究利用线粒体COI基因序列作为分子标记,对采自中国沿岸的4个金乌贼地理群体进行研究,以期阐明其群体遗传结构和遗传多样性现状,为金乌贼的资源保护及合理开发提供基础资料和科学依据。
1 材料与方法
1.1 样品采集
实验所用金乌贼样品于2014-2015年分别采自青岛、乳山、日照、舟山,共计4个群体117只金乌贼(见图1,表1)。所有样品经形态学鉴定后均取肌肉保存于95%酒精溶液中待用。
1.2 提取DNA
取金乌贼肌肉组织约100 mg,经蛋白酶K消化后,使用酚-氯仿抽提法提取DNA[24]。

图1 金乌贼采样地点图

表1 金乌贼群体样品信息及遗传多样性指数
Note:①Sampling sites;②Dates of collection;③Sample sizes;④Number of haplotypes;⑤Haplotype diversity(h);⑥Nucleotide diversity(π);⑦Mean number of pairwise differences(k)
1.3 PCR扩增
根据GenBank上金乌贼序列(No. AB699283.1)设计引物SEF: 5’-ACGAAGAGAGTTAGGTAAG-3’; SER: 5’- TGTGAGACAATATGTGAAA-3’。PCR反应体系为25μL,包括1.25 UTaqDNA聚合酶,各种反应组分的终浓度为200 nmol/L的正反向引物;200 μmol/L的dNTP,10 mmol/L Tris (pH=8.3),50 mmol/L KCL和1.5 mmol/L MgCl2;反应条件为94°C预变性3 min;94°C变性45 s,50°C退火45 s,72°C延伸45 s,35个循环;72°C延伸10 min。以上反应均设阴性对照以排除DNA污染的情况。取2μL PCR扩增产物进行1%琼脂糖凝胶电泳检测(U=5V/cm),使用回收试剂盒对目的条带进行回收纯化,对回收纯化后的产物送至上海桑尼生物科技有限公司进行双向测序。
1.4 数据分析
测序均采用正反链双向测序,为保证序列的准确性,使用Clustal X[25]将测得的COI基因片段序列进行比对、编辑和分析,并对结果辅以人工校正;使用ARLEQUIN v.3.5[26]计算单倍型数目、多态位点、插入/缺失、转换、颠换等遗传多样性指数;同时通过软件ARLEQUIN v.3.5[26]根据Nei的公式[27]计算核苷酸多样度(π)、单倍型多样度(h)、两两序列比对差异数(k)以及相应的标准差。

2 实验结果
2.1 群体遗传多样性
所分析的4个群体共117只金乌贼的线粒体COI基因序列长度为562 bp,共检测到29个多态位点,包括17个简约信息位点,5处颠换、25处转换。变异多发生在密码子第一位点,第二位点仅存在2个变异位点,第三位点未检测到变异位点。核苷酸组成分析结果显示,金乌贼COI序列A+T较为丰富,远高于C+G的含量(A: 27.57%; T: 36.98%; C: 19.06%; G: 16.38%)。
在117只金乌贼样品中共检测到27个单倍型。4个群体中单倍型数量最少的为RZ群体,单倍型最丰富的为ZS群体,RS和QD群体的单倍型数均为8个。其中群体间共享单倍型为7个,占单倍型总数的25.9%。仅单倍型3与单倍型5为4个群体共享。共享单倍型3的金乌贼个体数最多,共67个个体拥有此单倍型。在20个独享单倍型中,QD群体4个;RS和RZ群体各3个;ZS群体10个。
4个群体中,青岛、乳山、日照群体单倍型多样度、核苷酸多样度及两两序列比较的平均碱基差异数较低,其中,舟山群体单倍型多样度(h=0.956)、核苷酸多样度(π=0.006)以及两两序列比较的平均碱基差异数(k=3.453)均高于其它3个群体(见表1)。
2.2 群体遗传结构
以虎斑乌贼(Sepiapharaonis)为外群,使用软件MEGA 5.0[23]基于TrN+G[24]进化模型构建单倍型邻接关系树。结果显示,虎斑乌贼与金乌贼明显分开,系统发育树的拓扑结构比较简单,谱系结构不明显,没有检测到与采样地点相吻合的分支,即单倍型分支并不与地理分支相对应(见图2)。单倍型最小跨度树也未检测到明显的谱系结构(见图3)。
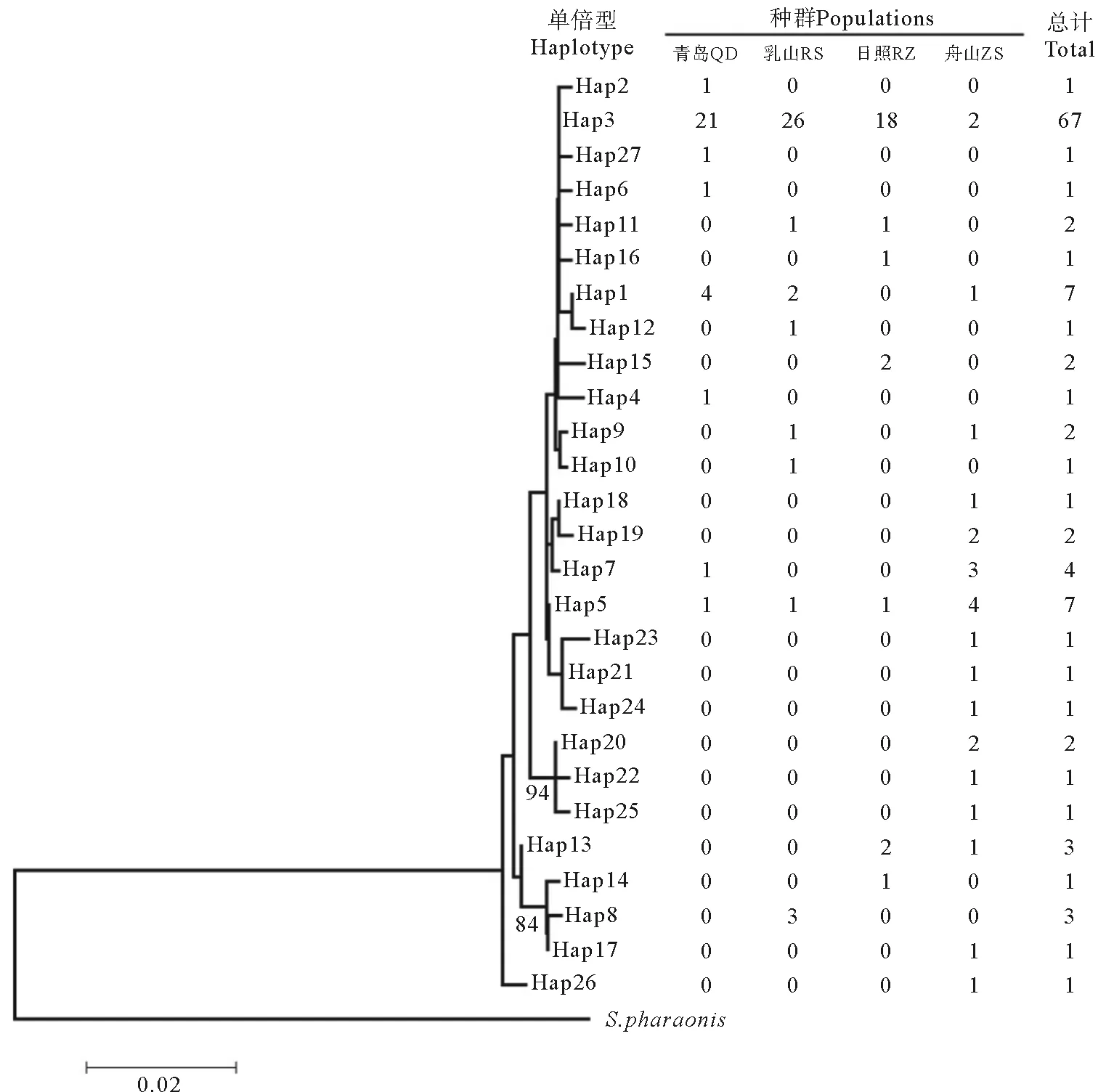
图2 金乌贼单倍型分布及以虎斑乌贼为外群构建的金乌贼单倍型系统发育树
两两群体遗传分化指数(FST)经过Bonferroni校正后除RS与QD、RS与RZ群体外,其余均显著或极显著。结果显示,ZS群体与其他群体之间的FST值(0.186~0.292)远高于QD、RS和RZ群体之间的FST值(-0.007~0.039)。此外,确切P检验的结果显示,QD、RS和RZ群体间均存在随机交配,而ZS群体与其他群体不存在随机交配(见表2)。

(圆圈面积与单倍型频率成正比,短划线代表单倍型间的核苷酸替换数。Sizes of circles were proportional to haplotype frequency. Perpendicular tick marks on the lines joining haplotypes represent the number of nucleotide substitutions.)

表2 金乌贼两两群体间遗传分化指数(FST)及确切P检验值结果Table 2 Pairwise FST (below diagonal) and non-differentiation exact test significance values (above diagonal) among S. esculenta populations
注:*Bonferroni校正后显著(P<0.05),**Bonferroni校正后极显著(P<0.01)。
Note:*P<0.05 after Bonferroni correction ,**P<0.01 after Bonferroni correction.
在进行分子方差分析(AMOVA)时,先将QD、RS、RZ和ZS四个群体作为一个组群进行分析,结果显示85.15%的遗传变异来源于群体内个体间,14.85%的遗传变异来源于群体间;将4个群体按照地理分支进行分组,即将地理距离较近的3个群体QD、RS、RZ组成一组,将ZS群体单独分组,结果显示大多数变异来自群体内(71.55%),组群间的分化较大但不显著(ΦST=0.285,P>0.05)。
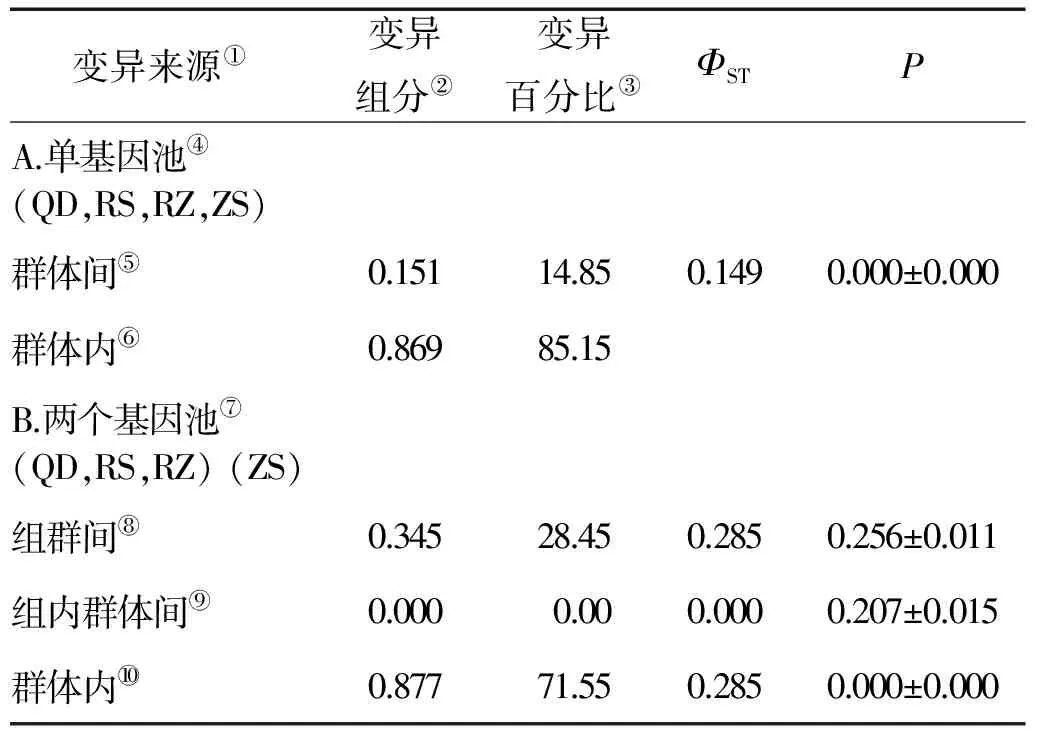
表3 金乌贼群体的AMOVA分析
Note:①Source of variation;②Variance components;③Percentage variation;④One gene pool;⑤Among populations;⑥Within populations;⑦Two gene pools;⑧Among groups;⑨Among populations within groups;⑩Within populations
3 讨论
遗传多样性是物种分化和生命进化的基础,是物种多样性的重要组成部分,通常情况下物种的遗传多样性水平代表着其对变化环境的适应能力,它不仅是生物多样性的内在表现形式,还是物种保持进化潜能的基本条件,决定着生物多样性的形成、发展或多样性的降低[33]。
群体中单倍型多样度(h)是评价该群体遗传分化和多态程度的重要指标之一,h值越大,群体遗传多样性越高。本研究4个群体中仅舟山群体单倍型多样度较高(0.960),青岛、乳山及日照群体单倍型多样度均较低(0.478~0.536)。Zheng[19]使用微卫星标记对2004年采自日照的金乌贼群体进行遗传学研究,结果显示日照群体处于较高的遗传多样性,与我们的研究结果相悖,这可能与大规模的金乌贼增殖放流相关。通常,用于繁育增殖放流苗种的亲体数量有限,人工繁育苗种的遗传多样性较野生群体低,被投入自然海域的放流苗种通过与野生群体之间的基因交流从而降低野生群体的遗传多样性。近年来,大量人工繁育的金乌贼苗种被投入山东沿岸海域,自然海域的野生金乌贼群体因此受到影响。相反,至今尚未有关于浙江省沿岸海域金乌贼增殖放流的相关报道,金乌贼资源的补充依靠自然群体,种群相对稳定,其单倍型多样度和核苷酸多样度处于较高水平。因此,在以后的金乌贼增殖放流工作中,应当注意繁育过程中亲本数量。
通过构建以虎斑乌贼为外群的单倍型系统发育树可以看出,金乌贼各群体间互相混杂,没有显示出明显的谱系结构,系统发育树并未出现与采样地点相对应的分支。同样,单倍型最小跨度树中也未检测到明显的谱系结构。
生物种群遗传分化主要决定于种群内的遗传漂变与种群间的基因流的动态平衡。Wright[34]提出的遗传分化理论认为:当遗传分化指数(FST)的值介于0~0.05之间表示种群间遗传分化程度低;0.05~0.15为分化程度中等;0.15~0.25为分化程度高;大于0.25表明种群遗传分化程度极大。本研究各群体间遗传分化指数FST值显示,舟山群体与山东沿岸的3个群体之间遗传分化较大,而山东沿岸的3个群体之间遗传分化较小。同时,确切P检验的结果也显示,舟山群体与其它3个群体间不存在随机交配现象,说明舟山群体与山东沿岸金乌贼群体之间基因流动较弱。这可能与金乌贼的生活习性有关,金乌贼为一年生头足类,喜集群,卵为沉性,游泳能力较弱,仅每年8—11月幼体自沿岸产卵场向越冬场进行越冬洄游,次年4—5月达到性成熟的成体自越冬场向产卵场做生殖洄游。有学者报道东海、黄海金乌贼的越冬场大致可分为:对马岛西南海域、黄海中部海域和台湾北部海域[8]。舟山群体与其他群体的在地理位置上相距较远,同时舟山群体的越冬场可能不用于其他3个群体,以至于舟山群体无论在产卵期还是越冬期都与其他3个群体没有交流,导致舟山群体与其他群体遗传分化较大。而青岛、乳山和日照地理位置较近,群体间交流频繁,此外,由于近年来山东省大规模开展金乌贼增殖放流活动,各地之间金乌贼苗种的交易导致山东沿岸海域的金乌贼群体相互混杂,山东沿岸的3个群体遗传分化较小。
4 结语
山东沿岸的金乌贼资源经过增殖放流的养护与补充,资源量得到一定程度的恢复,但种群的遗传多样性显著降低,这将会降低野生金乌贼的环境适应能力以及抵御病害的能力。因此,在金乌贼资源养护工作中,应当注意资源增殖对野生种群的遗传学影响,通过增加繁殖亲本数量以提高放流苗种的遗传多样性。
[1] Natsukari Y, Tashiro M.Neritic squid resources and cuttlefish resources in Japan [J]. Marine Behaviour and Physiology, 1991, 18: 149-226.
[2] 齐钟彦, 马继同, 王祯瑞, 等. 黄渤海的软体动物[M]. 北京: 农业出版社, 1989: 257-258. Qi ZY, Ma J Z, Wang Z R, et al. Mollusca of Huanghai and Bohai Seas [M]. Beijing: Agriculture Press, 1991, 18: 149-226.
[3] 郝振林. 金乌贼的行为习性及标志技术的研究 [D]. 青岛: 中国海洋大学, 2007. Hao Z L. Studies on the Behavior and Marking Technology ofSepiaesculenta[D]. Qingdao: Ocean University of China, 2007.
[4] 郝振林, 张秀梅, 张沛东. 金乌贼的生物学特性及增殖技术 [J]. 生态学杂志, 2007, 26(4): 601-606. Hao Z L, Zhang X M, Zhang P D. Biological characteristics and multiplication techniques ofSepiaesculenta[J]. Chinese Journal of Ecology, 2007, 26: 601-606.
[5] 严利平, 李建生. 东海区经济乌贼类资源量评估[J]. 海洋渔业, 2004, 26: 189-192. Yan L P, Li J S. Assessment of commercial cuttlefish biomass in the East China Sea [J]. Marine Fisheries, 2004, 26: 189-192.
[6] 日照市水产研究所. 金乌贼全人工育苗在我国首获成功[J]. 现代渔业信息, 2010, 25: 37. Rizhao Fisheries Research Institute. The first successful artificial breeding ofSepiaesculentain China [J]. Journal of Modern Fisheries Information, 2010, 25: 37.
[7] 山东省海洋与渔业厅. 2016年我省放流金乌贼125万头[EB/OL]. (2016-07-19) [2016-09-23]. http: //www. hssd. gov. cn/pub/sdhyyyt/xwzx/stdt/201607/t20160719_827000. html. Shandong Provincial Oceanic and Fishery. Our province has released 1250 thousands golden cuttlefish in 2016 [EB/OL]. (2016-07-19) [2016-09-23]. http: //www. hssd. gov. cn/pub/sdhyyyt/xwzx/stdt/201607/t20160719_827000. html.
[8] 山东省海洋与渔业厅. 我省增殖放流资源回捕生产旺季来临[EB/OL]. (2007-10-26)[2016-09-23]. http: //www. hssd. gov. cn/pub/sdhyyyt/xwzx/stdt/201412/t20141219_35852. html. Shandong Provincial Oceanic and Fishery. Recapturing season of our province’s stock enhancement is coming[EB/OL]. (2007-10-26) [2016-09-23]. http: //www. hssd. gov. cn/pub/sdhyyyt/xwzx/stdt/201412/t20141219_35852. html.
[9] 山东省海洋与渔业厅. 我省海洋资源增殖效果显著[EB/OL]. (2012-12-05) [2016-09-23]. http: //www. hssd. gov. cn/pub/sdhyyyt/xwzx/stdt/201412/t20141219_39409. html. Shandong Provincial Oceanic and Fishery. The marine stock enhancement of our province has been very effective [EB/OL]. (2012-12-05) [2016-09-23]. http: //www. hssd. gov. cn/pub/sdhyyyt/xwzx/stdt/201412/t20141219_39409. html.
[10] Schramm Jr H L, Piper R G. Uses and effects of cultured fishes in aquatic ecosystems [J]. American Fisheries Society Symposium, 1995, 15: 17-42.
[11] Escalante M A, García-De-León F J, Dillman C B, et al. Genetic introgression of cultured rainbow trout in the Mexican native trout complex [J]. Conservation Genetics, 2014, 15: 1063-1071.
[12] Norris A T, Bradley D G, Cunningham E P. Microsatellite genetic variation between and within farmed and wild Atlantic salmon (Salmosalar) populations [J]. Aquaculture, 1999, 180: 247-264.
[13] Iguchi K, Watanabe K, Nishida M. Reduced mitochondrial DNA variation in hatchery populations of ayu (Plecoglossusaltivelis) cultured for multiple generations [J]. Aquaculture, 1999, 178: 235-243.
[14] O'Donnell T P, Denson M R, Darden T L. Genetic population structure of spotted seatroutCynoscionnebulosusalong the south‐eastern USA [J]. Journal of Fish Biology, 2014, 85: 374-393.
[15] Watanuki N, Kaneuchi S, Fujita T, et al. Entry of immature cuttlefishSepiaesculentainto basket traps [J]. Fisheries Science, 1998, 64: 707-710.
[16] Wada T, Takegaki T, Mori T, et al. Sperm displacement behavior of the cuttlefishSepiaesculenta(Cephalopoda: Sepiidae) [J]. Journal of Ethology, 2005, 23: 85-92. [17] 陈四清, 刘长琳, 庄志猛, 等. 饵料、盐度对金乌贼幼体生长的影响[J]. 渔业现代化, 2008, (06): 23-25. Chen S Q, Liu C L, Zhuang Z M, et al. Effect of feeds and water salinity on growth of golden sepia (Sepiaesculenta) [J]. Fishery Modernization, 2008, (06): 23-25.
[18] 雷舒涵, 吴常文, 高天翔, 等. 金乌贼和曼氏无针乌贼胚胎发育及其盐度耐受能力的比较研究[J]. 中国水产科学, 2011, 18: 350-359. Lei S H, Wu C W, Gao T X et al. A comparative study ofSepiaesculentaandSepiellamaindronion embryonic development and ability of salinity tolerance [J]. Journal of Fishery Sciences of China, 2011, 18: 350-359.
[19] Zheng X, Ikeda M, Kong L, et al. Genetic diversity and population structure of the golden cuttlefish,Sepiaesculenta(Cephalopoda: Sepiidae) indicated by microsatellite DNA variations [J]. Marine Ecology, 2009, 30: 448-454.
[20] 刘云国, 刘贤德, 高焕, 等. 水产生物DNA分子标记技术[M]. 北京: 科学出版社, 2009: 59-62. Liu Y G, Liu X D, Gao H, et al. Aquatic DNA Molecular Marker Technology [M]. Beijing: Science Press, 2009: 59-62.
[21] 常抗美, 李焕, 吕振明, 等. 中国沿海7个长蛸(Octopusvariabilis)群体COI基因的遗传变异研究[J]. 海洋与湖沼, 2010, (03): 307-314. Chang K M, Li H, Lv Z M et al. Genetic variation in different populations ofOctopusvariabilisin China coastal waters based on the COI gene analysis [J]. Oceanologia et Limnologia Sinica, 2010, (03): 307-314.
[22] 吕振明, 李焕, 吴常文, 等. 中国沿海六个地理群体短蛸的遗传变异研究[J]. 海洋学报: 中文版, 2010, 32: 130-138. Lv Z M, Li H, Wu C W et al. Genetic variation ofOctopusocellatuspopulations in China’s coastal waters based on the COI gene analysis [J]. Acta Oceanologica Sinica, 2010, 32: 130-138.
[23] 刘连为, 许强华, 陈新军. 基于线粒体COI和Cytb基因序列的北太平洋柔鱼种群遗传结构研究[J]. 水产学报, 2012, 36: 1675-1684. Liu L W, Xu Z Q, Chen X J. Population genetic structure ofOmmastrephesbartramiiin the North Pacific Ocean based on the COI and Cytb gene sequences analysis [J]. Journal of Fisheries of China, 2012, 36: 1675-1684.
[24] Miya M, Nishida M. Organization of the mitochondrial genome of a deep-sea fish,Gonostomagracile(Teleostei: Stomiiformes): first example of transfer RNA gene rearrangements in bony fishes[J]. Marine Biotechnology, 1999, 1: 416-426.
[25] Jeanmougin F, Thompson J D, Gouy M, et al. Multiple sequence alignment with Clustal X [J]. Trends in Biochemical Sciences, 1998, 23: 403-405.
[26] Excoffier L, Lischer H E L. Arlequin suite ver 3. 5: a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10: 564-567.
[27] Nei M. Molecular Evolutionary Genetics [M]. New York, Columbia University Press, 1987.
[28] Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28: 2731-2739.
[29] Tamura K, Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees [J]. Molecular Biology and Evolution, 1993, 10: 512-526.
[30] Weir B S, Cockerham C C. EstimatingF-statistics for the analysis of population structure [J]. Evolution, 1984, 38: 1358-1370.
[31] Rice W R. Analyzing tables of statistical tests[J]. Evolution, 1989, 43: 223-225.
[32] Excoffier L, Smouse P E, Quattro J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data[J]. Genetics, 1992, 131: 479-491.
[33] 施立明. 遗传多样性及其保存[J]. 生物科学信息, 1990, 2: 158-164. Shi L M. Genetic diversity and its conservation [J]. Bioscience Communication, 1990, 2: 158-164.
[34] Wright S. Ecolution and the genetics of populations: Variability within and among natural populations [J]. Chicago: University of Chicago Press, 1978.
责任编辑 朱宝象
Genetic Variation of DifferentSepiaesculentaPopulations in China Coastal Waters Based on Mitochondrial COI Gene Analysis
SHAN Bin-Bin1, SONG Na1, LIU Shu-De2, TU Zhong2, WANG Xiao-Mei3, 4, GAO Tian-Xiang5, HAN Zhi-Qiang5, ZHANG Xiu-Mei1
(1. College of Fisheries, Ocean University of China, Qingdao 266003, China; 2. Shandong Hydrobios Resources Conservation and Management Center, Yantai 264003, China; 3. Resources and Eco-environmental Research Center, Chinese Academy of Fishery Sciences, Beijing 100141, China; 4. Scientific Observing and Experimental Station of Fishery Remote Sensing of Ministry of Agriculture, Chinese Academy of Fishery Sciences, Beijing 100141, China; 5. College of Fisheries, Zhejiang Ocean University, Zhoushan 316022, China)
Mitochondrial COI gene fragment ofSepiaesculentafrom Rushan, Qingdao, Rizhao and Zhoushan were sequenced. A total of 27 haplotypes were detected in 117 individuals. The haplotype diversity and nucleotide polymorphism indexesranged from 0.478 to 0.956 and from 0.001 to 0.006, respectively. The results indicated thatS.esculentafrom Zhoushan had a higher genetic diversity than other populations. The genetic differentiation index (Fst) showed that genetic differentiation among Rushan, Qingdao and Zhoushan populations ranged from -0.007 to 0.039 while the genetic differentiation between Zhoushan population and others were relatively larger, ranging from 0.186 to 0.292. The results of AMOVA also revealed that there were two groups, populations along the coast of Shandong Province (Rushan, Qingdao and Rizhao) and Zhoushan. The results clarified the loss of genetic diversity of wildS.esculentapopulations along the coast of Shandong Province. The long-term extensive hatchery releasing program might have affected the genetic diversity of wildS.esculentapopulations.
Sepiaesculenta; COI gene; genetic diversity; genetic structure; stock enhancement
农业部渔业遥感科学观测试验站开放课题项目(OFSOESFRS201506);国家自然科学基金项目(41676153)资助 Supported by Scientific observing and experimental Station of fishery remote sensing, Ministry of Agriculture (OFSOESFRS201506);National Natural Science Foundation of China (41676153)
2016-08-01;
2016-11-10
单斌斌(1993-),男,博士生,研究方向:渔业资源增殖学。E-mail:shanbinbin@yeah.net
❋❋ 通讯作者:E-mail:xiumei1227@163.com
S931.5
A
1672-5174(2017)05-050-07
10.16441/j.cnki.hdxb.20160277
单斌斌,宋娜,刘淑德,等.基于线粒体COI基因序列的金乌贼群体遗传学研究[J].中国海洋大学学报(自然科学版),2017, 47(5): 50-56.
SHAN Bin-Bin, Song Na, LIU Shu-De, et al.Genetic variation of differentSepiaesculentapopulations in China coastal waters based on mitochondrial COI gene analysis[J].Periodical of Ocean University of China, 2017, 47(5): 50-56.
