氮沉降对温带森林土壤甲烷氧化菌的影响
2017-03-09张丹丹莫柳张丽梅徐星凯
张丹丹, 莫柳张丽梅,徐星凯
1 中国科学院生态环境研究中心城市与区域生态国家重点实验室, 北京 100085 2 中国科学院大学, 北京 100049 3 中国科学院大气物理研究所大气边界层物理和大气化学国家重点实验室, 北京 100029 4 华中农业大学资源与环境学院, 武汉 430070
甲烷(CH4)是仅次于水蒸气和二氧化碳的第三大温室气体,对全球气候变暖的贡献率接近20%(IPCC,2007)。由于人类活动的加剧,大气中的CH4浓度已大幅增长,造成大气CH4浓度的升高的原因不仅是其源增加的结果,也与其汇减少有关[1]。水分不饱和的土壤作为大气CH4重要的汇,每年可以从大气中吸收(氧化)大约20—60Tg的CH4[2-3],其微小变化都可能影响全球的CH4平衡[4]。森林土壤更被视为其中最为有效的汇[5-6],但由于环境因素变化如降水、温度、pH、树种、氮沉降以及森林管理方式等的影响,森林土壤氧化大气CH4的能力具有很高的变异性,因此,深入研究环境因素影响森林土壤CH4氧化的驱动机制对全面评估和调控森林土壤甲烷氧化能力具有重要意义[7- 13]。
近一个半世纪以来,由于化石燃料燃烧和农业化肥的大量投入等向大气中排放的含氮化合物激增,导致大气的氮沉降大幅增加[14]。森林生态系统是氮沉降大面积的直接承受者[15],据调查,温带森林的氮沉降量已高于15kg N hm-2a-1,有些地区甚至高于50kg N hm-2a-1[16],氮沉降因而成为森林生态系统备受关注的环境因子,并针对氮沉降对森林土壤甲烷氧化的影响开展了大量工作。但目前多数研究主要针对CH4气体吸收通量或吸收动力学这些表观特征来揭示氮沉降对森林土壤甲烷氧化的影响,对于这一过程中起关键作用的土壤微生物的作用机制研究相对较少,国内在这方面的研究更是鲜有报道。参与森林土壤甲烷氧化的微生物主要是甲烷氧化菌(Methanotroph)。甲烷氧化菌是一类以CH4为唯一碳源和能源的细菌,其通过甲烷单加氧酶(Methane monooxygenase,MMO)将CH4首先氧化为甲醇并启动系列酶将CH4最终代谢成二氧化碳和水[17]。根据生理生化特征以及对CH4代谢途径的差异,可将甲烷氧化菌分为type I和type II两种类型,其分别属于γ-变形菌纲(γ-Proteobacteria)和α-变形菌纲(α-Proteobacteria)[18]。有关农业、森林以及湿地土壤等中甲烷氧化菌数量和群落组成已有较多的研究报道,并发现土壤甲烷氧化菌的数量、群落组成及其活性会受土壤pH、湿度、温度、大气CH4浓度、植被类型以及施肥等环境因素的影响[19- 25]。但关于森林土壤甲烷氧化菌应对氮沉降增加的响应机制的研究相对较少,国内更是缺乏相关报道。国外已有的几篇报道也主要是通过室内的短期施氮培养来研究氮素增加对森林土壤甲烷氧化菌群落组成的影响[26-27],而关于野外长期氮沉降增加对森林土壤甲烷氧化菌数量和群落组成的影响仍有待进一步研究。
长白山阔叶红松混交林是位于我国东北部重要的温带森林生态系统。本研究基于长白山温带森林长期氮沉降模拟实验平台,采用实时荧光定量PCR和克隆测序以及土壤属性测定相结合方法,研究了长期施加不同形态氮肥后,森林土壤甲烷氧化菌的数量和群落组成随季节变化的特征及其与土壤基质属性的关系,以期揭示长期氮沉降增加影响温带森林土壤甲烷氧化的微生物机制。
1 材料和方法
1.1 土壤采集和理化性质的测定
本研究土壤样品采自长白山阔叶红松混交林长期氮沉降模拟实验平台,该控制实验始于2006年,位于长白山北坡底部(42°24′N,127°06′E),该地区属于典型的受季风影响的温带大陆性山地气候,水热同期,夏季短暂多雨,冬季寒冷漫长,年平均温度为2.5—5.5℃,年降水量600—900mm[28-29]。土壤类型为山地暗棕壤,冷凉淋溶土,以火山灰为成土母质,经有机质积累和弱酸性淋溶过程形成[30],主要植被有红松(Pinuskoraiensis)、椴树、蒙古栎和水曲柳等。土壤样品采样时选择野外控制实验中长期施加高剂量(45kg N hm-2a-1)的3种不同形态氮素((NH4)2SO4、NH4Cl和KNO3)及未施用氮素的处理(对照)为研究对象,每个处理均设有4个重复小区;有关森林剖面土壤属性和野外施氮控制实验参见[31-32]。根据长期积累数据规律以及当地的气候特征分别在夏季雨热同期(8月)和秋季干旱期(10月)两个典型时期采集土壤样品:每个小区利用不绣钢土钻随机选取3点采集0—10cm深度的土壤,充分混匀后置于冰盒带回实验室处理。将采集的土壤样品分成两份,一份随即以液氮速冻保存于-80℃冰箱,用于分子生物学分析;另一份中存放于4℃用于pH值、铵态氮、硝态氮和可溶性有机碳测定。
土壤pH 值的测定采用去离子水以水土比2.5∶1(w/w)浸提,并用Delta 320 pH仪测定;土壤铵态氮和硝态氮采用1mol/L KCl溶液,以水土比5∶1(w/w)浸提后,采用连续流动分析仪(SAN++, SKALAR, the Netherlands)测定;土壤可溶性有机碳采用 0.5mol/L K2SO4溶液,以水土比为5∶1浸提后,通过总有机碳分析仪(Shimadzu TOC-Vcsh/TN, Kyoto, Japan)测定[33]。
1.2 土壤DNA和定量PCR分析
土壤总DNA提取采用MoBio UltracleanTMSoil DNA 试剂盒(San Diego, CA)进行,所有操作依照产品说明书进行,细胞破碎在FastPrep上进行,破碎强度为5.0m/s,时间为45s。所获得的DNA样品经10倍稀释后直接用于下游实验。
以甲烷氧化菌的pmoA基因为靶基因,以pmoA基因的特异引物A189(5′-GGNGACTGGGACTTCTGG和mb661(5′-CCGGMGCAACGTCYTTACC)[34]利用实时荧光定量 PCR 对不同处理土壤中甲烷氧化菌的丰度进行定量分析。定量 PCR的扩增反应使用 SYBR® Premix Ex TaqTM试剂盒(TaKaRa),25μL定量PCR反应体系中包括2×SYBR Premix Ex TaqTM12.5μL,正反向引物(浓度为10μmol/L)各0.5μL,DNA模板2μL,其余用ddH2O补足至25μL。Real-time PCR反应在ICycler IQ 5 thermocycler (Bio-Rad Laboratories, Hercules, CA, USA)上运行,其扩增程序如下:94℃预变性5min;94℃变性45s,63℃退火45s,72℃延伸1min,之后每个循环退火温度降低1℃,共5个循环; 94℃变性45s,57℃退火45s,72℃延伸1min,共30个循环;在83℃收集反应荧光信号,数据分析采用iCycler软件[24]。
1.3 甲烷氧化菌的群落组成分析
甲烷氧化菌的群落组成通过对pmoA基因进行克隆测序分析,pmoA基因普通PCR扩增引物和扩增程序同1.2中的real-time PCR。PCR产物经过Wizard SV Gel & PCR Clean-Up System(Promega, USA)试剂盒切胶纯化后,连接到pGEM-T Easy Vector上(Promega, Madison, WI, USA),再通过热击法将载体转化入大肠杆菌JM109感受态细胞(TaKaRa, Japan),取转化液涂布到含有氨苄青霉素(Ampicillin)/IPTG/X-Gal的LB (Luria-Bertani)培养基上,37℃下培养16—18h。随机选取若干白色克隆子,采用菌体扩增方式,用pGEM-T Easy Vector通用引物T7/SP6扩增外源插入片段。通过1%浓度的琼脂糖凝胶电泳法检验含有插入片段的阳性克隆子。
每个克隆文库随机挑选30个阳性克隆子进行测序。所获得的目的基因序列通过ClustalX2,mouthur进行序列分析,按照93%同源划分可操作分类单元(Operational Taxonomic Units, OTUs)[35],并把正确长度的序列提交NCBI数据库进行Blast序列对比,下载最相似菌种序列作为系统发育树的参考序列。通过Clustal W和Mega 5.0建立Neighbor-Joining系统发育树,系统发育树各分支置信度由自举分析方法(Bootstrap)检验,重复1000次。
1.4 数据处理
本研究中的统计分析在SPSS 19.0软件中进行,其中多组数据间的差异分析采用单因素方差分析与多重比较分析(Duncan,P<0.05);相关分析采用Spearman相关分析;克隆文库数据的分析采用Redundancy analysis(RDA)分析样品间群落组成与不同形态氮沉降增加下土壤基质属性的关系,在CANOCO 4.5软件中进行。
2 结果
2.1 土壤理化性质
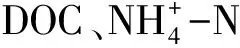

表1 土壤基本理化属性(平均数±标准误差)
平均数±标准误差;同列相同小写字母a分别表明夏季和秋季土壤性质在不同处理之间无显著性差异,同列大写字母A, B表明不同处理之间土壤性质的显著差异性(P<0.05)
2.2 甲烷氧化菌的数量及其与土壤基质特性之间的关系

图1 夏季和秋季不同施肥处理间土壤甲烷氧化菌pmoA 基因丰度Fig.1 The abundance of soil methanotrophic pmoA gene under different nitrogenous compound amendments in Summer (August) and Fall (October)小写字母a:pmoA基因丰度在夏季(8月)雨热同期时期各处理之间差异无显著性;大写字母A, B:pmoA基因丰度在秋季(10月)干旱期各处理之间的有差异显著性(P < 0.05)

2.3 甲烷氧化菌的群落组成

图2 夏季和秋季施氮处理样地甲烷氧化菌群落组成的变化Fig.2 The community composition of methanotroph under different nitrogenous compound amendments in Summer (August) and Fall (October)CK:对照组,未施氮处理;HS:施加硫酸铵,(NH4)2SO4处理;HCl:施加氯化铵,NH4Cl处理;HK:施加硝酸钾,KNO3处理
通过克隆测序技术,对甲烷氧化菌的群落组成进行了分析。对两个季节所采集的样品一共构建了32个pmoA基因克隆文库,每个克隆文库随机挑取约30个克隆子进行测序,共获得742条有效序列。在93%的序列相似水平上,甲烷氧化菌pmoA基因序列一共被划分为112个OTU,在系统发育树上形成2个主要分支,分别对应Type I型(磷酸核酮糖氧化途径)和Type Ⅱ型(丝氨酸氧化途径)甲烷氧化菌,如图3所示。其中,属于Type I型的序列可进一步形成4个分支,参考Kolb等人[36]的分类经验,研究将这4个分支命名为:Methylobacter-group (Type I)、Methylococcus-group (Type I)、Methylohalobius-group (Type I)和upland soil cluster γ(Type I)。其中,upland soil cluster γ(Type I) 序列主要来自于旱地土壤和森林土壤[19,36],但本研究的供试样品只检测到Methylobacter-group (Type I)和Methylococcus-group (Type I),未检测到upland soil cluster γ(Type I)和Methylohalobius-group (Type I)的序列,可能由引物的偏差所致(图3)。属于Type II的序列也形成两个小分支,即Methylosinus-group (Type II)和Methylocapsaspp. (Type II)(图3),于供试土壤样品中均被检测到。


图3 甲烷氧化菌pomA 基因系统发育树Fig.3 Phylogenetic tree of methanotroph pomA gene sequences CK8(10):8(10)月份对照,CK土壤样品;HS8(10):8(10)月份施加硫酸铵,(NH4)2SO4处理土壤样品;HCl8(10):8(10)月份施加氯化铵,NH4Cl处理土壤样品;HK8(10):8(10)月份施加硝酸钾,KNO3处理土壤样品;分号后的数字:OTU的序号;括号里的数字:该处理代表OTU的序列条数
对pmoA基因为代表的甲烷氧化菌微生物群落组成和土壤基本属性进行冗余分析(Redundancy analysis, RDA),结果显示甲烷氧化菌群落组成与盐浸提有机碳/矿质氮比值呈显著正相关关系(r=0.49,P<0.05),与土壤湿度和pH值无相关性(图4)。此外,RDA分析结果显示夏季(8月)样品和秋季(10月)样品被RDA Axis 1轴分开,表明采样时间即季节变化对甲烷氧化菌的群落组成有显著影响(PerMANOVA,P< 0.1, 图4)。
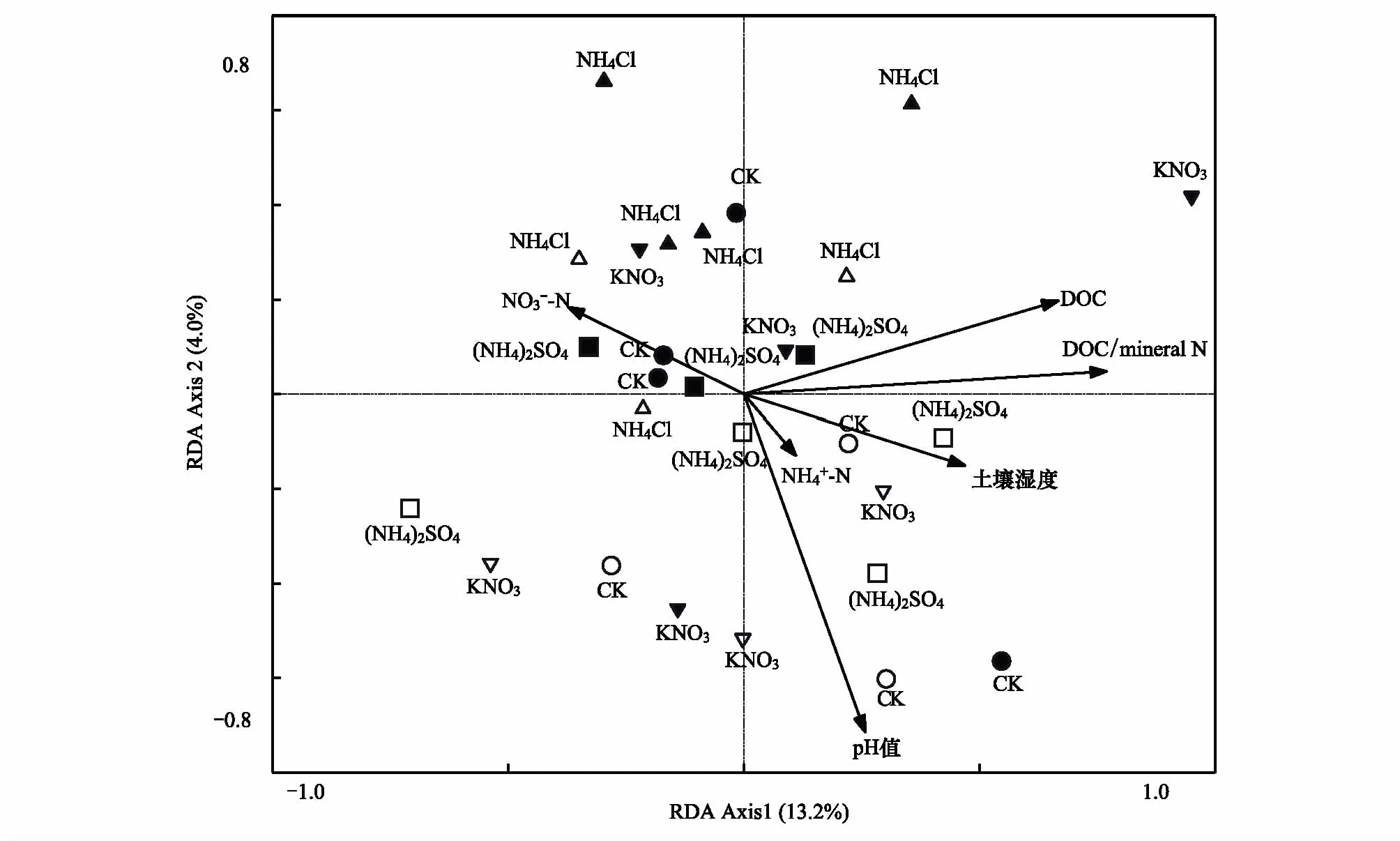
图4 甲烷氧化菌群落组成与环境因子的RDA分析Fig.4 Redundancy analysis (RDA) between methanotroph community and environmental variables 空心图标:8月份土壤样品;实心图标:10月份土壤样品;○:8月份对照,CK土壤样品;□:8月份施加硫酸铵,(NH4)2SO4处理土壤样品;△:8月份施加氯化铵,NH4Cl处理土壤样品;▽:8月份施加硝酸钾,KNO3处理土壤样品;●:10月份对照,CK样品;■:10月份施加硫酸铵,(NH4)2SO4处理土壤样品;▲:10月份施加氯化铵,NH4Cl处理土壤样品;▼:10月份施加硝酸钾,KNO3处理土壤样品
3 讨论
3.1 甲烷氧化菌的数量对不同形态氮沉降增加的响应

3.2 甲烷氧化菌的组成对不同形态氮沉降增加的响应
根据甲烷氧化菌的生理生化特征以及对CH4代谢途径和亲和力的差异,可将甲烷氧化菌分为Type I型和Type II型两种类型,分别属于γ-Preoteobacteria和α-Preoteobacteria纲;Type I型甲烷氧化菌通过磷酸核酮糖途径氧化CH4,对CH4亲和力低,但具有较强的繁殖能力,属于r-策略者[44],其常见类群包括:甲基单胞菌属(Methylomonas)、甲基球菌属(Methylococcs)等;而Type Ⅱ型利用丝氨酸途径氧化CH4,对CH4的亲和力高,属于k-策略者,包括甲基弯曲菌(Methylosinus)和甲基孢囊菌属(Methylocystis)等;此外,人们将类似于Type I型荚膜甲基球菌(Methylococcuscapsulatus)的甲烷氧化菌定义为X型,其代谢途径与Type I型相同[17-18,45]。本研究中检测到的pmoA基因序列分为Type I型和Type Ⅱ型两类甲烷氧化菌,主要以Type I型甲烷氧化菌为主,其相对比例大于70%(图2),造成这种现象的原因可能是:Type I型甲烷氧化菌在低CH4高氧气环境中为优势种群,而Type Ⅱ型甲烷氧化菌则在高CH4低氧气环境中起主导作用[46]。此外,已有的研究发现Type I型甲烷氧化菌对土壤环境要求较苛刻,而Type Ⅱ型甲烷氧化菌有较强的生存能力,因此施氮过程会较大程度影响Type I型甲烷氧化菌的活性和群落组成,而对Type Ⅱ型甲烷氧化菌的群落组成无显著影响[2,47]。
3.3 季节变化对甲烷氧化菌响应氮素输入的影响
RDA分析结果表明森林土壤盐浸提有机碳/矿质氮比值与甲烷氧化菌群落组成呈显著相关性,再次印证不同施氮处理是导致甲烷氧化菌群落组成差异的主要因素。此外,RDA结果显示夏季(8月)样品和秋季(10月)样品被RDA Axis 1轴分开,表明甲烷氧化菌的群落组成在不同季节有明显变化,与该样地中甲烷通量随季节变化的趋势和之前有关季节和温度变化显著影响甲烷氧化菌的群落组成和甲烷的产生与氧化能力的研究报道一致[8,20,32]。但本研究中施氮处理对甲烷氧化菌的丰度和群落组成的影响在夏季不显著,而在秋季均表现出明显影响。长白山阔叶红松混交林地属于温带大陆季风型气候,夏季平均温度保持在25℃左右,而秋季平均温度在5—15℃范围内[32]。虽然嗜热性甲烷氧化菌相继被发现[48-49],但多数甲烷氧化菌生长的适宜温度在20—30℃之间。长白山地区夏季较适宜的温度更有利于甲烷氧化菌的生长和活性,该影响可能超过施氮本身的影响,从而使得其对长期施氮的响应不显著。此外,也有不少研究表明,氮肥施用可通过促进植物生长促进根系分泌更多碳源供给甲烷氧化菌从而促进甲烷氧化能力[50-51];夏季植物生长旺盛对氮肥的吸收利用较快,从而减弱了氮肥对土壤中甲烷氧化菌的抑制作用。
4 结论
本研究对长期施加不同形态氮素模拟沉降增加条件下温带森林土壤中甲烷氧化菌的数量和群落组成的季节性演变特征及其与土壤基质属性的耦合关系进行研究,结果表明,夏季不同施氮处理对甲烷氧化菌pmoA基因丰度无显著性差异,而秋季施加NH4Cl和(NH4)2SO4处理降低甲烷氧化菌的丰度。无论夏季和秋季,施加不同形态氮的土壤中甲烷氧化菌均以适应低CH4但有氧环境的Type I型为主(相对丰度在70.6%—85.4%之间),作为I型甲烷氧化菌的优势类群,Methylobacter-group (Type I)占Type I型的55.1%—91.7%,在夏季不同形态氮处理间无显著差异,但在秋季施加(NH4)2SO4和NH4Cl处理中显著低于对照处理。长白山地区夏季较适宜的温度更有利于甲烷氧化菌的生长,以及夏季植物生长旺盛为甲烷氧化菌提供更多碳源,从而使得施氮对甲烷氧化菌的影响不如秋季显著。这些结果解释了以往观测到的施铵态氮肥显著降低秋季林地甲烷净吸收通量,而在夏季无显著影响的观测结果,一定程度上揭示了氮沉降影响森林土壤甲烷氧化季节性差异的潜在的微生物机制。
致谢:中国科学院大气物理研究所王迎红博士、吴浩浩同学帮助样品采集和土壤性质分析测定,中国科学院微生物研究所郑勇博士和中国科学院生态环境研究中心王雅琪同学帮助微生物分析,特此致谢。
[1] Hütsch B W, Webster C P, Powlson D S. Methane oxidation in soil as affected by land use, soil pH and N fertilization. Soil Biology and Biochemistry, 1994, 26(12): 1613- 1622.
[2] Henckel T, Jäckel U, Schnell S, Conrad R. Molecular analyses of novel methanotrophic communities in forest soil that oxidize atmospheric methane. Applied and Environmental Microbiology, 2000, 66(5): 1801- 1808.
[3] Lau E, Ahmad A, Steudler P A, Cavanaugh C M. Molecular characterization of methanotrophic communities in forest soils that consume atmospheric methane. FEMS Microbiology Ecology, 2007, 60(3): 490- 500.
[4] Bradford M A, Ineson P, Wookey P A, Lappin-Scott H M. Role of CH4oxidation, production and transport in forest soil CH4flux. Soil Biology and Biochemistry, 2001, 33(12/13): 1625- 1631.
[5] Mer J L, Roger P. Production, oxidation, emission and consumption of methane by soils: A review. European Journal of Soil Biology, 2001, 37(1): 25- 50.
[6] Kolb S. The quest for atmospheric methane oxidizers in forest soils. Environmental Microbiology Reports, 2009, 1(5): 336- 346.
[7] Peichl M, Altaf A M, Ullah S, Moore T R. Carbon dioxide, methane, and nitrous oxide exchanges in an age-sequence of temperate pine forests. Global Change Biology, 2010, 16(8): 2198- 2212.
[8] Mohanty S R, Bodelier P L E, Conrad R. Effect of temperature on composition of the methanotrophic community in rice field and forest soil. FEMS Microbiology Ecology, 2007, 62(1): 24- 31.
[9] Saari A, Rinnan R, Martikainen P J. Methane oxidation in boreal forest soils: kinetics and sensitivity to pH and ammonium. Soil Biology and Biochemistry, 2004, 36(7): 1037- 1046.
[10] Menyailo O V, Abraham W R, Conrad R. Tree species affect atmospheric CH4oxidation without altering community composition of soil methanotrophs. Soil Biology and Biochemistry, 2010, 42(1): 101- 107.
[11] Zhang W, Mo J M, Zhou G Y, Gundersen P, Fang Y T, Lu X K, Zhang T, Dong S F. Methane uptake responses to nitrogen deposition in three tropical forests in southern China. Journal of Geophysical Research: Atmospheres, 2008, 113(D11): D11116.
[12] Wu X, Brüggemann N, Gasche R, Papen H, Willibald G, Butterbach-Bahl K. Long-term effects of clear-cutting and selective cutting on soil methane fluxes in a temperate spruce forest in southern Germany. Environmental Pollution, 2011, 159(10): 2467- 2475.
[13] Dutaur L, Verchot L V. A global inventory of the soil CH4sink. Global Biogeochemical Cycles, 2007, 21(4): GB4013.
[14] Galloway J N, Townsend A R, Erisman J W, Bekunda M, Cai Z, Freney J R, Martinelli L A, Seitzinger S P, Sutton M A. Transformation of the nitrogen cycle: recent trends, questions, and potential solutions. Science, 2008, 320(5878): 889- 892.
[15] Churkina G, Trusilova K, Vetter M, Dentener F. Contributions of nitrogen deposition and forest regrowth to terrestrial carbon uptake. Carbon Balance and Management, 2007, 2: 5.
[16] Dalal R C, Allen D E. Greenhouse gas fluxes from natural ecosystems. Australian Journal of Botany, 2008, 56(5): 396- 407.
[17] Hanson R S, Hanson T E. Methanotrophic bacteria. Microbiology and Molecular Biology Reviews, 1996, 60(2): 439- 471.
[18] Semrau J D, DiSpirito A, Yoon S. Methanotrophs and copper. FEMS Microbiology Reviews, 2010, 34(4): 496- 531.
[19] Knief C, Lipski A, Dunfield P F. Diversity and activity of methanotrophic bacteria in different upland soils. Applied and Environmental Microbiology, 2003, 69(11): 6703- 6714.
[20] 丁维新, 蔡祖聪. 土壤甲烷氧化菌及水分状况对其活性的影响. 中国生态农业学报, 2003, 11(1): 94- 97.
[21] Börjesson G, Sundh I, Svensson B. Microbial oxidation of CH4at different temperatures in landfill cover soils. FEMS Microbiology Ecology, 2004, 48(3): 305- 312.
[22] Henckel T, Roslev P, Conrad R. Effects of O2and CH4on presence and activity of the indigenous methanotrophic community in rice field soil. Environmental Microbiology, 2000, 2(6): 666- 679.
[23] Tsutsumi M, Kojima H, Uemura S, Ono K, Sumida A, Hara T, Fukui M. Structure and activity of soil-inhabiting methanotrophic communities in northern forest of Japan. Soil Biology and Biochemistry, 2009, 41(2): 403- 408.
[24] Zheng Y, Zhang L M, Zheng Y M, Di H J, He J Z. Abundance and community composition of methanotrophs in a Chinese paddy soil under long-term fertilization practices. Journal of Soils and Sediments, 2008, 8(6): 406- 414.
[25] Shrestha M, Shrestha P M, Frenzel P, Conrad R. Effect of nitrogen fertilization on methane oxidation, abundance, community structure, and gene expression of methanotrophs in the rice rhizosphere. The ISME Journal, 2010, 4(12): 1545- 1556.
[26] Mohanty S R, Bodelier P L E, Floris V, Conrad R. Differential effects of nitrogenous fertilizers on methane-consuming microbes in rice field and forest soils. Applied and Environmental Microbiology, 2006, 72(2): 1346- 1354.
[27] Jang I, Lee S, Zoh K D, Kang H. Methane concentrations and methanotrophic community structure influence the response of soil methane oxidation to nitrogen content in a temperate forest. Soil Biology and Biochemistry, 2011, 43(3): 620- 627.
[28] 郭忠玲, 郑金萍, 马元丹, 李庆康, 于贵瑞, 韩士杰, 范春楠, 刘万德. 长白山各植被带主要树种凋落物分解速率及模型模拟的试验研究. 生态学报, 2006, 26(4): 1037- 1046.
[29] 郝占庆, 张健, 李步杭, 叶吉, 王绪高, 姚晓琳. 长白山次生杨桦林样地: 物种组成与群落结构. 植物生态学报, 2008, 32(2): 251- 261.
[30] 耿世聪, 陈志杰, 张军辉, 娄鑫, 王秀秀, 戴冠华, 韩士杰, 于丹丹. 长白山三种主要林地土壤甲烷通量. 生态学杂志, 2013, 32(5): 1091- 1096.
[31] Xu X K, Han L, Wang Y S, Inubushi K. Influence of vegetation types and soil properties on microbial biomass carbon and metabolic quotients in temperate volcanic and tropical forest soils. Soil Science and Plant Nutrition, 2007, 53(4): 430- 440.
[32] Xu X K, Han L, Luo X B, Han S J. Synergistic effects of nitrogen amendments and ethylene on atmospheric methane uptake under a temperate old-growth forest. Advances in Atmospheric Sciences, 2011, 28(4): 843- 854.
[33] 鲁如坤. 土壤农业化学分析方法. 北京: 中国农业科技出版社, 2000.
[34] Costello A M, Lidstrom M E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments. Applied and Environmental Microbiology, 1999, 65(11): 5066- 5074.
[35] Zheng Y, Yang W, Sun X, Wang S P, Rui Y C, Luo C Y, Guo L D. Methanotrophic community structure and activity under warming and grazing of alpine meadow on the Tibetan Plateau. Applied Microbiology and Biotechnology, 2012, 93(5): 2193- 2203.
[36] Kolb S, Knief C, Dunfield P F, Conrad R. Abundance and activity of uncultured methanotrophic bacteria involved in the consumption of atmospheric methane in two forest soils. Environmental Microbiology, 2005, 7(8): 1150- 1161.
[37] Murrell J C, McDonald I R, Bourne D G. Molecular methods for the study of methanotroph ecology. FEMS Microbiology Ecology, 1998, 27(2): 103- 114.
[38] Adamsen A P S, King G M. Methane consumption in temperate and subarctic forest soils: rates, vertical zonation, and responses to water and nitrogen. Applied and Environmental Microbiology, 1993, 59(2): 485- 490.
[39] 胡荣桂. 氮肥对旱地土壤甲烷氧化能力的影响. 生态环境, 2004, 13(1): 74- 77.
[40] Dam B, Dam S, Kim Y, Liesack W. Ammonium induces differential expression of methane and nitrogen metabolism-related genes inMethylocystissp. strain SC2. Environmental Microbiology, 2014, 16(10): 3115- 3127.
[42] Schnell S, King G M. Mechanistic analysis of ammonium inhibition of atmospheric methane consumption in forest soils. Applied and Environmental Microbiology, 1994, 60(10): 3514- 3521.
[43] Hütsch B W. Methane oxidation in arable soil as inhibited by ammonium, nitrite, and organic manure with respect to soil pH. Biology and Fertility of Soils, 1998, 28(1): 27- 35.
[44] Steenbergh A K, Meima M M, Kamst M, Bodelier P L E. Biphasic kinetics of a methanotrophic community is a combination of growth and increased activity per cell. FEMS Microbiology Ecology, 2009, 71(1): 12- 22.
[45] 梁战备, 史奕, 岳进. 甲烷氧化菌研究进展. 生态学杂志, 2004, 23(5): 198- 205.
[46] Amaral J A, Knowles R. Growth of methanotrophs in methane and oxygen counter gradients. FEMS Microbiology Letters, 1995, 126(3): 215- 220.
[47] Krause S, Meima-Franke M, Hefting M M, Bodelier P L E. Spatial patterns of methanotrophic communities along a hydrological gradient in a riparian wetland. FEMS Microbiology Ecology, 2013, 86(1): 59- 70.
[48] Bodrossy L, Murrell J C, Dalton H, Kalman M, Puskas L G, Kovacs K L. Heat-tolerant methanotrophic bacteria from the hot water effluent of a natural gas field. Applied and Environmental Microbiology, 1995, 61(10): 3549- 3555.
[49] Dunfield P F, Yuryev A, Senin P, Smirnova A V, Stott M B, Hou S B, Ly B, Saw J H, Zhou Z M, Ren Y, Wang J M, Mountain B W, Crowe M A, Weatherby T M, Bodelier P L E, Liesack W, Feng L, Wang L, Alam M. Methane oxidation by an extremely acidophilic bacterium of the phylum Verrucomicrobia. Nature, 2007, 450(7171): 879- 882.
[50] Bodelier P L E, Roslev P, Henckel T, Frenzel P. Stimulation by ammonium-based fertilizers of methane oxidation in soil around rice roots. Nature, 2000, 403(6768): 421- 424.
[51] Schimel J. Rice, microbes and methane. Nature, 2000, 403(6768): 375- 377.
