基于荧光定量PCR和高通量测序技术的葡萄园土壤细菌群落结构多样性分析
2016-12-23王晓雯洪振瀚刘安瑞罗立新
王晓雯,洪振瀚,刘安瑞,罗立新
(华南理工大学,生物科学与工程学院,广东广州510006)
基于荧光定量PCR和高通量测序技术的葡萄园土壤细菌群落结构多样性分析
王晓雯,洪振瀚,刘安瑞,罗立新
(华南理工大学,生物科学与工程学院,广东广州510006)
土壤细菌在葡萄园生态系统中起着重要作用,具有十分重要的生态功能。以贵州镇远葡萄园基地根际、非根际土壤为研究对象,在对土壤的基本理化指标及酶活检测的基础上,采用荧光定量PCR(Q-PCR)和Illum ina M iseq高通量测序技术对葡萄园根际和非根际土壤的细菌菌群结构进行分析。结果表明,葡萄园根际土壤细菌16S rRNA基因的拷贝数在1.38×108~1.17×109copies/g之间,高于非根际土壤。IlluminaM iseq高通量测序显示,葡萄园根际土壤细菌呈多样性分布且优势菌群突出,主要涵盖了变形菌门(Protecbacteria),放线菌门(Actinbacteria),酸杆菌门(Acidobacteri),厚壁菌门(Firm icute),绿弯菌门(Chloroflexi),蓝藻门(Cyanobacteria),泉古菌门(Crenarchoeo),拟杆菌门(Bacteroidetes),芽单胞菌门(Gemmatimonadete),硝化螺旋菌门(Nitrospirae)。其中变形菌门和放线菌门为优势菌群,两者相对丰度之和占到了53.33%~67.28%。葡萄园根际土壤的主要优势细菌属包括乳球菌属(Lactococcus),节细菌属(Arthrobacter),Candidatus nitrososphaera,亚硝化螺菌属(Nitrososphaera),热单胞菌属(Thermomonas),Phycicoccus,泛菌属(Pantoea),芽孢杆菌属(Bacillus),埃希氏杆菌属(Escherichia),鞘脂单胞菌属(Sphingomonas),和溶杆菌属(Lysobacter)。α多样性分析表明,不同位点的根际土壤细菌多样性比较接近,而根际土壤细菌多样性低于非根际土壤。冗余分析(RDA)进一步分析表明,葡萄园土壤细菌群落多样性与土壤养分及酶活存在不同程度的相关关系,群落结构多样性受多种因素的影响。
葡萄园土壤; 根际土壤; 细菌多样性; 高通量测序; 荧光定量PCR
土壤根际是一个特殊的生态系统,它与周围非根际土壤在化学、生物学等特性上具有显著差异[1]。它易受植物根系的影响,是有益微生物及有害微生物与宿主植物发生复杂相互作用的一个微小区域,蕴含着丰富的微生物资源[2]。其中,细菌是根际土壤中数量最丰富、种类较多的微生物类群,广泛参与抑制病原体,氮循环,土壤矿化和养分吸收过程,它的含量和组成会对植株产生显著的影响[3-4]。有研究表明,有益的细菌类群能够促进植株的生长,果实的发育[3];有害的细菌类群则可能引起各种病虫害的发生[5]。近年来兴起的高通量测序方法为全面认识土壤微生物提供了新的契机,研究者们对根际微生物的研究和应用越来越深入[5-6]。
葡萄又称提子,多年生落叶藤本植物,褐色枝蔓细长,根系发达,可深达数米以下,在我国有大量的栽培,主要分布在贵州、甘肃、云南、宁夏、河北、新疆及吉林等地。其中贵州镇远拥有得天独厚的气候资源,是著名美酒产区,适宜原生态刺葡萄种植,葡萄产量高,粒大,着色度好,主要用于加工成葡萄酒。然而,对于该区葡萄园的微生物组成知之甚少,尤其是根际,研究该葡萄园土壤的微生物,对于评价该区酿酒葡萄基地的品质具有重要意义。由此,本研究选取贵州金奇谷葡萄园为研究基地,对该葡萄园根际土壤的理化性质及主要酶活进行探究,并结合现代分子生物学手段RT-PCR和高通量测序等对根际葡萄园土壤微生物群落结构组成和大小进行研究,旨在了解该葡萄园土壤养分,酶活及根际微生物情况,以期对该葡萄园的土壤质量评价和葡萄的合理种植及优质生产提供理论依据。
1 材料与方法
1.1土壤样品采集
土壤样品于2015年6月25日采自贵州省镇远县清溪镇金奇谷产业园(E 108°42′,N 27°05′),在该葡萄种植基地随机选取3株葡萄植株,采用5点取样法,以葡萄植株为中心,沿行内挖取离树干30 cm左右,0~20 cm深度的土壤,作为根区土壤;根际土壤采用抖落法[7],先抖落根系上附着的大块土壤,然后在纸上轻拍根系或用镊子轻触,将根系上附着的0~4mm的粒状或粉状土壤取下收集,此为根际土,分别标记为GY1、GY2、GY3;同时选取该植株附近无根系存在的裸地土壤作为对照,此为非根际土壤样品,混样后标记为CK。土样装入无菌封口塑料袋,带回实验室。一部分土样于室内自然风干,研磨、过筛,置于4℃冰箱内保存以供土壤基本理化性质和土壤酶活测定,另一部分置于-20℃冰箱内保存,以供土壤微生物学指标分析。
1.2土壤理化性质及酶活测定
土壤理化性质按常规方法测定[8]。含水量的测定采用烘干法,pH值采用pH计测定(水土比=5∶1),有机质测定采用重铬酸钾外加热法,全氮采用凯氏定氮法测定,总磷用HClO4-H2SO4消解-钼蓝比色法测定,速效磷采用NaHCO3浸提-钼锑抗比色法。供试土壤理化性质见表1。
土壤脲酶采用苯酚钠比色法,以24 h后每1g土中的NH4+-N的mg数表示;淀粉酶采用3,5-二硝基水杨酸法测定,以24 h后1g土中麦芽糖的mg数表示;蔗糖酶采用3,5-二硝基水杨酸法测定,以24 h后1g土中葡萄糖的mg数表示;磷酸酶采用磷酸苯二钠比色法测定,以24 h后每1g土壤中酚的mg数表示;过氧化氢酶采用高锰酸钾滴定法测定,以每1g土壤消耗的0.02mol/L的高锰酸钾的mg数表示[9]。
1.3土壤总基因组DNA的提取和荧光定量PCR
每个样品取0.4g土样提取DNA,采用OMEGA公司的SoilDNA Kit进行土壤全基因组提取,经1%琼脂糖凝胶电泳测定DNA完整性、Nanodrop测定DNA纯度和浓度,以-20℃保存、备用。
荧光定量PCR(quantitative polymerase chain reaction,Q-PCR)采用Takara荧光定量试剂盒(SYBR®Prem ix Ex TaqTM II,Takara,Dalian,China)进行PCR扩增。以提取总DNA为模板,采用细菌特异引物338F/518R扩增16S rRNA,PCR扩增体系和程序参照参考文献[10]。最后利用7500 system SDS software软件构建标准曲线及对荧光定量数据进行分析。
1.4土壤样本的高通量测序
1.4.1PCR扩增
将稀释后的基因组DNA作为PCR模板,采用细菌特异引物341F/805R扩增16S rRNA-V4区,同时在上游引物的5'端添加带6个碱基长度Barcode序列区分样品。细菌扩增程序为95℃预变性2m in,95℃变性15 s,55℃退火15 s,72℃延伸30 s,30个循环后,72℃终延伸10m in,保持4℃直到反应完成。每个样品做3个重复,将同一样品的PCR产物用2%的琼脂糖凝胶进行电泳检测,再根据PCR扩增产物的浓度进行等浓度混样,最后使用GeneJET胶回收试剂盒(Thermo Scientific公司)回收产物。
1.4.2文库构建和上机测序
使用NEB Next®UltraTM DNA Library Prep Kit (New England Biolabs公司)建库试剂盒进行文库的构建,文库构建好后经过Qubit定量和文库检测,其结果合格后使用M iSeq 2500测序平台(诺禾致源,北京)进行上机测序。
1.5数据分析
根据Barcode序列和PCR扩增引物序列从下机数据中拆分出各样品数据后使用FLASH软件对每个样品的reads进行拼接,得到高通量测序原始数据(Raw Tags)。进一步去除嵌合体、两端引物以及非靶区域序列后得到有效数据(Effective Tags)。再利用Uparse软件对有效数据以97%的一致性进行OTU聚类,并利用Greengene数据库进行物种注释。然后根据OTU使用Qiime软件计算Observed-species,Chao1,Shannon,Simpson,ACE,Goods-coverage指数,使用R软件(Version 2.15.3)绘制稀释曲线,Rank abundance曲线,物种累积曲线并使用R包进行Alpha多样性和Beta多样性分析。然后用SPSS 18.0软件对细菌群落结构与土壤养分及酶活进行Pearson相关性分析。最后利用Canoco软件进行冗余分析(Redundancy analysis,RDA)。在进行RDA分析之前,先进行降趋对应分析(DCA),当DCA分析结果中排序轴梯度最大值小于3时选择RDA分析,从而对细菌群落相对丰度和土壤养分及酶活的关系进行进一步分析。
2 结果与分析
2.1葡萄园土壤理化性质及酶活(表1、表2)

表1 供试土壤的理化性质情况
土壤pH值、有机质、总氮、总磷和有效磷是土壤质量状况的重要指标。由表1可知,葡萄园3个根际土壤pH值处在7.96~7.98之间,属于弱碱性土壤;根际土壤的有机质处在6.66~22.36g/kg之间;根际土壤总氮(处在0.57~1.47mg/kg之间)明显高于非根际土(0.38mg/kg);根际总磷(处在0.43~0.56g/kg之间)高于非根际土(0.34g/kg);根际土有效磷(处在19.38~26.09mg/kg之间)明显高于非根际土(9.66mg/kg)。
蔗糖酶、淀粉酶、脲酶和磷酸酶是土壤中的几种主要水解酶类。从表2可知,根际土壤酶活与非根际酶活性存在一定差异。其中葡萄园根际土壤淀粉酶活明显高于非根际土壤,而过氧化氢酶活和脲酶酶活低于非根际土壤。

表2 供试土壤的酶活
2.2RT-PCR定量分析葡萄园土壤微生物群落大小
用RT-PCR技术对细菌16S rRNA基因拷贝数进行定量分析,16S rRNA基因扩增的标准曲线的斜率分别为-3.33,R2值为0.997,扩增效率分别为99.50%,基因拷贝数见图1。根际土壤细菌16S rRNA基因的拷贝数在1.38×108~1.17×109copies/g之间,高于非根际土壤。
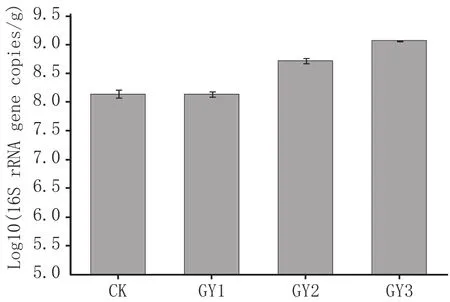
图1 葡萄园细菌16S rRNA基因拷贝数
2.3高通量测序分析葡萄园土壤细菌群落结构
2.3.1葡萄园土壤细菌测序序列及α多样性分析
通过对供试土壤宏基因组进行16S rRNA V4区高通量测序,共获得213382条原始序列,207327有效序列。根据97%水平下OTU的丰度,对有效序列利用QIIME软件处理操作分类单元,获得10935个OTUs分类,每个土壤样品的序列数及OTU数见表3。使用Rarefaction curve对样品进行评估,稀释曲线趋于饱和,证明此次测序的深度是合理的,继续测序不会再产生较多新的OTU(图2)。另外,表3也列出了依据OUT的根际土壤和非根际土壤α多样性指数。从Shannon指数及Simpson指数来看,根际土细菌群落的α多样性低于非根际土壤。

表3 葡萄园土壤测序序列和α多样性分析
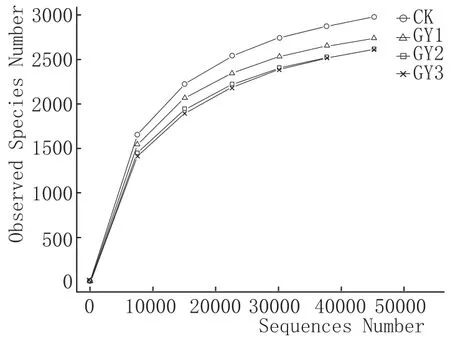
图2 土壤样品稀释性曲线
2.3.2葡萄园土壤细菌落组成及丰度
从门的分类水平看(图3a),葡萄园土壤的优势细菌类群为变形菌门(Protecbacteria),所占比例为33.42%~41.26%,其次为放线菌门(Actinbacteria),占16.07%~31.05%,其他的优势微生物有酸杆菌门(Acidobacteria)(占6.27%~12.28%),厚壁菌门(Firm icute)(占3.00%~9.18%),绿弯菌门(Chloroflexi)(占5.24%~8.88%),蓝藻门(Cyanobacteria)(占0.48%~4.53%);拟杆菌门(Bacteroidetes)(占1.53%~3.15%),芽单胞菌门(Gemmatimonadete)(占1.68%~2.74%),硝化螺旋菌门(Nitrospirae)(占1.26%~2.19%)。同时还检测到古菌中的泉古菌门(Crenarchoeota),占1%~4%,广古菌门(Euryarchaeota),占0.2%~0.8%。其中根际土变形菌门、厚壁菌门,蓝藻门,拟杆菌门的相对丰度明显低于非根际土壤,根际土放线菌门,泉古菌门,绿弯菌门,芽单胞菌门的相对丰度明显高于非根际土壤。
进一步从属的角度分析发现(图3b),根际土壤和非根际土壤分别鉴定出259个和276个细菌属。主要优势细菌属(排名前10属)包括乳球菌属(Lactococcus),节细菌属(Arthrobacter),Candidatus Nitrososphaera,亚硝化螺菌属(Nitrososphaera),热单胞菌属(Thermomonas),Phycicoccus,泛菌属(Pantoea),芽孢杆菌属(Bacillus),埃希氏杆菌属(Escherichia),鞘脂单胞菌属(Sphingomonas),和溶杆菌属(Lysobacter)。其中根际土壤中节细菌属,泛菌属,芽孢杆菌属,鞘脂单胞菌属丰度明显低于非根际土壤,而根际土壤中Candidatus Nitrososphaera丰度明显高于非根际土壤。另外,对比根际和非根际土壤细菌属可以发现,葡萄园根际和非根际土壤中各含有一些特有的低丰度细菌属(表4)。其中根际特有属4个,比如Sphingobium某些属具有降解苯酚,异丙隆等功能,Desulfosporosinus广泛参与硫还原过程;非根际特有属18个,并各自具有一些特殊生物学功能。
2.3.3葡萄园土壤细菌群落聚类分析和PCoA分析
根据各土壤样品的OTU聚类及其丰度,使用Unifrac距离的Unweighted算法对样品进行了聚类分析(图4a)和PCoA分析(图4b)。由UPGMA聚类树可以看出,根际土3个不同位点的样品聚在同一个分支,非根际土壤样品单独在一个分支,表明同一葡萄园不同位点的根际土壤细菌群落结构较为相似,不同于非根际土壤。由PCoA可以看出,第一轴得分63.44%,第二轴得分29.20%,两轴之和超过了80%,与非根际土壤相比,根际土壤3个不同位点位置相对集中,说明不同位点的根际土壤细菌群落结构更为相近。总的来说,聚类分析和PcoA分析结果一致,均表明根际土壤与非根际土壤细菌群落结构有所差异。
2.4葡萄园土壤细菌群落结构和土壤养分及酶活相关性分析
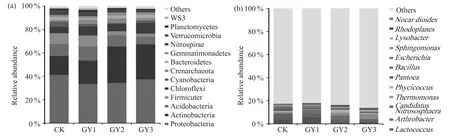
图3 葡萄园土壤细菌群落结构相对丰度
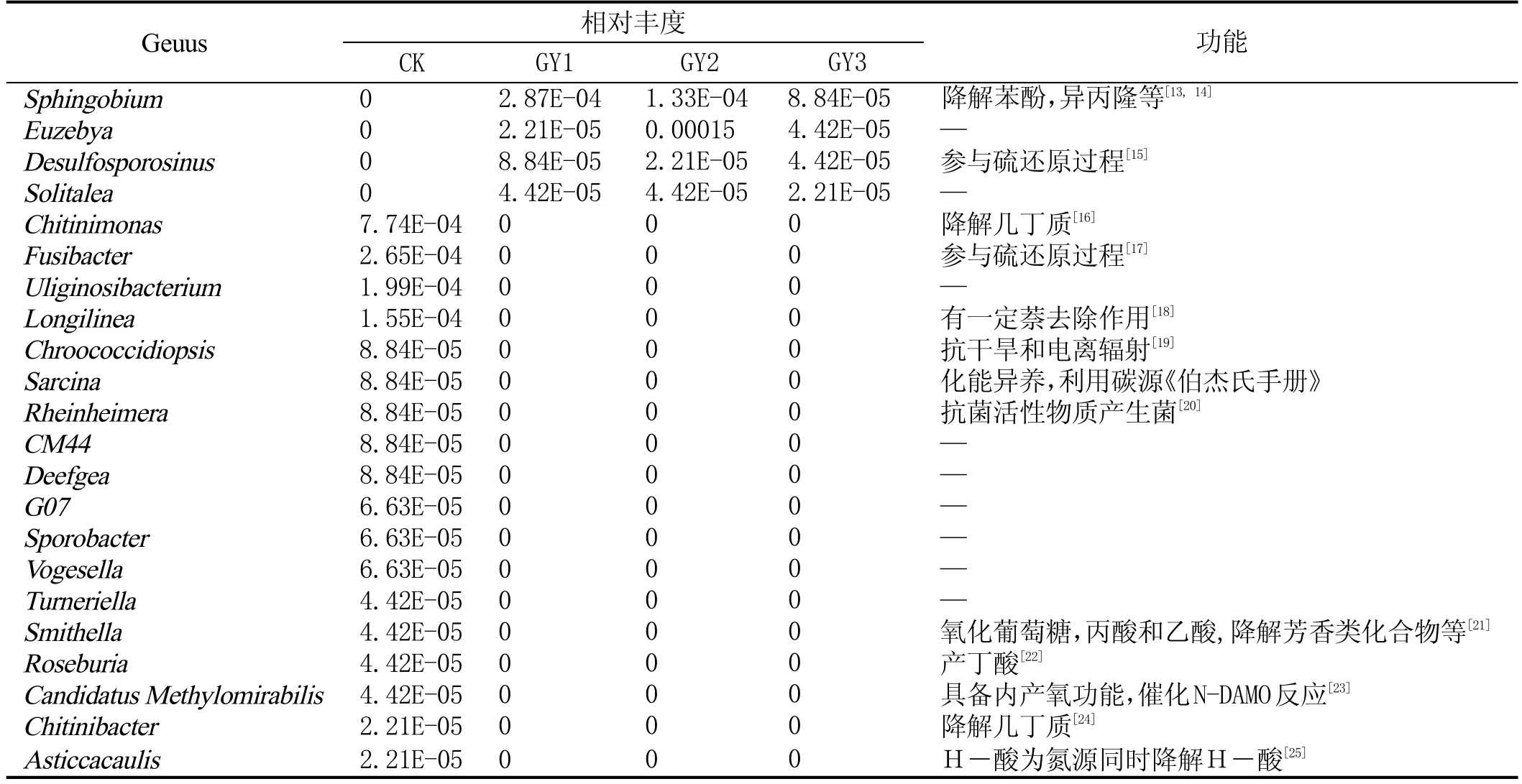
表4 葡萄园根际土壤和非根际土壤细菌差异属比较分析

图4 葡萄园土壤基于Unweighted Unifrac距离的β多样性分析
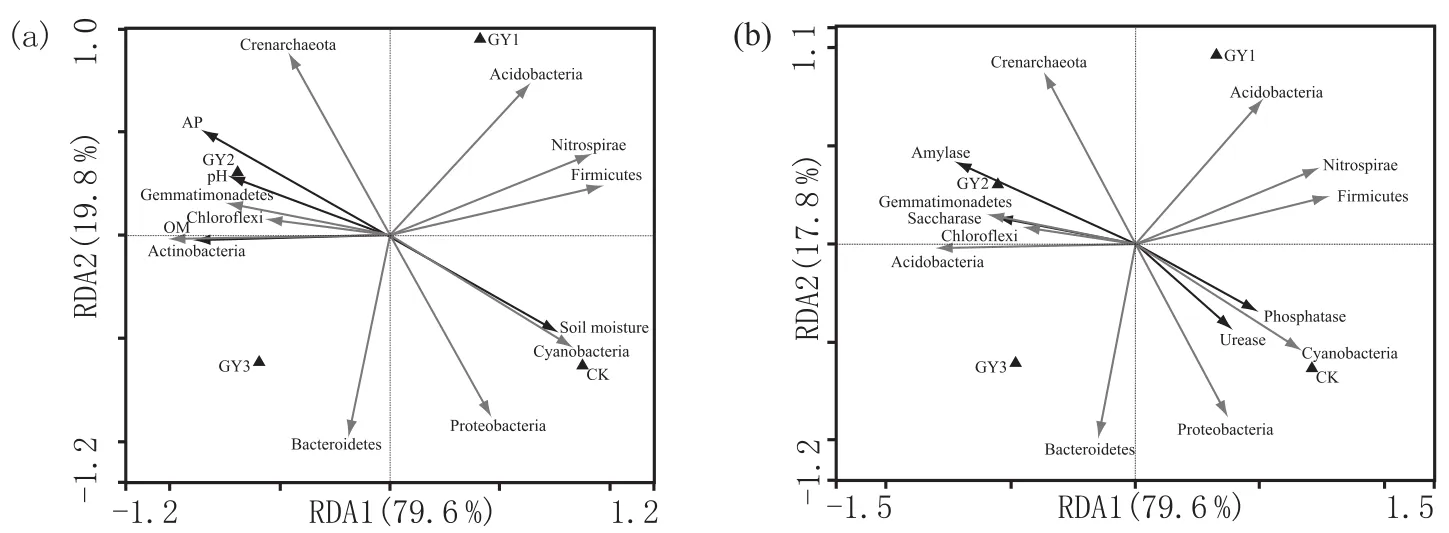
图5 葡萄园土壤因子与细菌群落多样性间RDA分析
运用Canoco4.5软件,将细菌群落结构的相对丰度分别和土壤养分及酶活进行RDA排序(图5)。前两个轴得分分别为79.6%和17.8%,两者之和接近于100%,这说明本研究选择的土壤养分和酶活能较好地解释葡萄园土壤细菌群落,而且各细菌类群与各因子间的相关系数较高。结果显示,参与RDA分析的各因子在根际和非根际土壤细菌群落组成之间所起的作用是不同的。其中土壤放线菌门、绿弯菌门、芽单胞菌门和泉古菌门与葡萄园土壤的pH值、有机质、有效磷含量、淀粉酶和蔗糖酶呈现正相关关系,变形菌门、厚壁菌门和蓝藻门与葡萄园土壤水分、脲酶和磷酸酶呈较好的正相关关系。另外,土壤水分与pH值及脲酶酶活呈显著的负相关关系(p<0.05),有效磷与淀粉酶呈显著的正相关关系(p<0.01)。
3 讨论
3.1葡萄园土壤养分及酶活评价
葡萄植株生长需要从土壤中吸收养分,土壤养分作为根际微生态系统中的重要因素之一,其含量对葡萄植株生长产生重要影响。葡萄最适宜的土壤pH6.5~7.5,本文选取的几个样点pH7.90~7.95之间,与最适pH值比较接近。另外,该葡萄园根际土平均含水量为9.67%,有机质为14.51g/kg,达到果园土壤养分肥力指标有机质的三级水平;总氮含量为1.01mg/kg,仅为果园土壤养分肥力指标四级水平,总体含量偏低;总磷和有效磷含量分别为0.48g/kg和21.71mg/kg,达到果园土壤养分肥力指标有效磷一级水平[11]。总体来看,该葡萄园根际土壤有机质、总氮含量偏低,有效磷含量较高,因此该葡萄园应调整施肥措施,提高土壤中有机质和氮的含量,更好地促进葡萄植株生长。
土壤酶活在一定程度上也能表征土壤的质量,一般认为,土壤酶活性是生物与非生物活性的总和,不仅受土壤微生物的影响,还受环境等非生物因素的影响[12]。本文中葡萄园根际土壤各酶活性各异,其中不同位点的葡萄园根际土壤的脲酶和过氧化氢酶活比较接近,这说明空间位置对这两种酶活影响不大,但对另外几种酶影响较大。另外,根际土的淀粉酶活明显高于非根际土,根际土的磷酸酶活明显低于非根际土壤,这表明淀粉酶活与酸酶活可能对根际效应比较敏感,受根系分泌物的影响可能比较大。由此看出,尽管是同一葡萄园土壤,土壤酶活对根际土不同空间位点以及非根际土壤做出了不同的表现,这进一步说明土壤酶活受多种因素的影响。
3.2葡萄园根际土壤和非根际土壤细菌群落结构
本研究通过对16S rDNA-V4区进行高通量测序,分析了该葡萄园根际土和非根际土细菌的群落结构。在97%的相似度水平上,将获得的OUT分别从门到属的水平上进行物种注释,归类为42个门,130纲,213个目,249个科,295个属。分析表明,葡萄园土壤的优势细菌类群为变形菌门,其次为放线菌门和酸杆菌门,其他的优势菌群包括厚壁菌门、绿弯菌门、蓝藻门、拟杆菌门、芽单胞菌门等,这与Kayla N.Burns等[3]对不同区域葡萄园细菌群落结构研究结果类似。另外,从门的分类水平来看,该葡萄园根际土壤和非根际的细菌群落组成相似,最丰富的3个优势种群均为变形菌门,放线菌门和酸杆菌门,其中变形菌门的丰度最高,其次是放线菌门,这与韩亚飞等[4]对杨树根际和非根际土壤细菌多样性研究相一致。这表明土壤根际细菌群落多样性虽然受根际效应的影响,但在门分类水平上差异并不是很大。
从属的分类水平看,根际和非根际细菌群落的种类和数量均具有一定差异,由α多样性指数显示,根际土壤细菌丰度低于非根际土壤细菌群落丰度,这与韩亚飞等[4]对连作杨树根际和非根际细菌丰度结果相一致,但与汪其同等[1]对杨树根际和非根际的细菌群落结构多样性结果相反,其结果发现根际土壤细菌丰度高于非根际土壤细菌群落丰度。在所有检测到的土壤细菌属中,相对丰度大于2%的有5个,包括乳球菌属(Lactococcus),节细菌属(Arthrobacter),Candidatus Nitrososphaera,热单胞菌属(Thermomonas)和Phycicoccus。此外,在根际和非根际还有一些相对丰度较高但丰度差别较大的属,其中根际对非根际显著下调的属包括节细菌属,泛菌属,芽孢杆菌属,鞘脂单胞菌属等。在非根际土壤中存在一些特有的降解几丁质,产丁酸等的相对丰度较低的细菌属。汪其同等[1]认为,根际和非根际微生物的群落的趋异化发展,其原因最初归结于根系分泌。细菌群落结构的演变可能与葡萄根系的分泌物有一定关系,但鉴于葡萄根际微生物组成非常复杂,影响因素众多,需进一步加大样本量,充分的利用高通量测序技术,更为全面解析根际土壤与非根际土壤理化性质差异,以期对葡萄根际理化性质、微生物群落结构有一个更全面和更深度的认识。
[1]汪其同,朱婉芮,刘梦玲,等.基于高通量测序的杨树人工林根际和非根际细菌群落结构比较[J].应用与环境生物学报, 2015(5):29.
[2]AiraM,Bybee S,Domínguez J.Carnivory doesnotchange the rhizosphere bacterialcommunity of the plant Drosera intermedia[J].Applied Soil Ecology,2015(92):14-17.
[3]Burns K N,KluepfelDA,Strauss SL,etal.Vineyard soil bacterialdiversity and composition revealed by 16S rRNAgenes:Differentiation bygeographic features[J].Soil Biology and Biochem istry,2015(91):232-247.
[4]韩亚飞,伊文慧,王文波,等.基于高通量测序技术的连作杨树人工林土壤细菌多样性研究[J].山东大学学报(理学版),2014 (5):1-6.
[5]张蕾,徐慧敏,朱宝利.根际微生物与植物再植病的发生发展关系[J].微生物学报,2016(8):1234-1241.
[6]纳小凡,郑国琦,彭励,等.不同种植年限宁夏枸杞根际微生物多样性变化[J].土壤学报,2016(1):241-252.
[7]Grayston S J,Wang SQ,CampbellCD,etal.Selective influence of plantspeciesonm icrobialdiversity in the rhizosphere[J]. Soil Biology and Biochemistry,1998,30(3):369-378.
[8]鲍士旦.土壤农化分析[M].3版.北京:中国农业出版社,2000:22-86.
[9] 关松荫.土壤酶及其研究法[M].北京:农业出版社,1986:274-339.
[10]LiP,LiS,Cheng L.M icrobiota dynam icsassociatedw ithenvironmental conditionsand potential rolesof cellulolytic communities in traditionalChinese cerealstarter solid-state fermentation[J].Appl Environ M icrobiol,2015,81(15):5144-5156.
[11]侍朋宝,陈海菊,张振文.山地酿酒葡萄园土壤理化性质分析[J].土壤,2009(3):495-499.
[12]陈汝,王海宁,姜远茂,等.不同苹果砧木的根际土壤微生物数量及酶活性[J].中国农业科学,2012(10):2099-2106.
[13]孙纪全,梁斌,黄星,等.Sphingobium属细菌土壤中降解异丙隆的特性[J].土壤学报,2011(2):383-388.
[14]许甜甜,陈军.Sphingobium sp.和Delftia sp.复合降解苯酚的研究[J].上海师范大学学报(自然科学版),2012(1):94-98.
[15]Senko JM,ZhanggX,M cdonough JT,etal.Metal reduction at low pH by a Desulfosporosinusspecies:implications for the biological treatmentof acidicm ine drainage[J].geomicrobiology Journal,2009,26(2):71-82.
[16]LiY,Zhu H,LaiQ,etal.Chitinimonasprasina sp.nov.,isolated from lakewater[J].International Journalof Systematic& Evolutionary M icrobiology,2014,64(9):3005-3009.
[17]Ben HW,Fraj B,Postec A,etal.Fusibacter tunisiensis sp.nov., isolated from an anaerobic reactorused to treatolive-m ill wastewater[J].International Journalof Systematic& Evolutionary M icrobiology,2012,62(Pt6):1365-1368.
[18]曹新垲,杨琦,郝春博.厌氧污泥降解萘动力学与生物多样性研究[J].环境科学,2012.33(10):3535-3541.
[19]刘洋,朱梦灵,徐瑶,等.中国蓝藻植物的新记录属——拟甲色球藻[J].水生生物学报,2013(3):413-417.
[20]李贺,林学政,何培清,等.南极抗细菌活性菌株的筛选及系统发育分析[J].微生物学通报,2011(2):264-269.
[21]Ariesyady H D,Ito T,Okabe S.Functionalbacterialand archaeal community structuresofmajor trophicgroups in a full-scaleanaerobic sludge digester[J].Water Research,2007,41 (7):1554-68.
[22] 沈丽,童卉琳.益生菌及早期进食对SD大鼠术后菌群失调的影响[J].青岛大学医学院学报,2011,47(6):507-508.
[23]沈李东.亚硝酸盐型甲烷厌氧氧化微生物特性研究进展[J].环境科学,2015(3):1133-1140.
[24]高聪,张阿磊,陈可泉,等.Chitinibactersp.GC72的筛选鉴定及其几丁质降解产物研究[J].食品与生物技术学报,2015(1):15-20.
[25] 黄丽萍,周集体,王竞,等.一株降解H-酸的细菌的分离筛选[J].环境科学研究,2000(2):6-8.
Analysisof BacterialCommunity Structure in Vineyard Soilby RT-PCR and High Throughput Sequencing
WANG Xiaowen,HONG Zhenhan,LIUAnruiand LUO Lixin
(Schoolof Biosciencesand Bioengineering,South China University of Technology,Guangzhouguangdong 510006,China)
Soilbacteria have importantecological functionsand play a very important role in vineyard ecological system.In this study,we performed quantitative PCR(Q-PCR)and Illum ina M iseq high-throughputsequencing to compare the basic physiochem ical parameters,enzymatic activities and bacterialgroupsbetween the rhizosphere and non-rhizosphere soil from a vineyard from Zhenyuan,Guizhou.Q-PCR results indicated that,the bacterial16S rRNAgene copy numbers in the rhizosphere soils ranged from 1.38×108to 1.17×109copies/g,higher than that in non-rhizosphere soil.Illum ina M iseq high-throughput sequencing results indicated that,the bacterial communities in rhizosphere vineyard soil mainly included Protecbacteria,Actinbacteria,Acidobacteria,Firm icute,Chloroflexi,Cyanobacteria,Crenarchoeo,Bacteroidetes,Gemmatimonadete,and Nitrospirae,among them,the dom inant bacterial taxa members were Proteobacteria and Actinobacteria which accounted for 53.33%~67.28%of total abundance.Atgenus level,Lactococcus,Arthrobacter,Candidatus Nitrososphaera,Nitrososphaera,Thermomonas, Phycicoccus,Pantoea,Bacillus,Escherichia,Sphingomonas,Lysobacter were the dominantbacteriagenera in rhizosphere vineyard soil.α-diversity analysis showed that,the bacterial community diversity of rhizosphere soilwas sim ilar in different site butwere lower than thatof the non-rhizosphere soil.Redundancy analysis showed that,therewere certain correlations between bacterial communities and nutrients and enzymeactivity in vineyard soil..
vineyard soil;rhizosphere soil;bacterialdiversity;high throughputsequencing;quantitative PCR(Q-PCR)
TS262.6;Q93-3;TS261.1;TS261.2
A
1001-9286(2016)11-0028-06
10.13746/j.njkj.2016279
华南理工大学“学生研究计划”(SRP)项目(105612015S533)。
2016-09-14
王晓雯,女,河南驻马店人,在读硕士研究生,研究方向为食品微生物发酵。
罗立新,男,广东兴宁人,教授,博士生导师,研究方向:工业微生物学,发酵工程,微生物分子生态学,E-mail:btlxluo@scut.edu.cn。
优先数字出版时间:2016-10-12;地址:http://www.cnki.net/kcms/detail/52.1051.TS.20161012.1505.016.htm l。
