水稻淡绿叶突变体HM133的遗传分析与基因定位
2016-12-07施勇烽贺彦郭丹吕向光黄奇娜吴建利
施勇烽 贺彦 郭丹 吕向光,2 黄奇娜 吴建利,*
(1中国水稻研究所 水稻生物学国家重点实验室/国家水稻改良中心, 杭州 310006; 2中国农业科学院 作物科学研究所, 北京 100081;# 共同第一作者; *通讯联系人, E-mail: beshangd@163.com)
水稻淡绿叶突变体HM133的遗传分析与基因定位
施勇烽1,#贺彦1,#郭丹1吕向光1,2黄奇娜1吴建利1,*
(1中国水稻研究所 水稻生物学国家重点实验室/国家水稻改良中心, 杭州 310006;2中国农业科学院 作物科学研究所, 北京 100081;#共同第一作者;*通讯联系人, E-mail: beshangd@163.com)
SHI Yongfeng, HE Yan, GUO Dan, et al. Genetic analysis and gene mapping of a pale green leaf mutantHM133 in rice. Chin J Rice Sci, 2016, 30(6): 603-610.
EMS诱导籼稻品种IR64获得淡绿叶突变体HM133。与野生型IR64相比,HM133播种后的第6周和第15周的光合色素含量以及抽穗期的净光合速率显著降低,气孔导度则明显上升;此外,突变体株高、每穗实粒数和结实率等农艺性状也较野生型显著下降。叶绿体超微结构分析表明,分蘖期HM133类囊体基粒片层形状不规则,堆叠凌乱、排列疏松。遗传分析表明HM133淡绿叶性状受单隐性核基因控制。通过分子标记将该基因定位于第3染色体长臂RM143和RM3684之间。该区间内包含编码镁螯合酶D亚基的基因OsCHLD。序列分析表明HM133中该基因第10外显子上有一个从G突变为A的单碱基变异,导致编码的氨基酸由精氨酸变成谷氨酸,推测OsCHLD基因即为控制HM133淡绿叶表型的候选基因。
水稻; 淡绿叶; 基因定位; 镁螯合酶; 光合作用
叶片是植物进行光合作用的主要场所。叶绿体是进行光合作用的重要细胞器,叶绿体发育异常、光合色素含量改变以及光合作用复合体的稳定性等都会影响影响植物的光合作用效率。常见的叶色变异大多与光合色素合成与降解,叶绿体的结构、组成和生理状态异常有关,通常会导致叶片出现白化、黄化、淡绿、黄绿和条纹等突变表型。在水稻[1,2]、玉米[3,4]、大麦[5]、拟南芥[6]和油菜[7]等植物中均有叶色突变体的报道。
目前水稻中已经鉴定了大量的叶色突变体(http://www.shigen.nig.ac.jp/rice/oryzabase/),分别涉及叶绿素代谢、光敏色素代谢以及叶绿体发育异常。其中,影响叶绿素生物合成途径相关基因的突变是研究报道最为深入的一类。对拟南芥的研究发现叶绿素生物合成途径共包括15步反应,涉及15种酶,并分离了 27个编码这些酶的基因[8]。水稻中目前也已克隆多个叶绿素合成途径中相关酶的基因,如OsCHLH、OsCHLD、OsCHLI、DVR、CAO1、CAO2、YGL1和PORB[2,9-14]。另外,四吡咯代谢分支途径即光敏色素合成途径中相关基因的突变也能造成叶色突变,例如水稻亚铁血红素加氧酶基因的变异能反馈作用于叶绿素合成途径,从而使叶绿素的合成发生异常[15,16]。除了约100个叶绿体基因组自身编码的蛋白外,叶绿体中含有大约3000个由核基因编码的蛋白,相关蛋白的突变会影响叶绿体的正常发育,导致叶色变异、植株发育迟缓等症状,如编码类囊体结合蛋白基因ZN突变可导致水稻产生斑马叶表型[17],编码PPR蛋白的基因YSA的突变致使水稻产生白化表型[18],编码叶绿体SRP43蛋白[19]和SRP54蛋白[20]基因的变异均导致水稻叶色变成淡绿色。
本研究利用籼稻IR64经EMS诱导后筛选获得一份叶色突变体HM133,对其开展表型性状、生理特性等方面的研究,同时对突变基因开展了遗传分析和基因定位,结合测序分析结果推测水稻镁螯合酶D亚基基因(OsCHLD)是控制HM133淡绿叶性状的候选基因。
1 材料与方法
1.1 供试材料
本研究采用的淡绿叶突变体HM133是由籼稻品种IR64经过EMS诱变得到。经过连续多代自交,该叶色突变性状无论种植于海南陵水或浙江富阳,在温室条件或大田环境下均能稳定遗传。2015年将该突变体种植于中国水稻研究所富阳实验基地,在成熟期随机选取野生型和突变体各3株考查株高、穗长、有效穗数、结实率和千粒重等农艺性状,取平均值进行分析。
1.2 群体构建及遗传分析
以突变体HM133为母本,分别与正常叶色品种IR24和热研1号配制杂交组合,观察F1的表型并收获F1单株种子,观察并统计HM133/IR24的F2群体中正常叶和淡绿叶水稻的株数,用于遗传分析。HM133/热研1号的F2群体中淡绿叶表型单株用于DNA提取和基因定位。
1.3 光合色素含量和光合参数测定
播种后第6周和第15周分别取野生型和突变体HM133倒2叶样品,称取约0.3 g,剪成0.3~0.5 cm长度的片段,在常温黑暗条件下用95%乙醇浸提48 h,然后用PerkinElmer分光光度计分别测定在470 nm、649 nm和665 nm三个波长处的光吸收值。播种后第6周和第15周各取3株设定生物学重复,参照Arnon和Wellburn等[21,22]的方法计算叶绿素和类胡萝卜素的含量。
于水稻抽穗期利用便携式光合测定仪LI-6400(LI-COR,USA)在晴好天气上午9:00-11:00,田间测定野生型和突变体HM133剑叶光合作用指标,光合参数设定参照Huang等[23]的方法。每株重复3次测定,取平均值。
1.4 叶绿体超微结构观察
选取野生型和突变体HM133分蘖期倒2叶叶片切成1 mm片段,在2.5% 戊二醛溶液中抽真空固定。参照Lv等[19]的方法进行样品制备,于浙江大学农业与生物技术学院用Tecnai G2F20 S-TWIN型透射电子显微镜(FEI,USA)观察叶绿体超微结构。
1.5 基因定位
采用简易法提取亲本和单株DNA[24],分别取HM133/热研1号F2群体正常叶和淡绿色叶单株各10株,以等量叶片构建野生型DNA池和突变体DNA池。利用水稻12条染色体上172对SSR标记,对亲本HM133和热研1号间进行多态性标记筛选,再将筛选到的标记用于野生型池和突变体池间的多态性分析,找到两池间有多态的标记,用于F2群体中淡绿叶单株的基因型分析,初步确定突变基因的位置。从Gramene数据库(http://www.gramene.org/)下载SSR标记引物序列,由上海生工生物工程有限公司进行合成。参照Shi等[25]的方法进行PCR和产物检测。
1.6 候选基因OsChlD的测序和验证
利用水稻基因组信息注释系统(http://rice.plantbiology.msu.edu),查询目标区间内的基因。根据OsChlD基因的cDNA序列设计引物(正向引物5′-TCTCTCCCTCCCCTCCCATG-3′,反向引物5′-GCTCTCCAGGATCACGAACTCT-3′),并对HM133、IR64和IR24进行PCR,由铂尚生物技术(上海)有限公司进行测序。
根据测序结果的比对,找到突变位点,在突变位点附近根据OsChlD基因组序列设计引物(正向引物5′-TGCTTGGCACCTTTATCACA-3′,反向引物5′-GCCATTCTTTTGGCTCTCAT-3′),对HM133、 IR24及其F2单株进行DNA扩增,扩增产物用限制性内切酶TaqⅠ进行酶切验证。
表1 实时定量PCR引物
Table 1. Primers used in real-time PCR.

基因Gene正向引物(5'-3')Forwardprimer(5'-3')反向引物(5'-3')Reverseprimer(5'-3')OsChlDGGAAAGAGAGGGCATTAGCAATACGATCAAGTAAGTGTTOsChlIAGTAACCTTGGTGCTGTGAATCCATCAACATTCAACTCTGOsChlHCTATACATTCGCCACACTTATCACACAACTCCCAAGHEMA1CGCTATTTCTGATGCTATGGGTTCTTGGGTGATGATTGTTTGGPORATGTACTGGAGCTGGAACAACAAGAGCACAGCAAAATCCTAGACGCAO1GATCCATACCCGATCGACATCGAGAGACATCCGGTAGAGCCab1RAGATGGGTTTAGTGCGACGAGTTTGGGATCGAGGGAGTATTTCab2RTGTTCTCCATGTTCGGCTTCTGCTACGGTCCCCACTTCACTpsaAGCGAGCAAATAAAACACCTTTCGTACCAGCTTAACGTGGGGAGpsbACCCTCATTAGCAGATTCGTTTTATGATTGTATTCCAGGCAGAGCrbcLCTTGGCAGCATTCCGAGTAAACAACGGGCTCGATGTGATArbcSTCCGCTGAGTTTTGGCTATTTGGACTTGAGCCCTGGAAGGYGL1CAGTCTCCAATGGCCACCTTGCTTTCATCAGTGGCTGGSPPCGGAGAGGAAACATAATGACATAGGCATTTGTCTTTGTCTCPPR1CTAAGACCGAATGACAAATGCGCACTGCCAACAAGAATACCDVRCGAGCCCAGGTTCATCAAGGTGCCCTCCCGATCTTGCCGAACTCCUbiquitinGCTCCGTGGCGGTATCATCGGCAGTTGACAGCCCTAG
1.7 RNA提取和RT-PCR
采用Trizol法提取IR64与淡绿叶突变体HM133分蘖期倒2叶的总RNA,利用ReverTra Ace qPCR RT Master Mix with gDNA Remover试剂盒(TOYOBO, 日本)将RNA反转录为cDNA。采用SYBR®PremixExTaqTMⅡ (Tli RNaseH Plus)试剂盒(TaKaRa,日本)和Thermal Cycle Dice®Real Time System(TaKaRa, 日本)进行实时定量PCR,分析叶绿素合成与代谢途径中相关基因的表达情况。以水稻Ubiquitin为内参基因,相关基因的特异性引物见表1。
2 结果与分析
2.1 HM133的表型及主要农艺性状
突变体HM133叶色在苗期、分蘖期均表现为淡绿色(图1),该表型伴随整个生育期。突变体HM133的主要农艺性状如有效穗数、穗长和千粒重与野生型相比未发生明显变化,但结实率、株高和每穗实粒数均显著低于野生型(表2)。

A—野生型IR64和突变体HM133幼苗表型;B—野生型IR64和突变体HM133分蘖期的表型。
A, Phenotype of the wide type IR64 and the mutantHM133 at the seedling stage; B, Phenotype of the wide type IR64 and the mutantHM133 at the tillering stage.
图1 野生型IR64和突变体HM133的不同时期植株表型
Fig. 1. Phenotype of the wide-type IR64 and the mutant HM133 at different growth stages.
2.2 HM133的光合色素含量和光合参数
在播种后第6周和第15周分别测定了突变体和野生型叶绿素和类胡萝卜素含量。第6周和第15周时突变体的总叶绿素含量与野生型相比分别下降了46.0%和47.7%,胡萝卜素含量下降了38.8%和47.6%,两个时期HM133的总叶绿素含量、叶绿素a和叶绿素b含量与野生型相比均存在极显著差异(表3)。
表2 野生型IR64和突变体HM133的主要农艺性状
Table 2. Agronomic traits of the wild-type IR64 and mutant HM133.

材料Material株高Plantheight/cm有效穗数No.ofproductivepanicles穗长Paniclelength/cm每穗实粒数Numberoffilledgrainsperpanicle结实率Seed-settingrate/%千粒重1000-grainweight/gIR64113.0±1.714.0±1.025.0±1.481.2±7.174.6±1.627.52±0.38HM133108.7±0.6*12.7±2.124.9±0.362.9±7.3*68.8±2.1*28.35±0.35
*HM133与IR64在0.05水平上差异显著。
*Significant difference between IR64 andHM133 at 0.05 level.
表3 不同生长时期HM133与IR64叶片光合色素含量的比较

Table 3. Comparison of photosynthetic pigment contents between HM133 and IR64 at different growth stages. mg/g
*HM133与IR64在0.05水平上差异显著;**HM133与IR64在0.01水平上差异显著。
*Significant difference between IR64 andHM133 at 0.05 level;**Significant difference between IR64 andHM133 at 0.01 level.
表4 抽穗期野生型IR64和突变体HM133的剑叶光合特性
Table 4. Photosynthetic parameters of flag leaf of IR64 and HM133 at the heading stage.

材料Material净光合速率Pn/(μmol·m-2s-1)气孔导度GS/(mol·m-2s-1)胞间CO2浓度Ci/(μmol·mol-1)蒸腾速率Tr/(mmol·m-2s-1)IR6414.45±1.780.57±0.13309.13±7.304.12±0.64HM13312.74±1.38*0.73±0.09*313.75±6.414.26±0.25
*HM133与IR64在0.05水平上差异显著。
*Difference between IR64 andHM133 was significant at 0.05 level.
为分析突变体光合色素含量的降低对光合作用的影响,抽穗期对IR64和HM133的光合参数进行测定,发现突变体的净光合速率降低了11.8%,气孔导度上升了26.3%,而胞间CO2浓度和蒸腾速率没有显著差异(表4)。
2.3 叶绿体超微结构观察
分蘖期分别对野生型和淡绿叶突变体HM133叶片进行叶绿体结构的电镜观察。结果显示野生型IR64的叶绿体基质浓密、基粒片层堆叠较厚、排列紧密(图2-A、B),而HM133的基粒数量明显减少,基粒片层形状不规则,片层堆叠较少、排列疏松(图2-C、D)。结果表明HM133叶绿体的发育受到了严重影响。
2.4 HM133的遗传分析
2.5 pglHM133基因定位及其候选基因预测
HM133/热研1号的F2群体中85株淡绿叶单株作为定位群体,并采用分池法构建了淡绿叶和正常叶色DNA池各1个。选用分布于12条染色体上的172对SSR标记,对HM133和热研1号进行多态性标记的筛选,获得的128对多态性标记用于淡绿叶与正常绿叶DNA池的多态性分析,其中第3条染色体上RM520、RM85和RM143表现DNA池的多态。进一步用这3个标记分析F2群体的85株淡绿叶单株,分别检测到20株、28株和4株重组个体,其中RM143与标记基因紧密连锁。为更准确定位该基因,根据已经公布的SSR引物在RM143附近找到RM16108、RM16103、RM16097和RM3684在亲本间有多态的标记,进一步分析发现仅标记RM3684检测到10株重组个体,其他3个标记均无交换发生,最终将该基因定位于第3染色体的长臂RM3684和RM143之间,物理距离约为3.1 Mb(图3-A)。
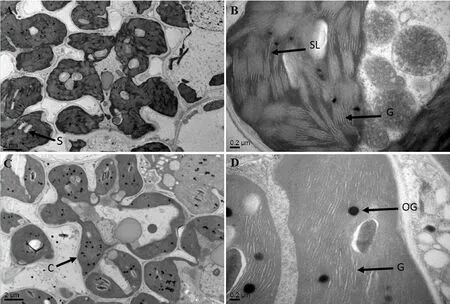
A和B-野生型IR64; C和D-突变体HM133。 S-淀粉粒; C-叶绿体; G-基粒; SL-基质片层; OG-嗜饿颗粒。
A and B, IR64; C and D,HM133. S, Starch granule; C, Chloroplast; G, Granum; SL, Stroma lamella; OG, Osmiophilic granule.
图2 野生型IR64和突变体HM133的叶绿体超微结构
Fig. 2. Chloroplast ultrastructure of IR64 and HM133.
通过水稻基因组信息注释系统(http://rice.plantbiology.msu.edu/)查询pglHM133目标区间内有超过200个候选基因,区间中包含已被Zhang等[10]注释为OsChlD基因(登录号为LOC_Os03g59640)。OsChlD编码镁螯合酶D亚基,参与叶绿素合成过程,对叶绿体发育具有重要作用。序列分析表明,IR64与IR24的OsChlD基因cDNA序列一致,而突变体HM133的第10外显子上存在一个单碱基突变,由G突变为A(图3-B),对应的氨基酸由精氨酸变为谷氨酸。该位点突变影响了限制性内切酶TaqⅠ 的特异性识别,用IR24/HM133的亲本、F1和F2群体进行验证,通过DNA扩增获得包含突变位点约193 bp大小片段,用TaqⅠ酶切后发现F2中淡绿叶单株和HM133的扩增片段不能被TaqⅠ酶切,而IR24能被TaqⅠ完全酶切,F1则被部分酶切,F2中正常叶单株有部分酶切和完全酶切两种情况(图3-C)。这一结果表明突变体HM133的淡绿叶表型可能是由OsChlD基因的单碱基替换导致。
2.6 叶绿素合成和叶绿体发育相关基因的表达
利用实时荧光定量PCR比较HM133及其野生型IR64中OsChlD基因和其他叶绿素合成、叶绿体发育相关基因的表达情况。结果表明,与野生型相比,突变体中编码镁螯合酶亚基的OsChlD、OsChlI和OsChlH的表达量明显下降,叶绿素合成相关基因HEMA1(谷氨酰t-RNA还原酶)、YGL1(叶绿素合成酶)、DVR(联乙烯还原酶)和CAO1(叶绿素酸酯氧化酶)的表达量也显著下降;叶绿体发育相关的psbA(D1蛋白)、psaA(光合系统Ⅰ叶绿素脱辅基蛋白)、rbcL(RUBP羧化酶大亚基)、SPP(基质加工肽酶)表达显著下调,而Cab1R和Cab2R(捕光叶绿素a/b结合蛋白)表达量显著上升;PORA(原叶绿素酸酯氧化还原酶)、rbcS(RUBP羧化酶小亚基)、PPR1(三角状五肽重复蛋白)的表达并没有显著变化(图4)。上述结果表明,OsChlD的变异导致与叶绿素合成以及叶绿体发育相关的众多基因的表达发生明显改变。
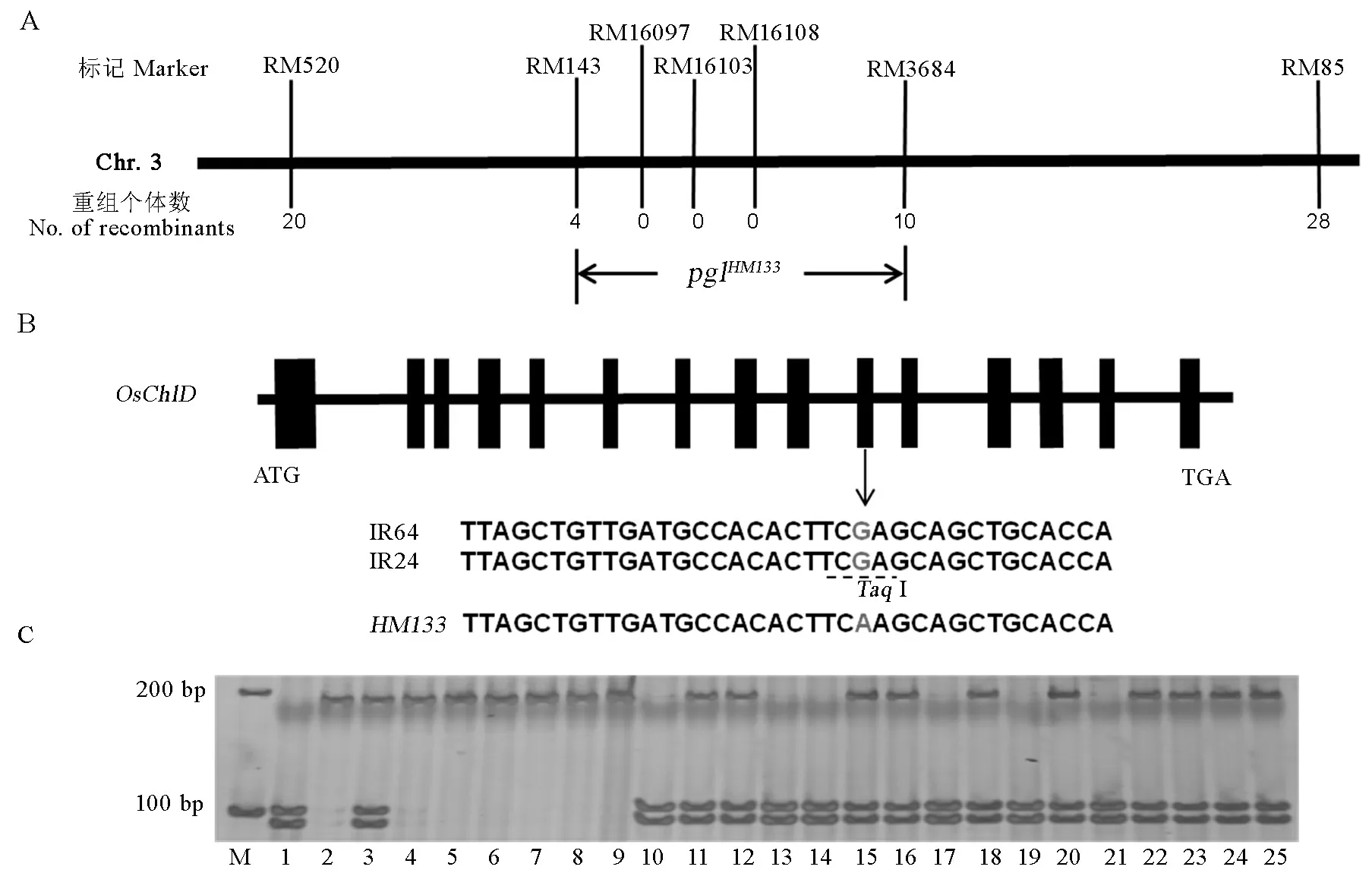
A-pglHM133初步定位; B-候选基因OsChlD序列分析. 灰色部分表示HM133的1541位碱基G突变为A; C-IR24/HM133 F2群体的TaqⅠ酶切验证. M-分子量标记; 1-IR24; 2-HM133; 3-F1; 4~9-F2中淡绿叶单株; 10~25-F2群体中正常叶单株。
A,Primary mapping ofyglHM133on chromosome 3; B, Sequence analysis of the candidate geneOsChlD. The gray letters indicate single base substitution from G to A at position 1541; C, Mutation base ofOsChlDconfirmed withTaqI restriction enzyme digestion. M, Molecular marker; 1, IR24; 2,HM133; 3, F1; 4-9, Pale green plant of F2; 10-25, Normal plant of F2.
图3 pglHM133定位及候选基因预测
Fig. 3. Location of pglHM133and candidate gene prediction.

图4 RT-PCR分析野生型IR64与HM133 中叶绿素合成、叶绿体发育相关基因的表达
Fig. 4. Expression of genes associated with chlorophyll biosynthesis and chloroplast development in IR64 and HM133 by real-time PCR.
3 讨论
植物中叶绿素的累积使叶片呈现绿色,叶绿素的生物合成需要包括镁螯合酶在内的15种酶的参与。镁螯合酶是由D、I和H三亚基组成的复合体,四吡咯化合物原卟啉Ⅸ在镁螯合酶催化下与镁离子螯合形成镁原卟啉Ⅸ[26]。植物叶色突变体中已鉴定了一些镁螯合酶的突变体,大麦的突变体xantha-g44和xantha-g45是由于编码镁螯合酶D亚基突变造成叶绿素合成受阻,分别产生黄叶和黄绿叶表型[5],拟南芥T-DNA插入突变体AtchlD-KO存在白化致死表型,水稻中突变体Chlorina-1和ygl7在D亚基基因的不同位点发生突变,ygl7在整个生育期均表现为黄绿叶,Chlorina-1仅幼苗期的前2~3周出现黄绿叶表型[10,13]。玉米镁螯合酶I亚基突变体Oy1为半显性突变体,表现为黄化致死[4],水稻和拟南芥的I亚基突变体则均表现为黄绿叶[6,10]。拟南芥和水稻镁螯合酶H亚基的突变体分别产生浅绿和黄绿叶表型[2,6]。
本研究中的淡绿叶突变体HM133是EMS诱变籼稻IR64获得,其叶绿素含量明显低于野生型IR64,灌浆期净光合速率明显下降,类囊体发育异常,遗传分析表明HM133淡绿叶性状受单隐性核基因控制。通过分子标记将pglHM133基因定位于第3染色体长臂RM143和RM3684之间,区间内包含编码镁螯合酶D亚基基因OsChlD,序列分析发现HM133中OsChlD基因第10外显子上存在G突变为A的单碱基变异,与已报道了另4个OsChlD等位基因Chl1[10]、ygl98[27]、ygl3[28]和ygl7[13]突变位点均不同,是一个新的OsChlD等位基因。这5个OsChlD突变体的表型不完全相同,可能由于不同的突变蛋白引起,也可能是受到遗传背景的影响。ygl7在日本晴背景下产生黄化叶,而在810S背景下则表现为黄绿叶[13],表明OsChlD控制叶色表型的机理非常复杂,受到遗传背景的影响。Deng等[13]研究表明,ygl7在810S遗传背景下,OsChlD、OsChlI和OsChlH的表达显著上升,在日本晴背景下的ygl7-NIL则与野生型的表达基本相同。本研究发现突变体中OsChlD、OsChlI和OsChlH的表达量较野生型显著下降,该结果与Deng等[13]的结果不同,可能与遗传背景的差异有关,也可能是突变位点的不同引起的。突变体HM133中核基因编码的HEMA1、YGL1、DVR、CAO1和叶绿体编码基因PsaA、PsbA、rbcL的表达量呈显著下降,而核基因编码的Cab1R和Cab2R的表达呈显著上升,表明OsChlD基因突变同样影响了其他叶绿素合成酶基因和叶绿体发育相关基因的表达。Deng等[13]研究发现OsChlD突变蛋白ygl7,能行使部分OsChlD功能,能促进光合作用和光能的高效利用,结实率等性状与野生型无明显差异。而本研究中突变体的净光合速率显著降低,株高、结实率和每穗实粒数也显著低于野生型,可能是由于OsChlD突变位点不同造成相应突变蛋白功能差异。另外,豌豆中ChlD亚基基因沉默植株中发现活性氧物质的积累[29],水稻中D亚基突变体是否存在同样的现象仍有待进一步研究。
[1] 李智强, 朱丹, 王志龙, 等. 水稻黄绿叶突变体djyg的遗传分析与基因定位. 中国水稻科学, 2015, 29: 601-609.
Li Z Q, Zhu D, Wang Z L,et al. Genetic analysis and gene mapping of a yellow-green leaf mutantdjygin rice.ChinJRiceSci, 2015, 29: 601-609. (in Chinese with English abstract)
[2] Jung K H, Hur J, Ryu C H, et al. Characterization of a rice chlorophyll-deficient mutant using the T-DNA gene-trap system.PlantCellPhysiol, 2003, 44: 463-472.
[3] 韩帅, 王立静, 钟世宜, 等. 一个新的玉米叶色突变体的遗传分析及基因定位. 玉米科学, 2012, 20: 26-28.
Han S, Wang L J,Zhong S Y, et al. Genetic analysis and gene mapping of a new leaf color mutant in maize.JMaizeSci, 2012, 20: 26-28. (in Chinese with English abstract)
[4] Sawers R J, Viney J, Farmer P R, et al. The maizeOilyellow1 (Oy1) gene encodes the I subunit of magnesium chelatase.PlantMolBiol, 2006, 60: 95-106.
[5] Axelsson E, Lundqvist J, Sawicki A, et al. Recessiveness and dominance in barley mutants deficient in Mg-chelatase subunit D, an AAA protein involved in chlorophyll biosynthesis.PlantCell, 2006, 18: 3606-3616.
[6] Mochizuki N,Brusslan J A, Larkin R, et al.Arabidopsisgenomesuncoupled5 (GUN5) mutant reveals the involvement of Mg-chelatase H subunit in plastid-to-nucleus signal transduction.ProcNatlAcadSci, 2001, 98: 2053-2058.
[7] 肖华贵, 杨焕文, 饶勇, 等. 甘蓝型油菜黄化突变体的叶绿体超微结构、气孔特征参数及光合特性. 中国农业科学, 2013, 46: 715-727.
Xiao H G, Yang H W, Rao Y,et al. Analysis of chloroplast ultrastructure, stomatal characteristic parameters and photosynthetic characteristics of chlorophyll-reduced mutant inBrassicanapusL.SciAgricSin, 2013, 46: 715-727. (in Chinese with English abstract)
[8] Beale S I. Green genes gleaned.TrendsPlantSci, 2005, 10: 309-312.
[9] Lee S, Kim J H, Yoo E S, et al. Differential regulation of chlorophyll a oxygenase genes in rice.PlantMolBiol, 2005, 57: 805-818.
[10] Zhang H, Li J, Yoo J H, et al. RiceChlorina-1 andChlorina-9 encode ChlD and ChlI subunits of Mg-chelatase, a key enzyme for chlorophyll synthesis and chloroplast development.PlantMolBiol, 2006, 62: 325-337.
[11] Wu Z, Zhang X, He B, et al. A chlorophyll-deficient rice mutant with impaired chlorophyllide esterification in chlorophyll biosynthesis.PlantPhysiol, 2007, 145: 29-40.
[12] Wang P, Gao J, Wan C, et al. Divinyl chlorophyll(ide) a can be converted to monovinyl chlorophyll(ide) a by a divinyl reductase in rice.PlantPhysiol, 2010, 153: 994-1003.
[13] Deng X J, Zhang H Q, Wang Y, et al. Mapped clone and functional analysis of leaf-color geneYgl7 in a rice hybrid (OryzasativaL. ssp.indica).PLoSOne, 2014, 9(6): e99564.
[14]Sakuraba Y, Rahman M L, Cho S H, et al. The rice faded green leaf locus encodes protochlorophyllide oxidoreductase B and is essential for chlorophyll synthesis under high light conditions.PlantJ, 2013, 74: 122-133.
[15] Li Q Z, Zhu F Y, Gao X, et al. Young Leaf Chlorosis 2 encodes the stroma-localized heme oxygenase 2 which is required for normal tetrapyrrole biosynthesis in rice.Planta, 2014, 240: 701-712.
[16] Chen H, Cheng Z, Ma X, et al. A knockdown mutation ofYELLOW-GREENLEAF2 blocks chlorophyll biosynthesis in rice.PlantCellRep, 2013, 32: 1855-1867.
[17] Li J, Pandeya D, Nath K, et al.ZEBRA-NECROSIS, a thylakoid-bound protein, is critical for the photoprotection of developing chloroplasts during early leaf development.PlantJ, 2010, 62: 713-725.
[18] Su N, Hu M L, Wu D X, et al. Disruption of a rice pentatricopeptide repeat protein causes a seedling-specific albino phenotype and its utilization to enhance seed purity in hybrid rice production.PlantPhysiol, 2012, 159: 227-238.
[19] Lv X G, Shi Y F, Xu X, et al.Oryzasativachloroplast signal recognition particle 43 (OscpSRP43) is required for chloroplast development and photosynthesis.PLoSONE, 2015, 10(11): e143249.
[20] Zhang F, Luo X, Hu B, et al.YGL138(t), encoding a putative signal recognition particle 54 kDa protein, is involved in chloroplast development of rice.Rice, 2013, 6(1): 7.
[21]Wellburn A R. The spectral determination of chlorophylls a and b, as well as total carotenoids, using various solvents with spectrophotometers of different resolution.JPlantPhysiol, 1994, 144: 307-313.
[22]Arnon D I. Copper enzymes in isolated chloroplasts. Polyphenoloxidase inBetavulgaris.PlantPhysiol, 1949, 24: 1-15.
[23]Huang Q N, Shi Y F, Zhang X B, et al. Single base substitution inOsCDC48 is responsible for premature senescence and death phenotype in rice.JIntegrPlantBiol, 2016, 58: 12-28.
[24]卢扬江, 郑康乐. 提取水稻DNA的一种简易方法. 中国水稻科学, 1992, 6(1): 47-48.
Lu Y J, Zheng K L. A simple method for isolation of rice DNA.ChinJRiceSci, 1992, 6(1): 47-48.(in Chinese with English abstract)
[25]Shi Y F, Chen J, Liu W Q, et al. Genetic analysis and gene mapping of a new rolled-leaf mutant in rice (OryzasativaL.).SciChinaCLifeSci, 2009, 52: 885-890.
[26]Masuda T. Recent overview of the Mg branch of the tetrapyrrole biosynthesis leading to chlorophylls.PhotosynthRes, 2008, 96: 121-143.
[27]孙小秋, 王兵, 肖云华, 等. 水稻ygl98黄绿叶突变基因的精细定位与遗传分析. 作物学报, 2011, 37(6): 991-997.
Sun X Q, Wang B, Xiao Y H,et al. Genetic analysis and fine-mapping ofygl98 yellow-green leaf gene in rice.ActaAgronSin, 2011, 37(6): 991-997. (in Chinese with English abstract).
[28]Tian X Q, Ling Y H, Fang L K, et al. Gene cloning and functional analysis of yellow green leaf3 (ygl3) gene during the whole-plant growth stage in rice.Genes&Genom, 2013, 35: 87-93.
[29]Luo T, Luo S, Araujo W L, et al. Virus-induced gene silencing of pea CHLI and CHLD affects tetrapyrrole biosynthesis, chloroplast development and the primary metabolic network.PlantPhysiolBiochem, 2013, 65: 17-26.
Genetic Analysis and Gene Mapping of a Pale Green Leaf MutantHM133 in Rice
SHI Yong-feng1,#, HE Yan1,#, GUO Dan1, LV Xiang-guang1,2, HUANG Qi-na1, WU Jian-li1,*
(1State Key Laboratory of Rice Biology, China National Rice Research Institute, Hangzhou 310006, China;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China;#These authors contributed equally to this work;*Corresponding author, E-mail: beshangd@163.com)
The pale green leaf mutantHM133 was identified from an EMS-induced IR64 mutant bank. The contents of photosynthetic pigments including chlorophyll and carotenoid ofHM133 were reduced significantly at 6 weeks and 15 weeks after sowing when compared with IR64.The net photosynthetic rate ofHM133 was considerably lower than that of the wild-type IR64 at heading stage while the stomatal conductance was apparently increased. The agronomic traits including plant height, number of filled grain per panicle and seed-setting rate decreased significantly in the mutant compared with the wild-type. In addition, the mutant exhibited a less number of grana, irregular arrangement of thylakoid layer in the chloroplast at the tillering stage. Genetic and mapping analysis showed that the pale green phenotype was controlled by a single recessive gene located in the long arm of chromosome 3 between SSR markers RM143 and RM3684. The interval contains an ORFOsChlDencoding magnesium-chelatase D subunit. Sequence analysis revealed that the mutant allele carried a nucleotide substitution from G to A in the tenth exon ofOsChlD, which led to the substitution of glutamic acid for arginine acid. Therefore, it is deduced thatOsChlDis the candidate gene controlling the pale green leaf phenotype ofHM133.
Oryzasativa; pale green leaf; gene mapping; magnesium-chelatase; photosynthesis
2015-12-29; 修改稿收到日期: 2016-03-12。
国家863计划资助项目(2014AA10A603); 浙江省超级稻研究重点实验室项目(2013E10021)。
Q343.5; S511.032
A
1001-7216(2016)06-0603-08
施勇烽, 贺彦, 郭丹, 等. 水稻淡绿叶突变体HM133的遗传分析与基因定位. 中国水稻科学, 2016, 30(6): 603-610.
