细粒棘球蚴AgB1、AgB2、AgB4抗原表位的特性分析
2016-12-06安梦婷吾拉木马木提马海梅张峰波王红英丁剑冰
安梦婷, 吾拉木·马木提,, 马海梅,, 张峰波, 王红英, 赵 慧, 丁剑冰
(新疆医科大学1基础医学院, 乌鲁木齐 830011; 2省部共建国家重点实验室培育基地—新疆重大疾病医学重点实验室;
·基础医学研究·
细粒棘球蚴AgB1、AgB2、AgB4抗原表位的特性分析
安梦婷1, 吾拉木·马木提1,2, 马海梅1,2, 张峰波2,3, 王红英2, 赵 慧2,3, 丁剑冰2
(新疆医科大学1基础医学院, 乌鲁木齐 830011;2省部共建国家重点实验室培育基地—新疆重大疾病医学重点实验室;
3第一附属医院检验科, 乌鲁木齐 830054)
目的 分析新疆细粒棘球蚴AgB1、AgB2、AgB4蛋白的氨基酸序列,了解其蛋白二级结构特点,预测抗原表位,为包虫病的进一步免疫学诊断和研究提供理论支持。方法 利用生物信息分析软件DNAstar分析3种抗原的蛋白二级结构特点,运用在线软件IEDB对3种抗原各参数进行综合分析并预测其抗原表位。结果 EgAgB1、EgAgB2和EgAgB4分别是由78、90、91个氨基酸残基组成的不同多肽。其蛋白质二级结构中均有α-螺旋、β-折叠、转角区域和卷曲区域。综合分析各参数推测EgAgB1具有2个抗原表位,分别是14~23位氨基酸残基(DDGLTSTSRS)、37~42位氨基酸残基(RDPLGQ)。推测EgAgB2的2个抗原表位分别为41~49位氨基酸残基(DFFRNDPLG)、83~90位氨基酸残基(EEKDDDSk)。EgAgB4可能含有的2个抗原表位分别是43~50位氨基酸残基(RSDPLGQR)、84~89位氨基酸残基(EEEDDS)。结论 采用生物信息技术对新疆细粒棘球蚴AgB1、AgB2、AgB4进行B细胞表位特点的研究和分析,对更准确诊断包虫病具有重要意义。
细粒棘球蚴; AgB; 生物信息技术; B细胞表位
细粒棘球绦虫(Echinococcus granulosus Batsch 1786)是属带科、棘球属。成虫主要寄生于犬科食肉动物,而幼虫(棘球蚴)主要寄生于人和多种食草类家畜等动物,能够引起一种严重的人兽共患病[1-2],称棘球蚴病或包虫病(echinococcosis, hydatid disease, hydatidosis)。人类囊型包虫病已成为至关重要的公共健康问题[3-5]。囊型包虫病通常起病隐匿,几年内可以毫无症状。另外,目前还未出现有效预防该病的疫苗[6]。传统方法治疗囊型包虫病局限于手术切除囊肿,然而这种方法对于多器官囊肿或者脑组织脊髓等风险较大的部位是不切实际的[7]。
我国是棘球蚴病流行最严重的国家之一,主要存在于我国西部和北部广大农牧区,包括新疆、宁夏、青海、四川、甘肃、西藏和内蒙7个省区。据几个重点流行省区的不完全统计,全国受棘球蚴病威胁的人口总数约5 000万,患病人数为50~60万,而人群中最易受到感染的是学龄前儿童[8]。主要中间宿主是绵羊,其感染率为3.3%~90%,而家犬的感染率为7%~71%[8]。
EgAgB抗原存在于细粒棘球蚴囊液中,具有高度的抗原性和免疫原性[9],因此被广泛研究。研究显示,EgAgB是由多态多基因家族编码,由EgAgB1~EgAgB5 共5个亚基构成[10]。有研究通过调查不同地区细粒棘球蚴的特点,提出EgAgB至少有10种独特的基因,其中有3~4种不同的基因与EgAgB3和EgAgb4有关[11]。AgB 是一个组成复杂的抗原蛋白家族,如果使用单一来源或者单一组分抗原对疾病进行诊断,其敏感性和特异性都不稳定,甚至差异也较大[12]。EgAgB 在寄生虫中的作用机制还不明确,其可能在细粒棘球蚴脂质新陈代谢中起到一定作用。但是其在免疫调节活动中特别是固有免疫起到重要作用[13]。因此,本研究旨在通过研究EgAgB1、EgAgB2和EgAgB4抗原表位,为包虫病的免疫学诊断提供更进一步的理论支持。
1 材料与方法
1.1 EgAgB1、EgAgB2及EgAgB4抗原氨基酸序列 根据NCBI GenBank EgAgB1 的AAV52691、EgAgB2的ACC85503.1、EgAgB3的AAS88254.1进行抗原分析及表位预测。
1.2 EgAgB1、EgAgB2及EgAgB4抗原的二级结构预测对比分析 运用DNAstar软件包在计算机上进行蛋白质分析,将从GenBank中已经获得的EgAgB1、EgAgB2及EgAgB4氨基酸序列导入Protean软件,对其蛋白的二级结构和抗原表位进行分析预测,通过Garnier-Robson方法和Chou-Fasman方法分别分析EgAgB1、EgAgB2及EgAgB4抗原的α-螺旋,β-折叠,转角区域和卷曲区域。
1.3 EgAgB1、EgAgB2及EgAgB4抗原B细胞表位分析预测 使用IEDB(Immune Epitope Data. base, http://tools. Immuneepitope. Org/main/html/tcell_tools. Html)在线分析软件,将从GenBank中已经获得的EgAgB1、EgAgB2及EgAgB4氨基酸序列输入IEDB软件中,并分别运用Emini方法预测抗原表面可及性,Karplus-Schulz方法预测其骨架柔韧性,并综合分析亲水性,抗原性及线性表位等多种参数对其进行B细胞表位的分析预测,并预测其可能的B细胞表位区域。
2 结果
2.1 EgAgB1、EgAgB2及EgAgB4抗原Blast分析结果 测序后发现,EgAgB1、EgAgB2和EgAgB4分别是由78、90、91个氨基酸残基组成的多肽。
2.2 EgAgB1、EgAgB2及EgAgB4抗原二级结构预测分析 运用DNAstar软件,通过Garnier-Robson方法和Chou-Fasman综合方法分析EgAgB1、EgAgB2及EgAgB4抗原二级结构区域见图1和表1。
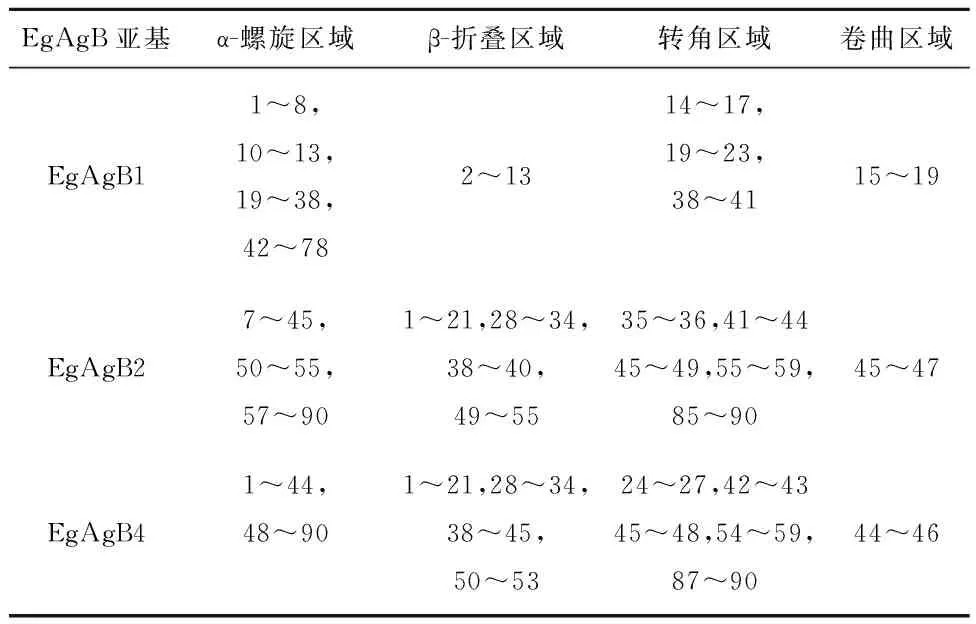
表1 EgAgB1、EgAgB2、EgAgB4抗原二级结构区域

图1 EgAgB1、EgAgB2和EgAgB4抗原的二级结构
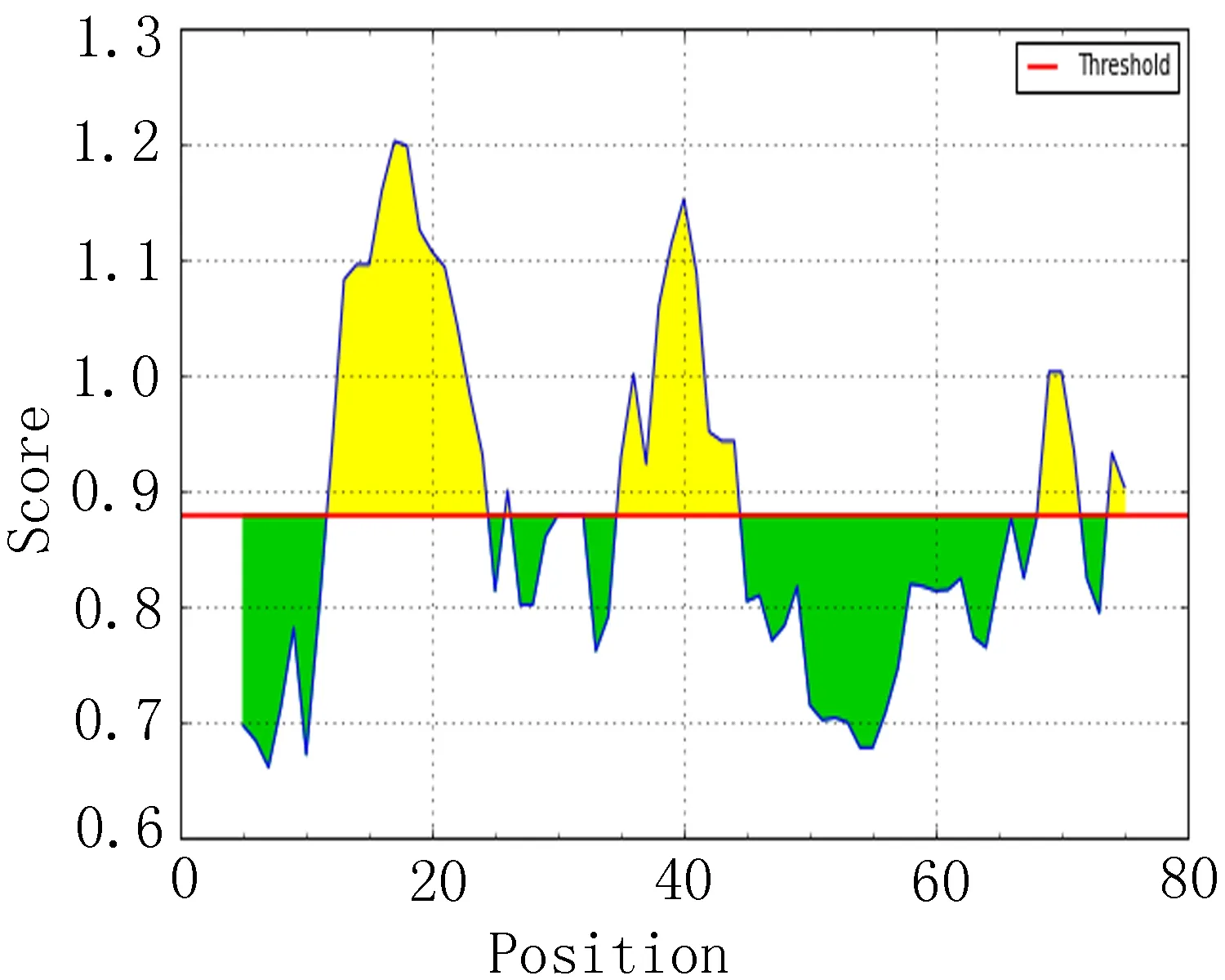
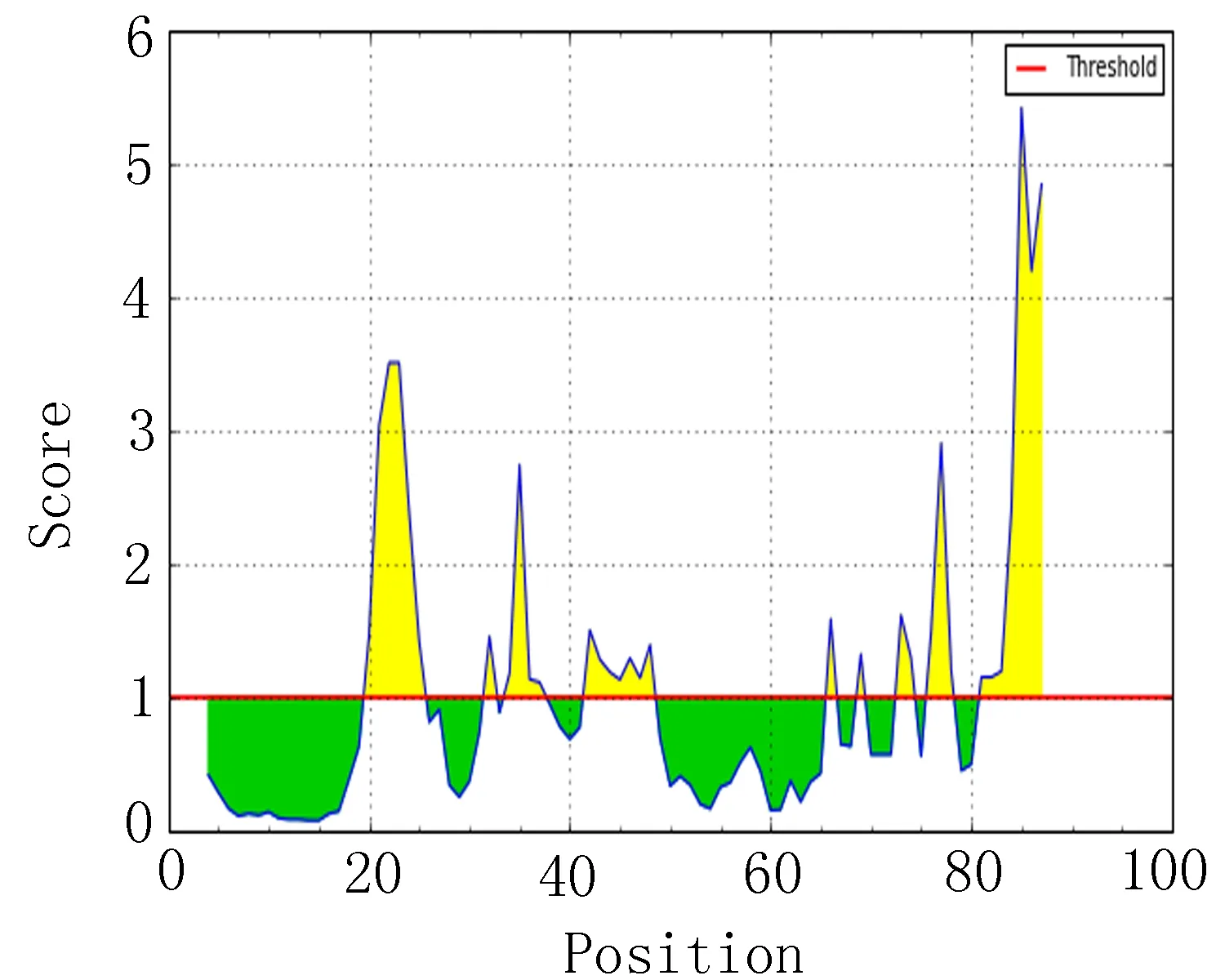
2.3 转角区域的分析比较 经IEDB软件分析转角区域(图2),分别列出EgAgB1、EgAgB2和EgAgB4转角区域指数较高的20个区段(表2),比较三者转角区域出现位置,发现 EgAgB2和EgAgB4 转角区域区段有较多相似性,而EgAgB1转角区域主要出现在12~24、35~44、67~72区段附近。
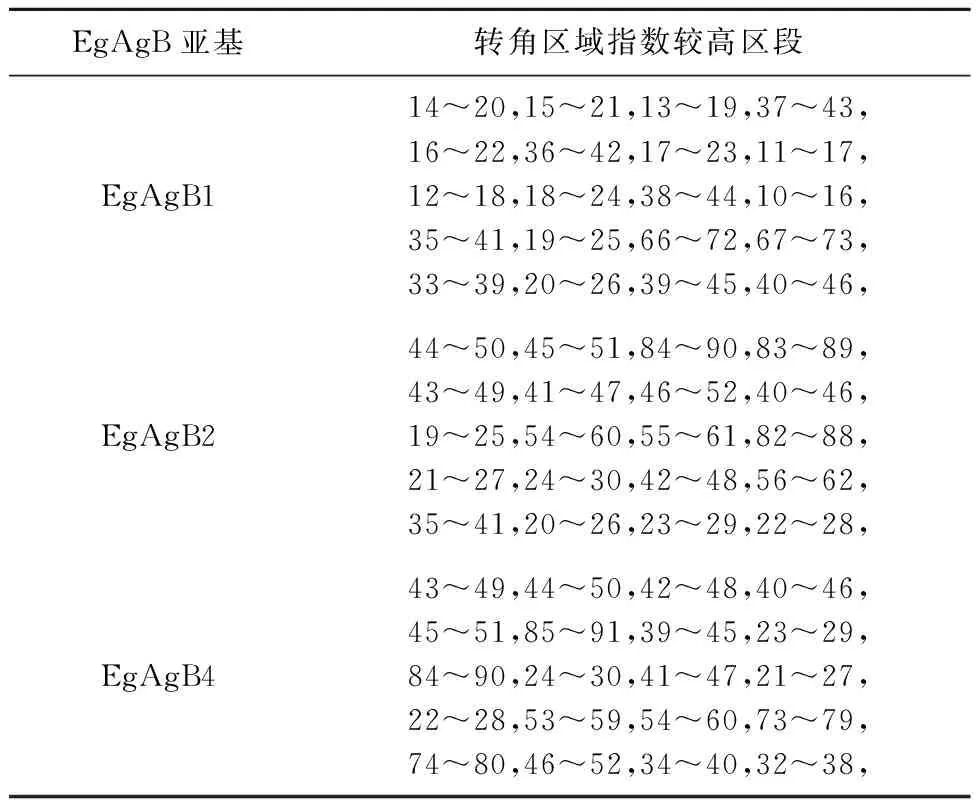
表2 EgAgB1、EgAgB2和EgAgB4转角区域比较
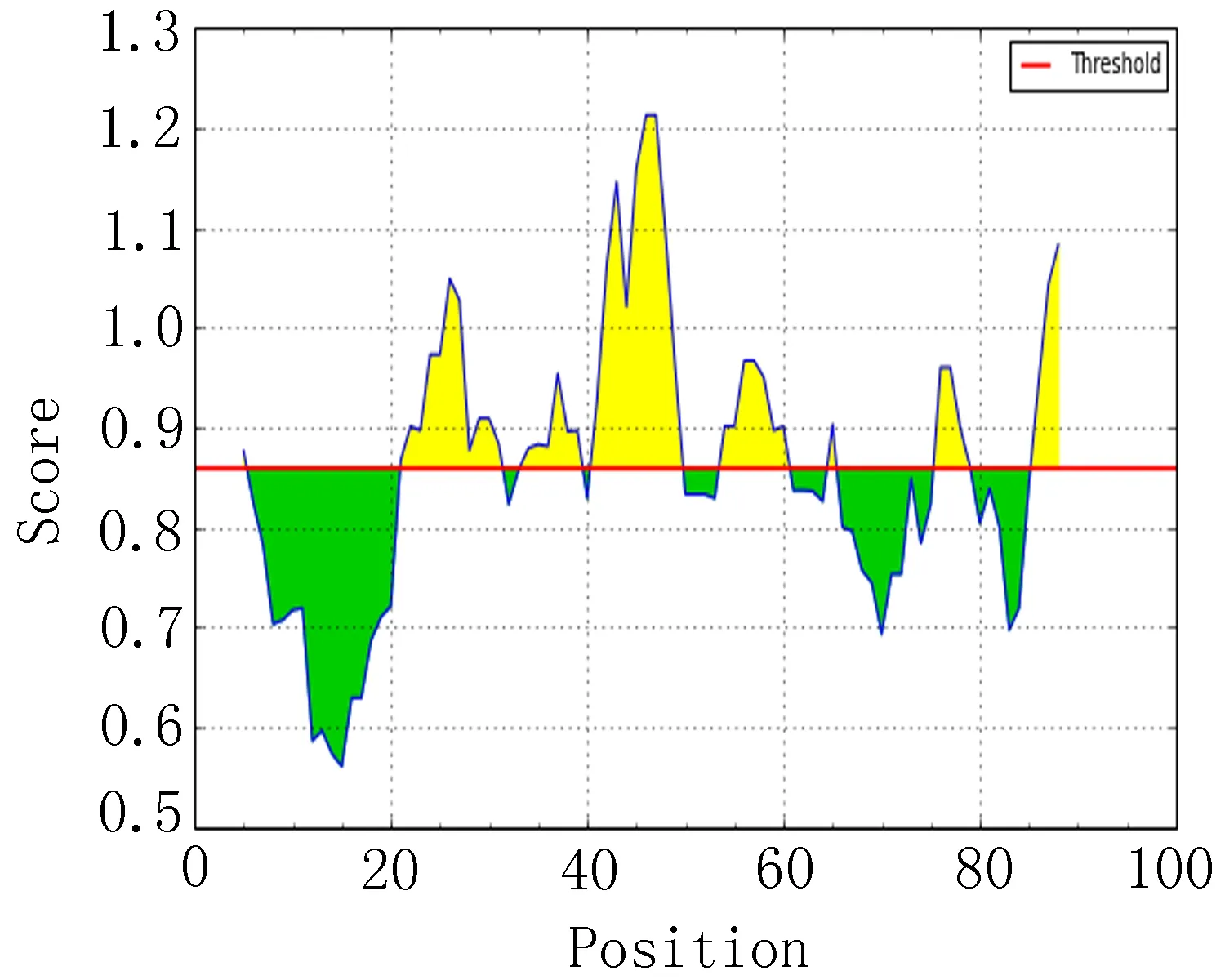
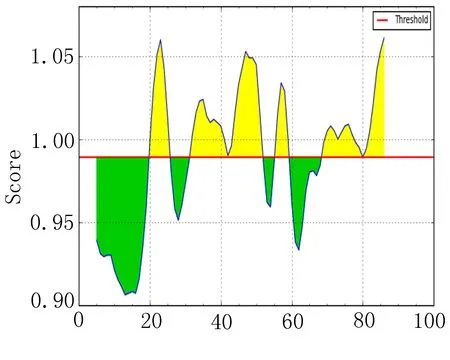
2.4 表面可及性分析比较 分析比较EgAgB1、EgAgB2和EgAgB4表面可及性较高的前20个区段(表3),并结合图3,EgAgB2和EgAgB4表面可及性较高区段较为相似,EgAgB2表面可及性指数较高可能出现在83~88、20~25和33~38区段附近。EgAgBg4主要是82~87、21~26和32~37区段。 EgAgB1表面可及性较高区域主要集中在59~63、18~23、32~37区段附近。

表3 EgAgB1、EgAgB2和EgAgB4表面可及性区段比较
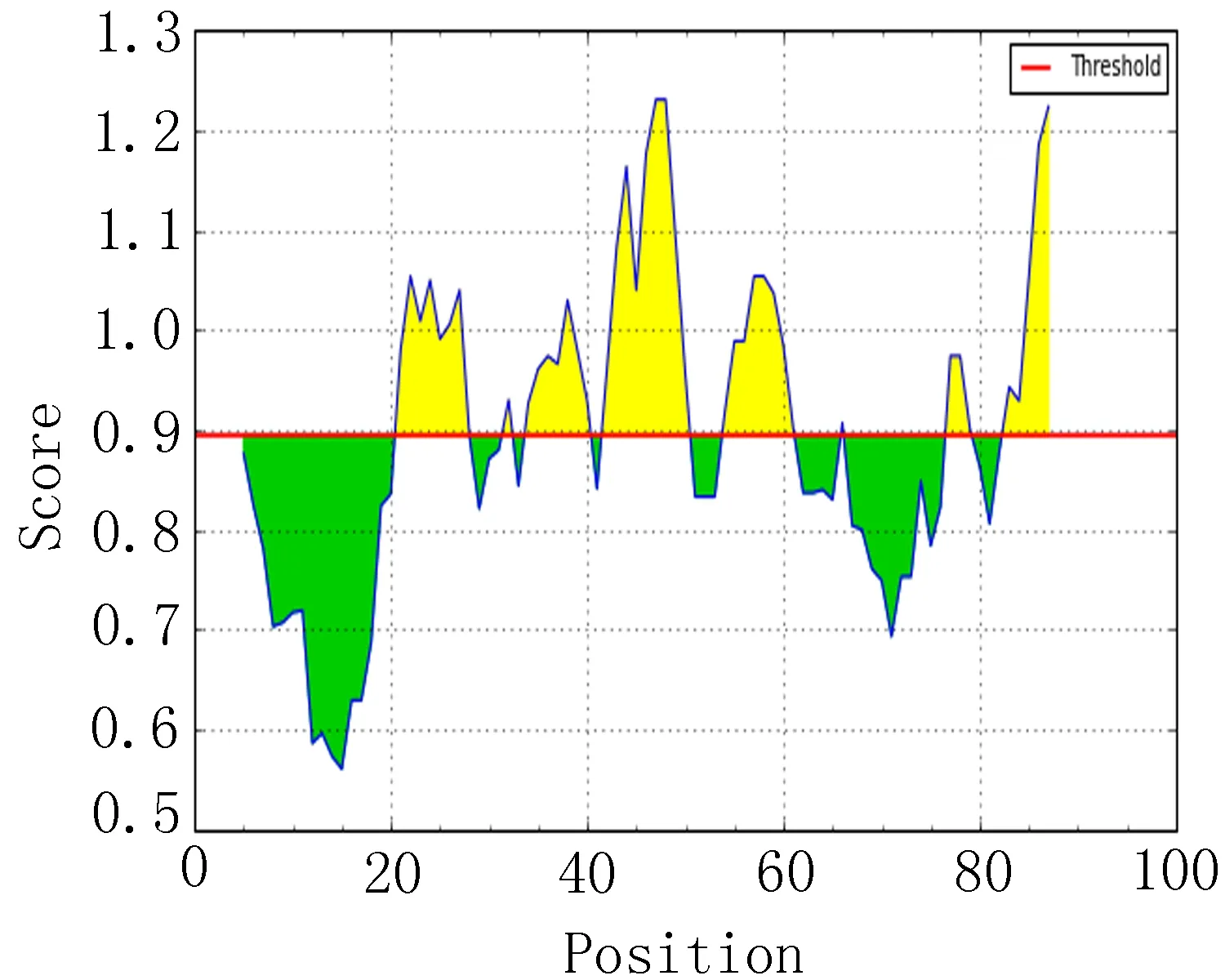
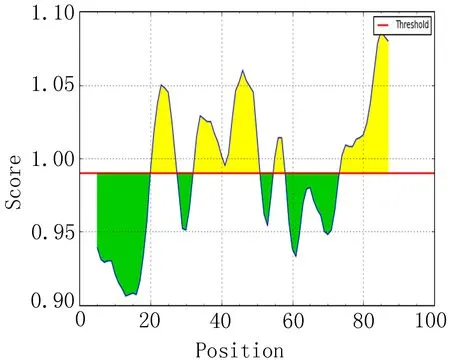
2.5 骨架柔韧性区段分析比较 分别获得EgAgB1、EgAgB2和EgAgB4骨架柔韧性指数较高的前20个区段(表4),结合图4,比较发现EgAgB1骨架柔韧性指数较高区段主要出现在17~23、38~44和11~17区段附近。EgAgB2主要在83~89、20~26和44~50区段附近。EgAgB4的骨架柔韧性指数较高主要位于82~88、43~49和20~26区段附近。
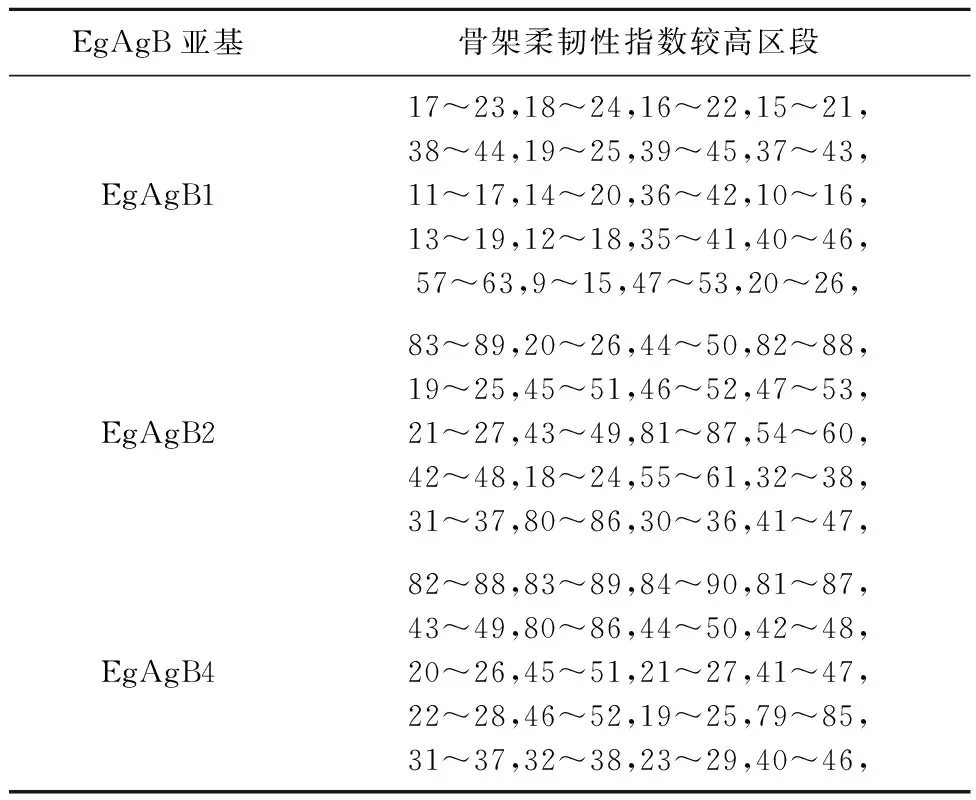
表4 EgAgB1、EgAgB2和EgAgB4柔韧性区段比较
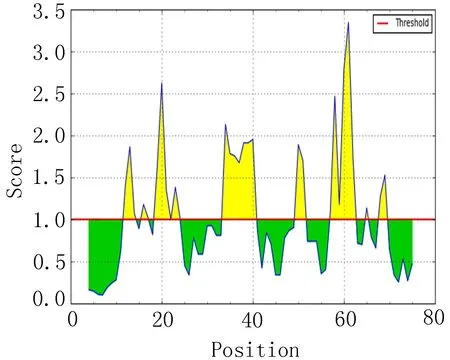
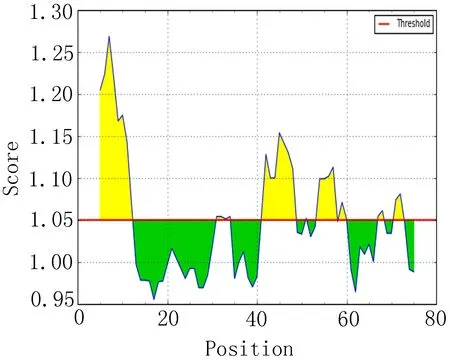
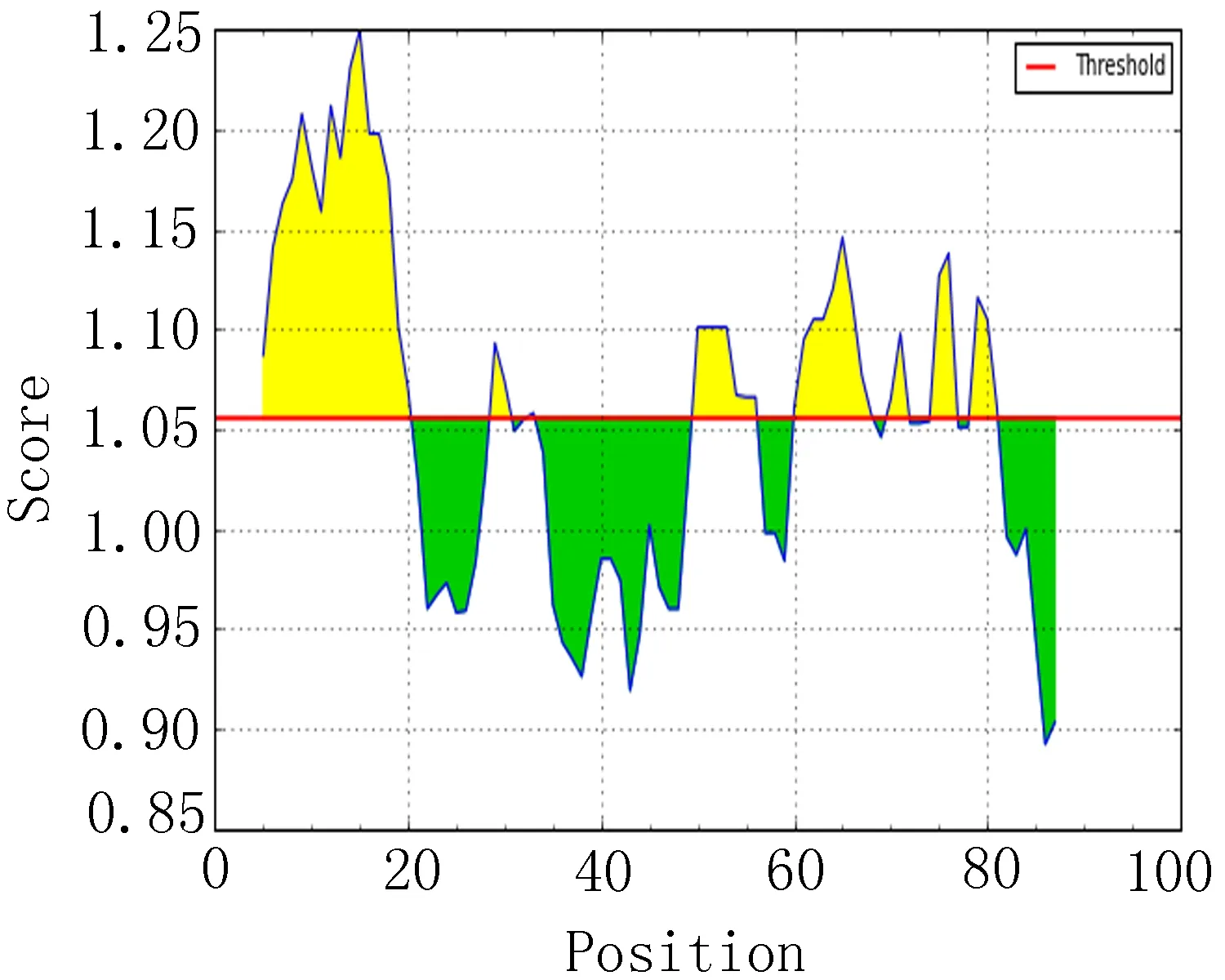
2.6 抗原区段的比较分析 分析比较EgAgB1、EgAgB2和EgAgB4抗原性较高的前20个区段(表5),结合图5,提示EgAgB1抗原性较高的区段主要集中在4~10、42~48和54~60区段。EgAgB2抗原性较高主要集中在12~18、62~68和73~79区段附近。EgAgB4抗原性较高区段在12~18和6~12附近。
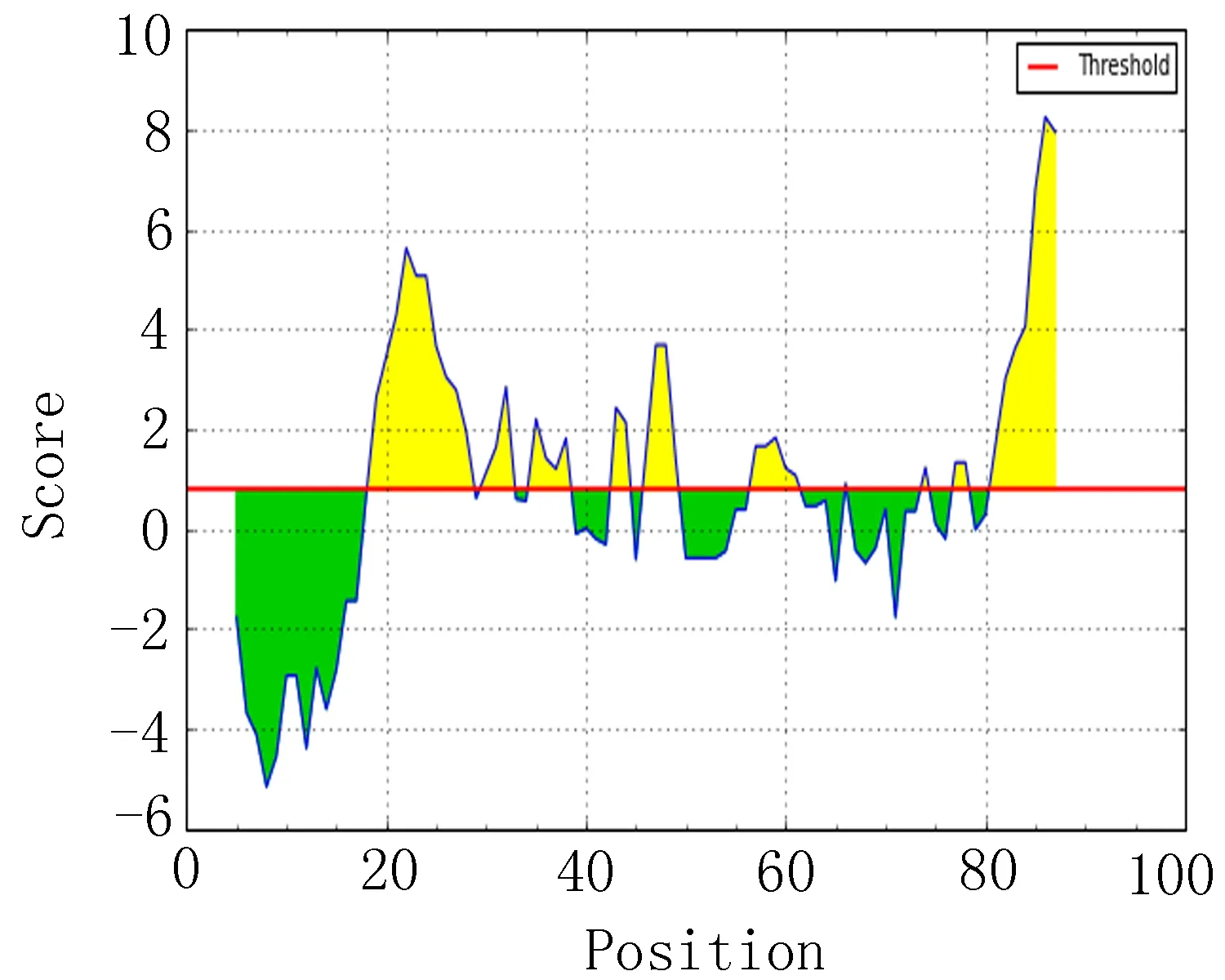
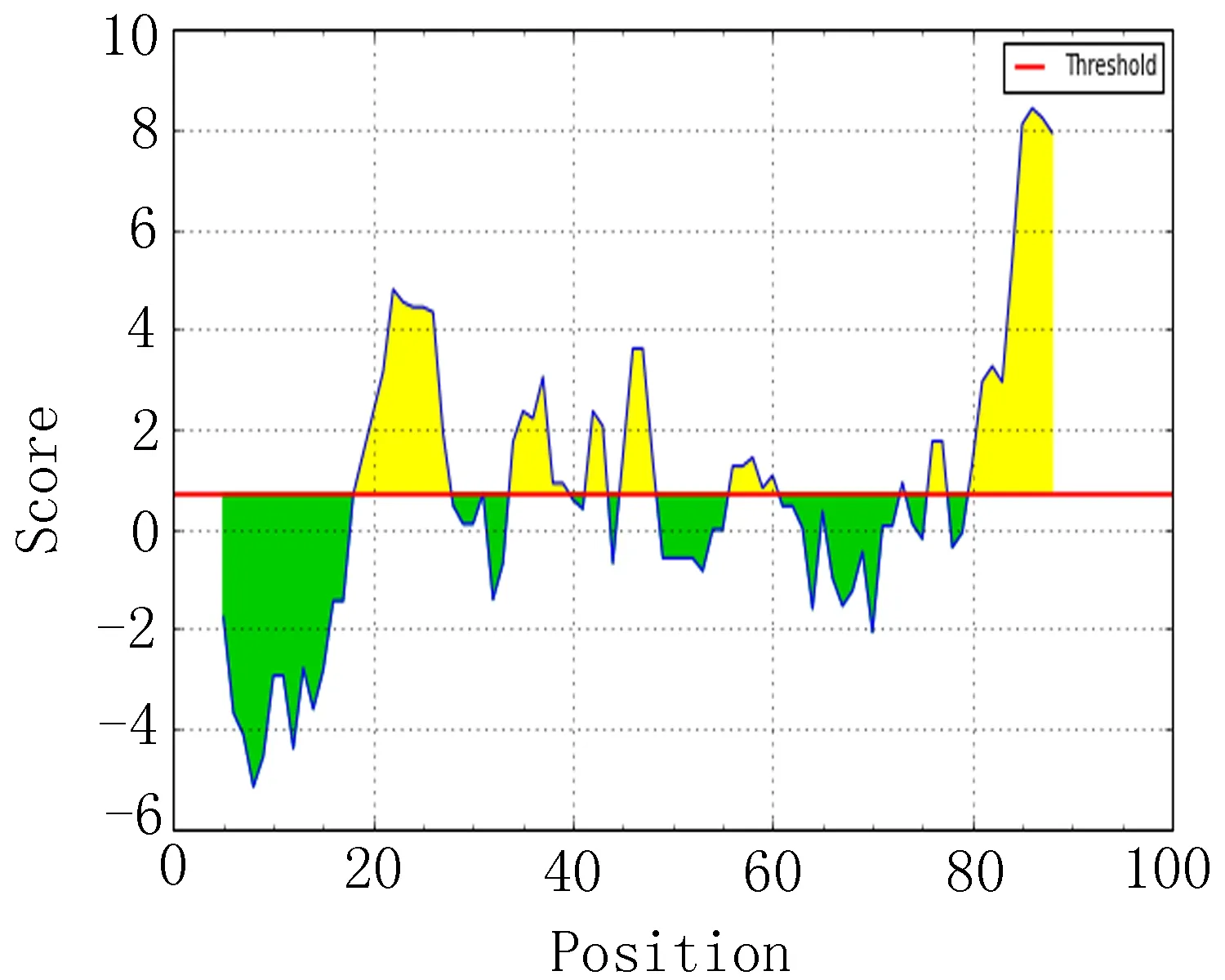
2.7 亲水性区段比较 分析EgAgB1、EgAgB2和EgAgB4亲水性指数较高的前20个区段(表6),结合图6,提示EgAgB1亲水性较高的区段主要集中在10~16、36~42和18~24附近。EgAgB2亲水性较高主要在83~89、19~25和44~50区段附近。EgAgB5的亲水性较高区段位于83~89、19~25和43~49附近。

表5 EgAgB1、EgAgB2和EgAgB4抗原区段比较
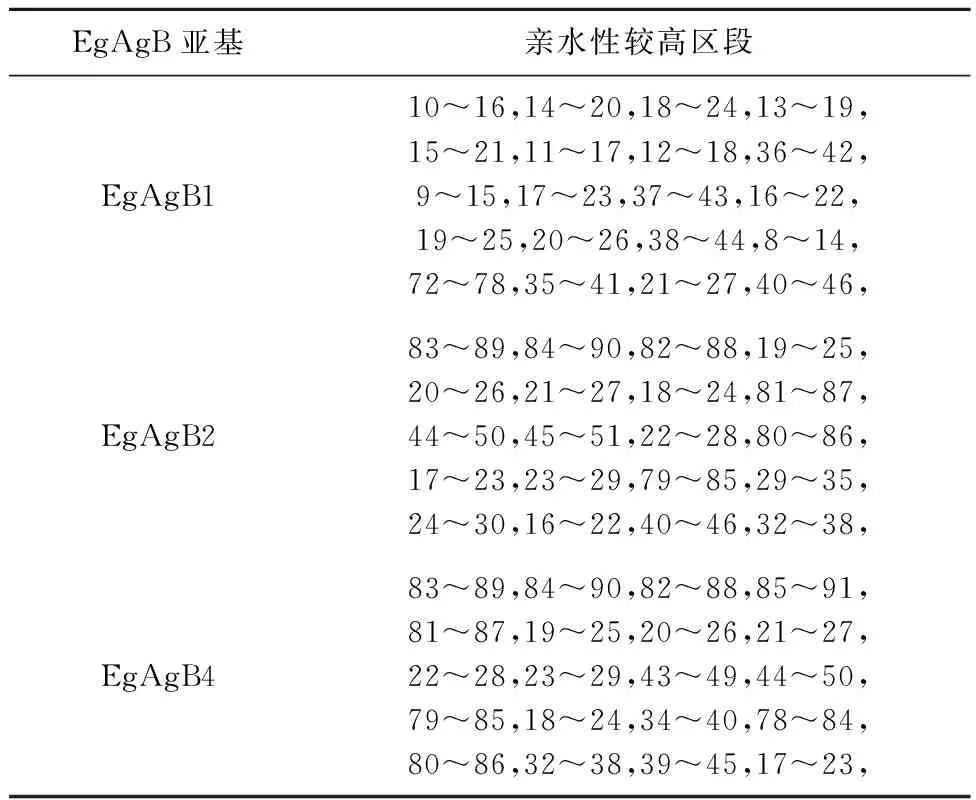
表6 EgAgB1、EgAgB2和EgAgB4亲水性区段比较
a: EgAgB1 b: EgAgB2 C: EgAgB4
图2 转角区域指数分析
2.8 B细胞抗原表位预测 综合分析EgAgB1、EgAgB2及EgAgB4的转角区域指数、表面可及性、骨架柔韧性指数、抗原性指数和亲水性指数,并结合DNAstar Protean软件对其蛋白质二级结构的分析,分别预测其可能的B细胞表位区域,综合分析各参数推测EgAgB1具有2个抗原表位,分别为14~23(DDGLTSTSRS)、37~42(RDPLGQ)。推测EgAgB2的2个抗原表位分别为41~49(DFFRNDPLG)、83~90(EEKDDDSk)。EgAgB4可能含有的2个抗原表位分别为43~50(RSDPLGQR)、84~89(EEEDDS)。
a: EgAgB1 b: EgAgB2 C: EgAgB4
图3 表面可及性指数分析
a: EgAgB1 b: EgAgB2 C: EgAgB4
图4 骨架柔韧性指数分析
a: EgAgB1 b: EgAgB2 C: EgAgB4
图5 抗原指数分析
a: EgAgB1 b: EgAgB2 C: EgAgB4
图6 新水性指数分析
3 讨论
有证据显示,EgAgB在同一虫体生命周期的不同发育阶段,不同组织基因表达水平也都明显不同。EgAgB1~EgAgB4主要在幼虫阶段表达,而EgAgB5表达于成虫阶段。并且,在幼虫阶段EgAgB1、EgAgB2、EgAgB4高表达于生发层,在其他发育阶段几乎不表达[11]。而EgAgB3在虫体所有阶段均有表达,并且表达水平在发育阶段呈现逐渐升高的趋势。比较EgAgB不同亚基的氨基酸序列,发现EgAgB1、EgAgB3、EgAgB5的氨基酸序列相似性较高[11]。Obal等[14]的研究显示,在提纯的EgAgB抗原中,EgAgB1的含量是最高的,而EgAgB4的含量较少,提示EgAgB2不是唯一含量最高的脂蛋白。Monteiro等[15]通过提取来自人和牛体内的寄生虫样本研究EgAgB抗原。结果也显示EgAgB1、EgAgB2、EgAgB3、EgAgB4在各样本中均有表达。张海涛等[16]研究发现EgAgB1在细粒棘球蚴的生发层大量表达。Jiang等[17]对EgAgB抗原的5个亚单位的血清反应性进行了分析,认为EgAgB1、 EgAgB2和EgAgB4是识别血清抗体的主要反应性亚单位。
运用生物信息学技术方法对蛋白质相关信息进行分析处理比较,并且预测其抗原表位,已经成为免疫学研究的重要手段之一。本研究采用DNAstar 软件和IEDB在线软件对EgAgB1、EgAgB2及EgAgB4蛋白质二级结构及B细胞表位特点进行了分析和预测。本研究发现,在蛋白质二级结构的分析中,EgAgB1、EgAgB2及EgAgB4抗原的α-螺旋的比例均较高,而EgAgB2和EgAgB4抗原的转角区域较EgAgB1比例稍高,提示EgAgB2和EgAgB4形成抗原表位的可能性会稍大一点。运用IEDB软件对蛋白质转角区域指数、表面可及性、骨架柔韧性指数、抗原性指数和亲水性指数进行分析,并分别选出20个指数较高的区段。亲水性指数越高暴露于表面的几率越大,成为抗原表位的可能性越大。表面可及性越大,蛋白质抗原中氨基酸残基被溶剂分子接触的可能性就越大。而蛋白质骨架柔韧性越高,发生扭曲折叠的几率就越大。抗原性好的肽段,可能作为抗原表位。综合分析各参数推测EgAgB1具有2个抗原表位:14~23(DDGLTSTSRS)和37~42(RDPLGQ)。推测EgAgB2的2个抗原表位分别为41~49氨基酸残基(DFFRNDPLG),83~90氨基酸残基(EEKDDDSk)。EgAgB4可能含有的2个抗原表位分别为43~50氨基酸残基(RSDPLGQR)、84~89氨基酸残基(EEEDDS)。从该结论中可以看出EgAgB2和EgAgB4的预测表位位置极为相近,与EgAgB1几乎不存在相似关系。计算出EgAgB2和EgAgB4相对应的氨基酸序列的相似度,得出2个抗原表位的相似度分别为57%和67%。有相关研究也表明EgAgB2和EgAgB4的氨基酸序列具有70%同源性[18],猜测EgAgB2和EgAgB4可能部分共同的抗原表位。
总之,通过对EgAgB1、EgAgB2及EgAgB4蛋白质二级结构及B细胞表位特点的分析和预测,根据蛋白质和RNA数据,推测EgAgB1、EgAgB2和EgAgB4在宿主体内细粒棘球蚴慢性感染中可能具有特殊且重要的作用,为进一步诊断包虫病提供理论依据。
[1] 赵慧,张峰波,庞楠楠. Tim-3/Galectin-9与Th1/Th2相关因子在人细粒棘球蚴感染中的表达研究[J]. 新疆医科大学学报, 2016, 39(3): 313-319.
[2] Cardona GA, Carmena D. A review of the global prevalence, molecular epidemiology and economics of cystic echinococcosis in production animals[J]. Vet Parasitol, 2013 , 192(1-3): 10-32.
[3] Ahmed ME, Eldigail MH, Elamin FM, et al. Development and evaluation of real-time loop-mediated isothermal amplification assay for rapid detection of cystic echinococcosis[J]. BMC Vet Res, 2016,12:202.
[4] Alvarez Rojas CA, Romig T, Lightowlers MW. Echinococcus granulosus sensu lato genotypes infecting humans--review of current knowledge[J]. Int J Parasitol, 2014,44(1): 9-18.
[5] Nakao M, Lavikainen A, Yanagida T, et al. Phylogenetic systematics of the genus Echinococcus (Cestoda: Taeniidae)[J]. Int J Parasitol, 2013,43(12-13):1017-1029.
[6] Ma X, Zhao H, Zhang F, et al. Activity in mice of recombinant BCG-EgG1Y162 vaccine for Echinococcus granulosus infection[J]. Hum Vaccin Immunother, 2016,12(1):170-175.
[7] Higuita NIA, Brunetti E, McCloskey C. Cystic echinococcosis[J].J Clin Microbiol ,2016, 54(3):518-523.
[8] 刘春燕,马秀敏,丁剑冰. 我国棘球绦虫感染的不同宿主状况[J].中国人兽共患病学报,2009, 25(6):586-588.
[9] Azizi H, Kazemi B, Bandehpour M, et al. Molecular Cloning and Expression an 8-kDa Subunit of Antigen B from G1 strain of Echinococcus granulosus[J]. Iran J Public Health, 2015 ,44(7):962-968.
[10] Wang H, Zhang F, Ma X, et al. Prokaryotic expression and identification of B- and T-cell combined epitopes of Em95 antigen of Echinococcus multilocularis[J]. Int J Clin Exp Pathol,2014, 7(8):5117-5122.
[11] Zhang W, Li J, Jones MK, er al. The Echinococcus granulosus antigen B gene family comprises at least 10 unique genes in five subclasses which are differentially expressed[J]. PLoS Negl Trop Dis, 2010,4(8):e784.
[12] Zhang F, Ma X, Zhu Y, et al.Identification, expression and phylogenetic analysis of EgG1Y162 from Echinococcus granulosus[J]. Int J Clin Exp Pathol,2014 ,7(9):5655-5664.
[13] Silva-lvarez V, Folle AM, Ramos AL, et al. Echinococcus granulosus Antigen B binds to monocytes andmacrophages modulating cell response to inflammation[J]. Parasit Vectors, 2016,9:69.[14] Obal G, Ramos AL, Silva V, et al. Characterisation of the native lipid moiety of Echinococcus granulosus antigen B[J]. PLoS Negl Trop Dis ,2012,6(5):e1642.
[15] Monteiro KM, Cardoso MB, Follmer C, et al. Echinococcus granulosus antigen B structure: subunit composition and oligomeric states[J]. PLoS Negl Trop Dis, 2012,6(3):e1551.
[16] 张海涛,马秀敏,吾拉木·马木提,等.细粒棘球绦虫抗原EgAgB8/I和EgAgB8/3亚单位基因在虫体发育过程中的阶段性表达[J].中国病原生物学杂志,2009,4(5):355-358.
[17] Jiang L,Zhang YG,Liu MX,et al. Analysis on the reactivity offive subunits of antigen B family in serodiagnosis of echinococcosis[J]. Exp Parasitol,2012,131(1):85-91.
[18] Arend AC, Zaha A, Ayala FJ, et al. The Echinococcus granulosus antigen B shows a high degree of genetic variability[J]. Exp Parasitol, 2004,108(1-2):76-80.
(本文编辑 杨晨晨)
Analysis of epitopes of AgB1, AgB2 and AgB4 epitopes of Echinococcus granulosus
AN Mengting1, Wulamu Mamuti1,2, MA Haimei1,2, ZhANG Fengbo2,3, WANG Hongying2, ZHAO Hui2,3, DING Jianbing2
(1DepartmentofImmunology,BasicMedicalCollege,Urumqi830011,2StateKeyLaboratoryIncubationBaseofXinjiangMajorDiseasesResearch,3DepartmentofClinicalLaboratoryoftheFirstAffiliatedHospital,Urumqi830054,China)
Objective To analyze EgAgB1, EgAgB2 and EgAgB4 amino acid sequence of the recombinant protein to understand their protein secondary structure prediction epitope to provide theoretical support for further immunological diagnosis and research on hydatid disease. Methods Bioinformatics analysis software DNAstar was used to analyze the protein secondary structure features of three antigens, and the online software IEDB was applied for analyze each of the three antigens parameters and predict their epitopes. Results EgAgB1, EgAgB2 and EgAgB4 are polypeptide consisting of 78, 90 and 91 amino acid residues respectively, all which possess α-helix, β-folded corner area and curling area in Protein secondary structure. Comprehensive analysis presumed EgAgB1 parameter contains two epitopes: amino acid fragment of 14-23 (DDGLTSTSRS) and 37-42 (RDPLGQ), EgAgB2 contains two epitopes: amino acid fragment of 41-49 (DFFRNDPLG) and 83-90 (EEKDDDSk), and EgAgB4 contains two epitopes: amino acid fragment of 43~50 (RSDPLGQR) and 84~89 (EEEDDS). Conclusion It is meaningful for more accurate diagnosis of hydatid disease that B cell epitopes feasures of EgAgB1, EgAgB2 and EgAgB4 were analyzed.
Echinococcus granulosus; AgB; bioinformatics; B cell epitope
国家自然科学基金(81160202,81460307,81660343); 新疆维吾尔自治区包虫病重点实验室基金项目(XJDX0202-2010-04)
安梦婷(1992-),女,在读硕士,研究方向:寄生虫免疫。
丁剑冰,女,博士,教授,博士生导师,研究方向: 感染免疫,E-mail:djbing002@sina.com。
R392.1
A
1009-5551(2016)12-1541-07
10.3969/j.issn.1009-5551.2016.12.015
2016-09-28]
