微流控PCR芯片的研究进展
2016-11-19何启迪黄丹萍黄冠陈缵光
何启迪 黄丹萍 黄冠 陈缵光

摘 要 基因是人类的遗传密码,人类个体之间只有万分之一的基因不相同,却导致了人与人之间丰富的差异。了解这种差异对于科学研究具有巨大的应用价值。PCR是基因研究中常用的手段之一,但传统PCR仪存在反应时间长、能量消耗大、不便于集成与携带等缺陷,微流控技术与PCR结合可以有效缩小反应体系,提高反应效率,且易于集成化与微型化。本文按照微流控PCR芯片的结构分类, 详细介绍了微池型、连续流动型PCR芯片,以及电泳、荧光、电化学和DNA杂交阵列等检测方法,并在最后进行了总结与展望。
关键词 微流控; 聚合酶链式反应; 芯片; 综述
1 引 言
2001年2月12日,中、美、日、德、法、英等国科学家和美国赛莱拉公司公布了人类基因组图谱和初步分析结果[1]。这是人类对自身奥秘的探索史上一个重要的里程碑。研究结果显示,人类99.99%的基因是相同的,仅万分之一的不同基因造就了个体间丰富的差异。了解这种差异对于基础生物学和医学等方面研究都具有巨大的应用价值。
在基因研究中,聚合酶链式反应(Polymerase chain reaction,PCR)是一种常用手段。PCR技术最先由Mullis等在1985年发明[2],并获得了1993年的诺贝尔化学奖[3]。PCR的过程包括3个步骤:变性、退火、延伸,需要在高低中3个不同的温度(高温变性:90℃~95℃,低温退火:55℃~60℃;中温延伸:70℃~72℃)下进行。除了反应区域温度的调节,实验中还涉及到反应物、酶、DNA模板溶液体积的精准测量,反应体系的混合,溶液的驱动和控制等。传统的PCR仪器中的反应是通过样品槽反应管样品之间的热传递实现的,存在反应时间长、反应体积大、能量消耗多、易产生副产物、不便于集成与携带等缺陷。
微流控(Microfluidics)是一门在μm~mm尺度下研究流体的处理与操控的技术[4],集成化的微流控芯片还可以将采样、稀释、反应、分离、检测、分析等多个步骤融为一体,已广泛用于化学、生物等各领域。本实验室对于微流控芯片在化合物分离检测[5]、药物筛选[6]等方面的应用均有研究。利用微流控芯片,传统PCR仪存在的各种问题都有了解决的可能。例如:微小的反应体系可以使热传递更迅速,显著提高扩增速度,同时避免非特异性扩增,提高反应效率;全封闭的体系可以减少频繁的手工操作引入的样本污染和溶液损失;系统微型便携,利于现场实时检测等。
2 微流控PCR芯片
用于制作微流控芯片的材料有硅、玻璃、石英、金属和有机聚合物如环氧树脂、聚甲基丙烯酸甲酯(PMMA)、聚碳酸酯(PC)和聚二甲基硅氧烷(PDMS)等[7]。其中,硅和玻璃具有良好的化学惰性和热稳定性,与硅相比,玻璃的机械强度更高,是制作PCR芯片的主要材料。PDMS生物相容性好,可塑性强,并且PDMS表面与很多材料表面都有很好的亲和力,易于实现可逆封装,可以用于制作生化分析器件[8]。但是PDMS的导热系数小,散热性差等特性在某些情况下不利于PCR反应的进行,可以与热传导性能好的玻璃形成混合芯片结构,同时改善PCR芯片的散热和光学性能。
除了利用软光刻技术对微流体通道进行微细精密的加工[9], 还可使用热压法、注塑法和激光烧灼法等传统方法以及3D打印等新兴手段制作芯片。微通道封装时,采用等离子表面处理或深紫外照射[10]后,迅速贴合,可以得到牢固和永久的封装。
根据芯片的结构差异可将PCR芯片分为静止PCR与动态PCR,或微池型PCR(Microchamber PCR, MCPCR)与连续流动PCR(ContinuousFlow PCR, CFPCR)。微池型PCR的特点在于,反应体系不流动,通过芯片整体升温降温的循环来实现反应;连续流动PCR则由反应液体循环流过不同温区进行扩增。在连续流动型PCR芯片中,反应溶液沿着微通道在3个固定的温度区连续流动,3个温度分别对应高温变性区、低温退火区和中温延伸区,在每个温区的流动时间和速度决定了反应时间。每流过3个温区为一次循环,PCR溶液按照所需的循环数依次流动,最后完成整个扩增反应。
2.1 微池型PCR芯片
微池型PCR芯片是传统PCR的微型化,有单反应池[11~15],液滴虚拟微池[16,17]及微池阵列[18~22]3种设计。 单反应池结构简单,1993年由Northrup等[11]首次报道,他们在硅基质上刻蚀出空腔,加入50 μL反应液进行PCR扩增。Giordano等[12]在2001年采用聚酰亚胺材料制作单池PCR芯片(如图1A所示),利用红外加热,在4 min内完成了对长度为500 bp的DNA片段的扩增。Neuzil等[16]采取以矿物油包裹PCR溶液的方式,形成了液滴虚拟微池来实现扩增(如图1B所示),可以有效减少反应溶液的挥发,他们设计的芯片能够快速地升温降温,最后通过融解曲线分析和毛细管电泳两种方法检验纯度。微池阵列是在单反应池的基础上增加反应通量得到。Matsubara等[18]在一块长3英寸、宽1英寸的硅片上刻蚀了1248个微孔,每个微孔容积为50 nL(如图1C所示)。PCR反应液用点样仪加入每个微池,再用石蜡油密封。Cai等[22]则是将PCR反应液通入芯片中,进行预载入,之后通过芯片多层之间的滑动使溶液进入微室阵列中,最后分别进行扩增与成像,免去了频繁加样的繁琐操作。微池型PCR的关键在于体系温度的快速切换,需要优化仪器的温度控制来缩短循环时间,其次因为反应体积微小,需要精密的加样仪器或是利用微流控芯片结构的特殊设计实现高通量。
2.2 蛇形通道PCR芯片
蛇形通道PCR芯片由Kopp等[23]在1998年首次提出,蛇形通道PCR芯片的经典形状是平行式(如图2A所示),随后Schaerli等[24]在2009年开发出了辐射式排布的蛇形通道芯片,同时结合了液滴技术,将纳升级的PCR反应液包裹在油相之中,沿着通道在温区间循环。Jiang等[25]在2014年同样设计了一款辐射式通道的芯片(图2B),利用太阳能加热,并通过智能手机装载的荧光检测器检测反应的进行。这种芯片的温区一般以高温中温低温的形式排布,加热元件有Cu[24]、Pt[26]、Al[27]等金属或是ITO[28, 29] (indium tin oxide,铟锡氧化物)电极等,为了提高芯片的温度控制效率和3个温区的精确控制,芯片还与温度传感器集成,并且通过热隔离槽来降低温区间的热交换。虽然在一些特别的模板和引物条件下,PCR反应中的退火和延伸温度可以一致,只需两个温区就可以进行PCR扩增,但是3个温区PCR芯片适用性更广。然而3个温区芯片存在的一个问题是,高温区解链产物在经过中温延伸区时可能会与DNA模板或互补链配对重新回到双链状态导致退火失败,降低扩增效率。而更改温区的排布会使得芯片的设计更为复杂。
2.3 螺旋通道PCR芯片
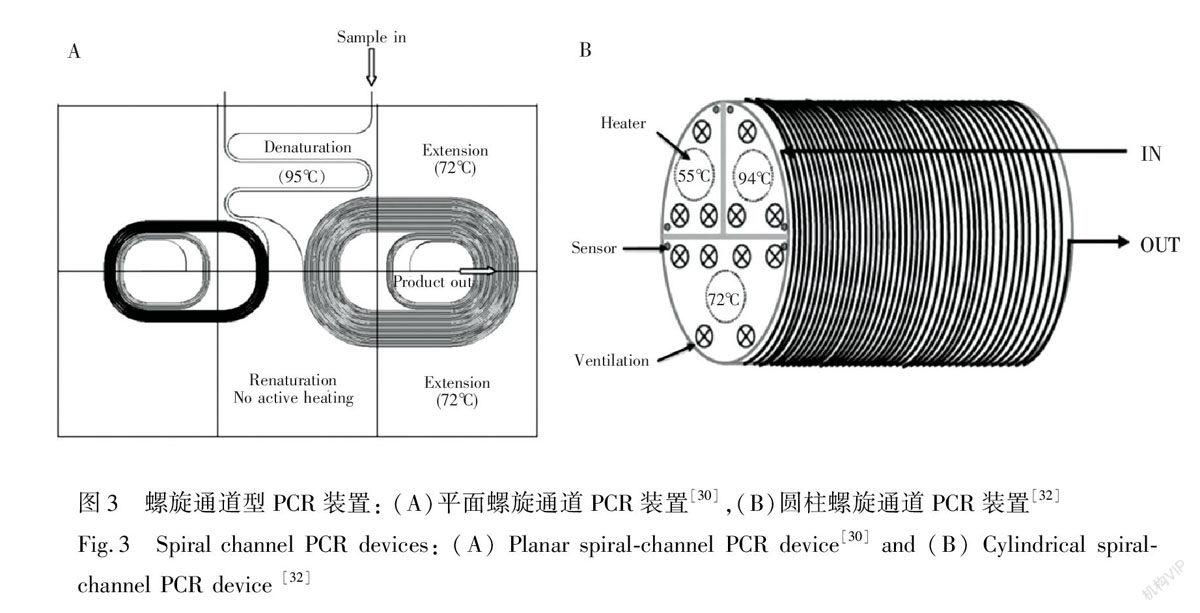
螺旋通道PCR芯片可以有效解决温区布局的问题。在这类芯片中,3个温区以扇形排布,反应溶液沿着通道盘旋流动,依次经过高低中3个温区。DNA的高温变性产物直接进入低温退火区,避免了在延伸区双链复性的可能,提高扩增效率。螺旋通道PCR芯片有平面式与圆柱式两种, Hashimoto等[30]研制的PCR芯片就是平面结构(如图3A所示),他们设计的芯片分左右两个区域,通道宽度为50 μm,右侧通道之间的距离为250 μm,左侧为50 μm。他们利用这款芯片对反应液流速进行了探索,最终可以分别在1.7和3.2别min内完成500 bp及997 bp的DNA片段的20次循环扩增与检测。Park等[31]将一条长3.5 m的弹性石英管在有3个温区的圆柱加热器上螺旋式缠绕33周,实现了PCR扩增。随后Dorfman等[32]也研制了类似的PCR设备(如图3B所示),他们利用不混溶的氟化溶剂将反应液分隔成液滴的形式,借助对界面性质的优化控制使得相邻液滴不会相互污染,以达到高通量的目的。Shu等[33]采用空气对液体片段进行隔离,发展了一种在螺旋通道微流控装置上进行的高通量快速核酸扩增方法,可以在一个液滴里同时扩增4种病原菌的靶基因,对实验中涉及的4种病原菌的基因组同时检测的灵敏度可以达到100 copies/μL。相比而言,平面式结构紧凑但每个循环的长度不一致,圆柱式体积比较大但是每个循环长度一致,流体控制较为简便。蛇形通道与螺旋通道PCR芯片的共同特点是,PCR的循环次数和时间受到整个通道的长度和流体速度的限制,一旦芯片结构固定,循环次数也相对固定。
2.4 振荡式PCR芯片
振荡式PCR是近年来随着微流控技术不断发展衍生的新方法。在振荡式装置中,循环次数不会受微通道长短限制。振荡式PCR装置由反应微通道、流体驱动器及加热器组成。PCR溶液在驱动器的控制下在温区间往复运动,循环次数可以随意调节,在各个温区的反应时间也可以通过改变各温区的长度和流动速度来优化(见图4)。Wang等[34]将1 μL PCR反应液以液滴的形式注入微通道中,反应液液滴两端以石蜡油封装,通过双向蠕动泵的控制使液滴在3个温区中循环,可以在15 min内完成对人乳头瘤细菌DNA的扩增。随后, 其他研究者们也设计了类似的芯片对黑色素瘤相关的酪氨酸酶基因[35],沙门氏菌、大肠杆菌、李斯特菌等[36]以及食物来源的病原体[37]的DNA进行了检测。振荡式PCR芯片与蛇形通道式PCR芯片有个共同点是,PCR反应溶液在循环过程中不得不在高温变性后穿过延伸区, 才能来到退火区,但是通过流体驱动器的调节,可以尽量缩短这段过程的时间,避免DNA双链复性。
2.5 闭环式PCR芯片
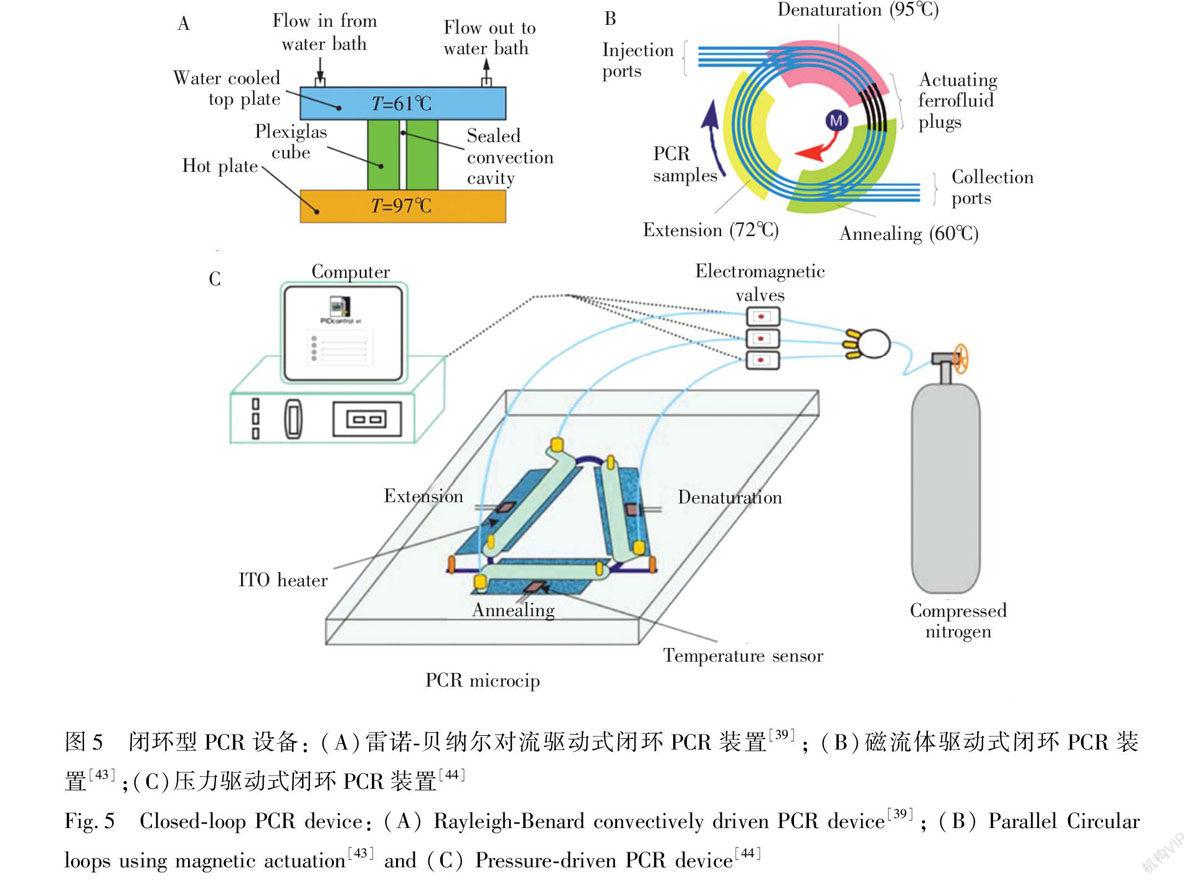
闭环式PCR芯片的原理与平面螺旋式PCR芯片类似,同样是通过驱动PCR溶液在平面内的环形通道内循环流过3个温区实现扩增反应。反应溶液依次经过高低中3个温区,避免了DNA复性影响扩增效率。根据驱动液体流动的原理可以分为温差环流[38]、对流驱动[39]和外力(如浮力[40,41]、磁力[42,43]、压力[44]等)驱动的闭环PCR。与螺旋式PCR不同的是,闭环式PCR每个循环都是在同一个环形通道中进行,因此每次循环的长度是一致的,芯片体积小,
图4 振荡型PCR装置[36]
Fig.4 Oscillatoryflow PCR device[36]利于集成化和平行化高通量扩增。Krishnan等[39]首次开发了一种利用雷诺贝纳尔(RayleighBenard)自然对流驱动方式进行的PCR设备(如图5A所示),PCR反应液在61℃和97℃的密闭空间内往复流动,DNA的扩增效率会受到雷诺数(Ra)和密闭腔体的高度与直径的比值(h/d)的影响。Sun等[43]将多个闭环通道合并在一块芯片内(如图5B所示),利用中心的磁铁同时对四重闭环内的磁性流体进行操纵,磁性流体继而推动反应液在环内流动,循环通过3个温区。Xu等[44]通过ITO电极加热控制3个温区的温度,采用压缩氮气作为驱动力,气体的释放由开关控制,反应液在通道内的移动同时需要微阀的配合(如图5C所示)。在这种设计的PCR芯片中,反应时间、循环次数、反应温度都是可以根据实际要求设置的,大大提高了芯片的适用性。
3 芯片PCR产物的检测与分析
在只具有扩增功能的PCR芯片中,反应完成后, 扩增产物一般采用离线的凝胶电泳[23,43,45~47]、毛细管电泳[12]等分离检测方法进行验证,这与传统PCR仪相比并没有优势,在线的检测方法在提高检测通量、减少交叉污染的风险等方面更具有前景,因此发展功能集成化的芯片也是研究者追求的方向。
3.1 毛细管电泳法
毛细管电泳(Capillary electrophoresis, CE)的原理是扩增产物在进入检测通道前, 先通过嵌入型染料进行标记,随后在电场的驱动下迁移运动,因不同片段长度的DNA分子迁移速度不同而被分离。检测器有紫外、荧光等。将PCR与毛细管电泳结合最早是由Mathies课题组[48]在1996年提出,他们将硅片PCR芯片与玻璃毛细管电泳芯片通过环氧树脂连接在起一次,PCR扩增后的产物经芯片的十字交叉口注入分离通道进行电泳。他们随后又利用类似的方法对人类基因组DNA进行扩增并且可以对性别进行鉴定[49];之后还对病原微生物的DNA进行了检测,检出限可以低至2~3个细菌[50]。近年来, PCR与CE的结合也逐渐朝着集成化的方向发展[51~53]。毛细管电泳因为结构简单、分析迅速,是最常用的分离检测方法,然而它的缺陷在于只能分辨DNA序列的大小而无法区分DNA序列的不同。
3.2 荧光检测法
荧光检测法也是目前微流控PCR芯片中常用的检测方法之一。研究者将芯片与荧光成像系统结合,不但可以在终点进行产物检测[22,54],还可以借助荧光探针和芯片结构实时监测反应的进行过程[27, 55~62]。荧光终点检测一般用于判断PCR的成功与否或进行半定量分析,但是实时荧光检测则可以通过循环阈值的方法进行准确的定量分析[27]。然而循环阈值定量方法会受到扩增效率的影响,Vogelstein等[63]在1999年提出了数字PCR(digital PCR)的概念,通过将样本分配到成百上千个反应单元中,每个单元只包含一个或多个拷贝的DNA分子,在每个反应单元中分别进行PCR扩增,扩增结束后对各个单元的荧光信号进行统计学分析,最终获得定量结果。反应单元越多,数字PCR的结果越准确,且不会受到扩增效率的影响。微池型PCR芯片与荧光检测相结合,进行数字PCR是目前的研究热点之一[17,20,64]。
在线荧光检测除终点检测、实时检测,还包括荧光融解曲线分析。这是一种可以与实时荧光PCR有机结合的检测技术,在PCR 结束后,利用现有的温度梯度,从退火温度缓慢升温至变性温度即可完成,具有灵敏、准确,且可以防止污染的优点。Crews课题组[65]采用这种分析技术,在20 min内完成了对人类唾液中的DNA的扩增与唾液来源的性别确认。
3.3 电化学方法
电化学方法是一种通过对电极的功能化修饰(如固定探针、酶、适配体等), 特异性地捕捉目标扩增产物,产生相应的电信号(电压、电流、电阻)的检测手段,具有灵敏性高、成本低等优点,而且可以通过微加工技术集成在微流控芯片上,因此最近也被发展用于PCR产物检测[13, 66~69]。Fang等[66]将蛇形通道PCR芯片与电化学检测相结合,在退火温区集成了三电极阵列,对每个循环进行实时的产物检测。电化学方法中所用到的探针可以是特定序列的一段核酸多聚物,通过与目标产物的特异性结合产生电信号的改变,因此电化学方法可以在一定程度上分辨产物的核酸序列。
3.4 DNA杂交微阵列
DNA杂交微阵列[70~75]是针对特定序列产物的一种检测手段,可以对扩增产物序列进行分析。通过精细的微加工技术,可以将PCR芯片与固定有大量寡核苷酸探针的微阵列相结合,PCR产物与探针完全互补,二者结合后则会产生荧光图样,通过这些荧光图样就可以获得扩增产物的序列信息。研究者们通过DNA杂交微阵列的方法在低丰度的DNA突变检测[70]、流感病毒检测[71]、HIV病毒基因分型[73]等方面进行了探索。
4 微流控PCR芯片分析应用实例
随着微机电加工技术的快速发展,微流控PCR芯片也在向结构简单化、功能集成化、便携式、一次性的方向发展,目前已有不少文献报道了可以在线完成完整的生物样本分离与分析的装置,为生物学和医药学等学科的研究带来了很多应用价值。
Sciancalepore等[35]在2011年构建了一款振荡型PCR芯片,也是第一款巢式PCR芯片。芯片由PDMS与玻璃基质两层键合而成,PDMS中包裹有一根玻璃毛细管,作为PCR反应的通道。毛细管中填充了硅化试剂玻璃基质表面通过电子束蒸发沉积的方式设置了3个钛/铅复合电极,作为微加热器,并与温度传感器结合,分别控制PCR过程中需要的3个温度,升/降温速度可以达到16℃/s。反应液滴通过毛细管两端的流体泵控制,在3个温区间循环。巢式PCR的特点在于引物包括内外两套,外引物完成第一次PCR后的产物作为内引物的模板,进行第二次PCR。这种扩增方式的特异性和灵敏性都明显高于普通的PCR。作者利用这款巢式PCR芯片在55 min内完成对酪氨酸激酶基因的扩增,可以用于恶性黑色素瘤的诊断,比传统PCR仪的速度提高了4倍。
Tian等[64]提出了一款利用负压协助完成DNA纯化和数字PCR检测的微流控芯片。芯片分为DNA纯化区与数字PCR区,以磁珠吸附生物样品裂解液中的DNA,并在磁铁的帮助下固定在纯化区,经过两步清洗后, 从磁珠上洗脱,与PCR试剂混合,再借助负压使溶液流入PCR区的微孔阵列中后,用硅油与未凝固的PDMS混合物冲走多余的样品,在温度循环的过程中,未固化的PDMS混合物也会逐渐凝固,起到封锁进样通道的作用。气压控制通道则注入水,使PCR区域保持湿润,避免试剂的蒸发。扩增完成后,通过对每个微孔中荧光强度的统计以及样品的稀释倍数可以对样品浓度进行定量。数字PCR的关键在于需要将样品溶液分配至成百上千个单独的反应孔中,使每个孔里只含有1~2个DNA模板或是没有,这款芯片利用了PDMS的透气性,在芯片外用注射器抽吸,使空腔内形成负压,从而完成了样品溶液的分配,摆脱了对自动加样器的依赖。
Chang等[76]设计了一种集样品制备、扩增、纯化、检测于一体的微池型微流控PCR芯片对假体关节液中的细菌进行了检测和分型,用于参考选择合适的抗生素,治疗在关节成形中的细菌感染等并发症。该课题组随后利用类似的芯片对幽门螺旋杆菌对喹诺酮类抗生素的耐药性诊断进行了研究[77]。他们将幽门螺旋杆菌特异性的引物结合在磁珠上,利用磁铁的吸附,在扩增完成后可以轻松地将扩增产物与剩余试剂分离,从而达到纯化的效果。利用两种不同的引物,芯片还可以同时进行单核酸多态性的研究,通过扩增产物可以分辨样品中的病原体是否是幽门螺旋杆菌,并判断是否出现了耐药性突变,为疾病的治疗提供了可靠的依据。
5 总结与展望
微流控PCR芯片的形式从最初单个微池或是简单的蛇形通道逐渐变得越来越丰富,并且在微阀、微泵等功能元件的组合下,功能也越来越完善,研究者们的思维也在逐步地扩展,出现了许多巧妙的设计。但是芯片的结构并非越复杂越好,繁琐的流体操作会耗费大量时间,也会对芯片制作带来麻烦,因此在完成实验目的的前提下, 尽量简化芯片结构才是重点。目前,PCR芯片在流体操作方面已经基本由机械化自动化的流体泵来完成,为了摆脱对流体泵的依赖,也有研究者提出了一些利用材料的表面张力自驱动的芯片,在样品制备方面也出现了不少可以直接利用功能结构在线提取和纯化的芯片,然而在在线检测方面仍旧有待改进。荧光检测是最为便捷和灵敏的检测手段,但是需要依赖于昂贵的光学仪器和摄像设备,毛细管电泳与凝胶电泳则相对廉价但是受到精度和灵敏度的限制,离线检测耗时耗力又易引入污染。所以,发展一种能够在线检测, 且低成本、高速度、高通量的检测方法, 是今后的研究重点,可以向电化学生物传感器的方向考虑。
由于芯片加工平台还不够普及,微流控PCR的技术也还不够成熟,微流控PCR芯片暂时还不能取代商品化的PCR仪,但是我们相信在更为精细的微机电加工、更为准确的温度与流体控制、更为灵敏的数码影像分析下,芯片的制作会越来越精巧,PCR的操作会更高效和便捷,基因分析的结果会更稳定和丰富。基于微流控芯片的突出优点和人们对新技术新方法的需求,微流控PCR芯片将在今后的基因研究中逐渐占据一席之地。
References
1 Scientists Announced the Latest Human Genome. http://www.people.com.cn/GB/guoji/25/95/20010212/393263.2001
科学家公布最新的人类基因组图谱. http://www.people.com.cn/GB/guoji/25/95/20010212/393263.2001
2 Saiki R K, Scharf S, Faloona F, Mullis K B, Horn G T, Erlich H A, Arnheim N. Science, 1985, 230(4732): 1350-1354
3 Mullis K B. Angew. Chem. Int. Ed. Engl., 1994, 33: 1209-1213
4 Whitesides G M . Nature, 2006, 442(7101): 368-373
5 Li X C, Chen Z G, Pan J B, Yang F, Li Y B, Yao M C. J. Chromatogr. A, 2013, 1291: 174-178
6 Zhang B B, Li Y B, He Q D, Qin J , Yu Y Y, Li X C, Zhang L , Yao M C, Liu J S, Chen Z G. Biomicrofluidics, 2014, 8(5): 054110
7 CHEN WenYuan, ZHANG WeiPing. Integrated Microfluidic Polymer PCR Chip. Shanghai: Profile of Shanghai Jiao Tong University Press, 2009: 35
陈文元,张卫平. 集成微流控聚合物PCR芯片, 上海: 上海交通大学出版社, 2009: 35
8 McDonald J C, Duffy D C, Anderson J R, Chiu D T, Wu H T, Schueller O J, Whitesides G M. Electrophoresis, 2000, D21(1): 27-40F
9 Deng T, Goetting L B, Hu J M, Whitesides G M. Sensor. Actuat. APhys., 1999, 75(1): 60-64
10 Dittrich P S, Tachikawa K, Manz A. Anal. Chem., 2006, 78(12): 3887-3907
11 Northrup M A, Ching M T, White R M. In: Proceeding of the 7th International Conference on Solid State Sensors and Actuators, Yokohama, Japan, 1993: 924-926
12 Giordano B C, Ferrance J, Swedberg S, Huhmer A F , Landers J P. Anal. Biochem., 2001, 291(1): 124-132
13 Lee T M, Carles M C, Hsing I M. Lab Chip, 2003, 3(2): 100-105
14 Shen K Y, Chen X F, Guo M, Cheng J. Sensor. Actuat. BChem., 2005, 105(2): 251-258
15 Wang S, Sun Y, Gan W, Liu Y, Xiang G, Wang D, Wang L, Cheng J, Liu P. Biomicrofluidics, 2015, 9(2): 024102
16 Neuzil P, Pipper J, Hsieh T M. Mol. Biosyst., 2006, 2(67): 292-298
17 Tanaka H, Yamamoto S, Nakamura A, Nakashoji Y, Okura N, Nakamoto N, Tsukagoshi K, Hashimoto M. Anal. Chem., 2015, 87(8): 4134-4143
18 Matsubara Y, Kerman K, Kobayashi M, Yamamura S, Morita V, Takamura Y, Tamiya E. Anal. Chem., 2004, 76(21): 6434-6439
19 Matsubara Y, Kerman K, Kobayashi M, Yamamura S, Morita Y, Tamiya E. Biosens. Bioelectron., 2005, 20(8): 1482-1490
20 Ottesen E A, Hong J W, Quake S R, Leadbetter J R. Science, 2006, 314(5804): 1464-1467
21 Prakash R, Kaler K V. Microfluid. Nanofluid., 2007, 3(2): 177-187
22 Cai D, Xiao M, Xu P, Xu Y C, Du W. Lab Chip, 2014, 14(20): 3917-3924
23 Kopp M U, Mello A J, Manz A. Science, 1998, 280(5366): 1046-1048
24 Schaerli Y, Wootton R C, Robinson T, Stein V, Dunsby C, Neil M A, French P M, deMello A J, Abell C, Hollfelder F. Anal. Chem., 2009, 81(1): 302-306
25 Jiang L, Mancuso M, Lu Z, Akar G, Cesarman E, Erickson D. Sci. Rep., 2014, 4: 4137
26 Schneegass I, Brautigam R, Kohler J M. Lab Chip, 2001, 1(1): 42-49
27 Tachibana H, Saito M, Shibuya S, Tsuji K, Miyagawa N, Yamanaka K, Tamiya E. Biosens. Bioelectron., 2015, 74: 725-730
28 Sun K, Yamaguchi A, Ishida Y, Matsuo S, Misawa H. Sensor. Actuat. BChem., 2002, 84(23): 283-289
29 Fukuba T, Yamamoto T, Naganuma T, Fujii T. Chem. Eng. J., 2004, 101(13): 151-156
30 Hashimoto M, Chen P C, Mitchell M W, Nikitopoulos D E, Soper S A, Murphy M C. Lab Chip, 2004, 4(6): 638-645
31 Park N, Kim S, Hahn J H. Anal. Chem., 2003, 75(21): 6029-6033
32 Dorfman K D, Chabert M, Codarbox J H, Rousseau G, de Cremoux P, Viovy J L. Anal. Chem., 2005, 77(11): 3700-3704
33 Shu B W, Zhang C S, Xing D. Anal Chim Acta, 2014, 826: 51-60
34 Wang W, Li Z X, Luo R, Lu S H, Xu A D, Yang Y J. J. Micromech. Microeng., 2005, 15(8): 1369-1377
35 Sciancalepore A G, Polini A, Mele E, Girardo S, Cingolani R, Pisignano D. Biosens. Bioelectron., 2011, 26(5): 2711-2715
36 Wang H Y, Zhang C S, Xing D. Microchim. Acta, 2011, 173(34): 503-512
37 Zhang C S, Wang H Y, Xing D. Biomed. Microdevices, 2011, 13(5): 885-897
38 Chen Z, Qian S, Abrams W R, Malamud D, Bau H H. Anal. Chem., 2004, 76(13): 3707-3715
39 Krishnan M, Ugaz V M, Burns M A. Science, 2002, 298(5594): 793-793
40 Agrawal N, Ugaz V M. Clin. Lab. Med., 2007, 27(1): 215
41 Wheeler E K, Benett W, Stratton P, Richards J, Chen A, Christian A, Ness K D, Ortega J, Li L G, Weisgraber T H, Goodson K E, Milanovich F. Anal. Chem., 2004, 76(14): 4011-4016
42 Sun Y, Kwok Y C, Nguyen N T. Lab Chip, 2007, 7(8): 1012-1017
43 Sun Y, Nguyen N T, Kwok Y C. Anal. Chem., 2008, 80(15): 6127-6130
44 Xu Z R, Wang X, Fan X F, Wang J H. Microchim. Acta, 2010, 168(12): 71-78
45 Nakano H, Matsuda K, Yohda M, Nagamune T, Endo I, Yamane T. Biosci. Biotechnol. Biochem., 1994, 58(2): 349-352
46 Agrawal N, Hassan Y A, Ugaz V M. Angew. Chem. Int. Edit., 2007, 46(23): 4316-4319
47 Guttenberg Z, Muller H, Habermuller H, Geisbauer A, Pipper J, Felbel J, Kielpinski M, Scriba J, Wixforth A. Lab Chip, 2005, 5(3): 308-317
48 Woolley A T, Hadley D, Landre P, deMello A J, Mathies R A, Northrup M A. Anal. Chem., 1996, 68(23): 4081-4086
49 Lagally E T, Emrich C A, Mathies R A. Lab Chip, 2001, 1(2): 102-107
50 Lagally E T, Scherer J R, Blazej R G, Toriello N M, Diep B A, Ramchandani M, Sensabaugh G F, Riley L W, Mathies R A. Anal. Chem., 2004, 76(11): 3162-317051 Kaigala G V, Huskins R J, Preiksaitis J, Pang X L, Pilarski L M, Backhouse C J. Electrophoresis, 2006, 27(19): 3753-3763
52 Beyor N, Yi L N, Seo T S, Mathies R A. Anal. Chem., 2009, 81(9): 3523-3528
53 Thaitrong N, Toriello N M, Del Bueno N, Mathies R A. Anal. Chem., 2009, 81(4): 1371-1377
54 Li Y Y, Zhang C S, Xing D. Microfluid. Nanofluid., 2011, 10(2): 367-380
55 Chen L, West J, Auroux P A, Manz A, Day P J. Anal. Chem., 2007, 79(23): 9185-9190
56 ZHAO ShuMi, ZHU Ling, ZHU CanCan, LI Yang, WANG HuaDong, ZHANG Long, DU DiWei,DENG GuoQing, WANG An, LIU Yong. Chinese J. Anal.Chem., 2014, 42(10): 1393-1399
赵树弥, 朱 灵, 朱灿灿, 李 阳, 王华东, 张 龙, 堵棣威, 邓国庆, 王 安, 刘 勇. 分析化学, 2014, 42(10): 1393-1399
57 Ramalingam N, Rui Z, Liu H B, Dai C C, Kaushik R, Ratnaharika B, Gong H Q. Sensor. Actuat. BChem., 2010, 145(1): 543-552
58 Hatch A C, Ray T, Lintecum K, Youngbull C. Lab Chip, 2014, 14(3): 562-568
59 Frey O, Bonneick S, Hierlemann A, Lichtenberg J. Biomed. Microdevices, 2007, 9(5): 711-718
60 Norian H, Field R M, Kymissis I, Shepard K L. Lab Chip, 2014, 14(20): 4076-4084
61 Sun Y, Zhou X, Yu Y. Lab Chip, 2014, 14(18): 3603-3610
62 Sun H, Olsen T, Zhu J, Tao J, Ponnaiya B, Amundson S A, Brenner D J, Lin Q. RSC Adv., 2015, 5(7): 4886-4893
63 Vogelstein B, Kinzler K W. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(16): 9236-9241
64 Tian Q C, Yu B D, Mu Y, Xu Y A, Ma C C, Zhang T, Jin W, Jin Q H. RSC Adv., 2015, 5(100): 81889-81896
65 Pjescic I, Crews N. Lab Chip, 2012, 12(14): 2514-2519
66 Fang T H, Ramalingam N, Dong X D, Ngin T S, Zeng X T, Lai Kuan A T L, Huat E Y P, Gong H Q. Biosens. Bioelectron., 2009, 24(7): 2131-2136
67 Defever T, Druet M, RocheletDequaire M, Joannes M, Grossiord C, Limoges B, Marchal D. J. Am. Chem. Soc., 2009, 131(32): 11433-11441
68 Ferguson B S, Buchsbaum S F, Wu T T, Hsieh K, Xiao Y, Sun R, Soh H T. J. Am. Chem. Soc., 2011, 133(23): 9129-9135
69 Yeung S S, Lee T M, Hsing I M. J. Am. Chem. Soc., 2006, 128(41): 13374-13375
70 Hashimoto M, Barany F, Soper S A. Biosens. Bioelectron., 2006, 21(10): 1915-1923
71 Sun Y, Dhumpa R, Bang D D, Handberg K, Wolff A. Diagn. Micr. Infec. Dis., 2011, 69(4): 432-439
72 Sun Y, Dhumpa R, Bang D D, Hogberg J, Handberg K, Wolff A. Lab Chip, 2011, 11(8): 1457-1463
73 Anderson R C, Su X, Bogdan G J, Fenton J. Nucleic Acids Res., 2000, 28(12): e60
74 Easley C J, Karlinsey J M, Bienvenue J M, Legendre L A, Roper M G, Feldman S H, Hughes M A, Hewlett E L, Merkel T J, Ferrance J P, Landers J P. P. Natl. Acad. Sci. USA, 2006, 103(51): 19272-19277
75 Jiang X, Shao N, Jing W, Tao S, Liu S, Sui G. Talanta, 2014, 122: 246-250
76 Chang W H, Wang C H, Lin C L, Wu J J, Lee M S, Lee G B. Biosens. Bioelectron., 2015, 66: 148-154
77 Chao C Y, Wang C H, Che Y J, Kao C Y, Wu J J, Lee G B. Biosens. Bioelectron., 2016, 78: 281-289
Advance in Research of Microfluidic Polymerase Chain Reaction Chip
HE QiDi, HUANG DanPing, HUANG Guan, CHEN ZuanGuang*
(School of Pharmaceutical Sciences, Sun Yatsen University, Guangzhou 510006, China)
Abstract Gene is the genetic code of human beings, and has only 0.1‰ difference among individuals but creates a weelthy divesity. Thus it has a huge application value to explore the difference. Polymerase chain reaction (PCR) is one of the technologies mostly used in genetic research. But traditional thermal cyclers are not only time and energyconsuming, but also difficult to be integrated and portable. The combination of microfluidics and PCR can decrease the reaction volume significantly and increase the reaction efficiency with easy integration and miniaturization. In this review, we briefly introduced microchamber PCR chip and continuousflow PCR chip according to their structures, and online detection methods including capillary electrophoresis, fluorescence, electrochemistry and DNA hybridization array. At last, we summarized the recent advances and pointed out the future development about microfluidic PCR chip.
Keywords Microfluidics; Polymerase chain reaction (PCR); Chip; Review
(Received 21 January 2016; accepted 25 February 2016)
This work was supported by the National Natural Science Foundation of China (No. 21375152)
