基于cpDNA序列分析不同生境芦苇种群的遗传结构
2016-11-17李毳
李毳
山西财经大学环境经济学院,山西 太原 030006
基于cpDNA序列分析不同生境芦苇种群的遗传结构
李毳
山西财经大学环境经济学院,山西 太原 030006
芦苇(Phragmites australis)是禾本科多年生植物,是生态幅极广的世界性物种之一。在长期适应不同生境的过程中,从形态到遗传产生了高度分化,形成种内丰富的生态型。芦苇自然种群遗传结构的研究有助于丰富进化遗传学理论并推动其在物种保护和生态恢复方面的应用。基于3个叶绿体DNA(cpDNA)的片断(rpl16,matK和trnL-F)对我国东北、西南和黄河中上游地区的水生和旱生芦苇的7个种群共96个个体进行遗传结构分析。结果表明旱生芦苇种群的遗传多样性为0.702,基因流为0.904;水生芦苇种群的遗传多样性为0.826,基因流为0.431。种群水平的遗传多样性为0.814,其中有49.0%的变异存在于种群之间,相对比较高;芦苇种群的基因流为0.520。虽然种群水平和物种水平的基因流都小于1.0,但这并不影响其物种的稳定性和遗传多样性,推测可能与芦苇的繁殖方式和种群数量庞大有关。
芦苇;生态型;叶绿体DNA;遗传多样性;基因流
LI Cui. Analysis of genetic diversity of Phragmites australis populations in different habitats based on cpDNA [J]. Ecology and Environmental Sciences, 2016, 25(8): 1315-1319.
芦苇(Phragmites australis)为禾本科多年生植物,是生态幅极广的世界性物种之一。在长期适应不同生境的过程中,从形态到基因产生了可以稳定遗传的差异(林文芳等,2007),形成种内不同的生态型。针对芦苇适应性机理的探讨基本是围绕表型、基因型和环境因子展开的(Qiu et al.,2016;Lambertini et al.,2012)。目前,对芦苇表型变异及遗传变异的生态型划分已进行了很多研究,但针对芦苇生态型变异规律及其可能形成机制的研究仍存在较多局限和分歧(庄瑶等,2010;王为东等,2010),分子证据与表型变异结合的分析相对较少,彻底阐明芦苇变异机制的报道更是少之又少(邱天,2012)。因此迫切需要加强不同生态型芦苇生理生态学和分子生态学等方面的研究(王萌等,2010)。对退化芦苇种群的分析探讨,进一步表明迫切需要加强分子生物学的研究,从微观水平上阐述环境因子对芦苇退化的影响(张玉峰等,2012)。
芦苇适应异质生境的主要策略是表型可塑性和遗传变异(邱天,2012),更多的研究表明,基因型是影响芦苇变异的主要因素。在同质种植实验中,芦苇在表型和种群结构上有显著差异,这种差异主要是由于遗传因素的作用,环境因子是次要因子,不同生态型之间的差异是遗传分化的结果(杨晓杰,2011;Hansen et al.,2007;Ostendorp,1991)。叶绿体DNA相对保守,既不像核基因组那样有较多的重复序列,也不像线粒体基因组那样重排事件频繁,cpDNA具有分子量小、多拷贝和结构简单的特点,有利于对叶绿体基因组进行分析(龚晓洁等,2009)。根据水热条件和生态环境的差异,中国芦苇产区可划分为5个地理区域:南方湖滨苇区、东部滨海苇区、北方沼泽苇区、西北干旱苇区和西南高原苇区(李必华等,1994)。大量文献报道了诸多生态因子对芦苇生物学特性的影响,结果均表明水分、盐分及二者交互作用对芦苇的影响较大,在综合水盐因子的研究中,发现水分对芦苇的影响大于盐分,水分是芦苇生态特征变化的关键驱动因子(贡璐等,2014),芦苇不同生态型遗传相似度与生境土壤含水量都呈极显著正相关(林文芳等,2007)。本研究中使用3个cpDNA片断:rpl16,matK和trnL-F,对我国东北、西南和黄河中上游地区旱地和水地的芦苇进行遗传结构分析,以期为芦苇生态型的鉴定、划分及其可能的形成机制提供更为可靠的参考数据。
1 材料与方法
1.1材料
本研究收集了野生芦苇7个种群共96个个体的新鲜叶片,包括水生芦苇(3个种群40个个体)和旱生芦苇(4个种群56个个体)两种生态型(详见表1)。样品采集按照常规采样方式,即每个种群10~20个个体的叶片,间距不少于100 m,采集的叶片放入棕色小纸袋,在袋子中加入适量硅胶,用于样品的干燥,然后以种群为单位收在一个大塑封袋中,带回实验室用于DNA提取。
1.2方法
1.2.1实验方法
本研究中植物总DNA提取采用CTAB法(Doyle et al.,1987)。改良后的方法如下:称0.1 g叶片,剪碎后用液氮研磨至粉末状;收集到2.0 mL离心管中,加入700 µL 2%的CTAB提取缓冲液、2% β一巯基乙醇,漩涡振荡混匀,65 ℃温育45 min;加入700 µL氯仿一异戊醇混合液(体积比24∶1),混匀呈乳浊状;12000 rpm离心5 min,取上清液。加入1/10体积的10% CTAB缓冲液,轻微振荡以混匀,重复抽提两次。加入1/10体积3 mol·L-1NaAc和等体积预冷的异丙醇,颠倒混匀,-20 ℃低温冰箱放置30 min以沉淀DNA。8000 rpm冷冻离心20 min,弃去上清液;分别用1 mL 70%和90%乙醇洗涤样品1次,8000 rpm冷冻离心20 min,干燥处理后用50 µL TE缓冲液溶解DNA,加入2 µLRNase(10 mg·mL-1)于37 ℃温育1 h以去除RNA,冰箱保存。使用的分子标记来自叶绿体的3个片段:rpl16、matK和trnL-F,引物序列见表2。PCR扩增反应如下:反应体系为25 µL,包括2×rTaq 12.5 µL,模板1 µL(~50 ng),引物0.8 µL(5 pmol),ddH2O 8.4 µL;反应条件:首先94 ℃变性5 min;然后进入循环复制扩增,即94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,共25个循环;结束后,72 ℃再延伸5 min。DNA序列的扩增和测序分别在9700 PCR仪和3730测序仪上进行(ABI,9700 thermal cycler and 3730xl Genetic Analyzer)。测序工作由上海生物工程有限公司完成。
1.2.2数据分析方法
对于测序获得的序列首先使用CLUSTAL X软件(Thompson et al.,1997)进行对位排列,然后导入MEGA 5(Tamura et al.,2011)中进行手工校对。最后将校正好的序列利用DnaSP version 5(Librado et al.,2009)进行DNA序列的单倍型统计,用于种群间的遗传多样性分析。本研究使用ARLEQUIN version 3.0(Excoffier et al.,2005)计算种群间的遗传多样性,遗传多样性指数包括种群内平均多样性指数(HS)、总遗传多样性(HT)、种群间遗传分化系数(Gst)和种群间基因流(Nm),软件所用参数为默认参数。此外,利用Network version 4.2.0.1(Bandelt et al.,1999)软件构建DNA序列单倍型的种群间网络关系图,参数同样为默认参数。
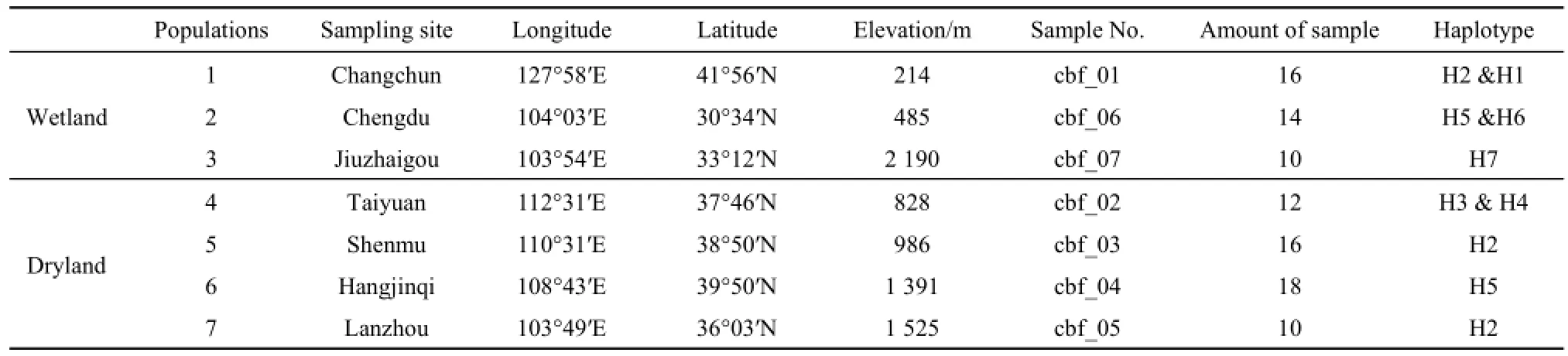
表1 样品分布信息和各种群叶绿体单倍型组成Table 1 Sampling sites of Phragmites australisin and composition of 7 choloroplast haplotypes indicated for each population
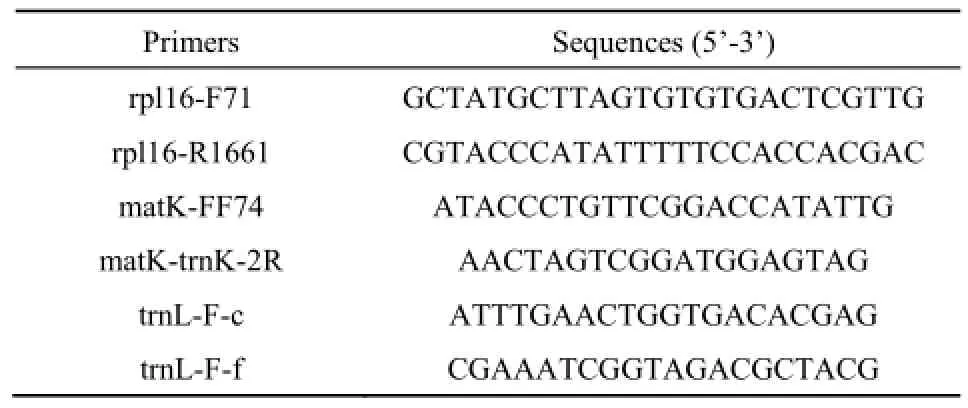
表2 扩增叶绿体rpl16、matK和trnL-F基因片段的引物序列Table 2 The primer sequences for amplifying the gene fragments of rpl16,matKand trnL-F from choloroplast genome in this study
2 结果
2.1cpDNA序列分析
从7个芦苇自然种群的96个个体中检测了3个cpDNA片段:rpl16、matK和trnL-F,序列对比后的长度分别为:654、877、917 bp。由于cpDNA具有结构简单、单亲遗传、无重组的特点(李丽卡等,2016),可以对3个片断进行联合分析,其片段总长度为2448 bp,总共定义了7个单倍型。该片段G+C含量为32.3%,含A/T 67.7%,说明此片断具有进化速率相对较快的特点(龚晓洁等,2009)。
2.2单倍型的种群分布
将校正好的序列利用DnaSP version 5进行DNA序列的单倍型统计,7个复合种群中共检测到7个单倍型,即H1~H7,见表1。单倍型H1、H6、H7仅分布于单个种群,分别是种群1、种群2、种群3,均属水地;单倍型H3、H4分布在旱地的种群4;单倍型H5分布于2个种群;单倍型H2分布频率最高,在旱地种群和水生种群都有分布。
2.3种群遗传结构和遗传分化
采用AARLEQUIN vversion 3.0进行种群间的遗传多样性分析(见表3),包括种群内平均多样性指数(HS)、总遗传多样性(HT)、种群间遗传分化系数(Gsst)和种群间基因流(NNm)。结果表明旱生芦苇种群的遗传多样性为0.7002,种群内的变异为0.452,种群间的变异为0.2550,占总变异的35.6%,基因流为0.9044;水生芦苇种群的遗传变异高于旱生芦苇,遗传多样性指数为0.826,种群内的变异为0..382,种群间的变异为00.444,占总变异的53.7%,基因流为0.4431。在物种水平,芦苇的遗传变异中有49.00%的变异存在于种群之间,近51.00%的变异存在于种群内,基因流为0.520。种群间49.00%%的遗传变异反映了芦苇对不同生境的适应情况。芦苇自然种群的遗传结构是物种长期进化的产物,在长期的进化过程中,各种群分布区的自然地理环境不同,自然选择压力也不同,而种群为适应特定的环境条件必然产生一定的分化。
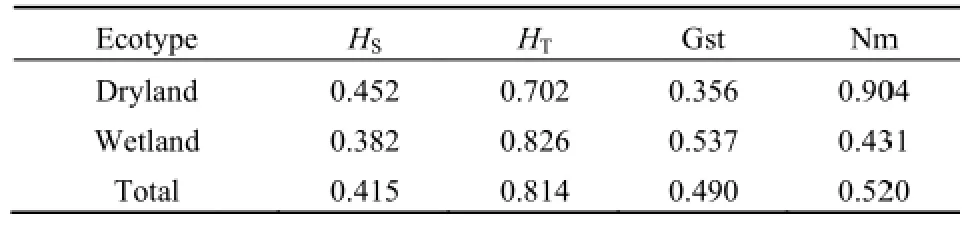
表3 两种生态型芦苇遗传多样性Table 3 Geenetic diversity of ttwo ecotypes of Phragmites australiis
2.4DNA序列单倍型的种群间网络关系图
用Netwwork version 4.2.0.1软件构建DNAA序列单倍型的种群间网络关系图,参数为默认参数。由图1可以看出单倍型H2、H3、H4、H5的遗传关系相对较近,且这几个单倍型在水地和旱地都有分布;而单倍型H1、H6、H7的遗传关系相对较远,且仅分布于水地,说明水生芦苇生境更复杂,遗传变异更为丰富。芦苇作为一种多年生水生草本植物,其典型生境为淡水或含盐沼泽、湖边,可适应多种生境条件,并演化为对干旱、盐渍或低温等陆生胁迫环境有较强抗性、具有基因型差异的不同生态型(王海波,22006)。水生芦苇是最古老的类群(龚晓洁等,22009),在长期的进化过程中,水生芦苇累积了更多的遗传变异,进化产生了差异较大且可以稳定遗传的单倍型(林文芳等,2007)。
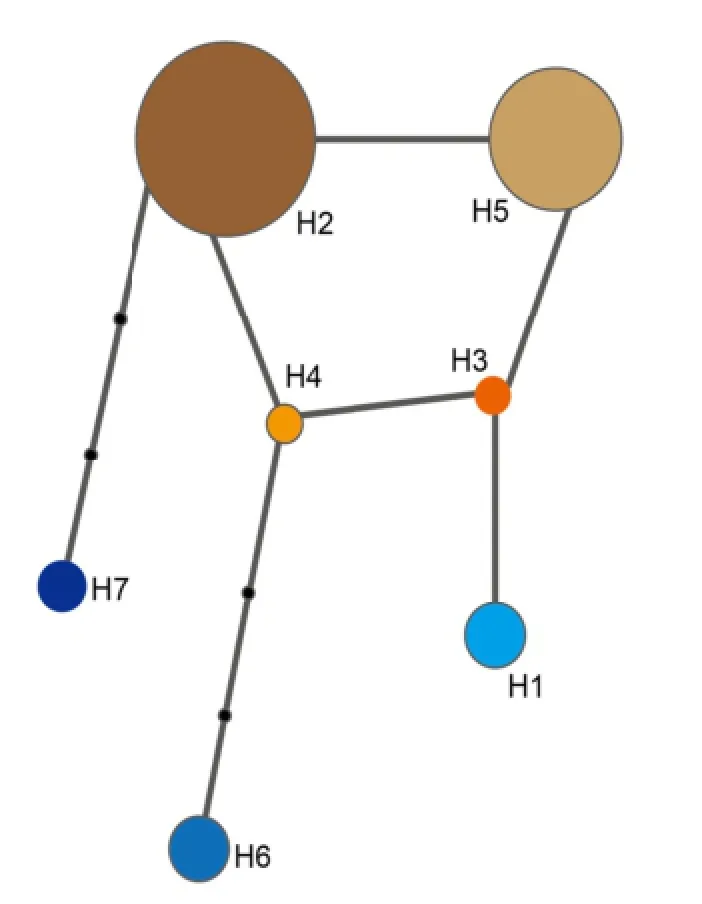
图1 基于叶绿体序列单倍型的Network关系图Fig. 1 Anetwork relationshhip based on chlorrotypes
3 讨论
3.1芦苇种群的遗传多样性
种群的遗传多样性水平在相当程度上制约着种群对环境的适应能力,因而可用于预测种群的发展趋势(潘莹等,1998)。如果种群内的基因型多样性较低,将不能为成功地适应新环境提供足够的表型变异,这可能是德国一些湖泊中具有较低遗传变异的芦苇种群正在退化的原因之一(Koppitz ett al.,1997)。遗传变异是芦苇适应异质环境的主要对策之一(邱天,2012),种群间的遗传变异反映芦苇对不同生境的适应情况。ISSR分子标记表明扎龙湿地芦苇种群存在遗传分化,种群间的变异占35.009%,表明芦苇种群的变异主要来源于种群内(杨晓杰等,2011)。张淑萍(2001)用RAPD分子标记方法研究了黄河湿地芦苇种群的基因多样性(0.2126),其中有29.008%的遗传多样性存在于种群间,各位点在种群间的分化较大,等位酶的结果也支持遗传多样性存在于种群间。龚晓洁(2009)利用cpDNA对芦苇种群进行研究,结果表明,种群的分化系数为0.609,遗传变异存在于种群间。本研究中,芦苇种群总的遗传变异为0.814,其中有49.0%的变异存在于种群之间,相对比较高,这可能与取样尺度有关,因为在较小的地理范围内物种的遗传分化程度较小。芦苇分布广泛,变异丰富,较小幅度和尺度的生境差异对种群间形态分化的影响力有限,大幅度的生境差异则可能导致种群间出现显著的形态差异(张淑萍等,2003)。有学者认为不同地理气候区间芦苇的形态变异是地理生态型,同一气候区内不同生境中芦苇的形态变异则是生境生态型(王萌等,2010)。本研究取样考虑不同地理气候区和生境两方面,经度、纬度跨度都比较大。
3.2芦苇种群间的基因流
影响物种的遗传多样性和种群间遗传分化的因素包括种群历史、繁育系统、基因突变、遗传漂变、基因流动、自然选择等(Nybom et al.,2000),种群间基因流大于1.0表明其能发挥均质化作用;小于1.0则表明基因流动成为遗传分化的主要原因;大小适中的基因流可以帮助维持种群的稳定,在一定程度上也能增强对变化环境的适应(Irwin et al.,2005)。本研究中,旱生芦苇种群的基因流为0.904,水生芦苇种群的基因流为0.431;在物种水平,芦苇种群的基因流为0.52。虽然种群水平和物种水平的基因流都小于1.0,但并不影响其物种的稳定性和遗传多样性,这可能与芦苇的繁殖方式和种群数量庞大有关。首先,芦苇是一种典型的无性系植物,同时具有有性繁殖和营养繁殖两种繁殖方式,天然种群主要依靠根茎繁殖补充更新(杨允菲等,1998;Eckert et al.,1999),芦苇各种群间也没有明显的异化趋势,其变异仍属于亚种到种间的范围(龚晓洁,2009);其次,广布种芦苇庞大的家族提供了较高的基因突变,虽然高等生物基因突变率非常低,平均10万到1亿个配子中才会出现1个基因突变,每个世代每个基因的平均突变率也只有0.0001~0.001,但是,由于每个物种拥有的个体很多,每个个体又有许多个基因位点,所以尽管个体的基因突变率很低,但物种整体的基因突变总和却是相当巨大的(贾竞波,2011)。
与旱生芦苇种群相比,水生芦苇种群虽然基因流较低,但是遗传多样性却较高,推测可能与其进化历史有关.水生芦苇起源较早,累积了较多的变异(龚晓洁等,2009;李建华等,2011);另外,水深和盐分对芦苇生态特征有明显影响(庄瑶等,2010),而湿地缺水严重,盐碱化加剧,导致芦苇群落退化(白军红等,2008),水生环境更为复杂,芦苇需要更强的适应能力。旱地芦苇和水地芦苇种群的单倍型分布、遗传多样性指数、基因流等有较明显区别,水因子确实是影响芦苇生态型的关键因子(贡璐等,2014)。克隆体植物的空间结构受到生境异质性、繁殖体传播、植物间相互作用、生物环境作用、外界干扰等因素的影响(Van Rossum et al.,2006)。芦苇变异丰富,从不同尺度、利用不同方法进行更广范围的研究,才能从本质上了解世界广布种的生物学、生态学和遗传结构特征。
BANDELT H J, FORSTER P, ROHL A. 1999. Median-joining networks for inferring intraspecific phylogenies [J]. Molecular Biology and Evolution, 16(1): 37-48.
DOYLE J, DICHKSON E. 1987. Preservation of plant samples for DNA restriction endonuclease analysis [J]. Taxon, 36(4): 715-722.
ECKERT C G, DORKEN M E, MITCHELL S A. 1999. Loss of sex in clonal populations of a flowering plant, Decodon verticillatus(Lythraceae) [J]. Evolution, 53(4): 1079-1092.
EXCOFFIER L, LAVAL G, SCHNEIDER S. 2005. Arlequin (version 3.0): an integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics Online, 1: 47-50.
HANSEN D L, LAMBERTINI C, JAMPEETONG A, et al. 2007. Clone-specific differences in Phragmitesaustralis: Effects of ploidy level and geographic origin [J]. Aquatic Botany, 86(3): 269-279.
IRWIN D E, BENSCH S, IRWIN J H, et al. 2005. Speciation by distance in a ring species [J]. Science, 307(5708): 414-416.
KOPPITZ H, KUHL H, HESSE K, et al. 1997. Some aspects of the importance of genetic diversity in Phragmites australis (cav) trin ex steudel for the development of reed stands [J]. Botanica Acta, 110(3): 217-223.
LAMBERTINI C, SORRELL B K, RIIS T, et al. 2012. Exploring the borders of European Phragmites within a cosmopolitan genus [J]. AoB Plants: pls020.
LIBRADO P, ROZAS J. 2009. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data [J]. Bioinformatics, 25(11): 1451-1452.
NYBOM H, BARTISH I. 2000. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants [J]. Perspectives in Plant Ecology Evolution & Systematics,3(2): 93-114.
OSTENDORP W. 1991. Damage by episodic flooding to Phragmites reeds in a prealpine lake: proposal of a model [J]. Oecololgia, 86(1): 119-124.
QIU T, JIANG L L, YANG Y F. 2016. Genetic and epigenetic diversity and structure of Phragmites australis from local habitats of the Songnen Prairie using amplified fragment length polymorphism markers [J]. Genetics and Molecular Research, 15(3): 10.4238/gmr.15038585.
TAMURA K, PETERSON D, PETERSON N, et al. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance, and maximum parsimony methods [J]. Molecular Biology and Evolution, 28(10): 2731-2739.
THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. 1997. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools [J]. Nucleic Acids Research, 25(24): 4876-4882.
VAN ROSSUM F, TRIEST L. 2006. Fine-scale genetic structure of the common Primula elatior (Primulaceae) at an early stage of population fragmentation [J]. American Journal of Botany, 93(9): 1281-1288.
白军红, 邓伟, 王庆改, 等. 2008. 松嫩平原湿地环境问题及整治方略[J].湿地科学, 6(1): 1-6.
龚晓洁, 闰广为, 浦铜良. 2009. 结合叶绿体基因mark/trnk序列和生境分析不同生态型芦苇的系统发育[J]. 兰州大学学报(自然科学版),45(3): 50-54.
龚晓洁. 2009. 基于cpDNA分析河西走廊地区4种生态型芦苇系统发育关系[D]. 兰州: 兰州大学: 32-33.
贡璐, 朱美玲, 塔西甫拉提·特依拜, 等. 2014. 塔里木盆地南缘旱生芦苇生态特征与水盐因子关系[J]. 生态学报, 34(10): 2509-2518.
贾竞波. 2011. 保护生物学(第二版)[M]. 北京: 高等教育出版社: 38-39.
李必华, 孙丕燀, 邢尚军. 1994. 山东滨海芦苇生物生态学特性初探[J].山东林业科技, (2): 1-6.
李建华, 李雪松, 田春元, 等. 2011. 大贵寺国家森林公园野生青檀居群的遗传多样性[J]. 生态环境学报, 20(12): 1799-1804.
李丽卡, 李象钦, 谢国文, 等. 2016. 基于cpDNA片段探讨中-日间断分布双花木属植物的系统发育学[J]. 生物技术通报, 32(1): 80-87.
林文芳, 陈林姣, 朱学艺. 2007. 用分子标记技术分析不同生态型芦苇的遗传多样性[J]. 植物生理与分子生物学学报, 33(1): 77-84
潘莹, 赵桂仿. 1998. 分子水平的遗传多样性及其测定方法[J]. 西北植物学报, 18(4): 645-653.
邱天. 2012. 普通芦苇适应异质生境策略的研究进展[J]. 长春师范学院学报: 自然科学版, 31(12): 63-66.
王海波. 2006. 不同生境两种生态型芦苇Rubisco蛋白分子的差异分析[D]. 兰州: 兰州大学: 1-2.
王萌, 王玉彬, 陈章和. 2010. 芦苇的种质资源及在人工湿地中的应用[J]. 应用与环境生物学报, 16(4): 590-595.
王为东, 王亮, 聂大刚, 等. 2010. 白洋淀芦苇型水陆交错带水化学动态及其净化功能研究[J]. 生态环境学报, 19(3): 537-543.
杨晓杰, 佟守正, 程加春. 2011. 扎龙芦苇遗传多样性ISSR分析[J]. 广西植物, 31(6): 725-729.
杨允菲, 郎惠卿. 1998. 不同生态条件下芦苇无性系种群调节分析[J].草业学报, 7(2): 2-10.
张淑萍, 王仁卿, 张治国, 等. 2003. 黄河下游湿地芦苇形态变异研究[J].植物生态学报, 27(1): 78-85.
张淑萍. 2001. 芦苇分子生态学研究[D]. 哈尔滨: 东北林业大学: 50-58.
张玉峰, 何彤慧, 张娟红, 等. 2012. 中国湿生芦苇群落退化现状研究[J].湿地科学管理, 8(1): 47-50.
庄瑶, 孙一香, 王中生, 等. 2010. 芦苇生态型研究进展[J]. 生态学报,30(8): 2173-2181.
Analysis of Genetic Diversity of Phragmites australis Populations in Different Habitats Based on cpDNA
LI Cui
Department of Environment and Economic, Shanxi University of Financial and Economic, Taiyuan 030006, China
Phragmites australis is perennial plant belonging to family Gramineae, and a species with very wide ecological amplitude worldwide. The phenotype and genetics of this plant highly differentiate and produce abundant ecotypes within species during long term adaptation to different habitats. Research on the spatial genetic structure in natural populations is important for both theoretical aspects of evolutionary genetics and their application in species conservation and ecological restoration. In this study, 7 populations including 96 individuals on wetland and dryland in northeast, southwest and middle-up stream of yellow river were analyzed for the genetic structure based on the molecular marker rpl16, matK and trnF of chloroplast genomic DNA. The results indicated that the genetic diversity and gene flow of P. australis populations in dryland were 0.702 and 0.904, respectively, in contrast, that in wetland were 0.826 and 0.431, respectively. The genetic diversity on the population level was 0.814, in which the high variants of 49.0% existed among populations and the gene flow of P. australis species was 0.520. Although the gene flow of the population level and species level was less than 1.0, the species stability and genetic diversity were not affected, suggesting that it was related to the reproductive strategy and massive populations of P. australis.
Phragmites australis; ecotype; chloroplast DNA; genetic diversity; gene flow
10.16258/j.cnki.1674-5906.2016.08.009
Q945.79; X17
A
1674-5906(2016)08-1315-05
山西省回国留学人员科研资助项目(2012-059);中国清洁发展机制基金赠款项目(1213116)
李毳(1968年生),女,副教授,博士研究生,主要研究方向为植物生态学。E-mail: aihl@sxu.edu.cn
2016-07-05
引用格式:李毳. 基于cpDNA序列分析不同生境芦苇种群的遗传结构[J]. 生态环境学报, 2016, 25(8): 1315-1319.
