应用依赖解旋酶DNA扩增技术检测转基因大豆
2016-11-11章晶晶张翠侠马超峰河北省食品检验研究院河北省食品安全重点实验室河北石家庄05007河北农业大学食品科技学院河北保定07000
章晶晶,王 赞,张翠侠,马超峰,周 巍,2,,张 岩(.河北省食品检验研究院,河北省食品安全重点实验室,河北 石家庄 05007;2.河北农业大学食品科技学院,河北 保定 07000)
应用依赖解旋酶DNA扩增技术检测转基因大豆
章晶晶1,王 赞1,张翠侠1,马超峰1,周 巍1,2,*,张 岩1
(1.河北省食品检验研究院,河北省食品安全重点实验室,河北 石家庄 050071;2.河北农业大学食品科技学院,河北 保定 071000)
目的:基于依赖解旋酶DNA恒温扩增(helicase-dependent isothermal DNA amplifi cation,HDA)技术,建立快速检测转基因大豆的方法。方法:采用以CaMV35S、NOS、CP4-EPSPS 3 种外源基因和内源基因Lectin(大豆凝集素基因)为目的片段设计4 对特异引物,建立反应体系,通过HDA方法对内源基因和3 种外源基因的检测特异性和灵敏度进行实验,并对结果进行同源性分析。结果:建立了转基因大豆HDA检测法,检测特异性良好,3 种外源基因的检出限为0.2%。结论:转基因大豆的HDA检测方法具有普通聚合酶链式反应的特异、灵敏等特点,并且对仪器要求更低,极适合基层实验室使用,具有广阔的应用前景。
依赖解旋酶DNA扩增;转基因大豆;检测
随着现代科技在农业生产中的应用,转基因作物近年来得到迅速发展。转基因食品在传统食品市场中份额不断加大并进入了食物链。转基因大豆是较早商业化生产的转基因作物之一,与非转基因大豆相比,转基因大豆在提高产量、抗逆能力、品质改良、解决人类面临的环境污染、温饱问题及资源短缺等方面日益显出巨大作用。同时,转基因作物的安全性也逐渐成为人们关注的焦点,具体包括食用安全性和生态安全性[1-2]。由于转基因食品的安全性短时间内难以确定,各国对转基因食品使用标签制度进行管理[3],所以急需建立一套准确、快速检测转基因食品的方法。
目前,对转基因大豆的实验室检测包括基于核酸的检测技术主要有聚合酶链式反应(polymerase chain reaction,PCR)方法[4]和基于PCR原理的其他检测方法如实时荧光PCR[5-7]、多重PCR[8]、巢式PCR[9-10]、等温环介导PCR[11-12],这些方法检测速度快,特异性高,但需要特定仪器,难以普及应用并且检测费用昂贵。基于蛋白检测的方法主要是酶联免疫法[13],此方法需要的抗体和抗原制备较为困难,价格昂贵。
近年来,一种新型核酸恒温扩增技术依赖解旋酶DNA恒温扩增(helicase-dependent isothermal DNA amplification,HDA)技术因其操作简单、结果准确、反应灵敏、无需特殊仪器、便于普及而广泛受到关注。HDA技术是核酸等温扩增技术的一种,主要利用解旋酶在恒温条件下解开DNA双链,同时DNA单链结合蛋白稳定解开的单链为引物提供结合的模板,之后由DNA聚合酶催化合成互补链。新合成的双链在解旋酶作用下又解成单链,并作为下一轮合成的模板进入上述循环扩增反应,最终实现靶序列的指数式增长[14]。HDA法目前已经应用于一些致病菌的检测[15-19]以及转基因成分检测[20],本研究拟采用以35S启动子(cauliflower mosaic virus 35S,CaMV35S)、NOS终止子(nopaline synthase,NOS)、5-烯醇丙酮酸莽草酸-3-磷酸合成酶(5-enolpyruvylshikimate-3-phosphate synthase,CP4-EPSPS)3 种外源基因和内标基因Lectin为目的片段设计4 对特异引物,建立快速检测转基因大豆的新方法。
1 材料与方法
1.1 材料与试剂
非转基因大豆、转基因大豆 国家标准物质中心。
十六烷三甲基溴化铵(hexadecyltrimethylammonium bromide,CTAB) 天津市科密欧化学试剂有限公司;三羟甲基氨基甲烷(tirs hydroxymethyl aminomethane,Tris) 天津市博迪化工有限公司;乙二胺四乙酸二钠(ethylene diaminetetraacetic acid disodium,Na2EDTA)北京鼎国昌盛生物技术有限责任公司;三氯甲烷、异戊醇、无水乙醇、氯化钠(均为分析纯) 天津市永大化学试剂有限公司;大肠杆菌解旋酶Ⅱ(UvrD) 上海富众生物科学有限公司;MutL蛋白、T4噬菌体基因32编码蛋白(T4 gp 32)、Bst聚合酶 美国NEB公司;dNTP,100 bp DNA Ladder Marker、三磷酸腺苷 大连宝生物技术公司。
1.2 仪器与设备
DXY-33A型电泳仪 北京市六一厂;Gel Doc XR凝胶成像系统 美国伯乐公司;6400型恒温金属浴 上海东升仪器有限公司。
1.3 方法
1.3.1 大豆DNA的提取
将大豆研磨成粉末状,称取100 mg大豆粉末,加入2 mL离心管中;加入1 000 μL CTAB提取液和2 μL RNase酶溶液,充分混匀,65 ℃孵育30 min,不时振荡;12 000 r/min室温离心5 min,将上清液转移至另一干净的2 mL离心管中;加入三氯甲烷-异戊醇(24∶1,V/V)溶液,轻缓颠倒数次后室温静置5 min,12 000 r/min室温离心10 min,将上清液转移至另一干净的1.5 mL离心管中;加入2 倍体积CTAB沉淀液,颠倒混匀后,室温静置1 h;加入400 μL 1 mol/L氯化钠溶解沉淀,56 ℃孵育15 min,期间轻摇离心管数次助溶,加入800 μL经-20 ℃预冷的无水乙醇,颠倒混匀后-20 ℃静置 1 h,15 000 r/min,室温离心10 min,小心弃去上清液;体积分数70%乙醇溶液洗涤沉淀2~3 次,干燥DNA;50 μL 0.1×TE溶解沉淀,4 ℃保存备用[21]。
1.3.2 引物设计
根据抗除草剂草甘膦的转基因大豆,转入外源基因有CaMV35S、NOS和CP4-EPSPS基因,以此类基因为检测目的片段,以Lectin基因作为内源基因检测DNA的提取质量,本实验就转基因成分的引物设计一般遵循目的靶序列较小控制在100~200 bp,一定程度上克服了深加工食品高温蒸煮等加工工艺对检测方法的限制。根据美国国家生物技术信息中心公布的外源基因和内源基因的准确序列,使用Primer Premier 5.0引物设计软件,并进行BLAST比对,设计4 对特异性扩增引物如表1所示。
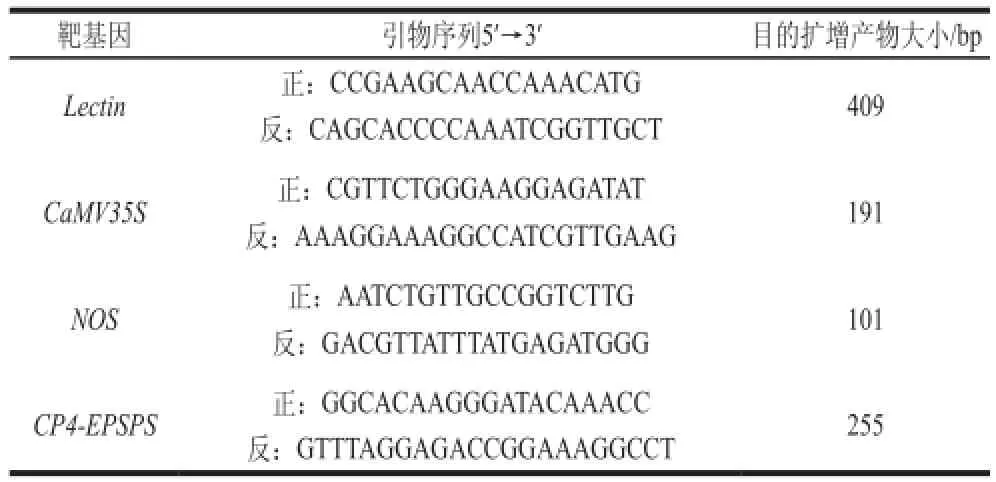
表1 引物序列与目的扩增产物大小Table 1 Primer sequences and corresponding lengths of PCR products
1.3.3 HDA扩增体系的建立
[22]优化并建立反应体系,反应体系为50 μL,采用两步法反应。
反应液Ⅰ:2 μL模板DNA,引物(10 μmol/L)各2 μL,用ddH2O补至25 μL。
反应液Ⅱ:10×buffer(100 mmol/L 二硫苏糖醇、350 mmol/L Tris-HAc、100 mmol/L MgSO4、1 mg/mL牛血清白蛋白)5 μL,dNTPs(10 mmol/L)4 μL,ATP(100 mmol/L)2 μL,Bst polymerase(8 000 U/mL)2 μL,UvrD helicase(100 mg/L)1 μL,MutL蛋白(600 mg/L)1 μL,T4 gp32(10 mg/mL)0.6 μL,海藻糖(5 mol/L)5 μL,用ddH2O补至25 μL。
反应液Ⅰ在95 ℃水浴5 min然后于55 ℃水浴退火3 min,取出后于室温将反应液Ⅱ加入反应液Ⅰ混匀,置65 ℃恒温扩增90 min。质量分数2%琼脂糖凝胶电泳(100 V)45 min检测产物。
1.3.4 HDA法检测转基因大豆特异性
利用表1所列外源基因CaMV35S、NOS、CP4-EPSPS的3 组引物和内源基因Lectin的一组引物,分别对转基因大豆和非转基因大豆进行HDA法检测。按照1.3.3节建立的HDA反应体系进行HDA法检测,验证HDA法检测的特异性。
1.3.5 HDA法检测转基因大豆的产物分析
按照1.3.3节建立的HDA反应体系,分别配制外源基因CaMV35S、NOS、CP4-EPSPS和内源基因Lectin的反应体系进行HDA扩增反应,反应结束后对扩增产物进行纯化并送专业测序机构测序,测序结果通过NCBI进行在线BLAST从而验证扩增产物的同源性。
1.3.6 HDA法检测转基因大豆的检出限
将非转基因大豆和转基因大豆按比例混合,混合后使转基因大豆所占比例为100%、10%、5%、2.5%、1%、0.5%、0.2%、0.1%、0.01%,使用高速粉碎机从低浓度到高浓度粉碎大豆样品,按照1.3.1节方法提取大豆基因组,并依据1.3.3节建立的HDA扩增体系进行PCR,确定非转基因大豆和转基因大豆混合样的检出限。
2 结果与分析
如图1所示,由于内源基因植物凝集素Lectin基因是大豆内源基因,转基因大豆和非转基因大豆均能够扩增出来409 bp的植物凝集素Lectin基因,符合理论要求,通过电泳结果证明与理论相符,因此,植物凝集素Lectin可以作为检测大豆转基因实验的内源参照基因,来达到检测整个检测体系是否正常运行的目的。
只有转基因大豆能够扩增出单一且清晰的191 bp的CaMV35S基因、101 bp的NOS基因、255 bp的CP4-EPSPS基因,而非转基因大豆没有扩增出相应的电泳条带,并且电泳条带清晰、无非特异性扩增现象,说明转基因大豆根据CaMV35S、NOS、CP4-EPSPS基因设计的引物特异性较强,可以用于转基因大豆的转基因成分的检测。
2.2 HDA法检测转基因大豆产物分析结果
扩增产物由上海生物工程公司进行测序,利用美国州立生物技术信息中心生物信息平台进行在线BLAST,对扩增产物进行同源性分析,结果均为100%,如表2所示,证明外源基因CaMV35S、NOS、CP4-EPSPS和内源基因Lectin的扩增产物为符合转基因大豆特异性要求的靶标基因,符合检测需要。
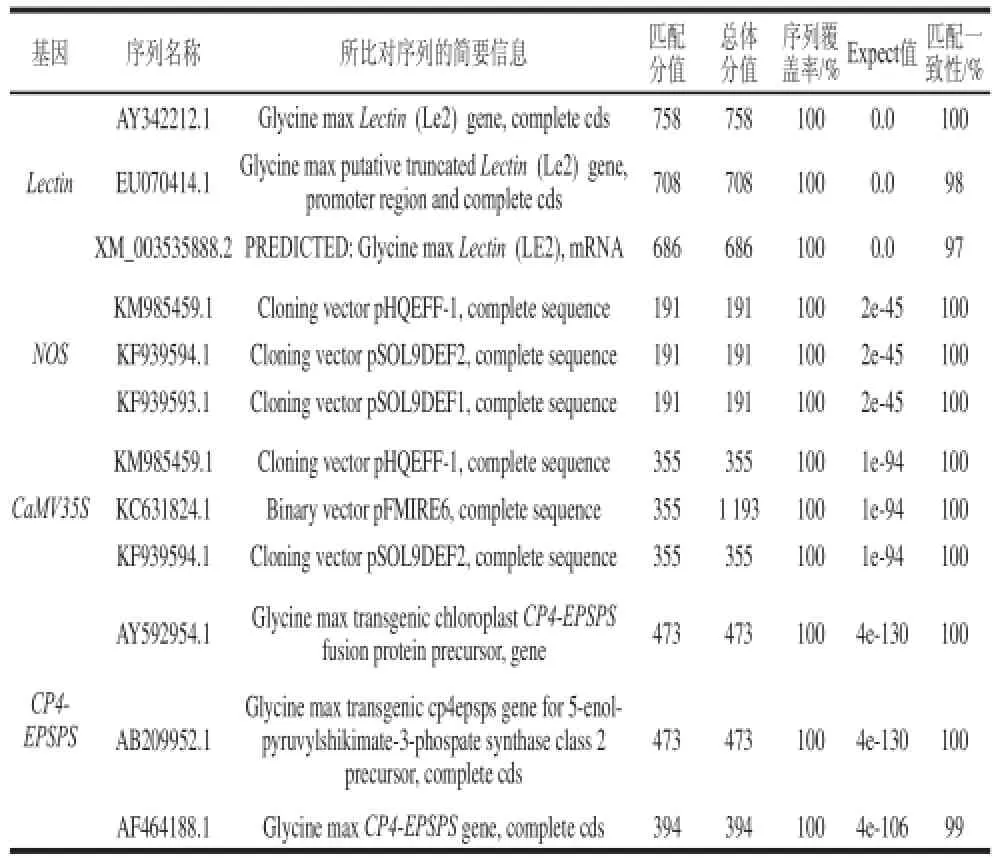
表2 HDA法检测转基因大豆扩增片断的比对结果Table 2 Homology analysis of HDA amplified fragments from genetically modified soybean by BLAST (basic local alignment search tool)
2.1 HDA法检测转基因大豆的特异性
利用设计的针对转基因大豆中常见的CaMV35S、NOS、CP4-EPSPS 3 种外源基因和大豆内源基因植物凝集素Lectin基因的4 对特异性引物进行HDA法检测,以非转基因大豆DNA作为阴性对照,检测这4 对引物的特异性。
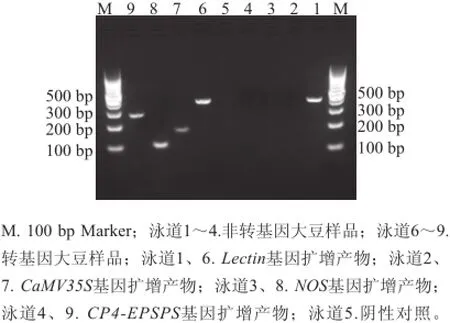
图1 HDA法检测转基因大豆的特异性Fig.1 Specificity of HDA for the detection of transgenetic soybean
2.3 HDA法检测转基因大豆的检出限
将已知的转基因大豆和已知的非转基因大豆按照不同比例混合,转基因大豆所占的比例分别为100%、10%、5%、2.5%、1%、0.5%、0.2%、0.1%、0.01%,按照1.3.1节建立的大豆基因组DNA提取方法提取大豆基因组,并配以非转基因大豆对照和阴性对照,按1.3.3节建立的HDA扩增体系进行扩增,确定大豆不同外源基因的检出限。
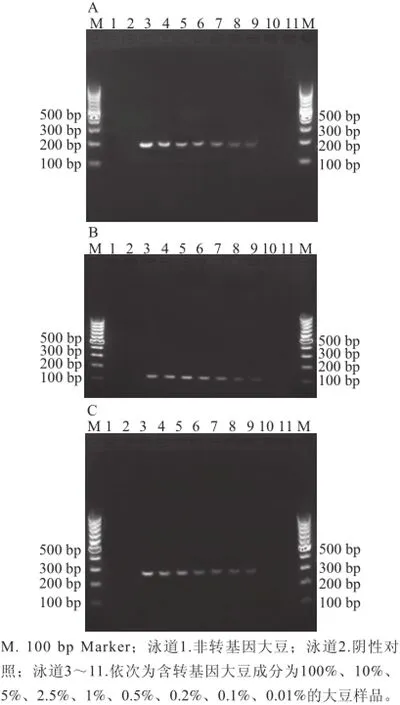
图2 HDA法检测转基因大豆外源基因CaMV35S(A)、NOS(B)、CP4 EPSPS(C)的检出限Fig.2 Electrophorograms showing the detection limit of HDA for CaMV35S (A), NOS (B) and CP4-EPSPS (C) genes
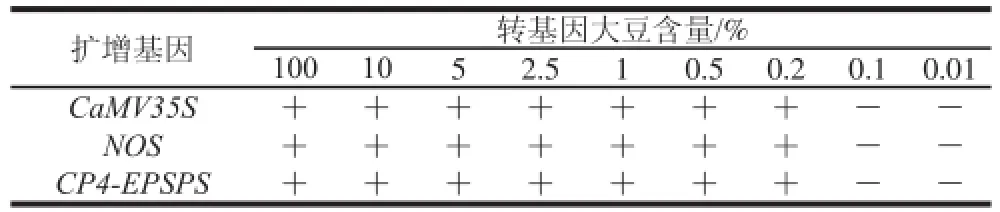
表3 HDA法检测转基因大豆的最出限Table 3 The detection limit of HDA for genetically modified soybean
由图2和表3可知,转基因大豆含量在100%、10%、5%、2.5%、1%、0.5%、0.2%时,外源基因CaMV35S、NOS、CP4-EPSPS均能够产生单一、清晰的目的基因片段条带,长度分别约为191、101、255 bp,符合特异性引物设计目的,而转基因大豆含量在0.1%、0.01%时则无清晰可见的目的电泳条带产生,因此,可以证明HDA法检测大豆不同外源基因的检出限为0.2%。
3 讨 论
目前转基因大豆的检测已经有了许多相关研究,黄昆仑等[9]用巢式和半巢式对大豆中的内源基因Lectin和转基因大豆中的外源基因进行了定量检测;励建荣等[4]通过设计特异性引物和探针对豆粕中外源基因CaMV35S、NOS、CP4-EPSPS进行了定性检测;王华[5]、叶可萍[7]等采用实时荧光定量PCR技术,通过使用特异的引物和探针对转基因大豆外源基因进行检测;Thongsri等[23]使用巢式PCR针对转基因大豆颗粒中提取的DNA作为模板梯度稀释成不同浓度梯度的DNA溶液进行扩增反应,但经典的半巢式PCR操作繁琐,还需进行两轮PCR反应,会大大增加污染的可能性;张秀丰等[8]建立了五重PCR反应体系用于检测转基因大豆;邵碧英等[11]据转基因大豆A2704-12品系的特异序列设计环介导等温扩增引物建立了转基因大豆A2704-12品系的LAMP检测方法,袁瑛娜等[12]以CP4-EPSPS基因的保守区域设计内、外引物和环引物通过采用改良环介导等温扩增技术检测抗草甘膦转基因大豆;曾有研究[24]报道使用HDA法可用于检测人乳头瘤病毒导致的宫颈癌;Zahradnik等[25]针对几种常用等温扩增方法在检测转基因木薯方面进行比较,发现针对转基因木薯的CaMV35S基因设计引物,使用HDA和环介导等温扩增方法并进行检测效果较好,并且HDA法检测限可以达到0.5%[25]。
目前常用的转基因大豆检测方法是实时荧光PCR技术,通过在线实时监测PCR反应并捕捉荧光信号强度来实现即时定量地检测待检样品中转基因成分,并且无需打开扩增体系这样封闭操作,也不会造成气溶胶污染的潜在污染情况的发生,毫无疑问是检测行业首推的检测方法;环介导等温扩增技术虽然也具备不需要昂贵的仪器设备这样的优势,但因其引物的灵敏度极高很容易造成假阳性,因此实际检测过程中也很难得到应用。在检测效率以及防止交叉污染方面,本研究建立HDA技术检测转基因大豆方法无法与荧光定量PCR相媲美,但HDA技术因其不需要昂贵的PCR仪,引物设计简单无需荧光探针,且操作简便,仅需要恒温水浴槽就能进行反应,所以在很多条件受限的基层检验检测机构还是有一定应用前景[26],同时HDA法假阳性率相对较低,很好地保证了检测结果的可信性。随着转基因技术的发展,转基因植物品系确实越来越多,本实验应用HDA技术检测转基因大豆成分只是针对CaMV35S、NOS、CP4-EPSPS等外源基因的筛查检测,无法做到品系鉴定,确实滞后于当前转基因检测技术的发展要求,相信通过筛查检测的实验积累和条件优化摸索可以为实现品系鉴定打下一定的技术基础。随着转基因作物种植面积的日益增加以及消费者对转基因产品关注度的日渐提高,使得转基因的检测任务也将日趋繁重,这就要求检测方法具备高特异性、高灵敏度和操作简便,因此HDA法用于转基因大豆的检测工作将会有很广泛的应用前景。
4 结 论
针对转基因大豆外源调控序列CaMV35S、NOS、CP4-EPSPS基因和目的基因Lectin序列设计引物,成功建立了检测转基因大豆中转基因成分的HDA方法。HDA法检测转基因大豆的特异性强,无非特异性扩增,方法检测检出限为0.2%。
参考文献:
[1] 刘佳, 刘志华, 徐广惠, 等. 抗草甘膦转基因大豆(RRS)根际微生物和土壤氮素转化的影响[J]. 农业环境科学学报, 2010, 29(7): 1341-1345.
[2] 陈笑芸, 汪小福, 周育, 等. 转基因大豆深加工食品DNA鉴定技术研究[J]. 中国食品学报, 2013, 13(4): 156-162.
[3] 刘欣, 张国丛, 周兴虎, 等. 转基因大豆MON89788实时荧光PCR检测方法的建立[J]. 食品科学, 2015, 36(4): 193-197. DOI:10.7506/ spkx1002-6630-201504038.
[4] 励建荣, 余春燕. 豆粕中转基因成分检测技术研究[J]. 中国粮油学报, 2007, 22(1): 114-117. DOI:10.3321/j.issn1003-0174.2007.01.029.
[5] 王华, 梁怀宇, 李华. SYBR Green实时荧光PCR高通量检测转基因大豆油[J]. 生物技术通报, 2011(8): 103-107. DOI:10.13560/j.cnki. biotech.bull.1985.2011.08.009.
[6] LERAT S, ENGLAND L S, VINCENT M L, et al. Real-time polymerase chain reaction quantification of the transgenes for roundup ready corn and roundup ready soybean in soil samples[J]. Journal of Agricultural and Food Chemistry, 2005, 53(5): 1337-1342. DOI:10.1021/jf048830.
[7] 叶可萍, 祝长青, 周光宏. 利用Taqman实时荧光PCR检测市售肉制品中转基因大豆成分[J]. 食品与发酵工业, 2010, 36(12): 160-164. DOI:10.13995/j.cnki.11-1802/ts.2010.12.037.
[8] 张秀丰, 苏旭东, 张伟, 等. 五重PCR检测转基因大豆[J]. 中国粮油学报, 2009, 23(3): 194-198.
[9] 黄昆仑, 罗云波. 用巢式和半巢式PCR检测转基因大豆Roundup Ready及其深加工食品[J]. 农业生物技术学报, 2003, 11(5): 461-466. DOI:10.3969/j.issn.1674-7968.2003.05.005.
[10] 闫伟, 徐桢惠, 龙丽坤, 等. 应用单管半巢式PCR技术筛查转基因食品[J]. 食品科学, 2015, 36(2): 194-197. DOI:10.7506/spkx1002-6630-201502037.
[11] 邵碧英, 陈文炳, 曾莹, 等. LA MP法检测转基因大豆A2704-12品系[J].食品科学, 2013, 34(24): 202-207. DOI:10.7506/spkx1002-6630-201324042.
[12] 袁瑛娜, 单潇潇, 王宗德, 等. 应用LAMP实时浊度法检测转基 因大豆[J]. 现代食品科技, 2011, 27(10): 1264-1267. DOI:10.13982/ j.mfst.1673-9078.2011.10.004.
[13] 白卫滨, 孙建霞, 姜桂传, 等. ELISA方法定量检测转基因大豆及其产品的研究[J]. 食品与发酵工业, 2008, 33(11): 103-106. DOI:10.13995/j.cnki.11-1802/ts.2007.11.029.
[14] CAO Y, KIM H J, LI Y, et al. Helica se-dependent amplification of nucleic acids[J]. Current Protocols in Molecular Biology, 2013, 104: 1511-1512. DOI:10.1002/04711427 27.mb1511s104.
[15] CHEN X, WU X, GAN M, et al. Rapid detection of Staphylococcus aureus in dairy and meat foods by combination of capture with silicacoate d magnetic nanoparticles and thermophilic helicase-dependent isothermal amplification[J]. Journal of Dairy Science, 2015, 98(3): 1563-70. DOI:10.3168/jds.2014-8828.
[16] BARREDA-GARCIA S, GONZALEZ-ALVAREZ M J, DELOS-SANTOS-ALVAREZ N, et al. Attomolar quantitation of Mycobac terium tuberculosis by asymmetric helicase-dependent isothermal DNA-amplification and electrochemical detection[J]. Biosens Bioelectron, 2014, 68: 122-128. DOI:10.101 6/ j.bios.2014.12.029.
[17] LI Y, KUMAR N, GOPALAKRISHNAN A, et al. Detection and species ide ntification of malaria parasites by isothermal tHDA amplification directly from human blood without sample preparation[J]. Journal of Molecular Diagnostics, 2013, 15( 5): 634-641. DOI:10.1016/j.jmoldx.2013.05.005.
[18] HUANG S, DO J, MAHALANABIS M, et al. Low cost extraction and isothermal amplification of DNA for infectious diarrhea diagnosis[J]. PLoS One, 2013, 8(3): e60059. DOI:10.1371/journal.pone.0060059.
[19] ECKERT C, HOLSCHER E, PETI T A, et al. Molecular test based on isothermal helicase-dependent amplification for detection of the Clostridium difficile toxin A gene[J]. Journal of Cl inical Microbiology, 2014, 52(7): 2386-2389. DOI:10.1128/JCM.00353-14.
[20] MOURA-MELO S, MIRANDA-CASTRO R, DE-LOS-SANTOSALVAREZ N, et al. Targeting helicase-depe ndent amplification products with an electrochemical genosensor for reliable and sensitive screening of genetically-modified organisms[J]. Analytical Chemistry, 2015, 87(16): 8547-8554. DOI:10.1021/acs.analchem.5b02271.
[21] 山东出入境检验检疫局, 汕头出入境检验检疫局, 广州出入境检验检疫局, 等. GB/T 19495.3—2004 转基因产品检测: 核酸提取纯化方法[S].
[22] 王建广, 雷质文, 刘云国, 等. 志贺菌依赖解旋酶DNA恒温扩增技术建立[J]. 中国公共卫生, 2012, 28(4): 550-552.
[23] THONGSRI Y, WONGLAKORN L, CHAIPRASERT A, et al. Evaluation for the clinical diagnosis of Pythium insidiosu m using a single-tube nested PCR[J]. Mycopathologia, 2013, 176(5/6): 369-376. DOI:10.1007/s11046-013-9695-3.
[24] BARBIERI D, VENTUROLI S, ROSL F, et al. Detection of high-risk human papillomavirus type 16 and 18 using isothermal helicase-dependent amplification[J]. Diagnostic Microbiology and Infe ctious Disease, 2014, 79(2): 178-182. DOI:10.1016/ j.diagmicrobio.2014.02.012.
[25] ZAHRADNIK C, KOLM C, MARTZY R, et al. Detection of the 35S promoter in transgenic maize via various isothermal amplification techniques: a practical approach[J]. Analytical and Bioanalytical Chemistry, 2014, 406(27): 6835-6842. DOI:10.1007/s00216-014-7889-2.
[26] YANG Z, MCLENDON C, HUTTER D, et al. Helicase-dependent isothermal amplification of DNA and RNA by using self-avoiding molecular recogni tion systems[J]. Chembiochem, 2015, 16(9): 1365-1370. DOI:10.1002/cbic.201500135.
Detection of Genetically Modified Soybean by Helicase-Dependent Isothermal DNA Amplification Assay
ZHANG Jingjing1, WANG Zan1, ZHANG Cuixia1, MA Chaofeng1, ZHOU Wei1,2,*, ZHANG Yan1
(1. Hebei Food Safety Key Laboratory, Hebei Food Inspection and Research Institute, Shijiazhuang 050071, China; 2. College of Food Science and Technology, Agricultural University of Hebei, Baoding 071000, China)
Objective: To establish a new rapid method to detect genetically modifi ed soybean based on helicase-dependent isothermal DNA amplification (HDA). Methods: With four highly specific sets of primers synthesized to target genes including Lectin in soybean and CaMV35S, NOS and CP4-EPSPS foreign genes, PCR reaction system was established. The specifi city and sensitivity of the PCR method were tested based on HDA and homology analysis was performed. Results: The helicase-dependent isothermal DNA amplifi cation method for the rapid detection of genetically modifi ed soybean has been established. The detection limits of CaMV35S, NOS and CP4-EPSPS genes in genetically modifi ed soybeans by HDA were all 0.2%. Conclusion: HDA is a rapid, user-friendly, specifi c and sensitive method for the detection of genetically modifi ed soybean, and is very suitable for use in grassroots laboratories with a broad prospect.
helicase-dependent isothermal DNA amplifi cation (HDA); genetically modifi ed soybean; detection
10.7506/spkx1002-6630-201604029
TS207.3
A
1002-6630(2016)04-0164-05
章晶晶, 王赞, 张翠侠, 等. 应用依赖解旋酶DNA扩增技术检测转基因大豆[J]. 食品科学, 2016, 37(4): 164-168.
DOI:10.7506/spkx1002-6630-201604029. http://www.spkx.net.cn
ZHANG Jingjing, WANG Zan, ZHANG Cuixia, et al. Detection on genetically modified soybean by helicase-dependent isothermal DNA amplification assay[J]. Food Science, 2016, 37(4): 164-168. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201604029. http://www.spkx.net.cn
2015-06-07
国家自然科学基金面上项目(31401584)
章晶晶(1988—),女,助理工程师,硕士,研究方向为生物化学与分子生物学。E-mail:zhangjj@qibebt.ac.cn
*通信作者:周巍(1983—),男,高级工程师,博士研究生,研究方向为农产品加工及贮藏工程。E-mail:zhouwei0311@163.com
