“温138”核桃硬壳初期转录组序列的初步研究
2016-10-28贾昌路徐崇志
贾昌路 徐崇志 张 锐,3*
(1 塔里木大学植物科学学院, 新疆 阿拉尔 843300)(2 新疆生产建设兵团塔里木盆地生物资源保护利用重点实验室, 新疆 阿拉尔 843300)(3 塔里木大学生命科学学院, 新疆 阿拉尔 843300)
“温138”核桃硬壳初期转录组序列的初步研究
贾昌路1徐崇志2张 锐2,3*
(1 塔里木大学植物科学学院, 新疆 阿拉尔 843300)(2 新疆生产建设兵团塔里木盆地生物资源保护利用重点实验室, 新疆 阿拉尔 843300)(3 塔里木大学生命科学学院, 新疆 阿拉尔 843300)
为了探究“温138”核桃硬壳出现露仁现象的根本原因,从分子水平出发,运用转录组测序技术,探索出硬壳中基因的功能。经测序后,共得到51 521 252个reads片段,包含4 636 912 680个核苷酸序列信息,对 reads 进行拼接组装后,共得到56 639条Unigene,序列信息长度达到了46 623 389 nt;将Unigene 和 COG、GO数据库进行比对表明,核桃硬壳转录组中的 Unigene 根据COG功能可分为 25 类;根据GO功能可分为生物过程、细胞组分和分子功能3大类53小类。
核桃;硬壳;转录组;基因功能
核桃(Juglans regia L.)又名胡桃、羌桃,为胡桃科胡桃属多年生落叶果,是世界著名的四大干果之一。核桃中含有较为丰富的蛋白质,被广泛认为是优质的植物蛋白资源,经研究表明,核桃蛋白有着与动物蛋白相近的营养价值[1]。中国是核桃发源地之一,在悠久的栽培历史中,由于核桃分布广范,地理条件和气候条件不同,加上人们长期的观察和选育,形成了极为丰富的种质资源,如:隔年核桃、薄皮核桃、穗状核桃等,分布范围包括河北、云南、新疆等地[2]。
所谓转录组,从广义上讲,是指生物体(包括生物体中的细胞、组织等)中出现的所有RNA的总和,包括能够编码蛋白质的RNA(即mRNA)和不能编码蛋白质的RNA(ncRNA,如rRNA、tRNA、mieroRNA等);从狭义上讲通常是指细胞所转录出的所有mRNA的总和[3-4]。随着科学技术的发展,人们在研究生物的过程中开始向后基因组时代挺进,陆续出现了转录组学、蛋白质组学、代谢组学等各种组学技术,在诸多组学中,以蛋白质组学和转录组学为研究重点,而转录组学又较蛋白质组学率先发展起来且应用广泛[5-6]。目前已有不少学者做过有关植物转录组的研究,2011年熊丽东[7]采用Solexa测序技术对红花转录组进行了测序,结果共得到153 769条红花Unigenes,随后对Unigenes进行了功能注释,结果显示:将Unigenes与nr库比对,得到相似基因65 536条;与Swiss-Prot库比对,得到相似基因56 843条;与KEGG库比对,得到相似基因35 056条;与COG库比对,得到相似基因11 010条。2012年李铁柱等[8]对杜仲幼果和成熟果实进行测序后,共获得了64 474个Unigene片段,随后将Unigene和COG库进行比对,发现共存在65 536条Unigene与COG库中的基因相似,与GO库进行比对,相似基因共有12 923条。2012年杨楠等[9]以红心蜡梅H29为材料,利用Illumina测序平台得到105个与花色合成相关的Unigene,对Unigene分析发现,其平均长度677 bp,在200~500 bp之间的有46个片段,在500~1 000 bp之间的有24个片段,大于1 000 bp的有21个片段。将Unigene与nr数据库进行比对,发现Unigene都具有较高的同源度(E值<10-5),其中存在45条片段与nr数据相比,同源度极高(E值<10-50)。
“温138”核桃是从原产于新疆的纸皮核桃的实生后代中选育出来的一个核桃品种,该品种与纸皮核桃相比,品质差异不大(通过化学测定方法对两个核桃品种中主要营养物质的测定结果)。但是,“温138”核桃不同于纸皮核桃的特性主要表现为“温138”核桃在成熟后,核桃硬壳包被不完全,出现露仁现象。为了研究“温138”核桃露仁的主要原因,本文借助于现有的高效测序技术——转录组测序技术,分析“温138”核桃硬壳中遗传信息,为从分子角度探究“温138”核桃露仁的真正原因奠定基础。
1 试验材料与方法
1.1试验材料
试验以新疆阿克苏地区温宿县木本粮油核桃林场种植的“温138”核桃为试验材料,于2014年6月13日进行采样,将采摘的核桃果实去青皮,用小刀将核桃切成片状,剔除核桃内的果仁,仅将核桃的内果皮保留,并迅速切碎,用液氮进行速冻,保存至-70℃冰箱,备用。
1.2核桃硬壳RNA提取
核桃硬壳中总RNA的提取采用pBIOZOL植物组织RNA提取试剂盒(BioFlux),具体操作参照说明书进行。
1.3核桃硬壳转录组的测序、数据组装及基因功能注释
转录组测序工作委托于深圳华大公司完成。对核桃硬壳形成过程中的转录组进行测序后,将测序图像数据经base calling转化为序列数据(raw reads),然后再通过filter_fq软件对转化后的数据进行过滤,得到最终所需的数据(clean reads),然后进行数据分析。对最终数据进行组装,得到所需要的Unigene,最后再对所得的Unigene进行分析、功能注释。
2 结果与分析
对核桃硬壳中的RNA进行测序后,共得到55 585 658条reads片段,利用filter_fq软件对reads进行过滤后共得到51 521 252个reads片段,其中包含有4 636 912 680个核苷酸序列信息,质量大于20%的碱基占97.94%,中间未知序列片段为0%,GC%含量达到了46.18%,由此可以看出,所测的转录组序列较为可靠,能够为数据组装提供很好的数据。
2.1核桃硬壳转录组数据组装
用over-lap的方法对51 521 252个reads片段进行拼接,共得到Contig片段94 301条,序列信息长度达到了39 186 004 nt,平均长度为416 nt;其中,长度在100-200 nt范围内的Contig片段达到了51 008条,比例占到了54.09%;在200-300 nt范围内的Contig片段达到了14 017条,比例占到了14.86%;在300-400 nt范围内的Contig片段达到了6 868条,比例占到了7.28%;在400-500 nt范围内的Contig片段达到了4 002条,比例占到了4.27%;大于等于500 nt的Contig片段达到了18 386条,比例占到了19.50%(如表1、图2所示)。由以上数据不难发现,用over-lap的方法对reads片段进行拼接后得到的Contig片段效果较好。

表1 核桃硬壳转录组Contig数据组装情况统计
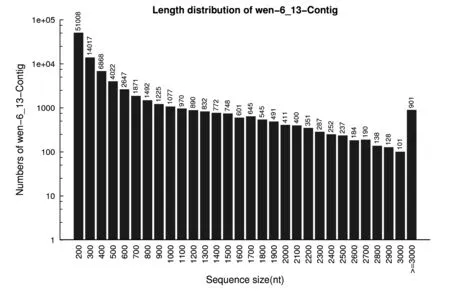
图2 核桃硬壳转录组Contig数据长度分布图
由reads片段拼接组装得到Contig片段后,再利用Contig片段数据进行进一步的拼接组装,得到Unigene。通过拼接组装后,共得到56 639条Unigene,序列信息长度达到了46 623 389 nt,平均长度为823 nt,N50为 1 543 nt;其中,长度在100~500 nt范围内的Unigene片段达到了30 701条,比例占到了54.20%;在500~1 000 nt范围内的Unigene片段达到了9 434条,比例占到了16.66%;在1 000~1 500 nt范围内的Unigene片段达到了6 444条,比例占到了11.38%;在1 500~2 000 nt范围内的Unigene片段达到了4 325条,比例占到了7.64%;大于等于2 000 nt的Unigene片段达到了5 735条,比例占到了10.13%(如表2、图3所示)。

表2 核桃硬壳转录组Unigene数据组装情况统计

图3 核桃硬壳转录组Unigene数据长度分布图
将Unigene 进行覆盖度分析,结果发现,有37 071条 Unigene 能够与测序的原始数据 reads 相对应,并且对应关系也各不相同,depth(测序深度——即reads碱基数与Unigene序列长度的比值)和coverage(覆盖度——即Unigene中有reads覆盖的碱基数与Unigene序列长度的比值)表示,其中,depth在0. 059 9-33 899. 706 6的范围内,coverage在4. 80%-100. 00%的范围内。对应长度在200-15 681 nt,GC%在14.85%-71.98%,不存在未知序列,说明通过拼接组装后得到的Unigene效果较好。
2.2Unigene的功能分类与分析
2.2.1Unigene 的COG功能的分类及分析
COG(Cluster of Orthologous Groups of proteins)是对基因产物进行直系同源分类的数据库,是基于细菌、藻类、真核生物具有完整基因组的编码蛋白、系统进化关系进行构建的,将Unigene和COG数据库进行比对,预测Unigene可能的功能并对其做功能分类统计,进而从宏观上了解相应物种的基因功能分布特征。
通过将样品的Unigene与COG数据库进行比对,发现共有43 521条Unigene与COG数据库中的基因具有相似性。将比对出具有相似性的Unigene进行分类,可以分为25类(如图4所示),并对每一类的基因数量进行统计(表3)。从图与表中可以看出,Unigene的COG功能比较全面,其中,涉及到普通功能的Unigene数量最多,为8 196条;而与核结构有关的基因只有10条,与细胞外结构有关的基因只有12条,其他相关功能的基因也有不同的差异,如表3所示。

图4 核桃硬壳转录组Unigene COG功能分类统计图

功能代号COG功能分类基因数量ARNAprocessingandmodification384BChromatinstructureanddynamics468CEnergyproductionandconversion1043DCellcyclecontrol,celldivision,chromosomepartitioning1498EAminoacidtransportandmetabolism1680FNucleotidetransportandmetabolism425GCarbohydratetransportandmetabolism2490HCoenzymetransportandmetabolism764ILipidtransportandmetabolism912JTranslation,ribosomalstructureandbiogenesis2243KTranscription4658LReplication,recombinationandrepair3987MCellwall/membrane/envelopebiogenesis1464NCellmotility320OPosttranslationalmodification,proteinturnover,chaperones3013PInorganiciontransportandmetabolism1326QSecondarymetabolitesbiosynthesis,transportandcatabolism968RGeneralfunctionpredictiononly8196SFunctionunknown1896
续上表

功能代号COG功能分类基因数量TSignaltransductionmechanisms3883UIntracellulartrafficking,secretion,andvesiculartransport989VDefensemechanisms314WExtracellularstructures12YNuclearstructure10ZCytoskeleton578
2.2.2Unigene 的GO功能分类与分析
Gene Ontology(简称GO)是一个国际标准化的基因功能分类体系,是通过一套动态更新的标准词汇表(controlled vocabulary)来全面描述生物体中基因和基因产物的属性。GO从三个方面对基因进行了描述,分别为基因的分子功能(molecular function)、细胞组分(cellular component)、参与的生物过程(biological process)。根据NR注释信息,使用Blast2GO软件得到Unigene的GO注释信息,然后用WEGO软件对所有Unigene做GO功能分类统计,从宏观上认识该物种的基因功能分布特征。
通过对样品Unigene的GO功能进行分析,共有7 419条Unigene与数据库中的基因呈现出相似性,且存在着单条Unigene与多种基因相对应的现象,并建立了一定的对应关系,从而可以得到3大类53小类的分类情况,结果如图5所示,并对每一小类的基因数进行统计,结果如表4所示。结合Unigene GO 功能分类图与分类表,可以看出,在核桃硬壳期转录组序列的GO功能中,参与生物过程的基因总数远高于表现出分子功能的基因总数,在三大主要基因功能中,调控细胞与细胞分裂的基因最多,达到了5 048个,而在基因的分子功能区中,具有连接功能的基因数最多,达到了3 675个,在生物过程区中,调控细胞过程的基因数最多,达到了4 717个,其他功能的基因数也存在着较大的差异,具体统计如表4所示。

图5 核桃硬壳转录组Unigene GO功能分类

OntologyClassNumberbiological_processbiologicaladhesion59biological_processbiologicalregulation1960biological_processcellularcomponentorganizationorbiogenesis1266biological_processcellularprocess4717biological_processdevelopmentalprocess1340biological_processestablishmentoflocalization1217biological_processgrowth357biological_processimmunesystemprocess416biological_processlocalization1304biological_processlocomotion9biological_processmetabolicprocess4520biological_processmulti-organismprocess767biological_processmulticellularorganismalprocess1324biological_processnegativeregulationofbiologicalprocess475biological_processpositiveregulationofbiologicalprocess421biological_processregulationofbiologicalprocess1801biological_processreproduction715biological_processreproductiveprocess672biological_processresponsetostimulus2667biological_processrhythmicprocess46biological_processsignaling821biological_processsingle-organismprocess3278cellular_componentcell5408cellular_componentcelljunction332cellular_componentcellpart5408cellular_componentextracellularmatrix7cellular_componentextracellularmatrixpart1cellular_componentextracellularregion510cellular_componentextracellularregionpart5cellular_componentmacromolecularcomplex726
续上表

OntologyClassNumbercellular_componentorganelle4181cellular_componentorganellepart1215cellular_componentsymplast332cellular_componentvirion1cellular_componentvirionpart1molecular_functionantioxidantactivity48molecular_functionbinding3675molecular_functioncatalyticactivity3670molecular_functionelectroncarrieractivity102molecular_functionenzymeregulatoractivity90molecular_functionmetallochaperoneactivity2molecular_functionmoleculartransduceractivity123molecular_functionnucleicacidbindingtranscriptionfactoractivity235molecular_functionnutrientreservoiractivity16molecular_functionproteinbindingtranscriptionfactoractivity21molecular_functionproteintag1molecular_functionreceptoractivity59molecular_functionstructuralmoleculeactivity188molecular_functiontransporteractivity491
3 讨论
2014年,蒋弘刚[10]对花椒皮刺中的转录组进行分析,共得到2*100 bp的原始数据,经过拼接组装后得到45 057条长度大于200 bp的Unigene,总长度为610 bp,N50为846 bp。将得到的Unigene与COG、GO数据库进行比对、注释,分别注释了70.26%,43.34%的Unigene。
2012年,李铁柱等[11]对杜仲的果实与叶片转录组数据进行分析,共得到54 471 338条reads,对reads进行拼接组装后,得到452 421条Contig,总长度达到了90 705 736 nt;对Contig进一步拼接组装后,得到49 610条Unigene,总长度达到了37 616 729 nt。随后,将Unigene与COG、GO数据库进行比对、注释,发现有与COG、GO数据库中的基因具有相似性的Unigene条数分别为125 934条、8 260条。
2013年,杜艳玲[12]运用 HiSeqTM 2000测序平台,对人参根、茎、叶的转录组序列进行组装,分别获得53 870,69 591,66 045条 Unigenes;序列的平均长度分别为553 nt,686 nt,644 nt。将人参根、茎、叶的Unigene分别与GO数据库进行比对,有 30 519、37 539、36 078 条Unigenes被归类到61个GO功能类别中;分别与COG数据库进行比对,有11 755、15 646、14 803条Unigenes 被归类到25个COG功能类别中。
实验对“温138”核桃硬壳中的转录组进行测序后,共得到51 521 252个reads片段,其中包含有4 636 912 680个核苷酸序列信息,对 reads 进行拼接组装,共得到94 301条Contig片段,序列信息长度达到了39 186 004 nt,平均长度为416 nt,在Contig 数据的基础上,进一步进行拼接组织,共得到56 639条Unigene,序列信息长度达到了46 623 389 nt,平均长度为823 nt,N50为 1 543 nt;将 Unigene与 COG 数据库进行比对,发现共有43 521条Unigene与COG数据库中的基因相似,与GO数据库进行比对,共有7 419条Unigene与GO库中的基因相似。
4 结论
与前人在转录组方面的研究相比,本次实验所得的转录组序列信息量大,且组装效果较好,得到的Unigene的可信度高,能够反映出“温138”核桃硬壳中基因的功能情况。
[1]刘玲,韩本勇,陈朝银.蛋白质研究进展[J].食品与发酵工业,2009,35(9):116-118.
[2]郗荣庭,张毅萍.中国果树志·核桃卷[M].1994:83-87
[3]史硕博,陈涛,赵学明.转录组平台技术及其在代谢工程中的应用[J].生物工程学报,2010,26(9):1187-1198.
[4]Costa V,Angelini C,De Feis I,Ciccodicola A.Uncovering the complexity of transcriptomes with RNA-Seq,J Biomed Biotechnol,2010,12:853-916.
[5]Lockhart D J,Winzeler E A.Genomics,gene express and DNA arrays.Nature,2000,405(6788): 827-836.
[6]张春兰,秦孜娟,王桂芝,等.转录组与RNA-Seq技术[J].生物技术通报,2012,12:51-56.
[7]熊丽东.红花转录组测序分析及其油体蛋白基因全长的获得[D].吉林:吉林农业大学,2011.
[8]李铁柱,杜红岩,刘慧敏,等.杜仲幼果和成熟果实转录组数据组装及基因功能注释[J].中国林业科技大学学报,2012,32(10):9-17.
[9]杨楠,赵凯歌,陈龙清.蜡梅花转录组数据分析及次生代谢产物合成途径研究[J].北京林业大学学报,2014,34(1):104-107.
[10]蒋弘刚.花椒皮刺分化转录组测序及数据分析[D].陕西:西北农林科技大学,2014.
[11]李铁柱,杜红岩,刘慧敏,等.杜仲果实和叶片转录组数据组装及基因功能注释[J].中国林业科技大学学报,2012,32(11):122-130.
[12]杜艳玲.人参根、茎、叶转录组测序及差异表达基因分析[D].吉林:长春中医大学,2013.
The Study of Transcriptome Sequences in Harden Endocarp of ‘Wen138’ Walnut
Jia Changlu1Xu Chongzhi2Zhang Rui2,3*
(1 College of Plant Science and Technology,Tarim University,Alar, Xinjiang 843300) (2 Xinjiang Production and Construction Corps Key Laboratory of Protection and Utilization of Biological Resources in Tarim Basin, Alar, Xinjiang 843300) (3 College of Life Science,Tarim University,Alar, Xinjiang 843300)
In order to explore the reason of bared nut for ‘Wen 138’ walnut, transcriptome sequencing technology were used, the function of genes in hard shell walnut was investigated. A total of 51, 521, and 252 reads fragment contains 4, 636, 912 and 680 nucleotides sequence were obtained, and got 56 639 Unigene fragment after splicing and assembling with the sequence information of 46 623 389 nt;Aligned with the COG database, Unigene in the transcriptome of ‘Wen 138’walnut could be broadly divided into 25 classes according to the function. The Unigene GO functions could be broadly divided into biological processes, cellular components and molecular function major major categories of 53 subsecs.
walnut; hard shell; transcriptome; gene ontology
2015-08-17
国家自然科学基金(31260469)
贾昌路(1991-),男,2014级硕士研究生,主要从事核桃露仁分子机理研究。E-mail:429315095@qq.com
�E-mail:zhrgsh@163.com
1009-0568(2016)03-0011-09
S664.1;Q789
ADOI:10.3969/j.issn.1009-0568.2016.03.003
