基于小生境改进的遗传算法求解多序列比对问题
2016-10-26张继成羊秋玲
张继成,羊秋玲
(1.长江大学工程技术学院,湖北荆州434020;2.海南大学信息科学技术学院,海南海口570228)
ZHANG Jicheng1,YANG Qiuling2
(1.Yangtze University College of Technology & Engineering,Jingzhou 434020;2.College of Information Science & Technology,Hainan University,Haikou 570228)
基于小生境改进的遗传算法求解多序列比对问题
张继成1,羊秋玲2
(1.长江大学工程技术学院,湖北荆州434020;2.海南大学信息科学技术学院,海南海口570228)
基于多核平台设计了一个求解多序列比对问题的改进遗传算法。该算法采用一致性函数作为个体的适应度函数,引进小生境技术,维持种群进化的多样性,以改善算法的整体搜索能力。考虑遗传算法本身具有较好的并行性,对其各算子针对多核平台进行了并行化设计。通过对BAliBASE中的测试例进行测试,与已有的算法相比取得了更优的结果,证明该算法是有效的。并行化设计使算法在多核平台的运行时间显著缩短,加速效果明显。
多序列比对;小生境遗传算法;多核;并行
ZHANG Jicheng1,YANG Qiuling2
(1.Yangtze University College of Technology & Engineering,Jingzhou 434020;2.College of Information Science & Technology,Hainan University,Haikou 570228)
0 引言
生物序列分析的一个关键问题之一就是进行多序列比对。运用多序列比对可以发现序列采用何种模式,挖掘出更多的保守区域,同时也可以运用到系统发育分析、结构预测等方面。在当前生物信息学中,众多研究者开始研究多序列比对问题[1-4]。如果采用人工对比,则工作量相对复杂,同时也要兼顾生物序列中的功能具有不确定性,因此无法用生物意义对比对的效果进行统一衡量,故人们把比对后各个序列之间差异的大小来主观地作为衡量的标准。已有的计算差异性的数据模型主要有3种[5]:一致性函数(consensus functions)、比对和函数(sum-of-pairs functions)、树函数(tree functions)。使用最普遍的是一致性函数,其分值一般简称为SP值,本文亦将采用此函数作为遗传算法的评价函数。遗传算法作为一种高效、通用的智能算法,在优化问题求解领域获得了广泛的应用。而对于遗传算法求解多序列比对问题,实现最完善的当属多序列比对软件包SAGA[6],它设计了22种不同的遗传算子,并针对这些遗传算子采取动态调度的方案来控制它们的使用,具有较好的通用性。但由于其重点在于兼顾通用的效果,因此在求解性能上不甚理想,对于规模稍大的工作量则更加难以胜任。因此也出现了一些改进的算法,如文献[7]设计了一个基于GC-GM的多序列比对穷举遗传算法(简称GC-GM-GA),取得了一定的改进效果。但GC-GM-GA仍然没有克服遗传算法的固有弱点,即过早收敛,这对求解质量仍然有极大影响,并且其穷举的策略,使算法本身复杂度增加,求解性能得不到保证。
为了提高对多序列比对问题的求解质量,本文设计了一个基于小生境技术的改进遗传算法,以小生境维持种群的多样性,达到防止算法过早收敛的目的,进而获得较好的求解效果。为了适应当前主流的多核平台,设计了针对多核平台的并行遗传算法,使之有更好的执行性能。通过对BAliBASE中的测试例进行测试,本文算法较已有算法求解质量更高,且并行化的效果也很明显,取得较好的加速比,从而证明算法的有效性。
1 问题描述
1.1多序列比对问题
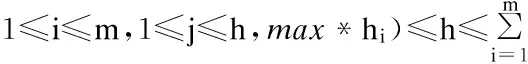
1.2多序列比对问题优化模型
多序列比对问题优化模型主要用于描述多序列的差异性,如引言所述,一致性函数(consensus functions)、比对和函数(sum-of-pairs functions)、树函数(tree functions)是最常用的3种主要用来计算差异性的数据模型。其中一致性函数会计算分值,分值的大小由当前多序列比对M和双序列比对库之间有多少相同的信息来决定,而双序列比对库则可以通过现存的双序列比对程序来产生,如果存在m条序列的双序列比对库,就不需要替代矩阵和空位罚分的选取。采用一致性函数的比对方法必须先建立双序列比对库,也就是m条序列的两两之间最佳比对结果,共有m(m-1)/2个双序列比对。因此1个多序列比对M的一致性函数可以定义为
(1)
式中,把序列qi和qj在M中的双序列比对记为Mij,其比对长度记为LENS(Mij),Mij与库中qi和qj最佳比对的一致性结果记为SC(Mij),其值的大小为对Mij中和库中的残基对进行比较得出的一致性数目,序列qi和qj的相同度记为ij。假如Q的一个比对Q*满足COST(Q*)=max(COST(M)),那么Q*为一个最优比对,这个问题是一个NP完全问题[8]。对于这种情况,很多学者近年来都在研究用近似算法来解决多序列比对问题,也收获了一定的成果,如文献[7]设计的一个基于GC-GM的多序列比对穷举遗传算法。
1.3小生境技术
在生物学上,小生境的产生有着重要的影响,它是指在某种环境中的一种组织结构或角色,它为新物种的产生创造了有利条件。遗传算法中的小生境是指种群中以某种形式描述的个体间相似程度达到一定水平的个体的集合[9]。小生境常用的实现方法为排挤型[10],其思想源自生物界中生物通过对有限资源的竞争来获取生存机会的现象。操作过程为:首先设置一排挤因子CF,在种群中随机选取1/CF个个体用来组成排挤成员(其实大多数实现并非是随机的选取个体,而是选择按适应度值大小降序排序后排名在前的1/CF个),其次比较排挤成员与新产生的个体的相似性,从而排挤掉一些适应度值相对较小的个体。这样种群中的个体也慢慢被分门别类,伴随着排挤过程的进行,一个个小的生存环境被产生,使种群丰富多彩。小生境的这种特性有效维持遗传算法的多样性,防止其过早收敛。
2 算法设计与实现
2.1小生境遗传算法
遗传算法发展较为成熟,具有稳定的框架。本文设计算法在原有框架的基础上,引进小生境操作,在种群每次迭代后实施。为平衡小生境在遗传算法在迭代过程中的作用,设计一个参数来控制调用小生境操作的次数。小生境遗传算法如图1所示。
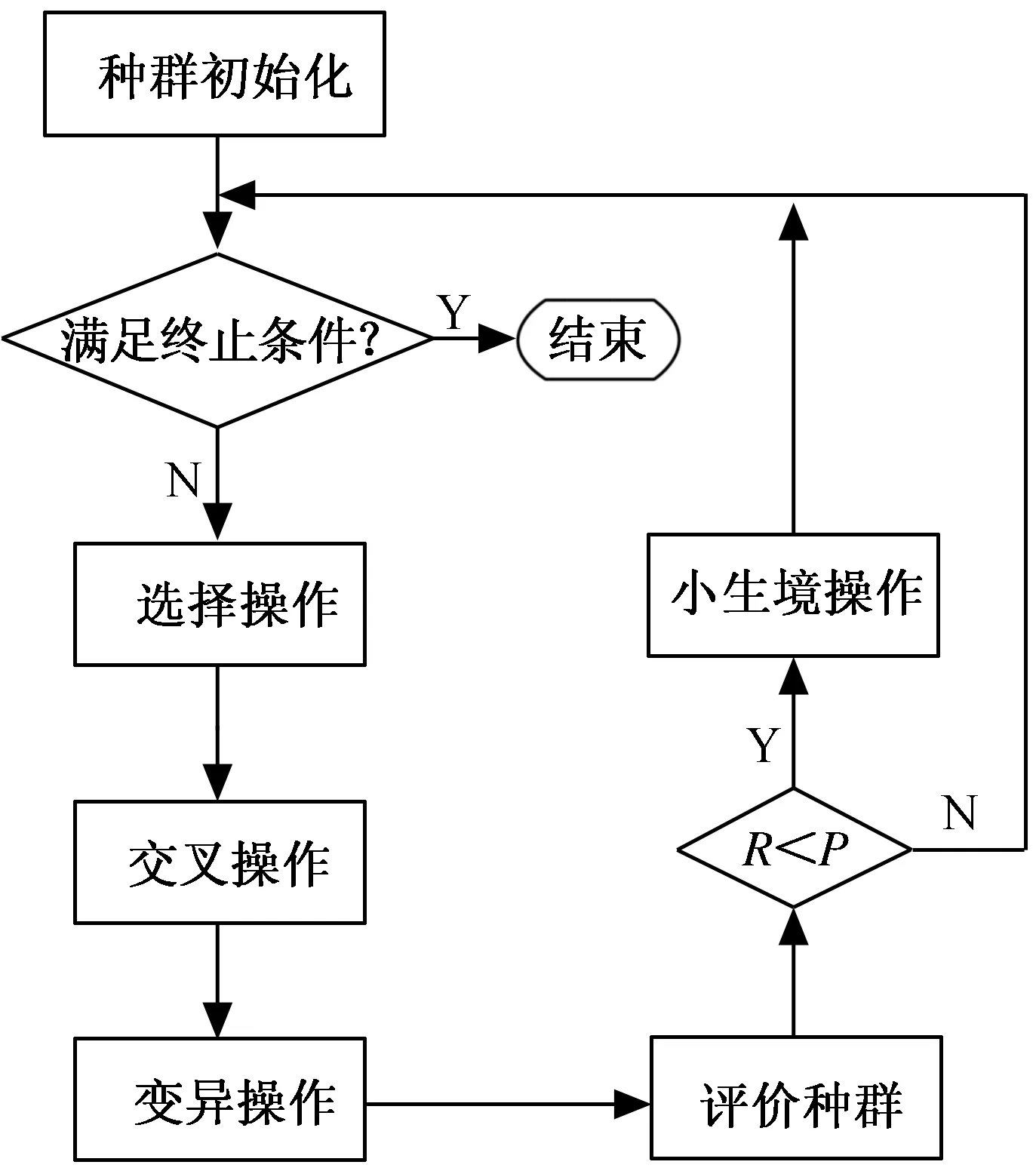
图1 小生境遗传算法
基于设计的框架,针对多序列问题,算法的各算子实现如下:
1)种群初始化。算法个体采用二维编码方式,通过随机插入空位的方式改变由m条序列组成的序列组,并使各序列长度均为h,m条序列分别称为M1,M2,…,Mn,其组成了规模为n的种群。
2)选择算子。算法采用了赌轮盘法结合精英策略选择下一代,首先根据适应度值排序,选取排名前10%的个体直接进入下一代,其余个体通过赌轮盘法产生。
3)交叉算子。算法采用两点交叉作为交叉算子,对任意的2条序列,随机选择序列的2点,然后将序列两点间的基因位进行交易。对生成的2个新个体,把适应度值高的且与已生成的个体相异的一个个体保留下来。
4)变异算子。算法的变异算子设计为:考虑一致性函数一致性信息的目的,随意选择1条序列并从中选择残基,将此残基对应位置的其他残基移到双序列上形成新的个体。
5)评价函数。为实现多序列比对一致性的最大化,建立如下的评价函数:
f(M)=max(COST(M))
(2)
其意义在于f(M)的函数值越大,则多序列比对一致性越高,因此其与式(1)的目标有着同一性。当算法执行结束时,则对应了一致性最大的多序列组。
6)小生境操作。选择操作前,按设定比例保存个体,并将其与变异操作后的种群合并以得到小生境操作的集合,然后计算两两个体间的差异性。本文中对应2个序列不同符号个数,当不相同符号数小于一定值时,被认为同一小生境,此时根据适应度值淘汰评价较差者。操作结束得到较为优秀且差异性较大的种群,此过程不断迭代至代数达到最大。
7)终止条件。设置1个最大迭代次数,当算法循环的次数达到最大迭代次数则停止执行。
2.2多核平台算法的并行化设计
多核CPU现在已经成为主流,对算法进行多核平台的并行化设计具有重要意义。考虑设计的小生境遗传算法具有良好的内在并行性,其选择、交叉、变异、个体评价及小生境操作均可并行计算,因此该算法非常适合在多核CPU上并行执行。为提高计算效率,笔者将其设计成并行形式,如图2所示。

图2 多核平台的并行小生境遗传算法
3 实验
BAliBASE基准多序列比对库有144个,共分5类蛋白质多序列比对测试例,该测试例库常用于多序列比对程序的性能测试。通常衡量多序列比对程序的性能采用SPS(Sum of Pairs Score)和CS(Column Score)分值,残基对准确对齐的比率称为SPS分值,所有序列准确对齐的比率(即对齐多少列)称为CS分值,两者值越大则结果越优。而为了方便比较,选择测试用例时与文献[7]一致,即在前2类中选择前10个测试例,后3类中选择5个测试例。本文算法标记为NGA,文献算法标记为GC-GM,实验结果表1所示。
表1实验结果比较
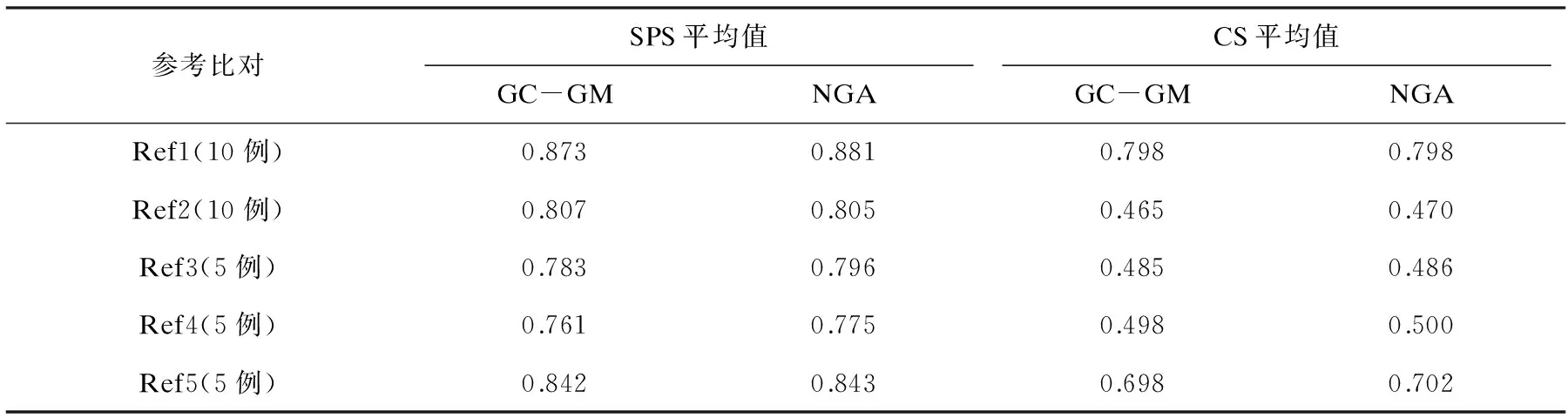
参考比对SPS平均值CS平均值GC-GMNGAGC-GMNGARef1(10例)0.8730.8810.7980.798Ref2(10例)0.8070.8050.4650.470Ref3(5例)0.7830.7960.4850.486Ref4(5例)0.7610.7750.4980.500Ref5(5例)0.8420.8430.6980.702
从表1结果可知,NGA取得的结果除Ref2(10例)中SPS平均值略小于GC-GM外,其他的结果均优于GC-GM。考虑遗传算法本身具有一定随机性,上述存在稍差的结果是可以接受的。因此总的来说,本文结果优于文献[7]的结果,这说明本文算法的有效性。
为验证并行化的有效性,对上述选取的测试用例分别用串行小生境遗传算法(SNGA)与并行小生境遗传算法(PNGA)进行求解,并在Dell机架式服务器PowerEdge 2950 III(配置Intel Xeon E5405 CPU,四核)上执行,对各问题分别求解5次最后时间,取平均值,最后通过加速比与效率衡量并行化的效果,结果如表2所示。
表2并行化实验结果
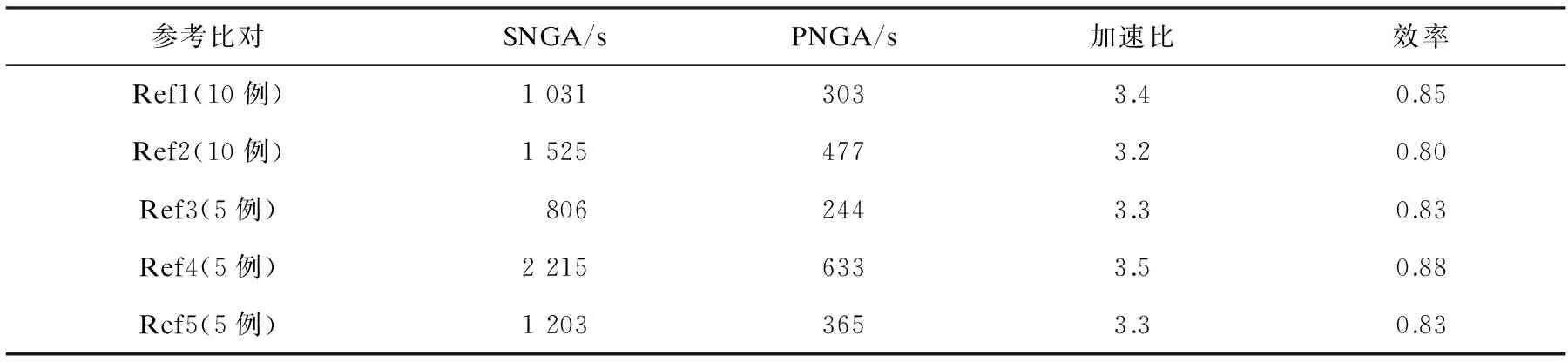
参考比对SNGA/sPNGA/s加速比效率Ref1(10例)10313033.40.85Ref2(10例)15254773.20.80Ref3(5例)8062443.30.83Ref4(5例)22156333.50.88Ref5(5例)12033653.30.83
从表2可知,算法并行化后取得了较好的加速比与计算效率,在包含4个CPU核芯的计算环境中,加速比达到了3以上,并行计算的加速效果明显,这说明了对算法并行化设计的有效性。
4 结语
针对求解难度大的多序列比对问题,设计了一个改进遗传算法对其进行求解。算法通过引进小生境技术,维持种群进化的多样性,改善算法的整体搜索能力。最后通过对BAliBASE中的测试例进行测试,结果较相关文献结果更优。而考虑遗传算法本身具有较好的并行性及当前多核平台的主流化,对算法进行了并行化设计,取得较好的加速比。
[1]ATTWOOD T K,PARRY-SMITH D J.生物信息学概论[M].罗静初,译.北京:北京大学出版社,2002.
[2]吴璟莉,王华,梁彬彬.求解创建者序列重建问题的单亲遗传算法[J].计算机应用与软件,2012,29(10):71-74.
[3]邹权,郭茂祖,韩英鹏,等.多序列比对算法的研究进展[J].生物信息学,2010,8(4):311-315.
[4]高峰,李防震,王珺,等.基于置换距离度量的蛋白质多序列比对算法性能评估[J].中山大学学报(自然科学版),2011,50(2):87-92.
[5]D GUSFIELD.Algorithms on strings,trees and sequences:computer science and computational biology[M].Cambridge:Cambridge University Press,1997.
[6]NOTREDAME C,HIGGINS D G.SAGA:Sequence alignment by genetic algorithm[J].Nucleic Acids Research,1996,24(8):1515-1524.
[7]张琎,张远.基GC-GM的多序列比对穷举遗传算法[J].计算机应用,2010,30(1):146-149.
[8]WANG L,JIANG T.On the complexity of multiple sequence alignment[J].Journal of Computational Biology,1994(4):337-348.
[9]李隽颖,楼晓俊.基于小生境遗传算法的分类优化方法[J].计算机应用研究,2012,29(5):1787-1790.
[10]李敏强,寇纪淞,林丹,等.遗传算法的基本理论与应用[M].北京:科学出版社,2004.
责任编辑:陈亮
An Improved Genetic Algorithm for Solving Multiple Sequence Alignment Problem Based on Niche Technology
An improved genetic algorithm based on the multi-core platform was presented for solving the multiple sequence alignment problem.The algorithm took consistency function as an individual fitness function,and introduced niche technology to maintain the diversity of population so as to improve the overall search capability of the algorithm.Considering the good parallelism of genetic algorithm,the operators were paralleled for the multi-core platform.BAliBASE test cases were used to test the algorithm,comparing with the existing algorithm.The better results showed that it was effective.Paralleling algorithm run significantly and reduced the running time on the multi-core platform with obvious acceleration effect.
multiple sequence alignment;niche genetic algorithm;multi-core;parallel
10.3969/j.issn.1671-0436.2016.04.008
2016- 07- 18
海南省重点研发资助项目(ZDYF2016153);长江大学工程技术学院基金项目资助(2016KY13)
张继成(1984—),男,硕士,讲师。
TP301.6
A
1671- 0436(2016)04- 0034- 05
