棉花GbRvd的亚细胞定位及其超表达烟草抗病性分析
2016-10-13杨占武吴金华李志坤王省芬吴立强张桂寅马峙英
杨占武,杨 君,张 艳,吴金华,李志坤,王省芬,吴立强,张桂寅,马峙英
棉花的亚细胞定位及其超表达烟草抗病性分析
杨占武,杨 君,张 艳,吴金华,李志坤,王省芬,吴立强,张桂寅,马峙英
(河北农业大学农学院/教育部华北作物种质资源研究与利用重点实验室,河北保定071001)
【目的】研究棉花NB-LRR家族基因的亚细胞定位及超表达烟草的黄萎病抗性,为解析介导的植物抗黄萎病机制提供依据。【方法】对真核表达载体pJCV52进行改造,在Ⅰ酶切位点插入,使其兼具亚细胞定位的功能,命名为GPJCV52。应用Gateway技术构建的亚细胞定位和超表达载体,并分别转化农杆菌GV3101。利用生物信息学和农杆菌介导的烟草瞬时表达技术对GbRvd分别进行亚细胞定位预测与观察。采用农杆菌介导法转化烟草,并通过组织培养技术获得转基因植株。运用PCR对超表达的烟草进行阳性植株筛选,且利用半定量RT-PCR对阳性转基因植株进行表达检测。制备棉花黄萎病菌孢子悬浮液,通过灌根法对T3代转基因烟草株系进行接种,利用5级标准统计发病情况并计算病情指数。【结果】利用在线分析软件ProtComp预测GbRvd为一种胞外(分泌)蛋白;GbRvd的跨膜预测结果显示其包含3个跨膜域;在线分析软件WoLF PSORT预测结果显示GbRvd在细胞膜、内质网及叶绿体的预测分值分别为7、3和1,表明GbRvd主要存在于植物细胞膜上。利用农杆菌介导的烟草瞬时表达技术进一步对GbRvd的亚细胞定位进行观察,结果显示在单独表达GFP蛋白的烟草细胞中,荧光信号在细胞核、细胞质和细胞膜均有分布;而GbRvd与GFP融合表达后的荧光信号主要分布于烟草表皮细胞质和细胞膜。综合生物信息学分析和亚细胞定位观察结果,表明细胞膜和细胞质是GbRvd的主要分布位置。利用农杆菌介导和组织培养获得了转基因烟草植株,通过对转基因烟草植株的PCR检测显示,阳性转基因烟草植株能够通过PCR扩增出与预期大小一致的目的条带,而野生型植株未出现相应条带;阳性转基因烟草植株半定量RT-PCR同样能够检测出与预期大小一致的目的条带,由此表明成功整合于烟草基因组并能够进行正常转录。对3个T3代超表达烟草株系的黄萎病抗性进行分析,结果显示超表达的烟草植株病情指数显著低于野生型对照植株,表明过表达可有效提高植株对黄萎病的抗性。【结论】GbRvd主要存在于植物细胞质和细胞膜,其超表达可显著提高烟草对黄萎病的抗性。
棉花;;亚细胞定位;转基因烟草;黄萎病
0 引言
【研究意义】黄萎病(Verticillium wilt)是影响棉花生产的主要病害之一,已成为制约中国棉花生产实现高产、稳产和优质的瓶颈,抗黄萎病机制研究是抗病育种领域的重要科学问题之一[1-2]。对棉花抗黄萎病相关基因的发掘和功能研究,不仅有助于全面阐述棉花抗黄萎病机制,还为棉花抗病遗传改良提供重要理论依据。【前人研究进展】目前,一些参与棉花抗黄萎病的重要基因已经被成功克隆和研究,如[3]、和[4]及[5]、[6]、[7]、[8]、[9]、[10]、[11]等。其中,编码蛋白属于NB-LRR(nucleotide-binding-leucine rich repeat)家族,而该家族通过免疫识别参与植物对多种病原的抗性,如病毒、细菌、真菌、昆虫、线虫等[12]。目前,大多数已知的R(resistance)蛋白来自该家族。NB-LRR家族蛋白含有3个独立的结构域:N端CC(coiled-coil)/TIR(Toll-interleukin 1 receptor),中心NB(nucleotide- binding,亦称为NB-ARC,nucleotide-binding adaptor shared by Apaf1, certain R genes and CED4)和C端富含亮氨酸重复序列(leucine-rich repeat,LRR)。关于NB-LRR介导的植物免疫机制主要归结为2种:其一,按照基因对基因假说,NB-LRR直接与病菌效应子(effector)互作,以使它们不能发挥致病作用[13];其二,间接保护机制,即NB-LRR识别病菌效应子与植物预警蛋白(guard protein)的结合,进而引发植物免疫反应[14]。拟南芥()基因组中包含大约150个NB-LRR基因,其中几个获得了较深入的研究,如(resistance to)被证明参与霜霉病(downy mildew)抗性[15],RPS4(resistance to4)和RRS1-R识别效应子PopP2引发植物对的抗性[16],以及RPS5能够被效应子AvrPphB激活[17]等。在棉花基因组中,仅二倍体亚洲棉()和雷蒙德氏棉()注释的NB-LRR家族基因就分别有280和391个[18],因此,研究NB-LRR基因的功能具有重要意义。【本研究切入点】目前,关于棉花NB-LRR家族基因的克隆和抗黄萎病功能的研究还鲜有报道。河北农业大学棉花遗传育种研究室前期从黄萎病菌()胁迫处理后的海岛棉()中筛选并克隆到一个编码CC-NB-LRR家族蛋白的基因——,并通过VIGS(virus induced gene silencing)技术初步证实其在棉花抗黄萎病过程中发挥重要作用[11]。【拟解决的关键问题】本研究通过生物信息学分析和亚细胞定位观察,综合确定GbRvd在植物细胞中的分布位置。利用遗传转化获得超表达的转基因烟草株系,并通过接菌处理和病情指数分析,进一步明确其抗病功能,为解析其抗黄萎病机制奠定基础。
1 材料与方法
1.1 材料
野生型本氏烟草()由清华大学刘玉乐教授馈赠,种植于温度25℃,湿度60%—70%,光照强度5 000 lx,16 h光照/8 h黑暗的光照培养室中。
落叶型黄萎病菌临西2-1由河北农业大学棉花遗传育种研究室分离鉴定并继代保存于PDA(potato dextrose agar)培养基。大肠杆菌DB3.1、DH5α及农杆菌GV3101由河北农业大学棉花遗传育种研究室保存。植物真核表达载体pJCV52由比利时根特大学VIB植物科学研究所馈赠,载体详细信息参见网站https:// gateway.psb.ugent.be/search;Gateway®载体pDONRTM207、携带编码绿色荧光蛋白(green fluorescent protein,GFP)基因质粒pGM-GFP和携带质粒pGM-GbRvd均由河北农业大学棉花遗传育种研究室保存。高保真酶2×PhantaTMMaster Mix购自南京诺唯赞生物科技有限公司;pGM-T载体、2×Taq PCR Master Mix、T4 DNA连接酶、限制性内切酶、质粒小提试剂盒及植物基因组DNA提取试剂盒均购自天根生化科技(北京)有限公司;BP ClonaseTMEnzyme Mix和LR ClonaseTMEnzyme Mix购自赛默飞世尔科技中国有限公司;胶回收试剂盒购自OMEGA Bio-Tek公司;其他生化试剂均为进口或国产分析纯。
引物合成以及DNA测序委托生工生物工程(上海)股份有限公司完成。
1.2 生物信息学分析
在线软件ProtComp Version 9.0(http://www. softberry.com/berry.phtml?topic=protcomp pl&group= program s&subgroup=proloc)和WoLF PSORT(http:// www.genscript.com/wolf-pso r.html)[19]用于蛋白的亚细胞定位预测;在线软件TMpred(http://www.ch. embnet.org/softwar e/TMPRED_form.html)用于蛋白的跨膜域分析。
1.3 亚细胞定位载体的构建
以质粒pGM-GFP为模板,利用引物SGFP-F:5′-GGGATGGTGAGCAAGGGCGAGGAG-3′和SGFP-R:5′-GGGCTTGTACAGCTCGT CCATGC-3′(下划线为Ⅰ酶切位点),扩增ORF序列(去终止子)。PCR反应体系(20 μL)为1 μL pGM-GFP质粒、1 μL SGFP-F、1 μL SGFP-R、10 μL 2×PhantaTMMaster Mix和7 μL去离子水。PCR反应程序为94℃5 min;94℃30 s,56℃30 s,72℃1 min,35个循环;72℃10 min。将目的片段进行胶回收,连接于pGM-T载体并转化大肠杆菌DH5α感受态细胞,菌落PCR筛选阳性克隆并测序。提取测序完全正确的单克隆质粒,经Ⅰ单酶切后用T4 DNA连接酶与相同酶切后的pJCV52载体连接(16℃过夜)。连接产物转化大肠杆菌DB3.1感受态细胞,并在含有庆大霉素(100 mg·L-1)的LB培养基平板上进行阳性克隆筛选并测序。测序正确的载体命名为GPJCV52,并用于后续亚细胞定位研究。
1.4 植物亚细胞定位及超表达载体的构建
以质粒pGM-GbRvd为模板,通过引物gRvd-F (5′-ATGGGGACTCACATCTTGAAATC-3′)和gRvd-R(5′-GCGCAAGATGTTTTGGAATATGA-3′)扩增含有Gateway通用接头(下划线表示)的ORF序列。PCR反应体系(20 μL)为1 μL pGM-GbRvd质粒、1 μL gRvd-F、1 μL gRvd-R、10 μL 2×PhantaTMMaster Mix和7 μL去离子水。PCR反应程序为94℃5 min;94℃30 s,56℃30 s,72℃3 min,35个循环;72℃10 min。回收目的条带进行BP反应,体系(5 μL)为2 μL PCR产物(>10 ng)、2 μL pDONRTM207(>150 ng)、1 μL BP ClonaseTMEnzyme Mix。体系转化方法:将体系混匀后,于PCR仪25℃反应10 h;加1 μL Proteinase K,37℃ 10 min终止反应;转化DH5α后进行阳性克隆筛选(庆大霉素)和测序。测序正确的质粒命名为pDONRTM207-GbRvd。通过LR反应将分别克隆至GPJCV52载体和pJCV52,反应体系(5 μL)为2 μL pDONRTM207-GbRvd(>150 ng)、2 μL GPJCV52或pJCV52(>150 ng)、1 μL LR ClonaseTMEnzyme Mix。体系转化方法同BP反应,阳性克隆筛选抗性变为壮观霉素(100 mg·L-1)。测序正确的载体分别命名为GPJCV52-GbRvd和pJCV52-GbRvd。构建超表达GFP的载体pJCV52-gfp的方法同GPJCV52-GbRvd,仅所用引物序列变为:gGFP-F(5′-ATGGTGAGCAAGGGCGAGGAG-3′;gGFP-R(5′-CGGTTGTACAGCTCGTCCATGC-3′)(下划线表示Gateway通用接头)。
1.5 农杆菌转化
载体转化农杆菌GV3101参考An[20]的方法。利用引物gGFP-F/gGFP-R和gRvd-F/gRvd-R分别对转化表达载体pJCV52-GbRvd、pJCV52-gfp和GPJCV52- GbRvd的农杆菌进行菌落PCR鉴定。
1.6 蛋白亚细胞定位
参考Sparkes等[21]方法进行目的蛋白在烟草瞬时表达。具有4片真叶的野生型本氏烟草生长用于农杆菌侵染。方法简述如下:农杆菌于含有壮观霉素和利福平(100 mg·L-1)的LB液体培养基中进行培养(28℃,200 r/min),至菌液OD600约为1.0时,通过离心(6 000 r/min,10 min)收集和清洗(灭菌去离子水)细胞,重悬于侵染液(2-(N-吗啡啉)乙磺酸9.76 g·L-1、葡萄糖50 g·L-1、Na3PO4·12H2O 0.76 g·L-1、乙酰丁香酮0.0196 g·L-1)并调整OD600≈0.2,置于室温待用。用1 mL注射器将重悬于侵染液中的农杆菌注入烟草叶背面,之后暗培养24 h,再正常培养2—3 d。撕取侵染后的烟草叶片下表皮,置于接有荧光激发模块U-RFL-T(Olympus,Tokyo,Japan)的BX51型显微镜(Olympus,Tokyo,Japan)下,通过Image-Pro®Plus 6.0 software(Media Cybernetics,Rockville,MD,USA)观察并拍照记录。
1.7 农杆菌介导的烟草遗传转化
活化含有真核表达载体pJCV52-GbRvd的农杆菌GV3101,用于侵染烟草叶片,具体方法如下:将活化的农杆菌于含有壮观霉素和利福平(100 mg·L-1)的LB液体培养基中进行培养(28℃,200 r/min),至菌液OD600约为1.0时,通过离心(6 000 r/min,10 min)收集和清洗(灭菌去离子水)细胞,重悬于侵染液(2-(N-吗啡啉)乙磺酸9.76 g·L-1、葡萄糖50 g·L-1、Na3PO4·12H2O 0.76 g·L-1、乙酰丁香酮0.0196 g·L-1),并调整OD600≈0.2;用1 mL注射器将重悬于侵染液中的农杆菌注入烟草叶背面,侵染后的烟草暗培养24 h,再正常光照培养2—3 d后用于组织培养。组织培养的具体方法如下:将农杆菌侵染的烟草叶片于70%的乙醇溶液中消毒1 min,1/1000的升汞溶液消毒3 min,之后用灭菌水清洗3—5次。在无菌条件下将消毒后的烟草叶片切成1 cm2大小,放入生芽培养基(2.15 g·L-1MS培养基、8 g·L-1琼脂、30 g蔗糖、0.1 mg·L-1吲哚丁酸、0.8 mg·L-16-苄氨基嘌呤、100 mg·L-1卡那霉素和100 mg·L-1头孢霉素)中培养(25℃,16 h光照/8 h黑暗)。3—4周后,将幼芽从基部与愈伤组织分离并插入生根培养基(2.15 g·L-1MS培养基、8 g·L-1琼脂、30 g蔗糖、0.5 mg·L-1吲哚丁酸、100 mg·L-1卡那霉素和100 mg·L-1头孢霉素)中培养。约10 d后,将生根幼苗移栽至营养土中继续培养。
1.8 转基因烟草检测
提取烟草基因组DNA。以其为模板,利用全长扩增引物RvdORF-F(5′-ATGGGGACTCACAT CTTGAAATC-3′)和RvdORF-R(5′-GCGCAAGATG TTTTGGAATATGA-3′)扩增,检测在转基因烟草中的插入情况。PCR反应体系和程序设置同1.4。
在烟草中的表达采用半定量RT-PCR检测。烟草RNA提取和反转录分别通过EasySpin植物RNA提取试剂盒和PrimeScript™ RT reagent Kit with gDNA Eraser,具体操作按照说明书进行。半定量引物为qRvd-F(5′-GGAAGGTTCGTAGGGTTTCT GTGC-3′)和qRvd-R(5′-CTACTGCCTCCTCCGAAAA GAACTC-3′)。烟草α作为内参,引物为EF1α-F(5′-ACGCTTGAGATCCTTAACCGC-3′)和EF1α-R(5′-ACGCTTGAGATCCTTAACCGC-3′)。反应体系为1 μL cDNA、1 μL正向引物、1 μL反向引物、10 μL 2×Taq PCR Master Mix和7 μL去离子水。PCR反应程序为94℃5 min;94℃30 s,60℃30 s,72℃30 s,25个循环;72℃10 min。反应结束后进行1%琼脂糖电泳检测并拍照。
1.9 转基因烟草抗病性鉴定
用T3代转基因烟草进行黄萎病抗性鉴定。参考Yang等[11]的方法进行黄萎病菌孢子悬浮液的制备。烟草种植于体积为600 mL的六棱钵中,生长约4周后用于灌根接菌,接菌量60 mL,浓度为1×107孢子/mL。操作如下:接菌前3 d停止对植株浇水,以使土壤含水量降低而易于菌液吸收;用医用注射器将孢子悬浮液注入距植株1 cm处培养土中,注射深度2 cm。接菌后的植株于温室条件下继续培养。20 d后检查植株发病情况,并按黄萎病温室苗期发病的5级制分级记录,即0级为植株无症状表现;1级为1—2片真叶发病;2级为3—4片真叶发病;3级为4片真叶以上发病或脱落;4级为全株枯死。病情指数(Disease index)= [Σ(发病级数×该级株数)]÷(调查总株数×4)×100。转基因烟草黄萎病抗病性鉴定试验共重复3次,每次处理不少于20株植株。使用GraphPad Prism®6软件进行差异显著性分析及制图。
2 结果
2.1 GbRvd亚细胞定位
利用ProtComp和WoLF PSORT 2个在线分析软
件对GbRvd进行亚细胞定位预测。ProtComp预测采用神经网络(neural nets)算法,结果显示,GbRvd为一种胞外(分泌)蛋白;WoLF PSORT是根据分选信号(sorting signals)、氨基酸组成(amino acid composition)和功能域(functional motif),采用简单的-nearest neighbor classifier算法进行的预测,获得GbRvd在细胞膜(plasma membrane)、内质网(endoplasmic reticulum)及叶绿体(chloroplast)的预测分值分别为7、3和1。此外,GbRvd的跨膜预测结果显示其包含3个跨膜域(图1):(1)从第2位氨基酸残基到第18位氨基酸残基;(2)从第355位氨基酸残基到第373位氨基酸残基;(3)从第584位氨基酸残基到第605位氨基酸残基。综合以上生物信息学分析结果,推测GbRvd主要存在于植物细胞膜上。
为进一步确认GbRvd在细胞中的位置,将其与GFP进行融合,在烟草表皮细胞中进行瞬时表达(图2)。在单独表达GFP蛋白的烟草细胞中,荧光分布于细胞核、细胞质和细胞膜;而对于GbRvd-GFP融合表达,荧光主要分布于烟草细胞膜和细胞质。综合预测和观察的结果,表明细胞膜和细胞质是GbRvd主要的分布位置。

①、②、③:3个跨膜域 ①, ②, ③: Three transmembrane domains

图2 GbRvd在烟草表皮细胞中的亚细胞定位
2.2 转GbRvd烟草植株的获得
为进一步分析的抗黄萎病功能,将其ORF克隆至超表达载体pJCV52并转入农杆菌。农杆菌浸染后的烟草叶片首先在生芽培养基上培养,3—4周后开始产生愈伤组织,再经过约10 d后愈伤组织分化出不定芽(图3-A);待不定芽分化出明显的茎和叶时,将其与愈伤组织分离并插入筛选生根培养基继续培养(图3-B);经过7—10 d,植株生出根(图3-C);在根伸长至2—3 cm时,将植株移栽至营养土中继续培养成完整、健壮的植株(图3-D)。

A:愈伤组织的形成;B:诱导不定根;C:生出不定根;D:转基因植株
利用的ORF全长扩增引物RvdORF-F和RvdORF-R对T2代转基因(OE)植株做进一步的检测,部分PCR扩增检测结果如图4所示:10个转基因株系均能扩增获得约3 000 bp大小的目的条带(ORF测序全长2 928 bp),而野生型(WT)植株未有目的带扩增出现。此外,半定量RT-PCR检测结果显示,相对于野生型,仅在转基因植株中转录(图5)。由此表明,成功整合进烟草染色体并正常转录,获得转基因烟草植株。
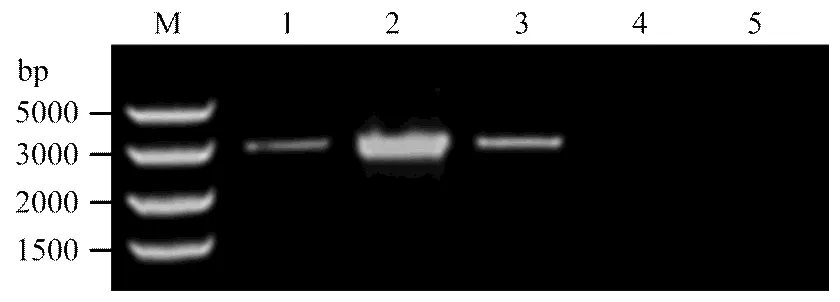
M:DNA maker DL5000;1—3:阳性植株;4:阴性对照;5:野生型

OE:过表达;WT:野生型 OE: Over-expression; WT: Wild type
2.3 转基因烟草的抗病性鉴定
通过对其中的3个转基因株系做进一步的抗黄萎病功能分析。野生型和转烟草植株接菌处理12 d后出现发病植株,20 d后野生型比转基因型叶片更加黄化、萎蔫(图6-A)。统计分析结果显示,超表达的烟草植株病情指数显著低于野生型对照(图6-B)。由此可见,过表达可以有效提高烟草植株对黄萎病的抗性。
3 讨论
面对病原菌的侵染,植物先天免疫系统(innate immune system)通常调用两道防御反应:依赖于细胞表面受体识别而启动的PTI(pathogen associated molecular patterns triggered immunity)和由NB-LRR等免疫受体(immune receptor)识别病菌效应子后的ETI(effector triggered immunity)[22]。为了更加有效地识别和启动ETI,NB-LRR等必须存在于植物细胞中更有利于发挥作用的位置。因此,亚细胞定位研究对于解析NB-LRR的抗病功能至关重要[23]。NB-LRR在植物免疫反应中发挥着识别病菌效应子和启动植物免疫的双重作用[24],因而,对于不同的NB-LRR在不同的功能时期其在植物细胞中的发生位置可能会明显不同。目前,已经有一些通过荧光蛋白标记对NB-LRR进行亚细胞定位的研究。一个蛋白序列中无标准核定位信号(nuclear localization signal,NLS)的烟草蛋白N,荧光标记显示其位于植物细胞核和细胞质中[25]。同样无NLS的大麦()蛋白MLA10,其也定位于细胞核和细胞质[26]。拟南芥RRS1-R有NLS和WRKY转录因子结构域,定位于细胞核[16]。另一个拟南芥NB-LRR蛋白RPS4,其含有NLS,定位于细胞核和细胞质。通过蔗糖梯度离心等分离技术,NS-LRR的其他亚细胞定位被发现,如RPS4、RPP1A、RPM1等,它们可能还存在于细胞膜、内质网和高尔基体等位置[27-29]。由此可见,NB-LRR家族蛋白在植物细胞中的分布与它们的蛋白功能域之间存在着不一致。NB-LRR在细胞中的位置较多样,可能与其识别不同病菌效应子和启动不同的免疫通路相关,亦或受不同的伴侣蛋白所调控,如WRKY[30]、SRFR1(Suppressor of)[31]等。GbRvd拥有NLS[11],但在本研究中其仅定位于细胞膜和细胞质,而未能在细胞核中发现。因此,推测GbRvd可能负责病原菌识别和向胞内传递免疫信号的功能,而进一步的激活植物免疫反应可能还需要其他蛋白辅助完成。

A:黄萎病菌侵染20 d后的烟草发病现象;B:病指统计分析。OE:超表达植株;WT:野生型;**表示OE组和WT组之间有显著差异(P<0.01,Student’s t-test)。图中数值为平均值±SE(n=3)
Zhao等[32]将玉米NB-LRR基因转入水稻中超表达,且有效地提高了水稻对细菌性条斑病(pv.)的抗性。Wang等[33]克隆了一个水稻NB-LRR基因,其超表达能够显著提高感病水稻品种对细菌性条斑病的抗性。Weaver等[28]将进行超表达,使拟南芥获得对霜霉病菌和丁香假单胞菌的广谱抗性。
在中国农业科学院棉花研究所测序的陆地棉TM-1基因组数据库中进行比对,结果显示与TM-1中同源性最高的基因CotAD_76302(locus: scaffold4990.1:14013:18271)的序列相似度仅为58%。在烟草和拟南芥等模式植物中未比对到的同源基因。本研究将转入烟草,并获得稳定表达植株。黄萎病菌处理后,超表达的烟草抗病性获得显著提高。异源超表达不仅进一步验证了的抗病功能,还为该类基因在未来农业中的应用提供了重要依据。
4 结论
棉花NB-LRR蛋白GbRvd主要分布于植物细胞质与细胞膜。获得转的烟草植株,其超表达显著提高了烟草对黄萎病的抗性,进一步证明介导棉花抗黄萎病反应,是一个重要的抗病因子。
References:
[1] 马存, 简桂良, 郑传临. 中国棉花抗枯、黄萎病育种50年. 中国农业科学, 2002, 35(5): 508-513.
Ma C, Jian G L, Zheng C L. The advances in cotton breeding resistance to fusarium and Verticillium wilts in China during past fifty years., 2002, 35(5): 508-513. (in Chinese)
[2] 潘家驹, 张天真. 棉花黄萎病抗性遗传研究. 南京农业大学学报, 1994, 17(3): 8-18.
Pan J J, Zhang T Z. Studies on the inheritance of resistance to Verticillum dahliae in cotton., 1994, 17(3): 8-18. (in Chinese)
[3] Zhang Y, Wang X F, Yang S, Chi J N, Zhang G Y, Ma Z Y. Cloning and characterization of a Verticillium wilt resistance gene fromand functional analysis in., 2011, 30(11): 2085-2096.
[4] Gao X Q, Wheeler T, Li Z H, Kenerley C M, He P, Shan L B. Silencingandcompromises cotton resistance to Verticillium wilt., 2011, 66(2): 293-305.
[5] Zhang B L, Yang Y W, Chen T Z, Yu W G, Liu T L, Li H J, Fan X H, Ren Y Z, Shen D Y, Liu L, Dou D L, Chang Y H. Island cottongene encoding a receptor-like protein confers resistance to both defoliating and non-defoliating isolates of Verticillium dahliae., 2012, 7(12): e51091.
[6] Gao X Q, Li F L, Li M Y, Kianinejad A S, Dever J K, Wheeler T A, Li Z H, He P, Shan L B. Cottonmediates Verticillium wilt resistance and cell death., 2013, 55(7): 586-596.
[7] Su X F, Qi X L, Cheng H M. Molecular cloning and characterization of enhanced disease susceptibility 1 () from., 2014, 41(6): 3821-3828.
[8] Li C, He X, Luo X Y, Xu L, Liu L L, Min L, Jin L, Zhu L F, Zhang X L. Cotton WRKY1 mediates the plant defense-to- development transition during infection of cotton byby activating JASMONATE ZIM-DOMAIN1 expression., 2014, 166(4): 2179-2194.
[9] Yang J, Ji L L, Wang X F, Zhang Y, Wu L Z, Yang Y N, Ma Z Y. Overexpression of 3-deoxy-7-phosphoheptulonate synthase gene fromenhancesresistance to Verticillium wilt., 2015, 34(8): 1429-1441.
[10] 杨君, 张艳, 王伟巧, 吴金华, 王国宁, 马峙英, 王省芬. 海岛棉. 植物遗传资源学报, 2015(3): 594-602.
Yang J, Zhang Y, Wang W Q, Wu J H, Wang G N, Ma Z Y, Wang X F, Cloning offromand validation of Verticillium wilt resistance in transgenic., 2015(3): 594-602. (in Chinese)
[11] Yang J, Ma Q, Zhang Y, Wang X F, Zhang G Y, Ma Z Y. Molecular cloning and functional analysis of, a gene inthat plays an important role in conferring resistance to Verticillium wilt., 2016, 575(2): 687-694.
[12] Gururani M A, Venkatesh J, Upadhyaya C P, Nookaraju A, Pandey S K, Park S W. Plant disease resistance genes: Current status and future directions., 2012, 78(51): 51-65.
[13] Flor H H. Current status of the gene-for-gene concept., 1971, 9(1): 275-296.
[14] McHale L, Tan X P, Koehl P, Michelmore R W. Plant NBS-LRR proteins: adaptable guards., 2006, 7(4): 1-11.
[15] Adnane N, Susanna A, Tarone A M, Huang Y S, Keyan Z, Studholme D J, Magnus N, Jones J D G. Genome-wide survey ofnatural variation in downy mildew resistance using combined association and linkage mapping., 2010, 107(22): 10302-10307.
[16] Deslandes L, Olivier J, Peeters N, Feng D X, Khounlotham M, Boucher C, Somssich I, Genin S, Marco Y. Physical interaction between RRS1-R, a protein conferring resistance to bacterial wilt, and PopP2, a type III effector targeted to the plant nucleus., 2003, 100(13): 8024-8029.
[17] Qi D, DeYoung B J, Innes R W. Structure-function analysis of the coiled-coil and leucine-rich repeat domains of the RPS5 disease resistance protein., 2012, 158(4): 1819-1832.
[18] Li F G, Fan G Y, Wang K B, Sun F M, Yuan Y L, Song G L, Li Q, Ma Z Y, Lu C R, Zou C S, Chen W B, Liang X M, Shang H H, Liu W Q, Shi C C, Xiao G H, Gou C Y, Ye W W, Xu X, Zhang X Y, Wei H L, Li Z F, Zhang G Y, Wang J Y, Liu K, Kohel R J, Percy R G, Yu J Z, Zhu Y X, Wang J, Yu S X. Genome sequence of the cultivated cotton., 2014, 46: 567-572.
[19] Horton P, Park K J, Obayashi T, Fujita N, Harada H, Adams-Collier C J, Nakai K. WoLF PSORT: protein localization predictor., 2007, 35(Web Server issue): 585-587.
[20] An G. Binary ti vectors for plant transformation and promoter analysis., 1987, 153(153C): 292-305.
[21] Sparkes I A, Runions J, Kearns A, Hawes C. Rapid, transient expression of fluorescent fusion proteins in tobacco plants and generation of stably transformed plants., 2006, 1(4): 2019-2025.
[22] Dodds P N, Rathjen J P. Plant immunity: towards an integrated view of plant-pathogen interactions., 2010, 11(8): 539-548.
[23] Cournoyer P, Dinesh-Kumar S P. Studying NB-LRR immune receptor localization by agroinfiltration transient expression., 2011, 712: 1-8.
[24] Chiang Y H, Coaker G. Effector triggered immunity: NLR immune perception and downstream defense responses., 2015, 11: e0183.
[25] Burch-Smith T M, Schiff M, Caplan J L, Tsao J, Czymmek K, Dinesh-Kumar S P. A novel role for the TIR domain in association with pathogen-derived elicitors., 2007, 5(3): e68.
[26] Shen Q H, Saijo Y, Mauch S, Biskup C, Bieri S, Keller B, Seki H, Ulker B, Somssich I E, Schulze-Lefert P. Nuclear activity of MLA immune receptors links isolate-specific and basal disease-resistance responses., 2007, 315(5815): 1098-1103.
[27] Wirthmueller L, Zhang Y, Jones J D G, Parker J E. Nuclear accumulation of the Arabidopsis immune receptor RPS4 is necessary for triggering EDS1-dependent defense., 2008, 17(23): 2023-2029.
[28] Weaver L M, Swiderski M R, Li Y, Jones J D G. TheTIR-NB-LRR R-protein, RPP1A; protein localization and constitutive activation of defence by truncated alleles in tobacco and., 2006, 47(6): 829-840.
[29] Boyes D C, Nam J, Dangl J L. Thedisease resistance gene product is a peripheral plasma membrane protein that is degraded coincident with the hypersensitive response., 1998, 95(95): 15849-15854.
[30] Haruhiko I, Nagao H, Akane M, Liu X Q, Akira N, Shoji S, Jiang C J, Hiroshi T. Blast resistance of CC-NB-LRR protein Pb1 is mediated by WRKY45 through protein-protein interaction., 2013, 110(23): 9577-9582.
[31] Li Z Y, Li S X, Bi D L, Cheng Y T, Li X, Zhang Y L. SRFR1 negatively regulates plant NB-LRR resistance protein accumulation to prevent autoimmunity., 2010, 6(9): 8871-8887.
[32] Zhao B Y, Lin X H, Poland J, Trick H, Leach J, Hulbert S. A maize resistance gene functions against bacterial streak disease in rice., 2005, 102(43): 15383-15388.
[33] Wang X M, Chen J, Yang Y, Zhou J, Qiu Y, Yu C L, Cheng Y, Yan C Q, Chen J P. Characterization of a novelGene Involved in bacterial blight resistance in rice., 2013, 31(3): 649-656.
(责任编辑 李莉)
Subcellular Localization and Verticillium Wilt Resistance Analysis of Cottonin Overexpressed Tobacco
YANG Zhan-wu, YANG Jun, ZHANG Yan, WU Jin-hua, LI Zhi-kun, WANG Xing-fen, WU Li-qiang, ZHANG Gui-yin, MA Zhi-ying
(College of Agronomy, Hebei Agricultural University/North China Key Laboratory for Crop Germplasm Resources of Ministry of Education, Baoding 071001, Hebei)
【Objective】In order to resolve the mechanism of GbRvd-mediated resistance to Verticillium wilt in cotton, subcellular localization and resistance of-overexpressed tobacco were studied.【Method】Expression vector pJCV52 was modified by insertinggene intoⅠenzyme site and the resulted vector was named as GPJCV52 which can be used for subcellular localization. Gateway-based constructs, made for subcellular localization and overexpression of GbRvd, were transformed intostrain GV3101 for tobacco transformation. Subcellular localization of GbRvd was predicted and observed by bioinformatics analysis and-mediated transient expression in tobacco leaf, respectively. Transformation of tobacco was made by-mediatedmethod, and final transgenic tobacco plants were obtained through tissue culture. PCR and semi-quantitative RT-PCR were used for screening positive transgenic plants and detecting gene expression levels, respectively.conidial suspension was prepared and incubated with T3generation of transgenic tobacco strains through the method of soil drench.The disease severity for plants was graded from 0 to 4 and then the disease index was calculated.【Result】ProtComp, an online analysis software, predicted that GbRvd is an extracellular (secreted) protein. Transmembrane prediction showed that GbRvd contains three transmembrane domains. On-line analysis software WoLF PSORT prediction results showed that GbRvd localization in the cell membrane, endoplasmic reticulum and chloroplast scores were 7, 3 and 1, respectively, indicating that GbRvd mainly existed in the plant cell membrane. In order to further confirm the location of GbRvd in the cell, expression of GbRvd fused with GFP was examined. Fluorescence signal of control GFP protein in tobacco cells was observed in the nucleus, cytoplasm and cell membrane, and fluorescence signals of GbRvd fused with GFP were mainly detected in the plasma membrane and cytoplasm of tobacco epidermal cells. The transgenic tobacco plants were produced by-mediated infection and tissue culture method.PCR detection results showed that the expected size of DNA band could be amplified in positive transgenic tobacco plants, and that could not appear in wild type plants. By semi-quantitative RT-PCR expected band could also be detected in positive transgenic tobacco plants, which indicates thatsuccessfully integrated to the tobacco genome and can be normally transcribed. Three independent T3transgenic tobacco lines were selected toanalyze Verticillium wilt resistance. The results showed that the disease index of transgenic lines was significantly lower than that of wild-type, indicating that overexpression of GbRvd can effectively improve plant resistance to Verticillium wilt. 【Conclusion】GbRvd mainly exist in plant cell plasma and membrane, and its overexpression can significantly increase the resistance of tobacco to Verticillium wilt.
cotton;; subcellular location; transgenic tobacco; Verticillium wilt
2016-04-29;接受日期:2016-08-29
国家“863”计划(2013AA102601)、国家自然科学基金(31301370)、河北省高等学校科学技术研究项目(QN2016009)
联系方式:杨占武,Tel:0312-7528415;E-mail:1091748213@qq.com。通信作者马峙英,Tel:0312-7528401;E-mail:mzhy@hebau.edu.cn
